Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Pulmonale strukturelle MRT mit freiatmender, selbstgesteuerter Ultrakurz-Echozeit-Bildgebung
In diesem Artikel
Zusammenfassung
Es wird ein Protokoll zur Erstellung hochauflösender struktureller Bilder der Lunge mit Hilfe der Ultrakurzechozeit (UTE) Magnetresonanztomographie (MRT) beschrieben. Dieses Protokoll ermöglicht die Aufnahme von Bildern mit einer einfachen MRT-Pulssequenz während der freien Atmung.
Zusammenfassung
Eine qualitativ hochwertige MRT der Lunge wird durch eine geringe Gewebedichte, eine schnelle MRT-Signalrelaxation sowie Atem- und Herzbewegungen erschwert. Aus diesen Gründen wird die strukturelle Bildgebung der Lunge fast ausschließlich mittels Computertomographie (CT) durchgeführt. Die CT-Bildgebung liefert jedoch ionisierende Strahlung und ist daher für bestimmte gefährdete Bevölkerungsgruppen (z. B. Kinder) oder für Forschungsanwendungen weniger gut geeignet. Als Alternative stößt die MRT mit ultrakurzen Echozeiten (UTE) auf Interesse. Diese Technik kann während der freien Atmung im Verlauf eines ~5-10-minütigen Scans durchgeführt werden. Informationen über Atembewegungen werden zusammen mit Bildern kodiert; Diese Informationen können verwendet werden, um Bilder selbst zu steuern. Durch das Self-Gating entfällt somit die Notwendigkeit einer fortschrittlichen MRT-Pulssequenzprogrammierung oder der Verwendung eines Atembalgs, was die Bildaufnahme vereinfacht. In diesem Protokoll werden einfache, robuste und recheneffiziente Akquisitions- und Rekonstruktionsmethoden für die Aufnahme einer qualitativ hochwertigen UTE-MRT der Lunge vorgestellt. Dieses Protokoll wurde für die Verwendung auf einem 3T-MRT-Scanner entwickelt, aber die gleichen Prinzipien können bei geringerer Magnetfeldstärke implementiert werden. Das Protokoll enthält empfohlene Parametereinstellungen für die radiale 3D-UTE-Bilderfassung sowie Anweisungen für die selbstgesteuerte Bildrekonstruktion, um Bilder in verschiedenen Atemphasen zu erzeugen. Durch die Implementierung dieses Protokolls können Benutzer hochauflösende UTE-Bilder der Lunge mit minimalen bis minimalen bis gar keinen Bewegungsartefakten erstellen. Diese Bilder können zur Beurteilung der Lungenstruktur verwendet werden, die für Forschungszwecke bei einer Vielzahl von Lungenerkrankungen eingesetzt werden kann.
Einleitung
Die hochauflösende Bildgebung der Lungenstruktur ist bei vielen Lungenerkrankungen ein wesentlicher Bestandteil der Diagnostik. In der Regel erfolgt dies mit Hilfe der Computertomographie (CT), die sich hervorragend eignet, um hochauflösende Bilder der Lungezu erstellen 1. Die CT-Bildgebung liefert jedoch eine nicht unerhebliche Dosis ionisierender Strahlung, was sie für die regelmäßige Wiederholungsbildgebung, die Bildgebung in mehreren verschiedenen Atemphasen oder die Bildgebung bestimmter Bevölkerungsgruppen (z. B. Kinder) ungeeignet macht. Die Magnetresonanztomographie (MRT) birgt nicht das gleiche Risiko ionisierender Strahlung und ist daher für solche bildgebenden Aufgaben zugänglich. Es ist jedoch schwierig, die Lunge mittels MRT abzubilden, da die Gewebedichte gering ist, die Atem- und Herzbewegung sowie die sehr schnelle Signalrelaxation 2,3,4.
Eine MRT-Technik, die in der Lage ist, diese Herausforderungen zu mildern, ist die MRT 4,5,6 mit ultrakurzer Echozeit (UTE). Bei der UTE-MRT wird das MRT-Signal unmittelbar nach der Signalanregung abgetastet, wodurch die Auswirkungen einer schnellen Signalrelaxation reduziert werden. Darüber hinaus wird bei dieser Technik der k-Raum von der Mitte nach außen abgetastet, was zu einer signifikanten Überabtastung im Zentrum des k-Raums führt. Dieses Oversampling in der Mitte des k-Raums macht dieses bildgebende Verfahren robust gegenüber Bewegungen. Zusätzlich zu dieser inhärenten Robustheit gegenüber Bewegung kodiert die wiederholte Abtastung des Zentrums des k-Raums Informationen über die Atembewegung, was das Selbstgating von Bildern ermöglicht 7,8,9. Dieses Self-Gating kann verwendet werden, um Bilder in einer Vielzahl von Atemphasen zu erzeugen. Da der Mensch den größten Teil der Atemphase bei der Exspiration verbringt, ist es üblich, ein Bild für die Endexspiration zu erstellen, da in dieser Phase die meisten Bilddaten erfasst werden.
Es gibt eine Vielzahl von Strategien für das respiratorische Self-Gating in der Lungen-MRT. Die erste Unterscheidung, die getroffen werden muss, ist bildbasiert vs. k-Space-basiertes Gating10 (Abbildung 1). Beim bildbasierten Gating wird ein Satz von Bildern mit hoher zeitlicher Auflösung erzeugt, indem kleine zeitliche Teilmengen der Bilddaten rekonstruiert werden. Anschließend wird die Position des Zwerchfells in diesen Bildern verwendet, um die Atmungsphase für einen gegebenen Satz von Bildprojektionenzu identifizieren 10, 11. Beim k-Raum-basierten Gating werden Daten aus dem Zentrum des k-Raums ("k0") untersucht 8,9,12. Die Signalintensität des Bildes ist in k0 kodiert, und somit variiert die Intensität des k0-Punktes mit der Atmung. Projektionen können so basierend auf der Intensität von k0 in verschiedene Atmungsphasen eingeteilt werden. Sowohl beim bildbasierten als auch beim k-Raum-basierten Gating werden Projektionen mit gleichrespiratorischen Phasen für die Bildrekonstruktion gruppiert. Es wurde vermutet, dass das bildbasierte Gating eine verbesserte Genauigkeit bei der Abschätzung der Atmungsphase bietet und dadurch Bilder mit reduzierter Unschärfe liefert10,13.
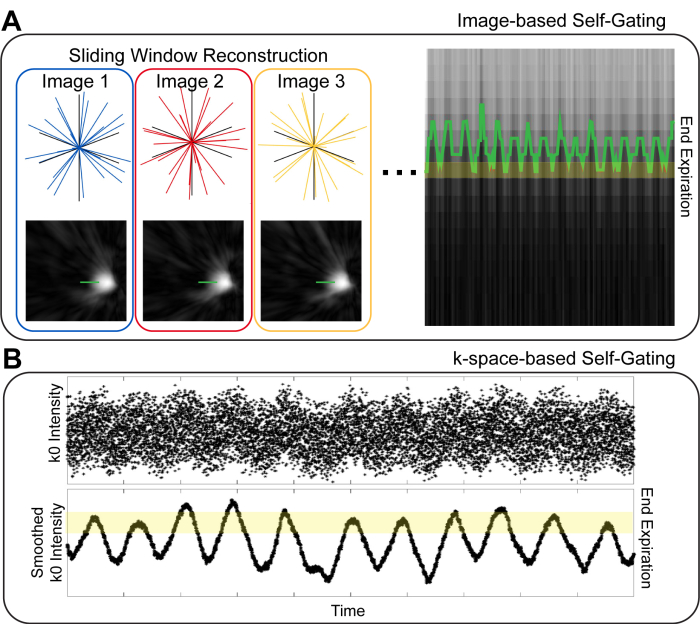
Abbildung 1: Bildbasierte und k-Raum-basierte Self-Gating-Techniken. (A) Beim bildbasierten Gating werden Bilder mit geringer räumlicher Auflösung und hoher zeitlicher Auflösung, die das Zwerchfell zeigen, aus zeitlichen Teilmengen der Gesamtdaten erzeugt. Mit Hilfe einer Linie über dem Zwerchfell kann die Atembewegung visualisiert und für die Bildrekonstruktion in einen Bin-Bereich aufgenommen werden. (B) Beim k-Raum-basierten Gating wird der erste Punkt auf einer zentrierten k-Raumprojektion ("k0") verwendet, um die Atembewegung zu visualisieren. Nach der Glättung von k0 sind Signalintensitätsunterschiede basierend auf dem Atemzyklus deutlich sichtbar und können zur Identifizierung verschiedener Atmungsphasen verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Sowohl bild- als auch k-raum-basiertes Gating können entweder mit hartem Gating oder weichem Gating durchgeführt werden11,14. Beim Hard Gating werden nur die Vorsprünge rekonstruiert, die der gewünschten Atmungsphase entsprechen. Diese Verwerfung unerwünschter Projektionen kann jedoch zu einem verringerten Signal-Rausch-Verhältnis (SNR) von Bildern und zu vermehrten Undersampling-Artefakten führen. Diese unerwünschten Effekte können durch die Verwendung von Soft Gating gemildert werden. Beim Soft-Gating werden alle Projektionen für die Bildrekonstruktion verwendet, Projektionen aus einer unerwünschten Atmungsphase werden jedoch so gewichtet, dass sie einen geringeren Einfluss auf das endgültige Bild haben. Auf diese Weise können Bilder mit minimalen Artefakten und hohem SNR rekonstruiert werden, während die Auswirkungen der Atembewegung weiterhin unterdrückt werden.
Durch die Kombination der UTE-MRT-Aufnahme mit dem Self-Gating nach der Aufnahme können qualitativ hochwertige Bilder erzeugt werden, die zwar nicht der CT entsprechen, aber einen Kontrast und eine Auflösung aufweisen, die sich der der CT-Bildgebung 6,15,16,17,18,19 annähern. Darin wird ein einfaches Protokoll zum Sammeln und Rekonstruieren von UTE-MRT-Bildern bereitgestellt, um qualitativ hochwertige Bilder der Lungenstruktur zu erzeugen.
Dieses Protokoll wurde hauptsächlich für 3T-MRT-Scanner geschrieben. 3T ist die gebräuchlichste Feldstärke, die für die Forschungs-MRT verwendet wird. Niedrigere Magnetfeldstärken wie 1,5T oder das kürzlich verfügbare 0,55 T20 können für eine verbesserte Bildqualität und Signalintensität in der Lunge sorgen, da die Signalrelaxation in der Lunge bei diesen Feldstärken langsamer ist.
Obwohl alle Anstrengungen unternommen wurden, um Klarheit und Einfachheit in diesem Protokoll und dem bereitgestellten Bildrekonstruktionscode zu schaffen, wird das Protokoll wahrscheinlich einen dedizierten MRT-Physiker (oder einen ähnlichen MRT-Experten) erfordern, um eine geeignete UTE-MRT-Sequenz auf dem MRT-Scanner zu erstellen. Die MRT-Sequenz sollte eine nicht-kartesische 3D-Kodierungsstrategie mit zentrierten k-Raum-Trajektorien implementieren. Beispiele hierfür sind radiale 3D- oder 3D-Spiralsequenzen (z. B. "FLORET")21,22 Bildsequenzen. Wichtig ist, dass die Reihenfolge der Projektionen eine gute zeitliche Stabilität aufweist: Über eine gegebene Teilmenge der Zeit sollten die Projektionen den gesamten Bereich des k-Raums23 abdecken. Beispiele für Projektionsordnungsstrategien mit guter zeitlicher Stabilität sind die goldenen Mittel oder die Halton-randomisierte archimedische Spirale. Wenn eine Projektionsordnung mit geringer zeitlicher Stabilität verwendet wird, werden beim Self-Gating nach der Erfassung große Bereiche des k-Raums weggelassen, was zu Bildartefakten führt. Schließlich sollte die Sequenz in der Lage sein, eine Echozeit (TE) von <100 μs zu erreichen. Die T2*-Relaxationszeit in der Lunge bei 3T beträgt <1 ms24, daher ist die Verwendung einer sehr kurzen TE für die Erzeugung qualitativ hochwertiger Bilder unerlässlich.
Protokoll
Alle Bildgebungen am Menschen wurden mit Genehmigung des KUMC IRB durchgeführt. Von allen Teilnehmern wurde eine schriftliche Einverständniserklärung eingeholt. Die Bilder in dieser Studie wurden im Rahmen eines generischen technischen Entwicklungsprotokolls aufgenommen, und die Ein-/Ausschlusskriterien waren bewusst weit gefasst. Einschlusskriterien: Alter ≥ 18 Jahre. Ausschlusskriterien: MRT kontraindiziert auf der Grundlage der Antworten auf den MRT-Screening-Fragebogen und Schwangerschaft. Das Zubehör und die Ausrüstung, die für diese Studie verwendet werden, sind in der Materialtabelle aufgeführt.
1. UTE-Bilderfassung
- Bereiten Sie die Bildgebungssequenz vor. Bereiten Sie die Bildgebungssequenz einmal vor und verwenden Sie dieselbe Sequenz für alle Teilnehmer.
- Stellen Sie die Parameter gemäß Tabelle 1 ein.
- Platzieren Sie ein MRT-Phantom in der Mitte des MRT und führen Sie die Bildgebungssequenz aus.
HINWEIS: Da diese Sequenz eine schnelle Gradientenleistung und viele HF-Impulse erfordert, ist es wichtig zu überprüfen, ob die Protokolleinrichtung vor dem Test an einem Menschen ausgeführt werden kann.
- Bereiten Sie den Teilnehmer auf die MRT vor. Verwenden Sie ein MRT-Sicherheitsscreening nach institutionellem Standard, um sicherzustellen, dass der Teilnehmer sicher in die MRT eintreten kann.
- Positionieren Sie den Teilnehmer auf der MRT-Liege und legen Sie eine Brustspirale über den Oberkörper des Teilnehmers. Positionieren Sie die Spule nahe am Kinn des Teilnehmers, um eine vollständige Abdeckung der Lungenspitzen zu gewährleisten.
- Bewegen Sie den Teilnehmer in den MRT-Scanner. Platzieren Sie den Positionierungsorientierungspunkt direkt unter dem Brustbein des Teilnehmers.
- Führen Sie einen Lokalisatorscan durch, um sicherzustellen, dass sich die Lungen des Teilnehmers innerhalb des Sichtfelds für den UTE-Scan befinden. Verschieben Sie nicht die Geometrie des UTE-Scans. Wenn sich die Lungen des Teilnehmers nicht im Sichtfeld befinden, bewegen Sie den Teilnehmer und nehmen Sie zusätzliche Lokalisationsscans auf, bis sich die Lungen vollständig im Sichtfeld befinden.
- Führen Sie die UTE-Sequenz aus. Während dieser Sequenz kann der Teilnehmer normal atmen.
- Exportieren Sie die Rohdaten aus dem Scanner. Abhängig von der verwendeten Bildsequenz kann der Scanner Bilder auf dem Scanner rekonstruieren oder nicht. Für die vorgeschlagene retrospektive Gating-Rekonstruktion werden Rohdaten benötigt, um zu bestimmen, ob Bilder auf dem Scanner erzeugt werden oder nicht. Beachten Sie, dass die Rohdaten groß sein werden (>10 GB).
- Exportieren oder berechnen Sie k-Raum-Trajektorien (d. h. die Position jedes Rohdatenpunkts im k-Raum).
HINWEIS: Bei einigen Bildgebungssequenzen können K-Raum-Trajektorien zusammen mit den Rohdaten auf dem MRT-Scanner gespeichert und direkt exportiert werden. Für andere Bildgebungssequenzen müssen die k-Raum-Trajektorien auf der Grundlage von Bildgebungsparametern berechnet werden.
| Parameter | Generische empfohlene Einstellungen | Hierin implementierte Einstellungen |
| Bildgebende Sequenz | Nicht-kartesische 3D-Trajektorien mit zentrierten k-Raum-Trajektorien | 3D-Radial mit Goldener Mittel-Projektion Reihenfolge |
| Sichtfeld | 400 x 400 x 400 mm3 | 400 x 400 x 400 mm3 |
| Matrix-Größe | Wie gewünscht für die Zielauflösung | 320 x 320 x 320 (1,25 mm isotrope Auflösung) |
| Bandbreite | Bei Bedarf für die Auslesedauer < 1,0 ms | 888 Hz/Pixel |
| TE | < 0,1 ms | 0,07 ms |
| TR | Minimum (Ziel 3 – 4 ms) | 3,5 ms |
| Flip-Winkel | Ungefähr 5° | 4.8° |
| Anzahl der Projektionen | Mindestens 100.000 | 1,35,386 |
| Dauer des Bildes | Mindestens 5 Minuten | 7 Minuten, 54 Sek. |
Tabelle 1: Empfohlene Einstellungen für die UTE-Bildgebung. Es werden generische empfohlene Einstellungen bereitgestellt, die zur Einrichtung des Protokolls verwendet werden können. Spezifische empfohlene Einstellungen, die für die Daten verwendet wurden, werden ebenfalls bereitgestellt, wie als repräsentative Ergebnisse dargestellt. Die Parameterspezifikationen sind bei allen Anbietern generisch, mit Ausnahme der Bandbreite. Einige große MRT-Anbieter geben die Bandbreite als Hz/Pixel an. Andere große MRT-Anbieter geben absolute Bandbreite an. Die empfohlene Bandbreite (888 Hz/Pixel) entspricht einer absoluten Bandbreite von 284.160 Hz.
2. UTE-Bildrekonstruktion mittels bildbasiertem respiratorischem Soft-Gating
HINWEIS: MATLAB-Code zum Ausführen der folgenden Schritte finden Sie unter https://github.com/pniedbalski3/UTE_Reconstruction.
- Importieren Sie Daten und k-Raum-Trajektorien in MATLAB. Der Code zum Importieren von MRT-Rohdaten ist für alle großen MRT-Anbieter verfügbar.
- Verwerfen Sie die ersten 1000 Projektionen, um sicherzustellen, dass sich die Daten in stationärer Magnetisierung befinden.
HINWEIS: Wenn die verwendete Bildgebungssequenz Dummy-Scans vor der Datenerfassung umfasst, kann dieser Schritt übersprungen werden. - Rekonstruieren Sie ein Bild mit niedriger Auflösung mit einer sehr kleinen Teilmenge von Daten.
- Rekonstruieren Sie das Bild mit einer ungleichmäßigen schnellen Fourier-Transformation auf eine Matrixgröße von 96 x 96 x 96.
- Verwenden Sie ca. 200 Projektionen, was Daten von 0,6 s bis 0,8 s entspricht.
- Rekonstruieren und speichern Sie Bilder von allen Spulenelementen sowie ein endgültiges, spiralkombiniertes Bild.
- Wählen Sie im resultierenden spiralkombinierten Bild eine koronale Schicht aus, die das Zwerchfell deutlich zeigt.
HINWEIS: Der bereitgestellte Code fordert den Benutzer auf, eine Scheibe auszuwählen, die die Membran enthält. - Nachdem Sie diese Scheibe ausgewählt haben, sehen Sie sich die einzelnen Spulenbilder für diese Scheibe an und wählen Sie ein oder zwei Spulenelemente aus, die die Membran am besten darstellen (Abbildung 2).
HINWEIS: Der bereitgestellte Code fordert den Benutzer auf, Spulenelemente auszuwählen. - Rekonstruieren Sie Bilder mit einem gleitenden Fenster , um Bilder mit einer zeitlichen Auflösung von ~0,5 s zu generieren (Abbildung 2).
- Rekonstruieren Sie nur die Daten aus den in Schritt 2.4 ausgewählten Spulenelementen.
HINWEIS: Obwohl alle Spulenelemente rekonstruiert werden können, werden nur die Elemente benötigt, die der Membran am nächsten sind, um die Membran zum Zwecke der Atemwegsselbstkontrolle zu visualisieren. Indem nur die Spulenelemente rekonstruiert werden, die dem Diagramm am nächsten sind, werden die Rekonstruktionszeit und der Rechenaufwand drastisch reduziert. - Verwenden Sie die ersten 200 Projektionen, um ein Bild mit einer ungleichmäßigen schnellen Fourier-Transformation zu rekonstruieren (Abbildung 2). Bewahren Sie nur die Scheibe auf, die die Membran zeigt (wie in Schritt 2.4 beschrieben).
HINWEIS: Letztendlich werden bis zu 1500 Bilder generiert; Es wird nur eine 2D-Schicht benötigt, um die Membranposition zu visualisieren, und das Speichern von 3D-Bildern für jede der Schiebefensterstufen wäre unerschwinglich. - Verschiebung um 100 Projektionen (d.h. das erste Bild wird mit den Projektionen 1-200 rekonstruiert. Die zweite wird mit den Projektionen 101 - 300) rekonstruiert und rekonstruiert ein zusätzliches Bild, wobei die in Schritt 2.4 ausgewählte Schicht gespeichert wird.
- Fahren Sie fort, bis alle Projektionen zum Generieren von Bildern verwendet wurden.
- Rekonstruieren Sie nur die Daten aus den in Schritt 2.4 ausgewählten Spulenelementen.
- Wählen Sie im ersten Bild des Schiebefensters eine Linie über der Blende aus. Stellen Sie sicher, dass die Leitung lang genug ist, um sich um 5-10 Voxel in die Lunge und um 5-10 Voxel in das Zwerchfell zu erstrecken.
- Visualisieren Sie die Atembewegung, indem Sie diesen Atemnavigator für alle Projektionen anzeigen.
- Bestimmen Sie die Position des Zwerchfells für alle Atemnavigationsgeräte. Es gibt eine Vielzahl von Möglichkeiten, dies zu tun, aber eine einfache Methode besteht darin, die Methode25 von Otsu zu verwenden, um die dunklere Seite (Lunge) von der helleren Seite (Zwerchfell) zu trennen.
- Verwenden Sie die Position des Zwerchfells, um Projektionen als zu einem bestimmten Atemwegsbehälter gehörend zu kennzeichnen. Wenn ein gegebener Atemnavigator das Zwerchfell an "Position 1" anzeigt, dann gehören die 200 Projektionen, die zur Erzeugung des Bildes für diesen Navigator verwendet wurden, zu "Bin 1".
HINWEIS: Da Bilder mit einem Schiebefenster mit einer Überlappung von 100 Projektionen erstellt wurden, können einige Projektionen als zu mehreren Klassen gehörend gekennzeichnet sein. Die grobe räumliche Auflösung von Schiebefensterbildern führt zu insgesamt ~4-6 Bins, die die gesamte Bandbreite der Inspiration bis zum Ablauf abdecken. - Wählen Sie den Abschnitt aus, der rekonstruiert werden soll, indem Sie bestimmen, welcher Abschnitt die größte Anzahl von Projektionen aufweist, die dem Endablauf entsprechen sollten.
- Alternativ können Sie Bilder für die gewünschten Atemphasen auf der Grundlage einer visuellen Inspektion des Atemnavigators rekonstruieren.
- Generieren von Gewichten für Soft-Gating14.
- Verwenden Sie einen Exponentialfilter , um den Projektionen innerhalb des primären Behälters eine Gewichtung von 1 und den Vorsprüngen in verschiedenen Atemwegsbehältern eine stark reduzierte Gewichtung zu verleihen.
- Verwenden Sie die Berkely Advanced Reconstruction Toolbox (BART; https://mrirecon.github.io/bart/)26,27, um ein hochauflösendes Bild am gewünschten Atemwegsbehälter zu rekonstruieren.
HINWEIS: BART ist eine frei verfügbare Toolbox für die MRT-Bildrekonstruktion.- Berechnen Sie Dichtekompensationsgewichte mithilfe der iterativen Dichtekombination.
- Skalieren Sie die Dichteausgleichsgewichte mit den Soft-Gating-Gewichten.
- Skalendaten basierend auf Dichtekompensation und Soft-Gating-Gewichten
- Führen Sie eine grundlegende ungleichmäßige schnelle Fourier-Transformation (NUFFT) durch, um die Spulenkombination zu erleichtern.
- Konvertieren Sie das NUFFT-Bild in einen gerasterten k-Raum, der für die Spulenkombination verwendet werden soll.
- Generieren Sie eine Spulenkombinationsmatrix und verwenden Sie sie, um Spulen sowohl für die Rohdaten als auch für den gerasterten k-Raum zu kombinieren.
- Schätzen Sie die Empfindlichkeit der Spule.
- Führen Sie eine parallele Imaging-Rekonstruktion des komprimierten Senses mit der gewichteten Dichtekompensation, kombinierten Spulendaten und Spulenempfindlichkeitskarten durch.
- Speichern Sie das endgültige Bild. Das NIFTI-Format ist einfach zu implementieren. Wenn das Bild auf ein PACs-System hochgeladen werden soll, kann ein DICOM-Format erforderlich sein.
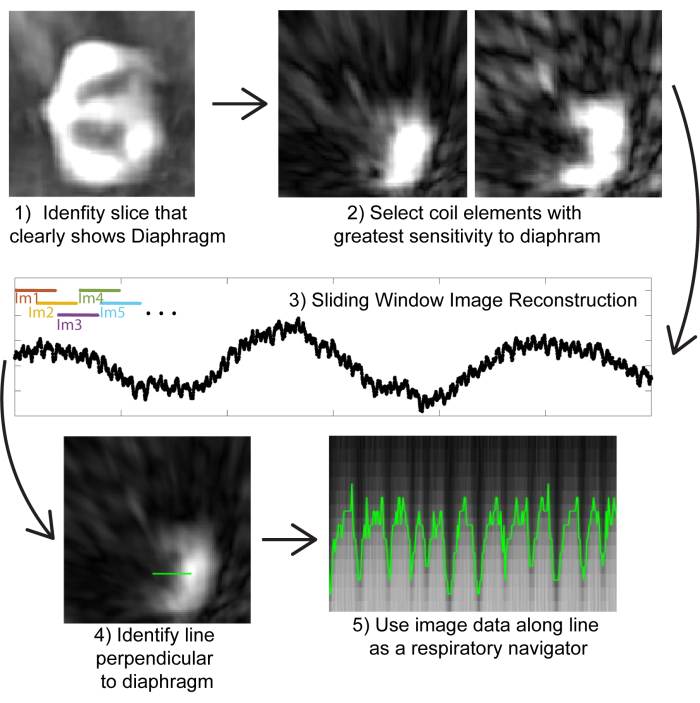
Abbildung 2: Bildbasiertes Self-Gating. (1) Identifizieren Sie anhand eines niedrig aufgelösten Bildes, das aus einer kleinen Anzahl von Projektionen rekonstruiert wurde (zur Recheneffizienz), eine koronale Schnitte, die das Zwerchfell deutlich zeigt. (2) Wählen Sie durch Untersuchen von Bildern von einzelnen Spulenelementen die Spulenelemente aus, die der Membran am nächsten sind. (3) Durchführen einer Schiebefensterrekonstruktion nur der Spulenelemente, die der Membran am nächsten sind (für die rechnerische Effizienz). Bilder können aus Teilmengen von 200 Projektionen (entsprechend ~0,8 s) erzeugt werden; Durch überlappende Projektionen kann eine pseudo-zeitliche Auflösung von ~0,5 s in Bildern erreicht werden. (4) Identifizieren einer Linie, die senkrecht zum Zwerchfell verläuft und als Atemnavigator verwendet werden soll. (5) Die Visualisierung der Bilddaten auf dieser Linie zeigt die Atembewegung, die zur Ablage von Bildern verwendet werden kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. UTE-Bildrekonstruktion mit k-Space-basiertem respiratorischem Soft-Gating
- Die Schritte 2.1 bis 2.4 sind so durchzuführen, dass das Spulenelement, das der Membran am nächsten liegt, identifiziert werden kann.
- Generieren Sie eine k 0-Zeitreihenverfolgung, indem Sie den absoluten Wert des ersten Punktes in der Projektion für alle Projektionen für das ausgewählte Windungselement verwenden. Dies ermöglicht die Visualisierung einer Atemwellenform.
- Normalisieren Sie in Schritten von 5000 Projektionen k0 durch die mittlere Signalintensität derselben k0 Punkte28. Dies verringert die Drift der Signalintensität im Laufe der Zeit und bietet eine verbesserte Möglichkeit, Projektionen quantitativ zu klassifizieren.
- Beschriften Sie jeden k0-Punkt als während der Inspiration oder des Ablaufs auftretend.
- Glätten Sie die k 0-Zeitreihe, und nehmen Sie die Ableitung, um die Steigung für jeden Punkt auf der Gating-Kurve zu bewerten.
- Beschriften Sie Inspirationspunkte basierend auf dem Zeichen der Steigung. Eine positive Steigung entspricht der Exspiration, während eine negative Steigung der Inspiration entspricht.
- Bin-Projektionen basierend auf der Signalintensität. Da die Atemtiefe variabel sein kann, basieren Bin-Projektionen auf der Signalamplitude und nicht auf der Position in der Atemphase.
HINWEIS: Eine einfache und schnelle Methode, um dies zu erreichen, ist die Implementierung von k-Means-Clustering, um verschiedene Signalintensitätsstufen zu identifizieren. - Identifizieren Sie für Bins, die zwischen Endinspiration und Endablauf liegen, Projektionen, die während der Inspiration und des Ablaufs basierend auf Schritt 3.4 auftreten.
- Schließen Sie die Bildrekonstruktion mit den Schritten in Schritt 2.10 bis Schritt 2.13 ab.
- Falls gewünscht, rekonstruieren Sie Bilder für alle Atemwegsbehälter und nicht nur am Ende des Ablaufs.
Ergebnisse
Repräsentative Ergebnisse (Abbildung 3) wurden mit den in Tabelle 1 gezeigten Einstellungen generiert. Die verwendete Bildgebungsdauer liefert qualitativ hochwertige Bilder, die von den meisten Teilnehmern toleriert werden können.
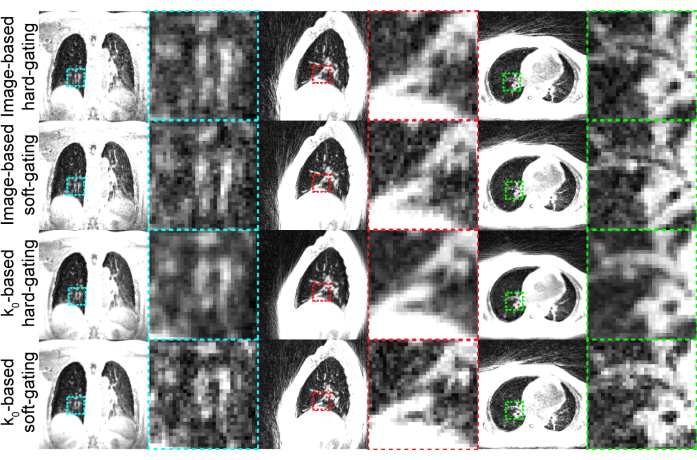
Abbildung 3: Repr?...
Diskussion
Bei der UTE-Bildgebung der Lunge können viele Variationen sowohl der Erfassung als auch der Rekonstruktion verwendet werden, um Bilder der Lunge zu erstellen. Dieses Protokoll konzentriert sich auf die einfache Implementierung und die Recheneffizienz. Die Bildgebung mit radialer 3D-UTE ist relativ einfach, da Bildgebungssequenzen in der Regel von den großen MRT-Anbietern erhältlich sind. MATLAB-basierte Tools werden für die Datenverarbeitung und das Self-Gating bereitgestellt. Da die...
Offenlegungen
Peter Niedbalski erhält Forschungsgelder von der National Scleroderma Foundation, der American Heart Association und dem NIH. Er ist Berater für Polarean Imaging Plc., ein Unternehmen, das die hyperpolarisierte 129Xe MRT-Technologie entwickelt.
Danksagungen
Die Entwicklung dieses Protokolls und die als repräsentative Ergebnisse gezeigten Bilder wurden von der Nationalen Sklerodermie-Stiftung unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Chest MRI Coil | Siemens, GE, Philips,, Other Clinical MRI Imaging Coil Vendor | N/A | A 26 - 32 channel Chest coil should be used |
| High Performance Workstation | HP, Apple, or other Computer Hardware company | N/A | A computer with a minimum of 64 GB of Memory is needed for image reconstruction |
| Matlab | Mathworks | R2016A or newer | A Matlab license is needed to run the provided computer code |
| MRI Phantom | Siemens, GE, Philips, or Other MRI Phantom Vendor | N/A | Any Phantom can be used to test the MRI sequence prior to its use in human subjects. |
| MRI Scanner | Siemens, GE, Philips, or Other Clinical MRI Scanner Vendor | N/A | The protocol was developed on a 3T scanner, but 1.5T or 0.55T would also work with minimal adaptation |
Referenzen
- Raju, S., Ghosh, S., Mehta, A. C. Chest ct signs in pulmonary disease: A pictorial review. Chest. 151 (6), 1356-1374 (2017).
- Biederer, J., et al. MRI of the lung (2/3). Why, when, how. Insights Imaging. 3 (4), 355-371 (2012).
- Biederer, J., et al. MRI of the lung (3/3)-current applications and future perspectives. Insights Imaging. 3 (4), 373-386 (2012).
- Johnson, K. M., Fain, S. B., Schiebler, M. L., Nagle, S. Optimized 3D ultrashort echo time pulmonary MRI. Magn Reson Med. 70 (5), 1241-1250 (2013).
- Ma, W., et al. Ultra-short echo-time pulmonary MRI: Evaluation and reproducibility in COPD subjects with and without bronchiectasis. J Magn Reson Imaging. 41 (5), 1465-1474 (2015).
- Roach, D. J., et al. Ultrashort echo-time magnetic resonance imaging is a sensitive method for the evaluation of early cystic fibrosis lung disease. Ann Am Thorac Soc. 13 (11), 1923-1931 (2016).
- Tibiletti, M., et al. Multistage three-dimensional UTE lung imaging by image-based self-gating. Magn Reson Med. 75 (3), 1324-1332 (2016).
- Weick, S., et al. Dc-gated high-resolution three-dimensional lung imaging during free-breathing. J Magn Reson Imaging. 37 (3), 727-732 (2013).
- Fischer, A., et al. Self-gated non-contrast-enhanced functional lung imaging (SENCEFUL) using a quasi-random fast low-angle shot (FLASH) sequence and proton MRI. NMR Biomed. 27 (8), 907-917 (2014).
- Tibiletti, M., et al. Respiratory self-gated 3D UTE for lung imaging in small animal MRI. Magn Reson Med. 78 (2), 739-745 (2017).
- Jiang, W., et al. Motion robust high resolution 3D free-breathing pulmonary MRI using dynamic 3D image self-navigator. Magn Reson Med. 79 (6), 2954-2967 (2018).
- Higano, N. S., et al. Retrospective respiratory self-gating and removal of bulk motion in pulmonary UTE MRI of neonates and adults. Magn Reson Med. 77 (3), 1284-1295 (2016).
- Metze, P., et al. Non-uniform self-gating in 2D lung imaging. Front Phys. 10, (2022).
- Gandhi, D. B., et al. Comparison of weighting algorithms to mitigate respiratory motion in free-breathing neonatal pulmonary radial UTE-MRI. Biomed Phys Eng Express. 10 (3), 035030 (2024).
- Fauveau, V., et al. Performance of spiral UTE-MRI of the lung in post-covid patients. Magn Reson Imaging. 96, 135-143 (2023).
- Metz, C., et al. Comparison of diagnostic quality of 3D ultrashort-echo-time techniques for pulmonary magnetic resonance imaging in free-breathing. Acta Radiologica. 64 (5), 1851-1858 (2023).
- Periaswamy, G., et al. Comparison of ultrashort TE lung MRI and HRCT lungs for detection of pulmonary nodules in oncology patients. Indian J Radiol Imaging. 32 (04), 497-504 (2022).
- Darçot, E., et al. Comparison between magnetic resonance imaging and computed tomography in the detection and volumetric assessment of lung nodules: A prospective study. Frontiers in Medicine. 9, 858731 (2022).
- Dournes, G., et al. 3D ultrashort echo time MRI of the lung using stack-of-spirals and spherical k-space coverages: Evaluation in healthy volunteers and parenchymal diseases. J Magn Reson Imaging. 48 (6), 1489-1497 (2018).
- Campbell-Washburn, A. E. 2019 American Thoracic Society Bear Cage winning proposal: Lung imaging using high-performance low-field magnetic resonance imaging. Am J Respir Crit Care Med. 201 (11), 1333-1336 (2020).
- Robison, R. K., Anderson, A. G., Pipe, J. G. Three-dimensional ultrashort echo-time imaging using a FLORET trajectory. Magn Reson Med. 78 (3), 1038-1049 (2017).
- Willmering, M. M., Robison, R. K., Wang, H., Pipe, J. G., Woods, J. C. Implementation of the FLORET sequence for lung imaging. Magn Reson Med. 82 (3), 1091-1100 (2019).
- Chan, R. W., Ramsay, E. A., Cunningham, C. H., Plewes, D. B. Temporal stability of adaptive 3D radial MRI using multidimensional golden means. Magn Reson Med. 61 (2), 354-363 (2009).
- Yu, J., Xue, Y., Song, H. K. Comparison of lung T2* during free-breathing at 1.5 T and 3.0 T with ultrashort echo time imaging. Magn Reson Med. 66 (1), 248-254 (2011).
- Otsu, N. A threshold selection method from gray-level histograms. IEEE Trans Syst Man Cybern. 9 (1), 62-66 (1979).
- Martin Uecker, F. O., et al. Berkely advanced reconstruction toolbox. Proc Intl Soc Magn Reson Med. 23, 2486 (2015).
- . . Bart Toolbox for Computational Magnetic Resonance Imaging. , (2024).
- Munidasa, S. . Treatment monitoring of pediatric cystic fibrosis lung disease using free breathing lung MRI. , (2024).
- Zhu, X., Chan, M., Lustig, M., Johnson, K. M., Larson, P. E. Z. Iterative motion-compensation reconstruction ultra-short TE (IMOCO UTE) for high-resolution free-breathing pulmonary MRI. Magn Reson Med. 83 (4), 1208-1221 (2020).
- Tan, F., et al. Motion-compensated low-rank reconstruction for simultaneous structural and functional UTE lung MRI. Magn Reson Med. 90 (3), 1101-1113 (2023).
- Bhattacharya, I., et al. Oxygen-enhanced functional lung imaging using a contemporary 0.55 T MRI system. NMR Biomed. 34 (8), e4562 (2021).
- Kim, M., et al. Feasibility of dynamic T2*-based oxygen-enhanced lung MRI at 3T. Magn Reson Med. 91 (3), 972-986 (2024).
- Klimeš, F., et al. 3D phase-resolved functional lung ventilation MR imaging in healthy volunteers and patients with chronic pulmonary disease. Magn Reson Med. 85 (2), 912-925 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten