Method Article
Ein Makrophagen-Tumor-Sphäroid-Co-Invasions-Assay
In diesem Artikel
Zusammenfassung
In dieser Arbeit stellen wir ein Protokoll vor, um die Interaktion von primären humanen Monozyten-abgeleiteten Makrophagen mit einem Tumorsphäroid in einer dreidimensionalen (3D) Kollagen-I-Matrix zu untersuchen, mit der Möglichkeit, den Einfluss löslicher und physikalischer Eigenschaften der Mikroumgebung auf die Zellinvasion zu vergleichen.
Zusammenfassung
Das Zusammenspiel von Immun- und Krebszellen und deren jeweiliger Einfluss auf die Metastasierung stellt einen wichtigen Aspekt der Krebsforschung dar. Bisher stehen nur wenige Protokolle zur Verfügung, die eine in vitro Annäherung an die in vivo Situation erlauben. In dieser Arbeit stellen wir einen neuartigen Ansatz zur Beobachtung des Einflusses menschlicher Makrophagen auf die Invasivität von Krebszellen vor, indem Tumorsphäroide von H1299 nicht-kleinzelligen Lungenkarzinomzellen verwendet werden, die in eine dreidimensionale (3D) Kollagen-I-Matrix eingebettet sind. Mit diesem Co-Kultivierungsaufbau haben wir den Einfluss der auf kleiner interferierende RNA (siRNA) basierenden Depletion regulatorischer Faktoren in Makrophagen auf die 3D-Invasion von Krebszellen aus dem Tumorsphäroid im Vergleich zu Kontrollen getestet. Diese Methode ermöglicht es, verschiedene Parameter, wie z.B. die Sphäroidfläche oder die Anzahl der eindringenden Krebszellen, zu bestimmen und so Unterschiede in der Invasion von Krebszellen zu erkennen. In diesem Artikel stellen wir das jeweilige Setup vor, diskutieren die anschließende Analyse, sowie die Vorteile und potenziellen Fallstricke dieser Methode.
Einleitung
Makrophagen sind ein wichtiger Teil des angeborenen Immunsystems und stellen die erste Verteidigungslinie bei vielen pathologischen Zuständen dar, wie z. B. bei Infektionen oder der Beseitigung von Zelltrümmern nach Verletzungen1. In den letzten Jahrzehnten war der Einfluss von Immunzellen auf das Fortschreiten von Krebs ein Aspekt vieler Studien. Dementsprechend wurde gezeigt, dass Makrophagen die Metastasierung erleichtern können, indem sie sich mit Primärtumoren assoziieren und zu tumorassoziierten Makrophagen (TAMs) werden2. Makrophagen können ihr Expressionsprofil verändern, wenn sie Krebszellen ausgesetzt sind, was das Entweichen von Metastasen aus dem Immunsystem begünstigt3. Darüber hinaus wurde gezeigt, dass Krebszellen Defekte in der extrazellulären Matrix (EZM), die von Makrophagen erzeugt werden, nutzen können, um aus dem Primärtumor zu entkommen, und dass ihr Verhalten auch durch die Aufnahme von sezernierten Faktoren, einschließlich extrazellulärer Vesikel (EVs), manipuliert wird4,5. Dieses Zusammenspiel von physikalischen und chemischen Aspekten erfordert die Entwicklung neuer Methoden, um den Einfluss von Immunzellen auf die Tumorausbreitung und die umgebende Mikroumgebung zu charakterisieren.
Es wurden verschiedene Ansätze entwickelt, um das Verhalten von Immunzellen im Rahmen der Metastasierung in vivozu untersuchen 6. Wie für alle Versuche, einschließlich Tierversuche, ist jedoch eine Lizenz für Tierversuche und eine Tiereinrichtung erforderlich; Diese Ansätze erfordern bereits viel Entwicklung und Vorbereitung. Darüber hinaus ist die Analyse oft kompliziert, insbesondere im Hinblick auf die Bildgebung lebender Zellen, da nicht alle Proben für die meisten mikroskopischen Aufbauten zugänglich sind. Die Entwicklung neuer Methoden, um Hypothesen zunächst unter qualifizierten In-vitro-Bedingungen zu testen, ist ebenfalls notwendig und zeitgemäß, da Wissenschaft und Gesellschaft eine tierversuchsfreie Forschung anstreben, um die Zahl der getöteten Tiere zu reduzieren.
In einer kürzlich erschienenen Publikation7 untersuchten wir den Einfluss primärer humaner Makrophagen auf die Invasivität des Eindringens von Krebszellen aus einem Tumorsphäroid. Zu diesem Zweck haben wir einen Kollagen-I-basierten Makrophagen-Tumor-Sphäroid-Co-Invasions-Assay etabliert. In diesem Zusammenhang wollten wir die Rolle der schnellen Recyclingwege der Membran-Typ-1-Matrix-Metalloproteinase (MT1-MMP) in Makrophagen aufklären. Wir untersuchten Makrophagen, die für das superprozessive Kinesin KIF16B abgereichert waren, das wir als Haupttreiber des MT1-MMP-Recyclings identifiziert haben, und deren Einfluss auf die invasiven Fähigkeiten von H1299-Grün fluoreszierendem Protein (GFP)-Zellen aus einem festen Sphäroid. KIF16B-depletierte Makrophagen zeigen reduzierte Spiegel des membrangebundenen MT1-MMP auf ihrer Oberfläche, während die H1299-Zellen selbst unbehandelt blieben.
Unseres Wissens wurde keine ähnliche Methode beschrieben, die eine vollständige Bildgebung und Analyse der Makrophagen-Tumor-Sphäroid-Co-Invasion ermöglicht. Obwohl wir uns in unserer jüngsten Veröffentlichung7 auf die Analyse von individuell wandernden Zellen in der Nähe des Sphäroids konzentriert haben, ermöglicht der Assay mehrere weitere Untersuchungen, wie z.B. die Anzahl der eindringenden Stränge, Proteine, die an der Interaktion zwischen Krebszellen und Makrophagen beteiligt sind, die Menge des Kollagen-I-Abbaus oder andere sphäroide Eigenschaften wie die Länge seines Umfangs.
Protokoll
Dieses Protokoll beinhaltet die Verwendung von primären menschlichen Makrophagen, die aus Blutproben von Spendern gewonnen wurden. Gemäß den ethischen Richtlinien des Universitätsklinikums Hamburg-Eppendorf wurden Blutproben entnommen und die Spender entschädigt. Die Weiterverarbeitung der Proben erfolgte nach vorgegebenen Sicherheitsrichtlinien (z.B. Behandlung von ungeprüften Proben). Die Arbeit mit primären humanen Makrophagen in dieser Studie wurde von der Ärztekammer Hamburg als unbedenklich beurteilt.
1. Erzeugung von H1299-Tumorsphäroiden im gerüstfreien Ansatz (3 Tage im Voraus)
- Kultivieren Sie Krebszellen in einem 37 °C Inkubator mit 5 % CO2 in 25 cm2 Zellkulturflaschen (siehe Materialtabelle) mit Krebszellmedium (Dulbeccos modifiziertes Adlermedium [DMEM] + 1 % Penicillin-Streptomycin [Pen-Streptokokken] + 10 % fötales Rinderserum [FBS] + 0,1 mM nichtessentielle Aminosäuren + 2 mM L-Glutamin) bis zu 80 % Konfluenz.
- Waschen Sie die Zellen einmal mit Dulbecco's phosphatgepufferter Kochsalzlösung (DPBS) und lösen Sie sie durch Zugabe von 1 mL Trypsin-Ethylendiamintetraessigsäure (EDTA) (0,25%, Phenol-Rot) für 2 min. Stoppen Sie die enzymatische Reaktion, indem Sie 2 mL Krebszellmedien hinzufügen und die Ablösung unter einem Lichtmikroskop überprüfen.
HINWEIS: Es ist wichtig, dass die Krebszellen getrennt werden und keine Krebszellcluster vorhanden sind. - Die Zellsuspension in ein 15 mL Reaktionsröhrchen überführen und bei 245 x g für 5 min zentrifugieren. Entfernen Sie anschließend die Trypsinlösung durch Pipettieren oder mit einer Pumpe, waschen Sie das Pellet anschließend durch Zugabe von 5 ml DPBS und zentrifugieren Sie es erneut unter den gleichen Bedingungen.
- Resuspendieren Sie das Pellet in 5 mL Krebszellmedium und zählen Sie die Zellen, z. B. mit einer Neubauer-Kammer. 8.000 H1299-GFP-Zellen werden in eine 96-Well-Platte mit extrem niedriger Adhäsion (siehe Materialtabelle) in einem Endvolumen von 25 μl Krebszellmedium überführt.
- Die Kammer wird 3 Tage lang im Inkubator bei 37 °C und 5 % CO2 inkubiert. Überprüfen Sie die Sphäroide unter dem Mikroskop auf Gleichmäßigkeit. Betrachten Sie keine Sphäroide mit einer geringeren Dichte oder nicht gebundene einzelne Krebszellen für weitere Experimente.
2. Präparation von primären humanen Makrophagen aus Spenderblutproben
- Wenn Spenderblutproben nicht sofort für die Zellisolierung verwendet werden, lagern Sie sie bei 4 °C unter Schütteln.
- Übertragen Sie 20 ml Blut aus einem Transfusionsbeutel in ein 50-ml-Reaktionsröhrchen.
- Bereiten Sie anschließend ein neues 50-ml-Röhrchen vor und fügen Sie 15 ml Lymphozytentrennmedium (LSM) hinzu.
- Geben Sie die 20 mL Blut vorsichtig und ohne Vermischung in jedes LSM-haltige Röhrchen und zentrifugieren Sie es bei 450 x g für 30 min bei 4 °C.
- Bereiten Sie in der Zwischenzeit ein neues 50-ml-Röhrchen vor und fügen Sie 10 ml kaltes Roswell Park Memorial Institute (RPMI)-1640-Medium hinzu.
- Nach der Zentrifugation wird die dichte, weiße Phase ("Buffy Coat") innerhalb des bluthaltigen Röhrchens in das vorbereitete RPMI-haltige Röhrchen überführt und das Volumen mit kaltem RPMI auf 50 mL addiert.
- Die Suspension wird bei 450 x g 10 min bei 4 °C zentrifugiert und der Überstand wird verworfen.
- Resuspendieren Sie die Zellen in 10 ml kaltem RPMI.
- Die Mischung erneut bei 450 x g für 10 min bei 4 °C zentrifugieren, den Überstand verwerfen und das Pellet in 50 mL RPMI resuspendieren.
- Zentrifugieren Sie die Probe erneut bei 450 x g für 10 min bei 4 °C, verwerfen Sie den Überstand und resuspendieren Sie das Zellpellet in 1,5 mL kaltem Monozytenpuffer (0,5 % Humanserumalbumin + 5 mM EDTA in 25 mL DPBS).
- Geben Sie die Zellsuspension auf Eis, fügen Sie 250 μl Anti-CD14-Mikrokügelchen hinzu und inkubieren Sie 15 Minuten lang auf Eis.
- Bereiten Sie eine Trennsäule vor, indem Sie einen Filter an einem magnetischen Abscheidergestell hinzufügen und 900 μl Monozytenpuffer zur Äquilibrierung hinzufügen.
- Gießen Sie die Zellsuspension auf den Filter und lassen Sie sie durch die Schwerkraft in ein Abfallrohr fließen.
- Waschen Sie die Säule, die die an die magnetischen Kügelchen gebundenen Zellen enthält, indem Sie 1 ml Monozytenpuffer hinzufügen.
- Bereiten Sie ein frisches 50-ml-Röhrchen mit 20 mL RPMI vor und setzen Sie das Abfallröhrchen unter der Säule wieder ein.
- Geben Sie 3 ml Monozytenpuffer in die Säule, nehmen Sie ihn aus dem Magnetgestell und bringen Sie den Stempel an.
- Drücken Sie die Zellen in das vorbereitete 50 mL Röhrchen und füllen Sie es bis zu 30 mL Volumen mit RPMI.
- Zählen Sie die Zellen (z.B. mit einer Neubauer-Zählkammer) unter einem Lichtmikroskop und passen Sie die Zellzahl an, indem Sie RPMI auf 2 x 106 Zellen/ml hinzufügen.
- 1 ml der Zellsuspension in jede Vertiefung einer 6-Well-Kammer säen und die Platte 2-4 Stunden lang bei 37 °C und 5 % CO2 inkubieren.
- Überprüfen Sie, ob die Zellen richtig haften, und ersetzen Sie den RPMI durch 1,5 ml Monomedium (20 % Humanserum + 1 % Pen-Streptokokken in RPMI).
- Wechseln Sie das Medium nach 24 Stunden Inkubation. Unter diesen Bedingungen differenzieren sich Monozyten innerhalb von 6 Tagen zu Makrophagen.
3. Vorbereitung der Makrophagen
- Am Tag des Experiments werden die Makrophagen mit einer für die einzelnen Kulturbedingungen geeigneten Menge Accutase abgelöst. Verwenden Sie zum Beispiel 500 μl Accutase, wenn die Zellen in einer 6-Well-Schale ausgesät werden. Inkubieren Sie die Zellen ca. 40 min in der Lösung, waschen Sie sie einmal mit 2 mL DPBS und zählen Sie sie mit einer Neubauer-Kammer.
- Für ein Sphäroid verdünnen Sie die gewünschte Anzahl von Makrophagen in 40 μl Kollagen I (2,5 mg/mL Rattenschwanzkollagen I) und mischen Sie sie, indem Sie das Reaktionsröhrchen kurz vortexen. In den durchgeführten Experimenten haben 200.000 Zellen/ml einen nachweisbaren Unterschied bei der Invasion von Krebszellen gezeigt. Bewahren Sie den Kollagen-Zell-Mix auf Eis auf, da er bei Raumtemperatur (RT) schnell zu polymerisieren beginnt.
4. Aufbau des Co-Invasions-Assays
HINWEIS: Abhängig vom Format der Bildgebungskammer, dem Zweck der Analyse oder dem jeweiligen Bildgebungsaufbau kann es erforderlich sein, das Tumorsphäroid von der Platte mit extrem niedriger Adhäsion zu übertragen und die Werte für Kollagen und Medien anzupassen. Die folgenden Schritte beschreiben die Einrichtung für eine Probe in einem 15-Well-μ-Objektträger.
- Waschen Sie zuerst das Sphäroid, indem Sie 300 μl DPBS hinzufügen. Schneiden Sie die Spitze einer blauen 1-ml-Pipettenspitze ab und nehmen Sie das Sphäroid mit DPBS auf. Nachdem sich das Sphäroid in der Spitze abgesetzt hat, schieben Sie die Pipette kurz an den Boden der Bildgebungskammer, um das Tumor-Sphäroid durch Oberflächenspannung zu übertragen.
HINWEIS: Es ist wichtig zu beachten, dass die Reibung an unveränderten Spitzen die Integrität des Sphäroids stören kann. - Entfernen Sie mit dem Sphäroid übertragene DPBS so weit wie möglich durch Pipettieren.
- Geben Sie schnell 40 μl Kollagen-I/Makrophagen-Mix (siehe Schritt 3.4) in die Bildgebungskammer, die das H1299-GFP-Sphäroid enthält.
- Übertragen Sie die Platte in einen Zellinkubator und inkubieren Sie die Platte 30 Minuten lang bei 37 °C und 5 % CO2 unter feuchten Bedingungen, um die Kollagenmischung vollständig zu polymerisieren.
- Nach der Polymerisation vorsichtig 25 μl Krebszellmedium in jede Vertiefung geben und die Platte 3 Tage lang im Inkubator unter den zuvor beschriebenen Bedingungen inkubieren. Bei Bedarf können Sie die lebende Probe zu den gewünschten Zeitpunkten mit einem Laser-Scanning-Mikroskop abbilden.
5. Zusätzliche Fixierung und Färbung
- Um eine Immunfluoreszenzfärbung durchzuführen, fixieren Sie die Makrophagen und H1299-GFP-Sphäroide in der Kollagen-I-Matrix. Entfernen Sie zunächst vorsichtig den Überstand von jeder Probe mit einer Pumpe, ohne die Matrix zu berühren.
HINWEIS: Um eine Zerstörung der Probe zu vermeiden, kann der Überstand auch durch manuelles Pipettieren ohne Verwendung einer Pumpe entfernt werden. - Geben Sie 50 μl 3,7 % Formaldehyd in DPBS in jede Vertiefung und inkubieren Sie die Proben 5 Minuten lang auf Eis. Entfernen Sie anschließend die Fixierlösung und fügen Sie erneut 50 μl frische Fixierlösung hinzu. Dieser Schritt ist wichtig, um einen gleichmäßigeren Formaldehydgehalt innerhalb der Vertiefung zu erreichen. Die Proben werden über Nacht bei 4 °C inkubiert.
HINWEIS: Abhängig von der Menge an Kollagen, die ich mische, können sich die Inkubationszeiten auf mehrere Stunden verlängern. - Entfernen Sie das Fixiermittel am nächsten Tag und waschen Sie die Proben durch wiederholte Entnahme von mindestens zwei DPBS, abhängig vom Volumen der Bildgebungskammer. Inkubieren Sie sie kurz für 5 Minuten auf Eis auf einem Shaker, bevor Sie das DPBS durch Pipettieren entfernen. Die Proben sind nun bereit für die Färbung.
- Färbeen Sie die Proben, indem Sie eine 1:100-Mischung aus 4',6-Diamidino-2-phenylindol (DAPI) (1 mg/ml, zum Färben von Zellkernen) und Phalloidin 568 (zum Färben von F-Aktin) mindestens 30 Minuten lang in DPBS einfärben und vor der Bildgebung zweimal mit DPBS waschen.
6. Bildgebung und Analyse verschiedener Parameter des Tumorwachstums
HINWEIS: Die folgenden Schritte können für feste Proben mit lebenden Zellen durchgeführt werden.
- Verwenden Sie für die Beobachtung des Tumor-Sphäroids ein inverses Laser-Scanning-Mikroskop, das mit einem 10-fach-Objektiv ausgestattet ist, das die Abbildung eines Z-Stapels von etwa dem unteren Drittel des Sphäroids ermöglicht.
HINWEIS: Aufgrund der Dicke der Probe kann auch ein 2-Photonen-Mikroskop von Vorteil sein. Für eine optimale Identifizierung einzelner Zellen kann eine höhere Laserleistung und Belichtungszeit erforderlich sein. - Kombinieren Sie die Stapel nach der Erfassung durch Z-Projektion mit Fiji, der ImageJ-Software (Fiji). Des Weiteren wird der resultierende Kanal des H1299-GFP-Signals durch Anwenden eines automatischen Schwellenwerts (z. B. Huang Auto-Threshold) verarbeitet und das Restrauschen durch Entfernen von Flecken im Bild in Abhängigkeit von der Menge des Hintergrunds und der Signalintensität entfernt.
- Messen Sie die Sphäroidparameter mit Fiji.
- Bereich: Messen Sie die Größe des Sphäroids mit dem Zauberstab-Werkzeug .
- Umfang: Beziehen Sie den Umfang in die Messeinstellungen ein.
- Durchmesser: Beziehen Sie den Feret-Durchmesser in die Messeinstellungen ein.
- Zirkularität: Beziehen Sie die Formdeskriptoren in die Messeinstellungen ein.
- Messen Sie die einzelnen Parameter der Krebszellen mit Fiji.
- Messen Sie die Anzahl der eindringenden Zellen.
- Wählen Sie mit dem Zauberstab-Werkzeug den zentralen Sphäroid und löschen Sie das Signal. Dadurch werden das Sphäroid sowie alle damit verbundenen Signale von Zellen, die noch an das Sphäroid gebunden sind, aus der Analyse entfernt.
- Verwenden Sie anschließend das Werkzeug Partikel analysieren oder quantifizieren Sie alle verbleibenden Einzelsignale.
HINWEIS: Das Ergebnis ist die Anzahl der eindringenden Krebszellen. Darüber hinaus könnte das Ergebnis auch den repräsentativen Bereich zeigen, der von den Krebszellen bedeckt ist, und je nach Größe des Bereichs als kollektive Invasion oder Einzelzellinvasion interpretiert werden. Um die Anzahl der eindringenden Krebszellen weiter zu validieren, konnte eine Maske der analysierten Partikel über den DAPI-Kanal gelegt und die Zellkerne gezählt werden.
- Messen Sie die Anzahl der eindringenden Zellen.
- Werten Sie die Ergebnisse außerdem mit einem Statistikprogramm wie GraphPad Prism oder Microsoft Excel aus.
HINWEIS: Dieses Protokoll hat sich sowohl als robust als auch als flexibel erwiesen. Es wurde in unserem Labor mehr als 50 Mal verwendet, immer mit primären menschlichen Makrophagen von mindestens 3 Spendern, um die Variabilität der Spender zu berücksichtigen. In seiner jetzigen Form hat sich gezeigt, dass das Protokoll zu einer Erfolgsquote von 90-95% führt. Da der Aufbau jedoch komplex ist, können auch unterschiedliche Ergebnisse eine Rolle spielen, da die Interaktion der Makrophagen für jedes Sphäroid individuell ist. Es wird daher empfohlen, das Experiment mindestens 3 Mal mit 5 Sphäroiden durchzuführen, um ein statistisch signifikantes Ergebnis zu erhalten.
Ergebnisse
Abbildung 1 zeigt einen H1299-GFP-Sphäroid, der an den jeweiligen Inkubationstagen in einer Kollagen-I-Matrix abgebildet wurde. Ein entsprechendes Hellfeldbild, das an Tag 0 aufgenommen wurde, zeigt auch die primären menschlichen Makrophagen, die mit dem Sphäroid kokultiviert wurden. Das repräsentative Bild, das an Tag 3 des Experiments aufgenommen wurde, wird nach der Verarbeitung vergrößert. Es werden verschiedene Parameter angegeben, die analysiert werden können, darunter die Anzahl der eindringenden Zellen, die Orte der kollektiven Invasion und der Sphäroidumfang. Die beigefügte Tabelle zeigt die Ergebnisse, die aus diesem Bild gewonnen wurden. Die Anzahl der erkannten Signale entspricht dem visuellen Eindruck. Es sind keine Störungen des zentralen Sphäroids sichtbar, was auf eine tatsächliche Invasion einzelner Krebszellen und nicht auf Zellen hindeutet, die aus Sphäroidtrümmern stammen. Die Ergebnisse verschiedener Sphäroide und Bedingungen können nun über statistische Analysen verglichen werden.
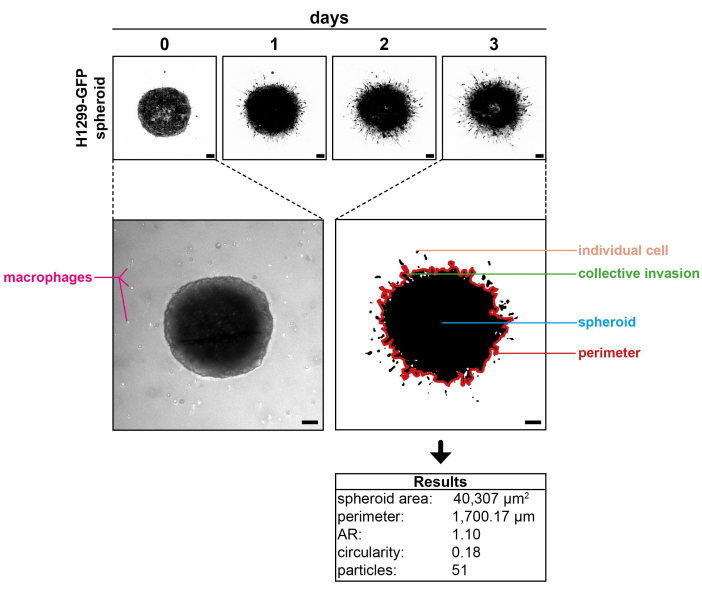
Abbildung 1: Repräsentative Ergebnisse. (Bild oben) H1299-GFP-Sphäroide, aufgenommen an Tag 0, Tag 1, Tag 2 und Tag 3 der Inkubation in einer Kollagen-I-Matrix. (Mittleres linkes Feld) Ein Hellfeldbild des Sphäroids, das die in Kokultur gezüchteten primären menschlichen Makrophagen zeigt. (Mittleres rechtes Feld) Ein repräsentatives Bild, das an Tag 3 aufgenommen wurde, vergrößerte sich nach der Verarbeitung und zeigt die verschiedenen Parameter, die analysiert werden können (Sphäroidumfang in rot; manuell nachgezeichnet zur besseren Visualisierung). (Untere Platte) Die Ergebnisse, die aus dem Bild in der Mitte rechts erhalten wurden. Hinweis: Makrophagen sind nicht sichtbar, da nur der GFP-Kanal aufgezeichnet wurde, um das Verhalten der Krebszellen aufzuklären. Maßstabsbalken: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Invasion von Krebszellen ist nach wie vor ein wichtiges, aber wenig untersuchtes Thema im Zusammenhang mit der Mitinvasion von Immunzellen wie Makrophagen. Die kollektive und individuelle Invasion von Krebszellen sind kritische Prozesse während der Metastasierung und es hat sich gezeigt, dass sie die Überlebensrate von Krebspatienten aufgrund der Vielzahl von Befall verschiedener Organe senken 8,9. In-vivo-Studien sind komplex und auf Laboratorien mit Zugang zu Tierhaltungseinrichtungen beschränkt. Darüber hinaus ist es auch schwierig, die Bedingungen in vivo zu kontrollieren oder einzelne Aspekte gezielt zu manipulieren. Daher ist ein besser zugängliches System nach wie vor unerlässlich, um grundlegende Fragen in der ersten Forschungslinie zu beantworten.
Mit der hier vorgestellten Methode haben wir eine Methode entwickelt, mit der i) das Verhalten von Immunzellen und ii) Krebszellen, iii) das Wachstum und die Entwicklung eines festen Sphäroids und iv) der Einfluss von Zellen in der umgebenden Tumormikroumgebung (TME) auf EZM-Komponenten weiter analysiert werden können. Es kann verwendet werden, um den Einfluss modifizierter (z.B. durch siRNA-Behandlung für bestimmte Regulatoren erschöpfter) Makrophagen auf die Invasivität einzelner Krebszellen von einem soliden Sphäroid aus zu vergleichen. Ob die physikalische Umlagerung der TME oder sezernierte Faktoren die Hauptursache für das beobachtete Verhalten von Krebszellen darstellt, ist derzeit unklar und muss genauer analysiert werden.
Darüber hinaus könnten auch die Krebszellen selbst manipuliert werden, zum Beispiel durch siRNA-Behandlung oder Knockout bestimmter Regulatoren. Darüber hinaus kann die Analyse durch den Vergleich der Anzahl der gezählten Zellkerne innerhalb des identifizierten Zellprofils verbessert werden, um eine genauere Bestimmung der eindringenden Krebszellen zu ermöglichen.
Wir haben diesen Assay verwendet, um den Einfluss von Makrophagen in der TME auf die Invasion von Tumorzellen zu bestimmen. Es sollte jedoch auch beachtet werden, dass Tumorzellen wahrscheinlich die Aktivität von Makrophagen beeinflussen, möglicherweise durch sezernierte Faktoren innerhalb des Mediums. Es wäre daher auch ein lohnendes Unterfangen, Veränderungen in den Makrophagen, wie z.B. einen veränderten Polarisationsstatus (M1 vs. M2), durch Immunfluoreszenzfärbung mit entsprechenden Antikörpern zu identifizieren. In der Vergangenheit hat der Vergleich zwischen Zellen, die in Monoschichten wachsen, und solchen, die in einer 3D-Umgebung kultiviert wurden, signifikante Unterschiede in ihren Expressionsprofilen gezeigt10.
Darüber hinaus könnte die fluoreszenzaktivierte Zellsortierung (FACS) von Makrophagen-Subpopulationen und deren Integration in das experimentelle Verfahren aufschlussreich sein. Nicht zuletzt könnte eine detailliertere Bildgebung der Kontaktbereiche, an denen Makrophagen mit Krebszellen interagieren, oder der sphäroiden Oberfläche des Tumors zur Identifizierung weiterer Mechanismen führen, die für die 3D-Invasion und -Interaktion relevant sind.
Bemerkenswert, was nicht vollständig kontrolliert werden kann, ist die genaue Positionierung des Sphäroids in der Mitte der Vertiefung nach der Zugabe des Kollagen-Mixes. Die Menge an Kollagen unter dem Sphäroid ist besonders schwer zu regulieren. Hier haben sich andere Verfahren etabliert, um eine präzise Positionierung des Sphäroids zu ermöglichen, z. B. auf Agaroseformen mit höheren Probenzahlen11. Da die Freisetzung von Zytokinen jedoch ein gemeinsamer Mechanismus ist, sind alle Sphäroide in diesem Multi-Sphäroid-Assay sezernierten Faktoren ausgesetzt, und die Anzahl der Immunzellen, die auf ein einzelnes Sphäroid wirken, ist schwer zu kontrollieren.
Eine grundlegende Einschränkung dieses Protokolls ist die Fähigkeit von Krebszelllinien, einheitliche Sphäroide zu bilden, die daher nur auf eine Untergruppe von Zelllinien anwendbar sind. Zum Beispiel bilden MeWo-Melanomzellen ungleichmäßige, blattartige 3D-Strukturen, aber keine einheitlichen Sphäroide.
Es sollte auch beachtet werden, dass der Assay sehr anpassungsfähig ist, da viele seiner Merkmale modifiziert werden können, wie z. B. das EZM-Material, die Zellzahl oder durch Zugabe bestimmter Faktoren wie Zytokine zum Überstand. Es sollte sich daher sehr gut für erste in vitro-Studien der Interaktion zwischen Krebszelle und Immunzelle eignen und kann auf die spezifische Forschungsfrage zugeschnitten werden, die derzeit bearbeitet wird.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Die Autoren danken Andrea Mordhorst für die hervorragende technische Unterstützung und Zellkultur sowie Martin Aepfelbacher für die kontinuierliche Unterstützung. Die Arbeiten zur Makrophageninvasion im SL-Labor werden gefördert durch die Deutsche Forschungsgemeinschaft (SFB 877/B13; LI925/13-1).
Materialien
| Name | Company | Catalog Number | Comments |
| 15 µ-Slide Angiogenesis | ibidi | 81506 | |
| Accutase | Invitrogen | 00-4555-56 | |
| Alexa Fluor 568 Phalloidin | ThermoFisher Scientific | A12380 | |
| CD14 MicroBeads | Miltenyi Biotec | 130-050-201 | |
| CO2 Incubator | Binder | ||
| Collagen I Rat Tail | Corning | 354236 | |
| DAPI | AppliChem | ||
| DMEM (1x) + GlutaMAX | Gibco | 31966-021 | |
| DPBS | Anprotec | MS01Y71003 | |
| FBS | Bio&Cell | FBS. S 0613 | |
| Fiji | NIH | ImageJ 2 Version: 2.3.0/1.53s | |
| Formaldehyde 37% | 252549-500ml | Sigma-Aldrich | |
| H1299-GFP cell line | |||
| Human serum albumin | Sigma-Aldrich | A5843 | |
| Leica TCS SP8 X | Leica | ||
| l-glutamine | Gibco | 25030-024 | |
| Lymphocyte Seperation Medium (LSM) 1077 | PromoCell | C-44010 | |
| MS Columns | Miltenyi Biotec | 130-042-201 | |
| nonessential amino-acids | Sigma-Aldrich | 11140050 | |
| Pen Strep | Gibco | 15140-122 | |
| RPMI-1640 | Gibco | 81275-034 | |
| TC-Platte 96 Well, BIOFLOAT, R | SARSTEDT | 83,39,25,400 | |
| VORTEX-GENE 2 | Scientific Industries |
Referenzen
- Lendeckel, U., Venz, S., Wolke, C. Macrophages: shapes and functions. ChemTexts. 8 (2), 12 (2022).
- Dallavalasa, S., et al. The role of tumor associated macrophages (TAMs) in cancer progression, chemoresistance, angiogenesis and metastasis - Current status. Curr Med Chem. 28 (39), 8203-8236 (2021).
- Pan, Y., Yu, Y., Wang, X., Zhang, T. Tumor-associated macrophages in tumor immunity. Front Immunol. 11, 583084 (2020).
- Boutilier, A. J., Elsawa, S. F. Macrophage Polarization States in the Tumor Microenvironment. Int J Mol Sci. 22 (13), 6995 (2021).
- Wenzel, E. M., et al. Intercellular transfer of cancer cell invasiveness via endosome-mediated protease shedding. Nat Commun. 15 (1), 1277 (2024).
- Anfray, C., Ummarino, A., Calvo, A., Allavena, P., Torres Andon, F. In vivo analysis of tumor-associated macrophages in the tumor microenvironment. Methods Mol Biol. 2614, 93-108 (2023).
- Hey, S., Wiesner, C., Barcelona, B., Linder, S. KIF16B drives MT1-MMP recycling in macrophages and promotes co-invasion of cancer cells. Life Sci Alliance. 6 (11), e202302158 (2023).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Shi, X., et al. Mechanism insights and therapeutic intervention of tumor metastasis: latest developments and perspectives. Signal Transduct Target Ther. 9 (1), 192 (2024).
- Li, G. N., Livi, L. L., Gourd, C. M., Deweerd, E. S., Hoffman-Kim, D. Genomic and morphological changes of neuroblastoma cells in response to three-dimensional matrices. Tissue Eng. 13 (5), 1035-1047 (2007).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. 160, e61392 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten