Method Article
Un test de co-invasion macrophage-tumeur sphéroïde
Dans cet article
Résumé
Ici, nous présentons un protocole pour étudier l’interaction des macrophages primaires dérivés de monocytes humains avec un sphéroïde tumoral dans une matrice tridimensionnelle (3D) de collagène I, avec la possibilité de comparer l’impact des propriétés solubles et physiques du microenvironnement sur l’invasion cellulaire.
Résumé
L’interaction entre les cellules immunitaires et cancéreuses et leur impact respectif sur les métastases représentent un aspect important de la recherche sur le cancer. Jusqu’à présent, seuls quelques protocoles sont disponibles qui permettent une approximation in vitro de la situation in vivo . Ici, nous présentons une nouvelle approche pour observer l’impact des macrophages humains sur le caractère invasif des cellules cancéreuses, en utilisant des sphéroïdes tumoraux de cellules de carcinome pulmonaire non à petites cellules H1299 intégrées dans une matrice tridimensionnelle (3D) de collagène I. Avec cette configuration de co-culture, nous avons testé l’impact de la déplétion des facteurs de régulation basée sur les petits ARN interférents (siRNA) dans les macrophages sur l’invasion 3D des cellules cancéreuses à partir du sphéroïde tumoral par rapport aux témoins. Cette méthode permet de déterminer différents paramètres, tels que la surface des sphéroïdes ou le nombre de cellules cancéreuses envahissantes, et ainsi, de détecter les différences d’invasion des cellules cancéreuses. Dans cet article, nous présentons la configuration respective, discutons de l’analyse ultérieure, ainsi que des avantages et des pièges potentiels de cette méthode.
Introduction
Les macrophages sont une partie importante du système immunitaire inné et représentent la première ligne de défense dans de nombreuses conditions pathologiques, telles que les infections ou l’élimination des débris cellulaires après des blessures1. Au cours des dernières décennies, l’impact des cellules immunitaires sur la progression du cancer a fait l’objet de nombreuses études. En conséquence, il a été démontré que les macrophages peuvent faciliter les métastases en s’associant aux tumeurs primaires et en devenant des macrophages associés aux tumeurs (TAMs)2. Les macrophages peuvent modifier leur profil d’expression lorsqu’ils sont exposés aux cellules cancéreuses, favorisant l’échappement des métastases du système immunitaire3. De plus, il a été démontré que les cellules cancéreuses peuvent utiliser les défauts de la matrice extracellulaire (MEC) générés par les macrophages pour échapper à la tumeur primaire et que leur comportement est également manipulé par l’absorption de facteurs sécrétés, y compris les vésicules extracellulaires (VE)4,5. Cette interaction d’aspects physiques et chimiques nécessite le développement de nouvelles méthodes pour caractériser l’impact des cellules immunitaires sur la propagation tumorale et le microenvironnement environnant.
Différentes approches ont été développées pour étudier le comportement des cellules immunitaires dans le contexte des métastases in vivo6. Cependant, comme pour toutes les expériences incluant des animaux, une licence pour l’expérimentation animale et une animalerie sont nécessaires ; Ces approches nécessitent déjà beaucoup de développement et de préparation. De plus, l’analyse est souvent compliquée, notamment en ce qui concerne l’imagerie de cellules vivantes, car tous les échantillons ne sont pas accessibles pour la plupart des configurations microscopiques. La mise au point de nouvelles méthodes pour tester d’abord des hypothèses dans des conditions in vitro qualifiées est également nécessaire et opportune, car la science et la société visent une recherche sans animaux afin de réduire le nombre d’animaux sacrifiés.
Dans une publication récente7, nous avons étudié l’impact des macrophages humains primaires sur le caractère invasif des cellules cancéreuses envahissantes d’un sphéroïde tumoral. À cette fin, nous avons mis au point un test de co-invasion de sphéroïdes de tumeurs à base de collagène I. Dans ce contexte, nous avons cherché à élucider le rôle des voies de recyclage rapide de la métalloprotéinase matricielle de type 1 membranaire (MT1-MMP) dans les macrophages. Nous avons testé des macrophages qui étaient épuisés pour la kinésine super processive KIF16B, que nous avons identifiée comme un moteur majeur du recyclage de MT1-MMP, et leur impact sur les capacités invasives des cellules H1299-Green Fluorescent Protein (GFP) d’un sphéroïde solide. Les macrophages appauvris en KIF16B présentent des niveaux réduits de MT1-MMP liés à la membrane à leur surface, tandis que les cellules H1299 elles-mêmes n’ont pas été traitées.
À notre connaissance, aucune méthode similaire n’a été décrite qui permette une imagerie et une analyse complètes de la co-invasion macrophage-tumeur-sphéroïde. Bien que nous nous soyons concentrés dans notre récente publication7 sur l’analyse de cellules migratrices individuelles éloignées du sphéroïde, le test permet de multiples études supplémentaires telles que le nombre de brins envahisseurs, les protéines impliquées dans l’interaction cellule-macrophage du cancer, la quantité de dégradation du collagène I ou d’autres propriétés sphéroïdes comme la longueur de son périmètre.
Protocole
Ce protocole implique l’utilisation de macrophages humains primaires dérivés d’échantillons de sang de donneurs. Selon les directives éthiques du centre médical universitaire de Hambourg-Eppendorf, des échantillons de sang ont été prélevés et les donneurs ont été rémunérés. Le traitement ultérieur des échantillons a été effectué conformément aux directives de sécurité données (p. ex., traitement d’échantillons non analysés). Les travaux sur les macrophages humains primaires dans cette étude ont été jugés non répréhensibles par Ärztekammer Hamburg, en Allemagne.
1. Génération de sphéroïdes tumoraux H1299 dans une approche sans échafaudage (3 jours à l’avance)
- Cultivez des cellules cancéreuses dans un incubateur à 37 °C avec 5 % de CO2 dans des flacons deculture cellulaire de 25 cm (voir le tableau des matériaux) avec un milieu de cellules cancéreuses (milieu d’aigle modifié de Dulbecco [DMEM] + 1 % de pénicilline-streptomycine [pen-streptomycine] + 10 % de sérum de veau fœtal [FBS] + 0,1 mM d’acides aminés non essentiels + 2 mM de L-glutamine) jusqu’à ce que 80 % de confluence soit atteinte.
- Lavez les cellules une fois avec la solution saline tamponnée au phosphate de Dulbecco (DPBS) et détachez-les en ajoutant 1 ml d’acide trypsine-éthylènediaminetétraacétique (EDTA) (0,25 %, rouge de phénol) pendant 2 min. Arrêtez la réaction enzymatique en ajoutant 2 ml de milieu cellulaire cancéreux et vérifiez le décollement au microscope optique.
REMARQUE : Il est important que les cellules cancéreuses soient séparées et qu’il n’y ait pas d’amas de cellules cancéreuses. - Transférez la suspension cellulaire dans un tube de réaction de 15 mL et centrifugez-la à 245 x g pendant 5 min. Ensuite, retirez la solution de trypsine par pipetage ou à l’aide d’une pompe, lavez ensuite la pastille en ajoutant 5 ml de DPBS et centrifugez-les à nouveau dans les mêmes conditions.
- Remettez la pastille en suspension dans 5 mL de milieu cellulaire cancéreux et comptez les cellules, par exemple avec une chambre Neubauer. Transférez 8 000 cellules H1299-GFP dans une plaque à très faible adhérence de 96 puits (voir le tableau des matériaux) dans un volume final de 25 μL de milieu cellulaire cancéreux.
- Incuber la chambre pendant 3 jours dans l’incubateur à 37 °C et 5 % de CO2. Vérifiez l’uniformité des sphéroïdes au microscope. Ne considérez pas les sphéroïdes à faible densité ou les cellules cancéreuses individuelles non attachées pour d’autres expériences.
2. Préparation de macrophages humains primaires à partir d’échantillons de sang de donneurs
- Si les échantillons de sang du donneur ne sont pas utilisés immédiatement pour l’isolement cellulaire, conservez-les à 4 °C, en les agitant.
- Transférez 20 ml de sang d’une poche de transfusion dans un tube de réaction de 50 ml.
- Par la suite, préparez un nouveau tube de 50 mL et ajoutez 15 mL de milieu de séparation des lymphocytes (LSM).
- Ajouter délicatement les 20 ml de sang dans chaque tube contenant du LSM sans mélanger et le centrifuger à 450 x g pendant 30 min à 4 °C.
- Entre-temps, préparez un nouveau tube de 50 ml et ajoutez 10 ml de milieu froid Roswell Park Memorial Institute (RPMI)-1640.
- Après la centrifugation, transférez la phase dense et blanche (« couche leucocytaire ») à l’intérieur du tube contenant du sang dans le tube contenant du RPMI préparé et augmentez le volume à 50 mL avec du RPMI froid.
- Centrifuger la suspension à 450 x g pendant 10 min à 4 °C et jeter le surnageant.
- Remettre les cellules en suspension dans 10 mL de RPMI froid.
- Centrifugez à nouveau le mélange à 450 x g pendant 10 min à 4 °C, jetez le surnageant et remettez la pastille en suspension dans 50 mL de RPMI.
- Centrifuger à nouveau l’échantillon à 450 x g pendant 10 min à 4 °C, jeter le surnageant et remettre en suspension la pastille cellulaire dans 1,5 mL de tampon monocytaire froid (0,5 % d’albumine sérique humaine + 5 mM d’EDTA dans 25 mL de DPBS).
- Mettez la suspension cellulaire sur de la glace, ajoutez 250 μL de microbilles anti-CD14 et incubez pendant 15 min sur de la glace.
- Préparez une colonne de séparation par ajout d’un filtre sur une grille de séparateur magnétique et ajoutez 900 μL de tampon monocytaire pour l’équilibrage.
- Versez la suspension cellulaire sur le filtre et laissez-la s’écouler dans un tube de vidage par gravité.
- Lavez la colonne, qui contient les cellules liées aux billes magnétiques, en ajoutant 1 mL de tampon monocytaire.
- Préparez un tube frais de 50 ml contenant 20 ml d’IPRP et replacez le tube de déchets sous la colonne.
- Ajoutez 3 ml de tampon monocytaire à la colonne, retirez-la du support magnétique et fixez le tampon.
- Pressez les cellules dans le tube de 50 ml préparé et remplissez-le jusqu’à 30 ml de volume avec RPMI.
- Comptez les cellules (p. ex., à l’aide d’une chambre de comptage Neubauer) au microscope optique et ajustez le nombre de cellules en ajoutant RPMI à 2 x 106 cellules/mL.
- Semez 1 mL de suspension cellulaire dans chaque puits d’une chambre à 6 puits et incubez la plaque pendant 2 à 4 h à 37 °C et 5 % de CO2.
- Vérifiez si les cellules ont bien adhéré et remplacez l’IPRP par 1,5 mL de monomilieu (20 % de sérum humain + 1 % de pen-streptocoque dans l’IPR).
- Changer de milieu après 24 h d’incubation. Dans ces conditions, les monocytes se différencient en macrophages en 6 jours.
3. Préparation des macrophages
- Le jour de l’expérience, détachez les macrophages avec une quantité appropriée d’accutase pour les conditions de culture individuelles. Par exemple, utilisez 500 μL d’accutase si les cellules sont ensemencées dans une boîte à 6 puits. Incuber les cellules pendant environ 40 minutes dans la solution, les laver une fois avec 2 mL de DPBS et les compter avec une chambre Neubauer.
- Pour un sphéroïde, diluez le nombre désiré de macrophages dans 40 μL de collagène I mélangé (2,5 mg/mL de collagène I de queue de rat) et mélangez-les en vortex brièvement le tube réactionnel. Dans les expériences menées, 200 000 cellules/mL ont montré une différence détectable sur l’invasion des cellules cancéreuses. Gardez le mélange de cellules de collagène sur de la glace, car il commencera à polymériser rapidement à température ambiante (RT).
4. Mise en place de l’essai de co-invasion
REMARQUE : Selon le format de la chambre d’imagerie, l’objectif de l’analyse ou la configuration d’imagerie respective, il peut être nécessaire de transférer le sphéroïde tumoral à partir de la plaque à très faible adhérence et d’adapter les valeurs de collagène et de milieu. Les étapes suivantes décrivent la configuration d’un échantillon dans une lame de μ à 15 puits.
- Tout d’abord, lavez le sphéroïde en ajoutant 300 μL de DPBS. Coupez l’extrémité d’une pointe de pipette bleue de 1 mL et prélevez le sphéroïde avec du DPBS. Une fois que l’aspiration du sphéroïde s’est installée dans la pointe, poussez brièvement la pipette au fond de la chambre d’imagerie pour transférer le sphéroïde tumoral par tension superficielle.
REMARQUE : Il est important de noter que le frottement aux extrémités non modifiées peut perturber l’intégrité du sphéroïde. - Éliminez autant que possible les DPBS résiduels transférés avec le sphéroïde par pipetage.
- Ajoutez rapidement 40 μL de mélange de collagène I/macrophage (voir étape 3.4) dans la chambre d’imagerie contenant le sphéroïde H1299-GFP.
- Transférez la plaque dans un incubateur cellulaire et incubez la plaque pendant 30 min à 37 °C et 5 % de CO2 dans des conditions humides pour polymériser complètement le mélange de collagène.
- Après la polymérisation, ajoutez soigneusement 25 μL de milieu de cellules cancéreuses dans chaque puits et incubez la plaque pendant 3 jours dans l’incubateur dans les conditions décrites précédemment. Si nécessaire, imagez l’échantillon vivant avec un microscope à balayage laser aux points de temps souhaités.
5. Fixation et coloration supplémentaires
- Afin d’effectuer une coloration par immunofluorescence, fixez les macrophages et les sphéroïdes H1299-GFP dans la matrice de collagène I. Tout d’abord, retirez soigneusement le surnageant de chaque échantillon à l’aide d’une pompe sans toucher la matrice.
REMARQUE : Pour éviter la destruction de l’échantillon, le surnageant peut également être éliminé par pipetage manuel sans utiliser de pompe. - Ajouter 50 μL de formaldéhyde à 3,7 % dans du DPBS dans chaque puits et incuber les échantillons pendant 5 minutes sur de la glace. Ensuite, retirez la solution fixatrice et ajoutez-y à nouveau 50 μL de solution de fixation fraîche. Cette étape est importante pour obtenir un niveau de formaldéhyde plus uniforme dans le puits. Incuber les échantillons pendant la nuit à 4 °C.
REMARQUE : Selon la quantité de collagène que je mélange, les temps d’incubation peuvent s’étendre à plusieurs heures. - Retirez le fixateur le lendemain et lavez les échantillons en les retirant à plusieurs reprises d’au moins deux fois DPBS en fonction du volume de la chambre d’imagerie. Incuber brièvement 5 min sur de la glace sur un shaker avant de retirer le DPBS par pipetage. Les échantillons sont maintenant prêts pour la coloration.
- Colorez les échantillons en ajoutant un mélange 1:100 de 4',6-diamidino-2-phénylindole (DAPI) (1 mg/mL, pour colorer les noyaux) et de phalloïdine 568 (pour colorer l’actine F) dans le DPBS pendant au moins 30 minutes et lavez-les deux fois avec du DPBS avant l’imagerie.
6. Imagerie et analyse de différents paramètres de croissance tumorale
REMARQUE : Les étapes suivantes peuvent être effectuées pour des échantillons d’imagerie de cellules vivantes fixes.
- Pour l’observation de la tumeur-sphéroïde, utilisez un microscope à balayage laser inversé équipé d’un objectif 10x qui permet d’imager un empilement z d’environ le tiers inférieur du sphéroïde.
REMARQUE : En raison de l’épaisseur de l’échantillon, un microscope à 2 photons peut également être avantageux. Pour une identification optimale des cellules individuelles, une puissance laser et un temps d’exposition plus élevés peuvent être nécessaires. - Après l’acquisition, combinez les piles par projection z à l’aide de Fiji, qui est le logiciel ImageJ (Fiji). De plus, traitez le canal résultant du signal H1299-GFP en appliquant un seuil automatique (par exemple, le seuil automatique de Huang) et supprimez le bruit résiduel en éliminant les taches sur l’image en fonction de la quantité d’arrière-plan et de l’intensité du signal.
- Mesurez les paramètres du sphéroïde à l’aide de Fidji.
- Surface : Mesurez la taille du sphéroïde à l’aide de l’outil Baguette .
- Périmètre : incluez le périmètre dans les paramètres de mesure.
- Diamètre : Incluez le diamètre du Féret dans les paramètres de mesure.
- Circularité : incluez les descripteurs de forme dans les paramètres de mesure.
- Mesurez les paramètres individuels des cellules cancéreuses à l’aide des Fidji.
- Mesurez le nombre de cellules envahissantes.
- Choisissez le sphéroïde central à l’aide de l’outil Baguette et supprimez le signal. Cela supprimera du sphéroïde ainsi que tous les signaux connectés des cellules, qui sont toujours attachés au sphéroïde, de l’analyse.
- Ensuite, utilisez l’outil Analyser les particules ou quantifiez tous les signaux individuels résiduels.
REMARQUE : Le résultat sera le nombre de cellules cancéreuses envahissantes. De plus, le résultat pourrait également montrer la zone représentative couverte par les cellules cancéreuses et être interprété comme une invasion collective ou une invasion unicellulaire, selon la taille de la zone. Pour valider davantage le nombre de cellules cancéreuses envahissantes, un masque des particules analysées a pu être posé au-dessus du canal DAPI, et les noyaux ont été comptés.
- Mesurez le nombre de cellules envahissantes.
- De plus, évaluez les résultats à l’aide d’un programme statistique tel que GraphPad Prism ou Microsoft Excel.
REMARQUE : Ce protocole s’est avéré à la fois robuste et flexible. Il a été utilisé dans notre laboratoire plus de 50 fois, toujours avec des macrophages humains primaires provenant d’au moins 3 donneurs, pour tenir compte de la variabilité des donneurs. Dans sa forme actuelle, il a été démontré que le protocole conduit à un taux de réussite de 90 à 95 %. Cependant, comme la configuration est complexe, des résultats variables peuvent également être un résultat, car l’interaction des macrophages pour chaque sphéroïde est individuelle. Ainsi, il est suggéré d’effectuer l’expérience au moins 3 fois avec 5 sphéroïdes pour obtenir un résultat statistiquement significatif.
Résultats
La figure 1 montre un sphéroïde H1299-GFP imagé aux jours respectifs d’incubation dans une matrice de collagène I. Une image en fond clair correspondante, prise au jour 0, montre également les macrophages humains primaires cocultivés avec le sphéroïde. L’image représentative enregistrée au jour 3 de l’expérience est agrandie après traitement. Différents paramètres pouvant être analysés sont indiqués, notamment le nombre de cellules envahissantes, les sites d’invasion collective et le périmètre des sphéroïdes. Le tableau ci-joint montre les résultats obtenus à partir de cette image. Le nombre de signaux détectés correspond à l’impression visuelle. Aucune perturbation du sphéroïde central n’est visible, ce qui indique une véritable invasion de cellules cancéreuses individuelles plutôt que des cellules dérivées de débris de sphéroïdes. Les résultats de différents sphéroïdes et conditions peuvent maintenant être comparés par analyse statistique.
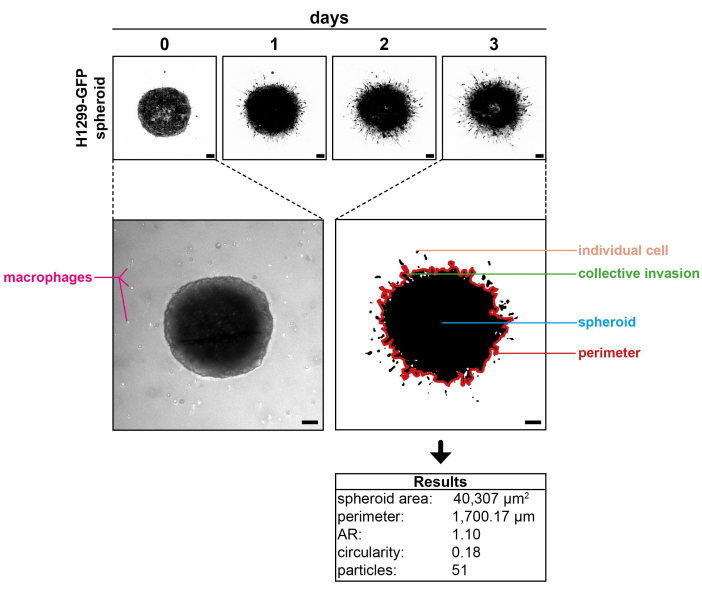
Figure 1 : Résultats représentatifs. (panneau du haut) Imagerie du sphéroïde H1299-GFP au jour 0, au jour 1, au jour 2 et au jour 3 de l’incubation dans une matrice de collagène I. (Panneau central gauche) Une image en fond clair du sphéroïde montrant les macrophages humains primaires cocultivés. (Panneau central droit) Une image représentative acquise au jour 3 agrandie après traitement, montrant les différents paramètres pouvant être analysés (périmètre du sphéroïde en rouge ; tracé manuellement pour une meilleure visualisation). (Panneau inférieur) Les résultats obtenus à partir de l’image du panneau central droit. Remarque : Les macrophages ne sont pas visibles, car seul le canal GFP a été enregistré pour élucider le comportement des cellules cancéreuses. Barres d’échelle : 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
L’invasion des cellules cancéreuses reste un sujet important mais peu étudié dans le contexte de la co-invasion de cellules immunitaires comme les macrophages. L’invasion collective et individuelle des cellules cancéreuses est un processus critique au cours des métastases et il a été démontré qu’elle réduit le taux de survie des patients atteints de cancer en raison de la multiplicité des infestations de différents organes 8,9. Les études in vivo sont complexes et sont limitées aux laboratoires ayant accès à des installations d’hébergement pour animaux. De plus, il est également difficile de contrôler les conditions in vivo ou de manipuler spécifiquement des aspects individuels. Par conséquent, la nécessité d’un système plus accessible reste essentielle pour répondre aux questions fondamentales de la première ligne de recherche.
Avec la méthode présentée ici, nous avons développé une méthode dans laquelle i) les cellules immunitaires et ii) le comportement des cellules cancéreuses, iii) la croissance et le développement d’un sphéroïde solide, et iv) l’impact des cellules du microenvironnement tumoral environnant (TME) sur les composants de la MEC peuvent être analysés plus en détail. Il peut être utilisé pour comparer l’impact des macrophages modifiés (par exemple, appauvris pour des régulateurs spécifiques par le traitement par siRNA) sur le caractère invasif de cellules cancéreuses individuelles à partir d’un sphéroïde solide. Il n’est actuellement pas clair si le réarrangement physique du TME ou les facteurs sécrétés représentent la cause principale du comportement observé des cellules cancéreuses et doit être analysé plus en détail.
En outre, les cellules cancéreuses elles-mêmes pourraient également être manipulées, par exemple par un traitement par siRNA ou l’élimination de régulateurs spécifiques. De plus, l’analyse peut être améliorée en comparant le nombre de noyaux comptés dans le profil cellulaire identifié afin de permettre une détermination plus précise des cellules cancéreuses envahissantes.
Nous avons utilisé ce test pour déterminer l’impact des macrophages dans le TME sur l’invasion des cellules tumorales. Cependant, il convient également de noter que les cellules tumorales sont susceptibles d’influencer l’activité des macrophages, éventuellement par le biais de facteurs sécrétés dans les milieux. Il serait donc également utile d’identifier les changements dans les macrophages, tels que l’altération de l’état de polarisation (M1 vs M2) par coloration par immunofluorescence à l’aide d’anticorps respectifs. Dans le passé, la comparaison entre les cellules cultivées en monocouches et celles cultivées dans un environnement 3D a montré des différences significatives dans leurs profils d’expression10.
De plus, le tri cellulaire activé par fluorescence (FACS) des sous-populations de macrophages et leur intégration dans la procédure expérimentale pourraient être instructifs. Enfin, une imagerie plus détaillée des zones de contact où les macrophages interagissent avec les cellules cancéreuses ou la surface du sphéroïde tumoral pourrait conduire à l’identification d’autres mécanismes pertinents pour l’invasion et l’interaction 3D.
Il est à noter que le positionnement précis du sphéroïde au centre du puits après l’ajout du mélange de collagène ne peut pas être entièrement contrôlé. La quantité de collagène sous le sphéroïde est particulièrement difficile à réguler. Ici, d’autres méthodes ont été mises au point pour permettre un positionnement précis du sphéroïde, par exemple au-dessus de moules d’agarose avec des échantillons de11 plus élevés. Cependant, comme la libération de cytokines est un mécanisme courant, tous les sphéroïdes de ce test multi-sphéroïde sont exposés à des facteurs sécrétés, et le nombre de cellules immunitaires agissant sur un seul sphéroïde est difficile à contrôler.
L’une des limites fondamentales de ce protocole est la capacité des lignées cellulaires cancéreuses à former des sphéroïdes uniformes, ne s’appliquant ainsi qu’à un sous-ensemble de lignées cellulaires. Par exemple, les cellules de mélanome MeWo forment des structures 3D inégales, semblables à des feuilles, mais pas de sphéroïdes uniformes.
Il convient également de noter que le test est très adaptable, car bon nombre de ses caractéristiques peuvent être modifiées, telles que le matériel ECM, le nombre de cellules ou en ajoutant des facteurs spécifiques tels que des cytokines au surnageant. Il devrait donc être parfaitement adapté aux études initiales in vitro de l’interaction cellule cancéreuse/cellule immunitaire et peut être adapté à la question de recherche spécifique actuellement abordée.
Déclarations de divulgation
Les auteurs déclarent qu’il n’existe pas d’intérêts concurrents.
Remerciements
Les auteurs tiennent à remercier Andrea Mordhorst pour l’excellent soutien technique et la culture cellulaire, ainsi que Martin Aepfelbacher pour son soutien continu. Les travaux sur l’invasion des macrophages dans le laboratoire SL sont soutenus par la Deutsche Forschungsgemeinschaft (CRC877/B13 ; LI925/13-1).
matériels
| Name | Company | Catalog Number | Comments |
| 15 µ-Slide Angiogenesis | ibidi | 81506 | |
| Accutase | Invitrogen | 00-4555-56 | |
| Alexa Fluor 568 Phalloidin | ThermoFisher Scientific | A12380 | |
| CD14 MicroBeads | Miltenyi Biotec | 130-050-201 | |
| CO2 Incubator | Binder | ||
| Collagen I Rat Tail | Corning | 354236 | |
| DAPI | AppliChem | ||
| DMEM (1x) + GlutaMAX | Gibco | 31966-021 | |
| DPBS | Anprotec | MS01Y71003 | |
| FBS | Bio&Cell | FBS. S 0613 | |
| Fiji | NIH | ImageJ 2 Version: 2.3.0/1.53s | |
| Formaldehyde 37% | 252549-500ml | Sigma-Aldrich | |
| H1299-GFP cell line | |||
| Human serum albumin | Sigma-Aldrich | A5843 | |
| Leica TCS SP8 X | Leica | ||
| l-glutamine | Gibco | 25030-024 | |
| Lymphocyte Seperation Medium (LSM) 1077 | PromoCell | C-44010 | |
| MS Columns | Miltenyi Biotec | 130-042-201 | |
| nonessential amino-acids | Sigma-Aldrich | 11140050 | |
| Pen Strep | Gibco | 15140-122 | |
| RPMI-1640 | Gibco | 81275-034 | |
| TC-Platte 96 Well, BIOFLOAT, R | SARSTEDT | 83,39,25,400 | |
| VORTEX-GENE 2 | Scientific Industries |
Références
- Lendeckel, U., Venz, S., Wolke, C. Macrophages: shapes and functions. ChemTexts. 8 (2), 12 (2022).
- Dallavalasa, S., et al. The role of tumor associated macrophages (TAMs) in cancer progression, chemoresistance, angiogenesis and metastasis - Current status. Curr Med Chem. 28 (39), 8203-8236 (2021).
- Pan, Y., Yu, Y., Wang, X., Zhang, T. Tumor-associated macrophages in tumor immunity. Front Immunol. 11, 583084 (2020).
- Boutilier, A. J., Elsawa, S. F. Macrophage Polarization States in the Tumor Microenvironment. Int J Mol Sci. 22 (13), 6995 (2021).
- Wenzel, E. M., et al. Intercellular transfer of cancer cell invasiveness via endosome-mediated protease shedding. Nat Commun. 15 (1), 1277 (2024).
- Anfray, C., Ummarino, A., Calvo, A., Allavena, P., Torres Andon, F. In vivo analysis of tumor-associated macrophages in the tumor microenvironment. Methods Mol Biol. 2614, 93-108 (2023).
- Hey, S., Wiesner, C., Barcelona, B., Linder, S. KIF16B drives MT1-MMP recycling in macrophages and promotes co-invasion of cancer cells. Life Sci Alliance. 6 (11), e202302158 (2023).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Shi, X., et al. Mechanism insights and therapeutic intervention of tumor metastasis: latest developments and perspectives. Signal Transduct Target Ther. 9 (1), 192 (2024).
- Li, G. N., Livi, L. L., Gourd, C. M., Deweerd, E. S., Hoffman-Kim, D. Genomic and morphological changes of neuroblastoma cells in response to three-dimensional matrices. Tissue Eng. 13 (5), 1035-1047 (2007).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. 160, e61392 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon