Method Article
Um ensaio de co-invasão de esferoides macrófagos-tumores
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para investigar a interação de macrófagos primários derivados de monócitos humanos com um esferóide tumoral em uma matriz tridimensional (3D) de colágeno I, com a possibilidade de comparar o impacto das propriedades solúveis e físicas do microambiente na invasão celular.
Resumo
A interação de células imunes e cancerígenas e seu respectivo impacto na metástase representa um aspecto importante da pesquisa do câncer. Até o momento, existem apenas alguns protocolos disponíveis que permitem uma aproximação in vitro da situação in vivo . Aqui, apresentamos uma nova abordagem para observar o impacto dos macrófagos humanos na invasividade das células cancerígenas, usando esferoides tumorais de células de carcinoma de pulmão de células não pequenas H1299 incorporadas em uma matriz tridimensional (3D) de colágeno I. Com essa configuração de co-cultivo, testamos o impacto da depleção baseada em RNA de interferência pequena (siRNA) de fatores regulatórios em macrófagos na invasão 3D de células cancerígenas do esferóide tumoral em comparação com os controles. Este método permite a determinação de diferentes parâmetros, como a área esferóide ou o número de células cancerígenas invasoras e, assim, detectar diferenças na invasão de células cancerígenas. Neste artigo, apresentamos a respectiva configuração, discutimos a análise subsequente, bem como as vantagens e possíveis armadilhas desse método.
Introdução
Os macrófagos são uma parte importante do sistema imunológico inato e representam a primeira linha de defesa em muitas condições patológicas, como infecções ou limpeza de detritos celulares após lesões1. Durante as últimas décadas, o impacto das células imunes na progressão do câncer tem sido um aspecto de muitos estudos. Assim, foi demonstrado que os macrófagos podem facilitar a metástase associando-se a tumores primários e tornando-se macrófagos associados a tumores (TAMs)2. Os macrófagos podem alterar seu perfil de expressão quando expostos a células cancerígenas, favorecendo o escape de metástases do sistema imunológico3. Além disso, foi demonstrado que as células cancerígenas podem utilizar defeitos na matriz extracelular (MEC) gerados por macrófagos para escapar do tumor primário e que seu comportamento também é manipulado pela captação de fatores secretados, incluindo vesículas extracelulares (EVs)4,5. Essa interação de aspectos físicos e químicos requer o desenvolvimento de novos métodos para caracterizar o impacto das células imunes na disseminação do tumor e no microambiente circundante.
Diferentes abordagens têm sido desenvolvidas para estudar o comportamento das células imunes no contexto de metástases in vivo6. No entanto, como para todos os experimentos, incluindo animais, é necessária uma licença para testes em animais e uma instalação animal; Essas abordagens já demandam muito desenvolvimento e preparação. Além disso, a análise é muitas vezes complicada, especialmente no que diz respeito à imagem de células vivas, pois nem todas as amostras são acessíveis para a maioria das configurações microscópicas. O desenvolvimento de novos métodos para testar hipóteses primeiro em condições in vitro qualificadas também é necessário e oportuno, pois a ciência e a sociedade visam pesquisas livres de animais para reduzir o número de animais sacrificados.
Em uma publicação recente7, investigamos o impacto dos macrófagos humanos primários na invasividade de células cancerígenas invasoras de um esferóide tumoral. Para isso, estabelecemos um ensaio de co-invasão de esferoides de macrófagos-tumor à base de colágeno I. Nesse contexto, objetivamos elucidar o papel das vias de reciclagem rápida da metaloproteinase da matriz do tipo 1 da membrana (MT1-MMP) em macrófagos. Testamos macrófagos que foram esgotados para a cinesina superprocessiva KIF16B, que identificamos como um dos principais impulsionadores da reciclagem de MT1-MMP, e seu impacto nas capacidades invasivas das células H1299-Green Fluorescent Protein (GFP) de um esferóide sólido. Os macrófagos depletados de KIF16B mostram níveis reduzidos do MT1-MMP ligado à membrana em sua superfície, enquanto as próprias células H1299 não foram tratadas.
Até onde sabemos, nenhum método semelhante foi descrito que permita imagens e análises completas da co-invasão macrófago-tumor-esferóide. Embora tenhamos nos concentrado em nossa recente publicação7 na análise de células migratórias individuais distantes do esferóide, o ensaio permite várias investigações adicionais, como o número de filamentos invasores, proteínas envolvidas na interação célula cancerígena-macrófago, quantidade de degradação do colágeno I ou outras propriedades esferóides, como o comprimento de seu perímetro.
Protocolo
Este protocolo envolve o uso de macrófagos humanos primários derivados de amostras de sangue de doadores. De acordo com as diretrizes éticas do Centro Médico Universitário Hamburg-Eppendorf, amostras de sangue foram coletadas e os doadores foram compensados. O processamento posterior das amostras foi realizado seguindo as diretrizes de segurança fornecidas (por exemplo, tratamento de amostras não testadas). O trabalho com macrófagos humanos primários neste estudo foi considerado inquestionável por Ärztekammer Hamburg, Alemanha.
1. Geração de esferoides tumorais H1299 em uma abordagem sem andaimes (3 dias de antecedência)
- Cultive células cancerígenas em uma incubadora a 37 ° C com 5% de CO2 em frascos de cultura decélulas 2 de 25 cm (ver Tabela de Materiais) com meio de células cancerígenas (meio de águia modificado de Dulbecco [DMEM] + 1% de penicilina-estreptomicina [pen-strep] + 10% de soro fetal bovino [FBS] + 0,1 mM de aminoácidos não essenciais + 2 mM de L-glutamina) até 80% de confluência.
- Lave as células uma vez com solução salina tamponada com fosfato de Dulbecco (DPBS) e separe-as adicionando 1 mL de ácido tripsina-etilenodiaminotetracético (EDTA) (0,25%, vermelho de fenol) por 2 min. Pare a reação enzimática adicionando 2 mL de meio de células cancerígenas e verifique o descolamento sob um microscópio óptico.
NOTA: É importante que as células cancerígenas estejam separadas e que nenhum aglomerado de células cancerígenas esteja presente. - Transferir a suspensão celular para um tubo de reação de 15 ml e centrifugar a 245 x g durante 5 min. Em seguida, remova a solução de tripsina pipetando ou usando uma bomba, lave o pellet posteriormente adicionando 5 mL de DPBS e centrifugue-os novamente nas mesmas condições.
- Ressuspenda o pellet em 5 mL de meio de células cancerígenas e conte as células, por exemplo, com uma câmara de Neubauer. Transfira 8.000 células H1299-GFP para uma placa de adesão ultrabaixa de 96 poços (consulte a Tabela de Materiais) em um volume final de 25 μL de meio de células cancerígenas.
- Incubar a câmara durante 3 dias na incubadora a 37 °C e 5% de CO2. Verifique a uniformidade dos esferóides sob o microscópio. Não considere esferóides com densidade mais baixa ou células cancerígenas individuais não anexadas para experimentos futuros.
2. Preparação de macrófagos humanos primários a partir de amostras de sangue de dadores
- Se as amostras de sangue do dador não forem imediatamente utilizadas para isolamento celular, armazená-las a 4 °C, com agitação.
- Transfira 20 mL de sangue de uma bolsa de transfusão para um tubo de reação de 50 mL.
- Em seguida, prepare um novo tubo de 50 mL e adicione 15 mL de meio de separação de linfócitos (LSM).
- Adicione cuidadosamente os 20 ml de sangue a cada tubo contendo LSM sem misturar e centrifugue-o a 450 x g durante 30 min a 4 °C.
- Enquanto isso, prepare um novo tubo de 50 mL e adicione 10 mL de meio frio Roswell Park Memorial Institute (RPMI)-1640.
- Após a centrifugação, transfira a fase densa e branca ("revestimento leucocitário") dentro do tubo contendo sangue para o tubo contendo RPMI preparado e adicione o volume a 50 mL com RPMI frio.
- Centrifugar a suspensão a 450 x g durante 10 min a 4 °C e rejeitar o sobrenadante.
- Ressuspenda as células em 10 mL de RPMI frio.
- Centrifugue novamente a mistura a 450 x g durante 10 min a 4 °C, elimine o sobrenadante e ressuspenda o sedimento em 50 ml de RPMI.
- Centrifugue a amostra novamente a 450 x g por 10 min a 4 ° C, descarte o sobrenadante e ressuspenda o pellet celular em 1,5 mL de tampão de monócitos frio (0,5% de albumina sérica humana + 5 mM de EDTA em 25 mL de DPBS).
- Coloque a suspensão celular no gelo, adicione 250 μL de microesferas anti-CD14 e incube por 15 min no gelo.
- Preparar uma coluna de separação por adição de um filtro num suporte separador magnético e adicionar 900 μL de tampão de monócitos para equilibrar.
- Despeje a suspensão da célula no filtro e deixe-a fluir para um tubo de resíduos por gravidade.
- Lave a coluna, que contém as células ligadas às esferas magnéticas, adicionando 1 mL de tampão de monócitos.
- Prepare um novo tubo de 50 mL contendo 20 mL de RPMI e recoloque o tubo de resíduos embaixo da coluna.
- Adicione 3 mL de tampão de monócitos à coluna, remova-o do rack magnético e anexe o carimbo.
- Pressione as células no tubo preparado de 50 mL e encha-o até 30 mL de volume com RPMI.
- Conte as células (por exemplo, usando uma câmara de contagem de Neubauer) sob um microscópio óptico e ajuste o número de células adicionando RPMI a 2 x 106 células/mL.
- Semear 1 ml da suspensão celular em cada poço de uma câmara de 6 poços e incubar a placa durante 2-4 h a 37 °C e 5% de CO2.
- Verifique se as células aderiram corretamente e substitua o RPMI por 1,5 mL de monomeio (20% de soro humano + 1% de pen-strep em RPMI).
- Mudar o meio após 24 h de incubação. Nessas condições, os monócitos se diferenciam em macrófagos em 6 dias.
3. Preparação de macrófagos
- No dia do experimento, separar os macrófagos com uma quantidade adequada de accutase para as condições individuais de cultura. Por exemplo, use 500 μL de accutase se as células forem semeadas em um prato de 6 poços. Incube as células por cerca de 40 min dentro da solução, lave-as uma vez com 2 mL de DPBS e conte-as com uma câmara de Neubauer.
- Para um esferóide, dilua o número desejado de macrófagos em 40 μL de colágeno I (2,5 mg / mL de colágeno I de cauda de rato) e misture-os por vórtice breve no tubo de reação. Nos experimentos realizados, 200.000 células/mL mostraram uma diferença detectável na invasão de células cancerígenas. Mantenha a mistura de células de colágeno no gelo, pois ela começará a polimerizar rapidamente à temperatura ambiente (RT).
4. Configuração do ensaio de co-invasão
NOTA: Dependendo do formato da câmara de imagem, do objetivo da análise ou da respectiva configuração de imagem, o esferóide tumoral pode precisar ser transferido da placa de adesão ultrabaixa e os valores de colágeno e meio adaptados. As etapas a seguir descrevem a configuração de uma amostra em uma lâmina de μ de 15 poços.
- Primeiro, lave o esferóide adicionando 300 μL de DPBS. Corte a ponta de uma ponta de pipeta azul de 1 mL e pegue o esferóide com DPBS. Após a aspiração do esferóide se estabelecer na ponta, empurre brevemente a pipeta para o fundo da câmara de imagem para transferir o tumor-esferóide por tensão superficial.
NOTA: É importante observar que o atrito nas pontas não modificadas pode perturbar a integridade do esferoide. - Remova o DPBS residual transferido com o esferóide tanto quanto possível por pipetagem.
- Adicione rapidamente 40 μL de mistura de colágeno I/macrófagos (consulte a etapa 3.4) à câmara de imagem que contém o esferóide H1299-GFP.
- Transfira a placa para uma incubadora de células e incube a placa por 30 min a 37 °C e 5% de CO2 em condições úmidas para polimerizar totalmente a mistura de colágeno.
- Após a polimerização, adicione cuidadosamente 25 μL de meio de células cancerígenas a cada poço e incube a placa por 3 dias na incubadora nas condições descritas anteriormente. Se necessário, faça a imagem da amostra viva com um microscópio de varredura a laser nos pontos de tempo desejados.
5. Fixação e coloração adicionais
- Para realizar a coloração por imunofluorescência, fixe os macrófagos e os esferoides H1299-GFP dentro da matriz de colágeno I. Primeiro, remova cuidadosamente o sobrenadante de cada sample com uma bomba sem tocar na matriz.
NOTA: Para evitar a destruição da amostra, o sobrenadante também pode ser removido por pipetagem manual sem usar uma bomba. - Adicione 50 μL de formaldeído a 3,7% em DPBS a cada poço e incube as amostras por 5 min em gelo. Em seguida, remova a solução fixadora e adicione 50 μL de solução fixadora nova novamente. Esta etapa é importante para obter um nível de formaldeído mais uniforme dentro do poço. Incubar as amostras durante a noite a 4 °C.
NOTA: Dependendo da quantidade de colágeno que misturo, os tempos de incubação podem se estender por várias horas. - Remova o fixador no dia seguinte e lave as amostras por remoção repetida de pelo menos duas vezes DPBS, dependendo do volume da câmara de imagem. Incube-os brevemente por 5 min no gelo em uma coqueteleira antes de remover o DPBS pipetando. As amostras estão agora prontas para coloração.
- Pinte as amostras adicionando uma mistura de 1:100 de 4',6-diamidino-2-fenilindol (DAPI) (1 mg/mL, para corar núcleos) e faloidina 568 (para corar F-actina) em DPBS por pelo menos 30 min e lave-as duas vezes com DPBS antes de fazer a imagem.
6. Imagem e análise de diferentes parâmetros de crescimento tumoral
NOTA: As etapas a seguir podem ser executadas para amostras fixas de imagens de células vivas.
- Para a observação do tumor-esferoide, use um microscópio de varredura a laser invertido equipado com uma objetiva de 10x que permite a obtenção de imagens de uma pilha z de aproximadamente o terço inferior do esferoide.
NOTA: Devido à espessura da amostra, um microscópio de 2 fótons também pode ser vantajoso. Para uma identificação ideal de células individuais, pode ser necessária maior potência do laser e tempo de exposição. - Após a aquisição, combine as pilhas por projeção z usando Fiji, que é o software ImageJ (Fiji). Além disso, processe o canal resultante do sinal H1299-GFP aplicando um limite automático (por exemplo, limite automático de Huang) e remova o ruído residual removendo as manchas da imagem, dependendo da quantidade de fundo e da intensidade do sinal.
- Meça os parâmetros esferóides usando Fiji.
- Área: Meça o tamanho do esferóide com a ferramenta Varinha .
- Perímetro: Inclua o perímetro nas configurações de medição.
- Diâmetro: Inclua o Diâmetro de Feret nas configurações de medição.
- Circularidade: inclua os descritores de forma nas configurações de medição.
- Meça os parâmetros individuais das células cancerígenas usando Fiji.
- Meça o número de células invasoras.
- Escolha o esferóide central com a ferramenta Varinha e exclua o sinal. Isso removerá o esferóide, bem como todos os sinais conectados das células, que ainda estão ligados ao esferoide, da análise.
- Em seguida, use a ferramenta Analisar partícula ou quantifique todos os sinais individuais residuais.
NOTA: O resultado será o número de células cancerígenas invasoras. Além disso, o resultado também pode mostrar a área representativa coberta pelas células cancerígenas e ser interpretado como invasão coletiva ou invasão unicelular, dependendo do tamanho da área. Para validar ainda mais o número de células cancerígenas invasoras, uma máscara das partículas analisadas pode ser colocada acima do canal DAPI e os núcleos contados.
- Meça o número de células invasoras.
- Além disso, avalie os resultados usando um programa estatístico como o GraphPad Prism ou o Microsoft Excel.
NOTA: Este protocolo demonstrou ser robusto e flexível. Ele foi usado em nosso laboratório mais de 50 vezes, sempre com macrófagos humanos primários de pelo menos 3 doadores, para levar em conta a variabilidade do doador. Em sua forma atual, o protocolo demonstrou levar a uma taxa de sucesso de 90-95%. No entanto, como a configuração é complexa, resultados variados também podem ser um resultado, pois a interação de macrófagos para cada esferóide é individual. Assim, sugere-se realizar o experimento pelo menos 3 vezes com 5 esferóides para obter um resultado estatisticamente significativo.
Resultados
A Figura 1 mostra um esferóide H1299-GFP fotografado nos respectivos dias de incubação em uma matriz de colágeno I. Uma respectiva imagem de campo claro, tirada no dia 0, também mostra os macrófagos humanos primários cocultivados com o esferóide. A imagem representativa registrada no dia 3 do experimento é ampliada após o processamento. Diferentes parâmetros que podem ser analisados são indicados, incluindo o número de células invasoras, locais de invasão coletiva e perímetro esferóide. A tabela incluída mostra os resultados obtidos com esta imagem. O número de sinais detectados corresponde à impressão visual. Nenhuma interrupção do esferóide central é visível, indicando invasão real de células cancerígenas individuais, em vez de células derivadas de detritos esferoides. Os resultados de diferentes esferóides e condições agora podem ser comparados por meio de análise estatística.
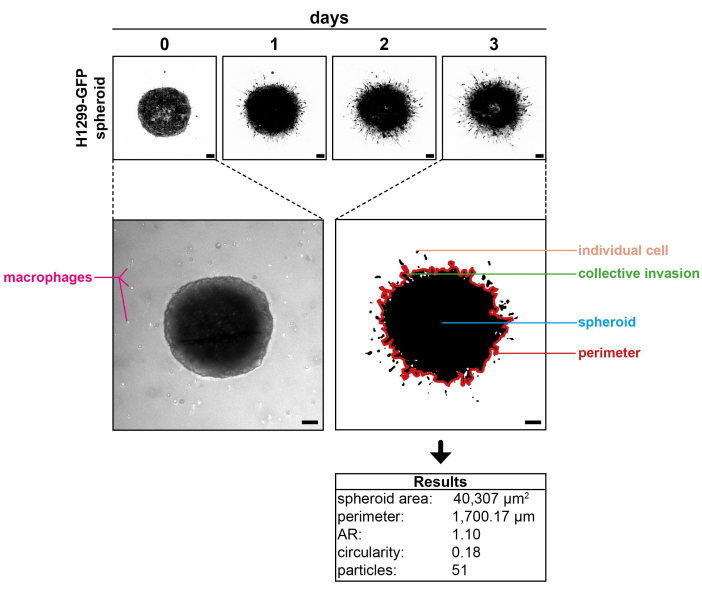
Figura 1: Resultados representativos. (Painel superior) Esferóide H1299-GFP fotografado no dia 0, dia 1, dia 2 e dia 3 de incubação em uma matriz de colágeno I. (Painel central esquerdo) Uma imagem de campo claro do esferóide mostrando os macrófagos humanos primários cocultivados. (Painel central direito) Uma imagem representativa adquirida no dia 3 ampliada após o processamento, mostrando os diferentes parâmetros que podem ser analisados (perímetro esferóide em vermelho; traçado manualmente para melhor visualização). (Painel inferior) Os resultados obtidos a partir da imagem no painel central direito. Nota: Os macrófagos não são visíveis, pois apenas o canal GFP foi registrado para elucidar o comportamento das células cancerígenas. Barras de escala: 100 μm. Clique aqui para ver uma versão ampliada desta figura.
Discussão
A invasão de células cancerígenas continua sendo um tópico importante, mas pouco estudado, no contexto de células imunes co-invasoras, como macrófagos. A invasão coletiva e individual de células cancerígenas são processos críticos durante a metástase e demonstraram diminuir a taxa de sobrevida de pacientes com câncer devido à multiplicidade de infestações de diferentes órgãos 8,9. Os estudos in vivo são complexos e restritos a laboratórios com acesso a instalações de alojamento de animais. Além disso, também é difícil controlar as condições in vivo ou manipular especificamente aspectos individuais. Portanto, a necessidade de um sistema mais acessível continua sendo essencial para responder a questões básicas na primeira linha de pesquisa.
Com o método aqui apresentado, desenvolvemos uma maneira pela qual i) células imunes e ii) comportamento das células cancerígenas, iii) crescimento e desenvolvimento de um esferóide sólido e iv) o impacto das células no microambiente tumoral circundante (TME) nos componentes da ECM podem ser analisados posteriormente. Ele pode ser usado para comparar o impacto de macrófagos modificados (por exemplo, esgotados para reguladores específicos pelo tratamento com siRNA) na invasividade de células cancerígenas individuais de um esferóide sólido. Se o rearranjo físico do TME ou os fatores secretados representam a principal causa do comportamento das células cancerígenas observadas atualmente não está claro e precisa ser analisado com mais detalhes.
Além disso, também as próprias células cancerígenas podem ser manipuladas, por exemplo, por tratamento com siRNA ou nocaute de reguladores específicos. Além disso, a análise pode ser melhorada comparando o número de núcleos contados dentro do perfil celular identificado para permitir uma determinação mais precisa das células cancerígenas invasoras.
Usamos este ensaio para determinar o impacto dos macrófagos no TME na invasão de células tumorais. No entanto, também deve ser observado que as células tumorais provavelmente influenciam a atividade dos macrófagos, possivelmente por meio de fatores secretados no meio. Portanto, também valeria a pena identificar alterações nos macrófagos, como estado de polarização alterado (M1 vs. M2) por coloração de imunofluorescência usando os respectivos anticorpos. No passado, a comparação entre células crescendo em monocamadas e aquelas cultivadas em um ambiente 3D mostrou diferenças significativas em seus perfis de expressão10.
Além disso, a classificação de células ativadas por fluorescência (FACS) de subpopulações de macrófagos e sua integração no procedimento experimental podem ser instrutivas. Por último, mas não menos importante, imagens mais detalhadas das áreas de contato onde os macrófagos interagem com as células cancerígenas ou com a superfície esferóide do tumor podem levar à identificação de outros mecanismos relevantes para a invasão e interação 3D.
Digno de nota que não pode ser totalmente controlado é o posicionamento preciso do esferóide no centro do poço após a adição da mistura de colágeno. A quantidade de colágeno sob o esferóide é especialmente difícil de regular. Aqui, outros métodos foram estabelecidos para permitir o posicionamento preciso do esferóide, por exemplo, em cima de moldes de agarose com maior número de amostras11. No entanto, como a liberação de citocinas é um mecanismo comum, todos os esferoides dentro deste ensaio multiesferóide são expostos a fatores secretados, e o número de células imunes que atuam em um único esferóide é difícil de controlar.
Uma limitação básica deste protocolo é a capacidade das linhagens de células cancerígenas de formar esferoides uniformes, sendo aplicável apenas a um subconjunto de linhagens celulares. Por exemplo, as células de melanoma MeWo formam estruturas 3D irregulares, semelhantes a folhas, mas sem esferóides uniformes.
Deve-se notar também que o ensaio é altamente adaptável, pois muitas de suas características podem ser modificadas, como o material da MEC, o número de células ou adicionando fatores específicos, como citocinas, ao sobrenadante. Deve, portanto, ser altamente adequado para estudos iniciais in vitro da interação entre células cancerígenas e células imunes e pode ser adaptado à questão de pesquisa específica que é abordada atualmente.
Divulgações
Os autores declaram que não existem interesses conflitantes.
Agradecimentos
Os autores gostariam de agradecer a Andrea Mordhorst pelo excelente suporte técnico e cultura celular e a Martin Aepfelbacher pelo apoio contínuo. O trabalho sobre invasão de macrófagos no laboratório SL é apoiado pela Deutsche Forschungsgemeinschaft (CRC877 / B13; LI925/13-1).
Materiais
| Name | Company | Catalog Number | Comments |
| 15 µ-Slide Angiogenesis | ibidi | 81506 | |
| Accutase | Invitrogen | 00-4555-56 | |
| Alexa Fluor 568 Phalloidin | ThermoFisher Scientific | A12380 | |
| CD14 MicroBeads | Miltenyi Biotec | 130-050-201 | |
| CO2 Incubator | Binder | ||
| Collagen I Rat Tail | Corning | 354236 | |
| DAPI | AppliChem | ||
| DMEM (1x) + GlutaMAX | Gibco | 31966-021 | |
| DPBS | Anprotec | MS01Y71003 | |
| FBS | Bio&Cell | FBS. S 0613 | |
| Fiji | NIH | ImageJ 2 Version: 2.3.0/1.53s | |
| Formaldehyde 37% | 252549-500ml | Sigma-Aldrich | |
| H1299-GFP cell line | |||
| Human serum albumin | Sigma-Aldrich | A5843 | |
| Leica TCS SP8 X | Leica | ||
| l-glutamine | Gibco | 25030-024 | |
| Lymphocyte Seperation Medium (LSM) 1077 | PromoCell | C-44010 | |
| MS Columns | Miltenyi Biotec | 130-042-201 | |
| nonessential amino-acids | Sigma-Aldrich | 11140050 | |
| Pen Strep | Gibco | 15140-122 | |
| RPMI-1640 | Gibco | 81275-034 | |
| TC-Platte 96 Well, BIOFLOAT, R | SARSTEDT | 83,39,25,400 | |
| VORTEX-GENE 2 | Scientific Industries |
Referências
- Lendeckel, U., Venz, S., Wolke, C. Macrophages: shapes and functions. ChemTexts. 8 (2), 12 (2022).
- Dallavalasa, S., et al. The role of tumor associated macrophages (TAMs) in cancer progression, chemoresistance, angiogenesis and metastasis - Current status. Curr Med Chem. 28 (39), 8203-8236 (2021).
- Pan, Y., Yu, Y., Wang, X., Zhang, T. Tumor-associated macrophages in tumor immunity. Front Immunol. 11, 583084 (2020).
- Boutilier, A. J., Elsawa, S. F. Macrophage Polarization States in the Tumor Microenvironment. Int J Mol Sci. 22 (13), 6995 (2021).
- Wenzel, E. M., et al. Intercellular transfer of cancer cell invasiveness via endosome-mediated protease shedding. Nat Commun. 15 (1), 1277 (2024).
- Anfray, C., Ummarino, A., Calvo, A., Allavena, P., Torres Andon, F. In vivo analysis of tumor-associated macrophages in the tumor microenvironment. Methods Mol Biol. 2614, 93-108 (2023).
- Hey, S., Wiesner, C., Barcelona, B., Linder, S. KIF16B drives MT1-MMP recycling in macrophages and promotes co-invasion of cancer cells. Life Sci Alliance. 6 (11), e202302158 (2023).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Shi, X., et al. Mechanism insights and therapeutic intervention of tumor metastasis: latest developments and perspectives. Signal Transduct Target Ther. 9 (1), 192 (2024).
- Li, G. N., Livi, L. L., Gourd, C. M., Deweerd, E. S., Hoffman-Kim, D. Genomic and morphological changes of neuroblastoma cells in response to three-dimensional matrices. Tissue Eng. 13 (5), 1035-1047 (2007).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. 160, e61392 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados