Method Article
대식세포-종양 스페로이드 동시 침습 분석
요약
여기에서는 3차원(3D) 콜라겐 I 매트릭스에서 1차 인간 단핵구 유래 대식세포와 종양 스페로이드의 상호 작용을 조사하기 위한 프로토콜을 제시하며, 미세환경의 용해성 및 물리적 특성이 세포 침입에 미치는 영향을 비교할 수 있습니다.
초록
면역 세포와 암 세포의 상호 작용과 전이에 대한 각각의 영향은 암 연구의 중요한 측면을 나타냅니다. 지금까지는 in vivo 상황의 in vitro 근사치를 허용하는 프로토콜은 몇 가지에 불과합니다. 여기에서는 3차원(3D) 콜라겐 I 매트릭스에 내장된 H1299 비소세포폐암종의 종양 스페로이드를 사용하여 인간 대식세포가 암세포의 침습성에 미치는 영향을 관찰하는 새로운 접근 방식을 제시합니다. 이 공동 배양 설정을 통해 우리는 대조군과 비교하여 종양 스페로이드에서 암세포의 3D 침입에 대한 대식세포의 작은 간섭 RNA(siRNA) 기반 조절 인자 고갈이 미치는 영향을 테스트했습니다. 이 방법을 사용하면 스페로이드 면적 또는 침입하는 암세포의 수와 같은 다양한 매개변수를 측정할 수 있으므로 암세포 침입의 차이를 감지할 수 있습니다. 이 기사에서는 각각의 설정을 제시하고 후속 분석과 이 방법의 장점과 잠재적 위험에 대해 설명합니다.
서문
대식세포는 선천면역계의 주요 부분이며, 감염이나 부상 후 세포 파편을 제거하는 것과 같은 많은 병리학적 상태에서 1차 방어선을 형성한다1. 지난 수십 년 동안 면역 세포가 암 진행에 미치는 영향은 많은 연구의 한 측면이었습니다. 따라서 대식세포는 원발성 종양과 결합하여 종양 관련 대식세포(TAM)가 되어 전이를 촉진할 수 있는 것으로 나타났습니다2. 대식세포는 암세포에 노출될 때 발현 프로파일을 변화시켜 면역 체계의 전이를 촉진할 수 있습니다3. 또한 암세포는 대식세포에 의해 생성된 세포외 기질(ECM)의 결함을 활용하여 원발성 종양에서 탈출할 수 있으며, 암세포의 행동은 세포외 소포체(EV)를 포함한 분비 인자의 흡수에 의해 조작되는 것으로 나타났습니다4,5. 이러한 물리적 및 화학적 측면의 상호 작용은 종양 확산 및 주변 미세 환경에 대한 면역 세포의 영향을 특성화하는 새로운 방법의 개발을 필요로 합니다.
생체 내 전이의 맥락에서 면역 세포 행동을 연구하기 위해 다양한 접근법이 개발되었습니다 6. 그러나 동물을 포함한 모든 실험은 동물실험 허가와 동물시설의 허가가 필요합니다. 이러한 접근 방식은 이미 많은 개발과 준비를 요구합니다. 또한, 대부분의 현미경 설정에서 모든 검체에 접근할 수 있는 것은 아니기 때문에 특히 살아있는 세포 이미징과 관련하여 분석이 복잡한 경우가 많습니다. 과학계와 사회가 희생되는 동물의 수를 줄이기 위해 동물을 사용하지 않는 연구를 목표로 하기 때문에 자격을 갖춘 시험관 조건에서 먼저 가설을 테스트하는 새로운 방법의 개발도 필요하고 시의적절합니다.
최근 발표된논문 7에서는 종양 스페로이드에서 암세포가 침입하는 암세포의 침입성에 대한 일차 인간 대식세포의 영향을 조사했습니다. 이를 위해 콜라겐 I 기반 대식세포-종양 스페로이드 동시 침습 분석을 확립했습니다. 이러한 맥락에서 우리는 대식세포에서 막형 1 매트릭스 금속단백분해효소(MT1-MMP)의 빠른 재활용 경로의 역할을 밝히는 것을 목표로 했습니다. MT1-MMP 재활용의 주요 동인으로 확인된 초진행성 키네신 KIF16B가 고갈된 대식세포와 고체 스페로이드에서 H1299-녹색 형광 단백질(GFP) 세포의 침습 능력에 미치는 영향을 테스트했습니다. KIF16B가 고갈된 대식세포는 표면에서 막으로 결합된 MT1-MMP의 수치가 감소한 반면, H1299 세포 자체는 처리되지 않았습니다.
우리가 아는 한, 대식세포-종양-스페로이드 동시 침입에 대한 완전한 이미징 및 분석을 가능하게 하는 유사한 방법은 설명되지 않았습니다. 최근 간행물7 에서는 스페로이드에서 멀리 떨어진 세포의 개별 이동 분석에 초점을 맞췄지만, 이 분석을 통해 침입하는 가닥의 수, 암세포-대식세포 상호 작용에 관여하는 단백질, 콜라겐 I 분해의 양 또는 둘레의 길이와 같은 기타 스페로이드 특성과 같은 여러 추가 조사를 수행할 수 있습니다.
프로토콜
이 프로토콜은 기증자의 혈액 샘플에서 유래한 1차 인간 대식세포의 사용을 포함합니다. 함부르크-에펜도르프 대학의료센터(University Medical Center Hamburg-Eppendorf)의 윤리 지침에 따라 혈액 샘플을 채취하고 기증자들에게 보상을 제공했습니다. 샘플의 추가 처리는 주어진 안전 지침(예: 테스트되지 않은 샘플의 처리)에 따라 수행되었습니다. 이 연구에서 1차 인간 대식세포에 대한 연구는 독일 함부르크의 Ärztekammer에 의해 이의가 없는 것으로 판단되었습니다.
1. scaffold-free 접근법에서 H1299 종양 스페로이드 생성(3일 전)
- 암세포 배지(Dulbecco's modified eagles' medium[DMEM] + 1% penicillin-streptomycin [pen-strep] + 10% 소 태아 혈청[FBS] + 0.1 mM 비필수 아미노산 + 2 mM L-글루타민)를 사용하여 80% confluence까지 5% CO2가 포함된 37 °C 인큐베이터에서 암세포를 배양합니다.
- Dulbecco의 인산염 완충 식염수(DPBS)로 세포를 한 번 세척하고 1mL의 트립신-에틸렌디아민테트라아세트산(EDTA)(0.25%, 페놀-레드)을 2분 동안 첨가하여 분리합니다. 2mL의 암세포 배지를 첨가하여 효소 반응을 중지하고 광학 현미경으로 박리를 확인합니다.
참고: 암세포가 분리되어 있고 암세포 클러스터가 존재하지 않는 것이 중요합니다. - 세포 현탁액을 15mL 반응 튜브로 옮기고 245 x g 에서 5분 동안 원심분리합니다. 그 후, 피펫팅 또는 펌프를 사용하여 트립신 용액을 제거하고 이후 DPBS 5mL를 첨가하여 펠릿을 세척한 후 동일한 조건에서 다시 원심분리합니다.
- 펠릿을 5mL의 암 세포 배지에 재현탁시키고 예를 들어 Neubauer 챔버로 세포를 계수합니다. 8,000개의 H1299-GFP 세포를 25μL의 암 세포 배지의 최종 부피에서 96웰 초저접착 플레이트( 재료 표 참조)로 옮깁니다.
- 인큐베이터에서 37 °C 및 5 % CO2 에서 3 일 동안 챔버를 배양합니다. 현미경으로 스페로이드의 균일성을 확인합니다. 추가 실험을 위해 밀도가 낮거나 개별 암세포가 부착되지 않은 스페로이드를 고려하지 마십시오.
2. 공여자의 혈액 샘플로부터 1차 인간 대식세포의 준비
- 기증자 혈액 샘플이 세포 분리에 즉시 사용되지 않는 경우 흔들면서 4°C에서 보관하십시오.
- 수혈 백에서 20mL의 혈액을 50mL 반응 튜브로 옮깁니다.
- 그런 다음 새 50mL 튜브를 준비하고 15mL의 림프구 분리 배지(LSM)를 추가합니다.
- 20mL의 혈액을 섞지 않고 각 LSM 함유 튜브에 조심스럽게 첨가하고 450 x g 에서 4°C에서 30분 동안 원심분리합니다.
- 그 동안 새 50mL 튜브를 준비하고 차가운 RPMI(Roswell Park Memorial Institute)-1640 배지 10mL를 추가합니다.
- 원심분리 후, 혈액 함유 튜브 내의 조밀한 흰색 상("버피 코트")을 준비된 RPMI 함유 튜브로 옮기고 차가운 RPMI로 부피를 50mL로 추가합니다.
- 현탁액을 450 x g 에서 4°C에서 10분 동안 원심분리하고 상층액을 버립니다.
- 10mL의 차가운 RPMI에 세포를 재현탁합니다.
- 혼합물을 450 x g 에서 4°C에서 10분 동안 다시 원심분리하고, 상층액을 버리고, 펠릿을 RPMI 50mL에 재현탁합니다.
- 4°C에서 10분 동안 450 x g 에서 샘플을 다시 원심분리하고, 상등액을 버리고, 세포 펠렛을 1.5mL의 저온 단핵구 완충액(25mL의 DPBS 중 0.5% 인간 혈청 알부민 + 5mM EDTA)에 재현탁합니다.
- 세포 현탁액을 얼음 위에 놓고 250μL의 anti-CD14 마이크로비즈를 첨가한 후 얼음 위에서 15분 동안 배양합니다.
- 자기 분리기 랙에 필터를 추가하여 분리 컬럼을 준비하고 평형을 위해 900μL의 단핵구 완충액을 추가합니다.
- 셀 현탁액을 필터에 붓고 중력에 의해 폐기물 튜브로 흐르도록 합니다.
- 마그네틱 비드에 결합된 세포가 포함된 컬럼을 1mL의 단핵구 완충액을 추가하여 세척합니다.
- 20mL RPMI가 들어 있는 새 50mL 튜브를 준비하고 컬럼 아래의 waste 튜브를 교체합니다.
- 컬럼에 단핵구 완충액 3mL를 추가하고 마그네틱 랙에서 제거한 다음 스탬프를 부착합니다.
- 준비된 50mL 튜브에 세포를 밀어 넣고 RPMI로 최대 30mL 부피를 채웁니다.
- 광학 현미경으로 세포를 계수하고(예: Neubauer 계수 챔버 사용) RPMI를 2 x 106 cells/mL에 추가하여 세포 수를 조정합니다.
- 1mL의 세포 현탁액을 6웰 챔버의 각 웰에 시딩하고 37°C 및 5%CO2에서 2-4시간 동안 플레이트를 배양합니다.
- 세포가 제대로 부착되었는지 확인하고 RPMI를 1.5mL 모노미디어(RPMI의 20% 인간 혈청 + 1% 펜 연쇄상구균)로 교체합니다.
- 배양 24시간 후에 배지를 교체하십시오. 이러한 조건에서 단핵구는 6일 이내에 대식세포로 분화합니다.
3. 대식세포 제제
- 실험 당일, 개별 배양 조건에 적합한 양의 accutase로 대식세포를 분리합니다. 예를 들어, 세포가 6웰 접시에 파종된 경우 500μL의 아큐타아제를 사용합니다. 용액 내에서 약 40분 동안 세포를 배양하고 2mL의 DPBS로 한 번 세척한 다음 Neubauer 챔버로 계수합니다.
- 하나의 스페로이드의 경우, 원하는 수의 대식세포를 40μL의 콜라겐 I 혼합물(2.5mg/mL 쥐꼬리 콜라겐 I)에 희석하고 반응 튜브를 짧게 와류로 혼합합니다. 수행된 실험에서 200,000 cells/mL는 암세포의 침입에 대해 감지 가능한 차이를 보여주었습니다. 콜라겐-세포 혼합물은 실온(RT)에서 빠르게 중합되기 시작하므로 얼음 위에 보관하십시오.
4. co-invasion assay의 설정
참고: 이미징 챔버의 형식, 분석 목적 또는 해당 이미징 설정에 따라 종양 스페로이드를 초저 접착 플레이트에서 전달해야 할 수도 있으며 콜라겐 및 배지 값을 조정해야 할 수도 있습니다. 다음 단계에서는 15웰 μ 슬라이드에서 하나의 샘플에 대한 설정을 설명합니다.
- 먼저 DPBS 300μL를 첨가하여 스페로이드를 세척합니다. 1mL 파란색 피펫 팁의 끝을 자르고 DPBS로 스페로이드를 취합니다. 스페로이드의 흡인이 팁에 정착한 후 피펫을 이미징 챔버 바닥으로 짧게 밀어 표면 장력에 의해 종양-스페로이드를 전달합니다.
알림: 수정되지 않은 팁의 마찰은 스페로이드의 무결성을 방해할 수 있다는 점에 유의하는 것이 중요합니다. - 피펫팅을 통해 스페로이드와 함께 전달된 잔류 DPBS를 최대한 제거합니다.
- 40μL의 콜라겐 I/대식세포 혼합물(3.4단계 참조)을 H1299-GFP 스페로이드가 포함된 이미징 챔버에 신속하게 추가합니다.
- 플레이트를 cell incubator로 옮기고 습한 조건에서 37°C 및 5% CO2 에서 30분 동안 플레이트를 배양하여 콜라겐 혼합물을 완전히 중합합니다.
- 중합 후 각 웰에 25μL의 암세포 배지를 조심스럽게 추가하고 앞서 설명한 조건에서 인큐베이터에서 3일 동안 플레이트를 배양합니다. 필요한 경우 원하는 시점에서 레이저 스캐닝 현미경으로 라이브 샘플을 이미지화합니다.
5. 추가 정착 및 염색
- 면역형광 염색을 수행하려면 콜라겐 I 매트릭스 내에서 대식세포와 H1299-GFP 스페로이드를 고정합니다. 먼저 매트릭스를 건드리지 않고 펌프로 각 샘플에서 상등액을 조심스럽게 제거합니다.
참고: 샘플 파괴를 방지하기 위해 펌프를 사용하지 않고 수동 피펫팅으로 상등액을 제거할 수도 있습니다. - DPBS의 3.7% 포름알데히드 50μL를 각 웰에 추가하고 얼음에서 5분 동안 샘플을 배양합니다. 그 후, 정착액을 제거하고 50μL의 새 정착액을 다시 첨가하십시오. 이 단계는 웰 내에서 보다 균일한 포름알데히드 수준을 달성하는 데 중요합니다. 샘플을 4°C에서 밤새 배양합니다.
참고: 제가 혼합하는 콜라겐의 양에 따라 배양 시간이 몇 시간까지 연장될 수 있습니다. - 다음 날 고정제를 제거하고 이미징 챔버의 부피에 따라 최소 2회 DPBS를 반복적으로 제거하여 샘플을 세척합니다. 피펫팅으로 DPBS를 제거하기 전에 셰이커의 얼음 위에서 5분 동안 잠시 배양합니다. 이제 샘플을 염색할 준비가 되었습니다.
- DPBS에 4',6-diamidino-2-phenylindole (DAPI) (1 mg/mL, 핵 염색용) 및 phalloidin 568 (F-actin 염색용)의 1:100 혼합물을 30분 이상 첨가하여 샘플을 염색하고 이미징 전에 DPBS로 두 번 세척합니다.
6. 종양 성장의 다양한 매개 변수의 이미징 및 분석
참고: 다음 단계는 고정된 라이브 셀 이미징 샘플에 대해 수행할 수 있습니다.
- 종양 스페로이드를 관찰하려면 스페로이드의 약 1/3 아래쪽에 있는 z 스택을 이미징할 수 있는 10x 대물렌즈가 장착된 도립 레이저 스캐닝 현미경을 사용하십시오.
참고: 샘플의 두께로 인해 2광자 현미경도 유리할 수 있습니다. 개별 세포를 최적으로 식별하려면 더 높은 레이저 출력과 노출 시간이 필요할 수 있습니다. - 획득 후 ImageJ 소프트웨어(Fiji)인 Fiji를 사용하여 z-투영으로 스택을 결합합니다. 또한 자동 임계값(예: Huang 자동 임계값)을 적용하여 H1299-GFP 신호의 결과 채널을 처리하고 배경 및 신호 강도에 따라 이미지의 얼룩을 제거하여 잔류 노이즈를 제거합니다.
- 피지를 사용하여 스페로이드 매개변수를 측정합니다.
- 영역: Wand 도구를 사용하여 스페로이드 크기를 측정합니다.
- 둘레: 둘레 를 측정 설정에 포함합니다.
- 지름: Feret의 지름 을 측정 설정에 포함합니다.
- Circularity: 측정 설정에 Shape Descriptors 를 포함합니다.
- Fiji를 사용하여 개별 암세포 매개변수를 측정합니다.
- 침입하는 세포의 수를 측정합니다.
- 지팡이 도구로 중앙 회전 타원체를 선택하고 신호를 삭제합니다. 이렇게 하면 스페로이드뿐만 아니라 여전히 스페로이드에 부착된 세포의 연결된 모든 신호가 분석에서 제거됩니다.
- 그런 다음 Analyze Particle(파티클 분석 ) 도구를 사용하거나 모든 잔류 개별 신호를 정량화합니다.
참고: 그 결과는 침입하는 암세포의 수입니다. 또한 그 결과는 암세포가 덮고 있는 대표 영역을 보여줄 수 있으며 영역의 크기에 따라 집단 침입 또는 단일 세포 침입으로 해석될 수도 있습니다. 침입하는 암세포의 수를 추가로 검증하기 위해 분석된 입자의 마스크를 DAPI 채널 위에 놓고 핵을 계수할 수 있습니다.
- 침입하는 세포의 수를 측정합니다.
- 또한 GraphPad Prism 또는 Microsoft Excel과 같은 통계 프로그램을 사용하여 결과를 평가합니다.
참고: 이 프로토콜은 강력하고 유연한 것으로 나타났습니다. 우리 실험실에서 50회 이상 사용되었으며, 항상 최소 3명의 기증자의 1차 인간 대식세포와 함께 기증자의 변동성을 설명하기 위해 사용되었습니다. 현재 형태에서 프로토콜은 90-95%의 성공률로 이어지는 것으로 나타났습니다. 그러나 설정이 복잡하기 때문에 모든 스페로이드에 대한 대식세포의 상호 작용이 개별적이기 때문에 다양한 결과가 결과일 수도 있습니다. 따라서 5개의 스페로이드로 실험을 3회 이상 수행하면 통계적으로 유의미한 결과를 얻을 수 있습니다.
결과
그림 1은 콜라겐 I 매트릭스에서 각 배양 일수에 이미지화된 H1299-GFP 스페로이드를 보여줍니다. 0일에 촬영한 각각의 명시야 이미지도 스페로이드와 공동 배양된 주요 인간 대식세포를 보여줍니다. 실험 3일차에 기록된 대표 이미지를 처리 후 확대한 것이다. 분석할 수 있는 다양한 매개변수가 표시되며, 여기에는 침입 세포의 수, 집단 침입 부위 및 회전 타원체 둘레가 포함됩니다. 포함된 표는 이 이미지에서 얻은 결과를 보여줍니다. 감지된 신호의 수는 시각적 인상과 일치합니다. 중앙 스페로이드의 붕괴는 보이지 않으며, 이는 스페로이드 파편에서 유래한 세포가 아닌 실제 개별 암세포 침입을 나타냅니다. 이제 통계 분석을 통해 다양한 스페로이드 및 조건의 결과를 비교할 수 있습니다.
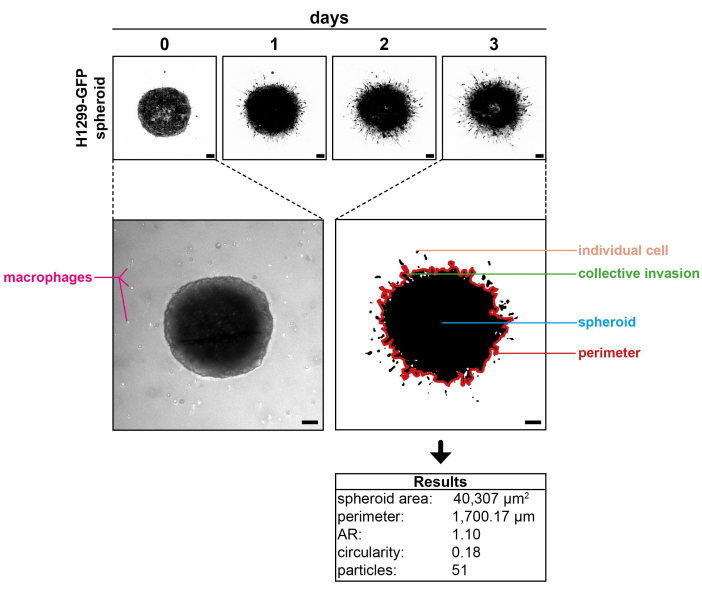
그림 1: 대표적인 결과. (상단 패널) 콜라겐 I 매트릭스에서 배양 0일, 1일, 2일, 3일에 이미지화된 H1299-GFP 스페로이드. (왼쪽 중간 패널) 공동 배양된 1차 인간 대식세포를 보여주는 스페로이드의 명시야 이미지. (오른쪽 중간 패널) 처리 후 3일차에 획득한 대표 이미지는 분석할 수 있는 다양한 매개변수를 보여줍니다(빨간색의 스페로이드 둘레, 더 나은 시각화를 위해 수동으로 추적). (하단 패널) 오른쪽 중간 패널의 이미지에서 얻은 결과입니다. 참고: 대식세포는 암세포 거동을 밝히기 위해 GFP 채널만 기록되었기 때문에 대식세포는 보이지 않습니다. 스케일 바: 100μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
암세포의 침입은 대식세포와 같은 면역세포의 동시 침입이라는 맥락에서 중요하지만 연구되지 않은 주제로 남아 있습니다. 집단 및 개별 암세포 침입은 전이 중 중요한 과정이며, 서로 다른 장기의 감염이 다양하기 때문에 암 환자의 생존율을 낮추는 것으로 나타났습니다 8,9. 생체 내 연구는 복잡하며 동물 사육 시설을 이용할 수 있는 실험실로 제한됩니다. 더욱이, 생체 내 조건을 제어하거나 개별 측면을 구체적으로 조작하는 것도 어렵습니다. 따라서 보다 접근하기 쉬운 시스템의 필요성은 첫 번째 연구 라인에서 기본적인 질문에 답하는 데 필수적입니다.
여기에 제시된 방법을 통해 i) 면역 세포 및 ii) 암세포 행동, iii) 고체 스페로이드의 성장 및 발달, iv) 주변 종양 미세환경(TME)의 세포가 ECM 구성 요소에 미치는 영향을 추가로 분석할 수 있는 방법을 개발했습니다. 이는 변형된(예: siRNA 처리에 의해 특정 조절자에 대해 고갈된) 대식세포가 고체 스페로이드에서 개별 암세포의 침습성에 미치는 영향을 비교하는 데 사용할 수 있습니다. TME의 물리적 재배열 또는 분비된 인자가 관찰된 암세포 행동의 주요 원인인지 여부는 현재 불분명하며 더 자세한 분석이 필요합니다.
또한, 암세포 자체도 예를 들어 siRNA 처리 또는 특정 조절자의 녹아웃에 의해 조작될 수 있습니다. 또한, 침습하는 암세포를 보다 정확하게 결정할 수 있도록 식별된 세포 프로파일 내에서 계수된 핵의 수를 비교함으로써 분석을 개선할 수 있습니다.
우리는 이 분석을 사용하여 TME의 대식세포가 종양 세포의 침입에 미치는 영향을 확인했습니다. 그러나 종양 세포는 배지 내에서 분비된 인자를 통해 대식세포의 활동에 영향을 미칠 가능성이 있다는 점도 유의해야 합니다. 따라서 각 항체를 사용한 면역형광 염색에 의한 변경된 편광 상태(M1 대 M2)와 같은 대식세포의 변화를 확인하는 것도 가치 있는 노력이 될 것입니다. 과거에는 단층에서 자라는 세포와 3D 환경에서 배양된 세포를 비교하면 발현 프로파일에서 상당한 차이가 나타났습니다10.
또한, 대식세포 하위 집단의 형광 활성화 세포 분류(FACS)와 이를 실험 절차로의 통합은 유익할 수 있습니다. 마지막으로, 대식세포가 암세포 또는 종양 스페로이드 표면과 상호 작용하는 접촉 영역에 대한 보다 상세한 이미징을 통해 3D 침입 및 상호 작용과 관련된 추가 메커니즘을 식별할 수 있습니다.
완전히 제어할 수 없는 점은 콜라겐 믹스를 첨가한 후 웰 중앙에 스페로이드를 정확하게 배치하는 것입니다. 스페로이드 아래의 콜라겐 양은 특히 조절하기가 어렵습니다. 여기서, 스페로이드의 정확한 위치, 예를 들어, 더 높은 샘플 수11을 갖는 아가로스 주형 위에 배치할 수 있도록 다른 방법이 확립되었습니다. 그러나 사이토카인의 방출이 일반적인 기전이기 때문에 이 다중 스페로이드 분석 내의 모든 스페로이드는 분비 인자에 노출되며 단일 스페로이드에 작용하는 면역 세포의 수를 제어하기 어렵습니다.
이 프로토콜의 한 가지 기본적인 한계는 암 세포주가 균일한 스페로이드를 형성할 수 있는 능력이므로 세포주의 하위 집합에만 적용할 수 있다는 것입니다. 예를 들어, MeWo 흑색종 세포는 3D 구조와 같은 고르지 않은 시트를 형성하지만 균일한 스페로이드는 없습니다.
또한 이 분석은 ECM 물질, 세포 수와 같은 많은 기능을 수정하거나 상층액에 사이토카인과 같은 특정 인자를 추가함으로써 수정할 수 있기 때문에 적응성이 매우 높다는 점에 유의해야 합니다. 따라서 암세포/면역세포 상호 작용에 대한 초기 체외 연구에 매우 적합해야 하며 현재 다루고 있는 특정 연구 질문에 맞게 조정할 수 있습니다.
공개
저자는 상충하는 이해관계가 존재하지 않는다고 선언합니다.
감사의 말
저자는 탁월한 기술 지원과 세포 배양을 제공해 준 Andrea Mordhorst와 지속적인 지원을 제공해 준 Martin Aepfelbacher에게 감사의 뜻을 전합니다. SL 실험실에서의 대식세포 침입에 대한 연구는 Deutsche Forschungsgemeinschaft(CRC877/B13; LI925/13-1).
자료
| Name | Company | Catalog Number | Comments |
| 15 µ-Slide Angiogenesis | ibidi | 81506 | |
| Accutase | Invitrogen | 00-4555-56 | |
| Alexa Fluor 568 Phalloidin | ThermoFisher Scientific | A12380 | |
| CD14 MicroBeads | Miltenyi Biotec | 130-050-201 | |
| CO2 Incubator | Binder | ||
| Collagen I Rat Tail | Corning | 354236 | |
| DAPI | AppliChem | ||
| DMEM (1x) + GlutaMAX | Gibco | 31966-021 | |
| DPBS | Anprotec | MS01Y71003 | |
| FBS | Bio&Cell | FBS. S 0613 | |
| Fiji | NIH | ImageJ 2 Version: 2.3.0/1.53s | |
| Formaldehyde 37% | 252549-500ml | Sigma-Aldrich | |
| H1299-GFP cell line | |||
| Human serum albumin | Sigma-Aldrich | A5843 | |
| Leica TCS SP8 X | Leica | ||
| l-glutamine | Gibco | 25030-024 | |
| Lymphocyte Seperation Medium (LSM) 1077 | PromoCell | C-44010 | |
| MS Columns | Miltenyi Biotec | 130-042-201 | |
| nonessential amino-acids | Sigma-Aldrich | 11140050 | |
| Pen Strep | Gibco | 15140-122 | |
| RPMI-1640 | Gibco | 81275-034 | |
| TC-Platte 96 Well, BIOFLOAT, R | SARSTEDT | 83,39,25,400 | |
| VORTEX-GENE 2 | Scientific Industries |
참고문헌
- Lendeckel, U., Venz, S., Wolke, C. Macrophages: shapes and functions. ChemTexts. 8 (2), 12 (2022).
- Dallavalasa, S., et al. The role of tumor associated macrophages (TAMs) in cancer progression, chemoresistance, angiogenesis and metastasis - Current status. Curr Med Chem. 28 (39), 8203-8236 (2021).
- Pan, Y., Yu, Y., Wang, X., Zhang, T. Tumor-associated macrophages in tumor immunity. Front Immunol. 11, 583084 (2020).
- Boutilier, A. J., Elsawa, S. F. Macrophage Polarization States in the Tumor Microenvironment. Int J Mol Sci. 22 (13), 6995 (2021).
- Wenzel, E. M., et al. Intercellular transfer of cancer cell invasiveness via endosome-mediated protease shedding. Nat Commun. 15 (1), 1277 (2024).
- Anfray, C., Ummarino, A., Calvo, A., Allavena, P., Torres Andon, F. In vivo analysis of tumor-associated macrophages in the tumor microenvironment. Methods Mol Biol. 2614, 93-108 (2023).
- Hey, S., Wiesner, C., Barcelona, B., Linder, S. KIF16B drives MT1-MMP recycling in macrophages and promotes co-invasion of cancer cells. Life Sci Alliance. 6 (11), e202302158 (2023).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Shi, X., et al. Mechanism insights and therapeutic intervention of tumor metastasis: latest developments and perspectives. Signal Transduct Target Ther. 9 (1), 192 (2024).
- Li, G. N., Livi, L. L., Gourd, C. M., Deweerd, E. S., Hoffman-Kim, D. Genomic and morphological changes of neuroblastoma cells in response to three-dimensional matrices. Tissue Eng. 13 (5), 1035-1047 (2007).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. 160, e61392 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유