Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Gene mundial y Análisis de Expresión utilizando una plataforma de microarrays de oligonucleótidos de pez cebra
Resumen
Microarrays de genes son herramientas poderosas en el perfil de expresión génica a nivel de todo el genoma. Esta tecnología tiene aplicación en una variedad de disciplinas biológicas como la biología del desarrollo y la toxicología. En este video, se detalla un protocolo para el análisis global de la expresión génica utilizando microarrays de oligonucleótidos de una plataforma completa para el pez cebra.
Resumen
Gen la tecnología de microarrays permite la medición cuantitativa y perfiles de expresión génica de los niveles de transcripción en forma todo el genoma. Gen la tecnología de microarrays se utiliza en numerosas disciplinas biológicas en una variedad de aplicaciones, incluyendo el análisis global de la expresión génica en relación con la etapa de desarrollo, a un estado de enfermedad, y en las respuestas tóxicas. En este documento, se incluye una demostración de un análisis global de la expresión génica utilizando una amplia pez cebra-específicas de la plataforma de microarrays de oligonucleótidos. La expresión de pez cebra plataforma de microarrays contiene 385.000 sondas, 60 pares de bases de longitud, interrogando a 37.157 blancos con hasta 12 sondas por objetivo. Para esta plataforma, toda la información genómica y de cDNA para el pez cebra se obtuvieron de diversas bases de datos genómicas incluyendo Ensembl (http://www.ensembl.org), VEGA (http://vega.sanger.ac.uk), UCSC ( http://genome.ucsc.edu), y ZFIN (http://www.zfin.org). Como resultado de esta gama de expresión ofrece una cobertura completa de la transcriptome pez cebra actual. Los microarrays de expresión pez cebra fue impreso por Roche NimbleGen (Madison, WI). Esta demostración técnica incluye el marcaje fluorescente de un producto de cDNA, hibridación de los productos etiquetados cDNA a la plataforma de microarray y análisis de matriz para la adquisición de señales mediante la estrategia de un solo color el análisis.
Protocolo
Parte 1: marcaje fluorescente de cDNA
El tinte Cy3 utilizados en el experimento de microarrays es sensible a la foto-degradación. El procedimiento debe llevarse a cabo en una mínima cantidad de luz. El protocolo de etiquetado es una adaptación del sistema Array CGH BioPrime Etiquetado Genómica Manual 1.
- Los reactivos para la reacción de marcaje se almacenan a -20 ° C. Desde el CGH array BioPrime Genómica sistema de etiquetado descongelar 2,5 veces el tampón Primer azar, mezcla de dCTP 10 veces, y el agua libre de nucleasa. También descongelar el tinte Cy3-dCTP. Después de descongelar, en pocas palabras centrífuga todos los reactivos antes de usar.
- Una reacción de marcaje por separado tendrá que ser completado para cada muestra de ADNc para ser analizados. En un 0,6 ml de pared gruesa del tubo se añaden 500 ng de cDNA, 40 ml Buffer 2,5 veces el Primer azar y el agua libre de nucleasa para alcanzar un volumen total de 86 microlitros.
- Mezclar bien y centrifugar brevemente. Incubar la reacción durante 5 minutos a 100 ° C en un termociclador.
- Inmediatamente la transferencia de reacción en una suspensión de baño de hielo durante 10 minutos.
- Para cada tubo se añaden 10 l de mezcla dCTP 10 veces, 2 l 1 mM Cy3-dCTP y 2 l Exo ADN polimerasa de Klenow. La polimerasa de ADN Klenow Exo se debe dejar en el congelador hasta que sea necesario y siempre se debe mantener en hielo.
- Mezcla de reacción bien y centrifugar brevemente.
- Incubar la reacción a 37 ° C durante 16 horas en un termociclador.
Parte 2: La precipitación de cDNA etiqueta
- Antes de continuar en el turno de protocolo en el sistema de hibridación NimbleGen y permitir que el sistema se equilibre a 42 ° C.
- Para cada reacción, el lugar de una columna Microcon en un tubo de 1.5 ml. Añadir 300 l SSC 0,1 X a cada columna seguidos por el contenido de la reacción de marcaje (100 l).
- Centrifugar el tubo que contiene la columna a 16.100 xg durante 10 minutos a 4 ° C.
- Deseche el flujo a través, que puede ser un poco de color rosa, y añadir otros 300 l SSC 0,1 X a la columna.
- Repetir la centrifugación a 16.100 xg durante 10 minutos a 4 ° C.
- Deseche el flujo a través y agregar un adicional de 300 l SSC 0,1 X a la columna.
- Centrifugar a 16.100 xg durante 12 minutos a 4 ° C.
- Deseche el flujo a través. El flujo a través Debe quedar claro después de este último lavado y usted debería ser capaz de ver la rosa cDNA marcado en la columna.
- Aplicar 100 l agua libre de nucleasa a la columna. Eliminar la columna del tubo de 1.5 ml y se invierte la columna en un tubo de 1.5 ml nuevo.
- Centrifugar a 2300 xg durante 2 minutos a 4 ° C. El cDNA marcado debe ser rehidratado en el agua que se acumula en la parte inferior del tubo.
- Use un NanoDrop ND-1000 espectrofotómetro de acuerdo a las instrucciones del fabricante para el cálculo de la concentración de ADN y evaluar la incorporación tinte mediante el cálculo de la base de tinte en la relación de nucleótidos a Cy3 relación es igual a la A 260 / A 550 multiplicado por 23,15. La base de relación de medio de contraste debe ser entre 50 y 80.
- Si cDNA si la etiqueta lo suficiente, alícuota de 6 mg de ADNc marcado en un tubo de 1.5 ml nuevo.
- Mezcle en el volumen de 0.1X de 3 M NaOAc, pH 5,2 y 1X volumen de isopropanol. Deje que la mezcla de incubación en la oscuridad durante 30 minutos a temperatura ambiente.
- A continuación centrifugar la muestra a 16.100 g durante 20 minutos a temperatura ambiente.
- Con cuidado, descartar el sobrenadante sin perturbar el precipitado de ADN, que debe ser de color rosado.
- Realizar un lavado de etanol mediante la adición de 500 l EtOH 70% y mezclar por inversión suave.
- Centrifugar la muestra a 16.100 xg durante 5 minutos a temperatura ambiente.
- Eliminar el sobrenadante, invierta el tubo, y permitir que el precipitado se seque durante 5 minutos. En este paso la bolita se puede almacenar a -20 º C si se desea o el protocolo se puede continuar con el paso de hibridación.
Parte 3: La hibridación
El procedimiento de hibridación es una adaptación de la Guía del usuario Roche NimbleGen matrices para dos análisis de expresión génica.
- Disolver el precipitado en 5 l de agua libre de nucleasa.
- Una vez que la pastilla se disuelve añadir componentes de la hibridación en la Tabla 1. Componentes de la hibridación son del Kit de hibridación Roche NimbleGen. Después de todos los componentes se añaden el volumen total ahora un total de 18 microlitros. Mezclar bien y brevemente girar el tubo para recoger el contenido en la parte inferior.
- El siguiente paso es desnaturalizar el cDNA marcado por colocarlo en un bloque de calor a 95 ° C durante 5 minutos.
- Transferir inmediatamente el tubo con el sistema de hibridación NimbleGen, mientras que las matrices se preparan en los siguientes pasos.
- Microarrays siempre deben manipularse con cuidado y almacenarse de acuerdo con las instrucciones del fabricante. Roche NimbleGen recomienda el almacenamiento de sus arrays de expresión en la oscuridad con desecante a temperatura ambiente.
- Cargar la matriz en la herramienta de alineación de precisión Mixer (PMAT), siempre en los accesorios del procesado NimbleGen matriz.
- Carga de un mezclador de X1 en el PMAT y retire la cinta adhesiva con una pinza. Cierre el PMAT segura. Aplique presión a la mesa de mezclas a través del agujero en el PMAT y abrir lentamente para que la matriz y un mezclador de retirarse de la PMAT.
- Firmemente ejercer presión con el rodillo en el exterior de la mesa de mezclas para asegurar el sello de la matriz es completa y para eliminar las burbujas. Coloque el complejo conjunto mezclador en el sistema de hibridación para calentar.
- Usando una pipeta de desplazamiento, agregue lentamente 15 l de la muestra de la etiqueta en el puerto de llenado. Este es el agujero situado en el centro de la mesa de mezclas. Aplique una presión lenta y constante de la pipeta de modo que la muestra se extienda de manera uniforme a través de la mezcla sin burbujas de aire. Limpie cualquier exceso de muestra de los orificios y sellar los agujeros de puerto con cinta adhesiva.
- Cierre el pestillo y empezar a mezclar de la solución de hibridación con el programa B en el sistema de hibridación NimbleGen. Permitir la hibridación de microarrays de 16-20 horas.
Parte 4: hibridación de Post-Lava y Análisis de Microarrays
El procedimiento de lavado y barrido es una adaptación de la Guía del usuario Roche NimbleGen matrices para análisis de expresión génica 2.
- Una vez que la hibridación está casi terminada preparar tampones de lavado como se indica en la Tabla 2. Pre-caliente IA tampón de lavado a 42 ° C en un baño de agua.
- Encienda el escáner GenePix serie 4000B y permitir que el escáner se caliente durante unos 15 minutos antes de su uso.
- Vierta la IA lavado pre-calentado de búfer en un plato.
- Quitar el complejo conjunto mezclador del sistema de hibridación NimbleGen y caer en la plantilla (incluido en los accesorios del procesado NimbleGen matriz). Sumerja asamblea en pre-calentado IA tampón de lavado y retirar con cuidado mezcladora pelando de diapositivas.
- Quitar matriz de plantilla y agite por 15 segundos en el tampón de lavado IA.
- Rápidamente la transferencia de matriz en un estante de la diapositiva en el IB temperatura ambiente tampón de lavado. Agite la gradilla para portaobjetos enérgicamente durante 2 minutos.
- La transferencia de la parrilla portaobjetos en el tampón de lavado II y agitar durante 1 minuto.
- La transferencia de la parrilla portaobjetos en el tampón de lavado III y agite por 15 segundos.
- La transferencia de la matriz en la diapositiva de secado y centrifugado giro de 2 minutos para eliminar toda la humedad. Proceder a la exploración.
- Es recomendable analizar la matriz inmediatamente, ya que el tinte es sensible a la luz y la capa de ozono.
- Cargar la matriz, código de barras hacia abajo, en el escáner de gama GenePix 4000B.
- Abra el software GenePix Pro y proceder a la exploración de la matriz. La Roche NimbleGen matrices de expresión génica utilizar una estrategia de un solo color hibridación. Como resultado de una sola longitud de onda es necesaria para la exploración. Exploración Cy3 señal de fluorescencia utilizando el láser de 532 nm. Abra el cuadro de diálogo Configuración de hardware y establecer el valor de PMT del láser de 532 nm a un valor de 500.
- En el cuadro de diálogo de configuración de hardware, sistema de energía al 100%, tamaño de píxel de 5 micras, las líneas de media a 1, y el foco puesto en 0 micras.
- Realizar una exploración previa y establecer el área de exploración.
- Realizar un análisis.
- Una vez que haya finalizado la digitalización comprobar el histograma de la imagen. Lo ideal sería que 1E-5 cuentas normalizado debe ser de 65.000 significado límite de saturación que ~ 1.2% de las características se encuentran saturados. Si el recuento de normalización están a menos de 1e-5, aumentar la ganancia PMT y diapositivas a escanear el. Si el recuento normalizado son mayores que 1e-5, disminuye la ganancia PMT y diapositivas a escanear el.
- Una vez que haya finalizado la digitalización guardar la imagen. Se recomienda elegir una convención de nomenclatura estándar para las imágenes para facilitar la referencia. La imagen ahora se pueden cargar en el programa de análisis de datos de preferencia para la normalización de datos y cálculo de los valores de intensidad.
Resultados representante
En primer lugar, puede comenzar a evaluar la eficacia de la reacción de marcaje fluorescente Cy3 del cDNA al comenzar el lavado con 0.1X SSC. Durante cada lavado posterior el flujo a través debería ser menos de color rosa con el último flujo a través de claro. Además, el Cy3 marcado cDNA producto debe ser visible en la columna durante estas etapas de lavado. Si el flujo a través de es de color rosa brillante en color o no hay ningún producto de color rosa en la columna, esta es una buena indicación de que la reacción de marcaje no procedió correctamente. La reacción de marcaje se puede evaluar cuantitativamente mediante el ND-1000 NanoDrop espectrofotómetro para determinar la concentración de la etiqueta de ADN y la base de proporción de medio de contraste (Figura 1). La reacción de marcaje habitualmente produce al menos 10 mg de ADN marcado en nuestro laboratorio. Incorporación de tinte se puede calcular mediante una sencilla base / medio de contraste en relación a la secuencia de nucleótidos Cy3 relación es igual a la A 260 / A 550 multiplicado por 23,15. Valores que van desde 50 hasta 80 indican incorporación tinte suficiente en la reacción de marcaje. Si la reacción de marcaje produce una baja concentración de ADN o la incorporación tinte pobres, la hibridación de la muestra en el micrófonoroarray no es recomendable y etiquetado de reacción se debe repetir.
Evaluación de la calidad de la hibridación puede llevarse a cabo mediante la evaluación del histograma de la imagen y la imagen escaneada de los microarrays (Figura 2). Hibridaciones aceptable tendrá un 1-2% de las características saturado (es decir, dando 1e-5 cuenta normalizado en el límite de saturación de 65.000) con el valor de PMT establecido cerca de 500. Si el valor de PMT tiene que ser ajustado drásticamente de 500 a alcanzar 1e-5 cuenta normalizado en el límite de saturación de 65.000, esto generalmente indica hibridación pobres del cDNA marcado con la micromatriz. Calidad de los datos estará compuesta por si estos datos se continúa a través de su análisis posterior. La calidad de la hibridación puede ser evaluada cualitativamente por la visualización de imágenes escaneadas de la matriz (Figura 3). El kit de Roche NimbleGen hibridación contiene una mezcla de oligo alineación de Cy3 y Cy5-etiquetados oligonucleótidos que hibridan 48mer a las características de alineación en las matrices. Evaluación cualitativa de la intensidad de los oligos de alineación con otras características de la matriz permite un control de calidad en la eficiencia de la hibridación del cDNA marcado con fluorescencia en la matriz. Tras el análisis anterior, un diagrama de dispersión de los datos analizados se puede utilizar para determinar los niveles relativos de expresión de genes y la identificación de genes que son expresados diferencialmente entre las muestras (Figura 4).
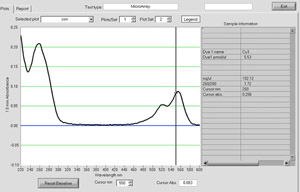
Figura 1. Un espectro NanoDrop de cDNA marcado con Cy3. Rendimiento de ADNc marcado y la incorporación de medio de contraste puede ser evaluada mediante un ND-1000 NanoDrop espectrofotómetro. Por favor, haga clic aquí para ver una versión ampliada de la figura 1.
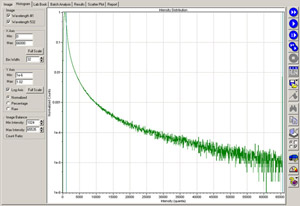
Figura 2. Un histograma GenePix de microarrays de análisis. Calidad de la hibridación puede ser evaluado por el histograma de la imagen. Hibridaciones aceptable tendrá un 1-2% de las características saturado de 1e-5 cuenta normalizado en el límite de saturación de 65.000 con el valor de PMT establecido cerca de 500. Por favor, haga clic aquí para ver una versión ampliada de la figura 2.

Figura 3. Una imagen escaneada de microarrays. Hibridación de calidad puede ser evaluada mediante la visualización de la imagen escaneada de la micromatriz. La imagen debe ser examinado para detectar la presencia de polvo u otros factores que interfieren en la prohibición de visualización de cada función y, por tanto, la prevención de un cálculo preciso de la intensidad de fluorescencia de la función. La intensidad de los oligos alineación puede ser comparada con las otras características de la matriz para evaluar la eficiencia de la hibridación.

Figura 4. Un gráfico de dispersión de un experimento de microarrays analizados. El objetivo de este procedimiento es identificar los genes que se expresan diferencialmente en las muestras. Microarrays de datos pueden ser analizados y ver en un gráfico de dispersión para visualizar los genes que son expresados diferencialmente.
Tabla 1. Componentes de Kit Roche NimbleGen hibridación agregar para el paso de hibridación 2.
| Componente | Volumen |
| Muestra (disuelto en agua) | 5 l |
| Tampón de hibridación 2X | 9 l |
| Una hibridación | 3,6 l |
| Alineación Oligo | 0,4 l |
| Volumen total | 18 l |
Tabla 2. Lave buffers que estar preparados para el lavado de las matrices siguientes hibridación.
| Componente | Tampón de lavado IA * | Tampón de lavado IB | Tampón de lavado II | Tampón de lavado III |
| De agua | 225 ml | 225 ml | 225 ml | 225 ml |
| 10 veces el tampón de lavado I, II o III | 25 ml | 25 ml | 25 ml | 25 ml |
| 1 M de TDT | 25 l | 25 l | 25 l | 25 l |
| Volumen total | 250 ml | 250 ml | 250 ml | 250 ml |
* Pre-caliente a 42 ° C
Discusión
Muchos protocolos de microarrays, incluyendo el que se detalla aquí, utilice colorantes fluorescentes como un medio de medir y cuantificar la hibridación. El tinte fluorescente Cy3 es sensible a la foto y la degradación de la capa de ozono. Se recomienda que el procedimiento se llevó a cabo en un mínimo de iluminación. Además, las pequeñas partículas de polvo pueden interferir con la hibridación y la exploración. Se debe prestar especial atención a asegurarse de que el área de trabajo esté limpia y libre d...
Materiales
| Name | Company | Catalog Number | Comments |
| Array Microcentrifuge Dryer | ISC Bioexpress | C-1303-T | |
| BioPrime Array CGH Genomic Labeling System | Invitrogen | 18095-011 | |
| Cyanine 3-dCTP | PerkinElmer, Inc. | NEL576 | |
| GenePix 4000B | Molecular Devices | GENEPIX 4000B | |
| Microcon YM-30 | EMD Millipore | 42411 | |
| NimbleGen Array Processing Accessories | Roche Group | 5223539001 | |
| NimbleGen Hybridization Kit | Roche Group | 5223474001 | |
| NimbleGen Hybridization System 4 | Roche Group | 5223652001 | |
| NimbleGen Wash Buffer Kit | Roche Group | 5223504001 | |
| X1 Mixers | Roche Group | 5223725001 |
Referencias
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados