Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Globale Genexpressionsanalyse Mit einem Zebrafisch Oligonukleotid-Microarray-Plattform
In diesem Artikel
Zusammenfassung
Gene Microarrays sind mächtige Werkzeuge in Genexpressions-Profiling bei einer genomweiten Ebene. Diese Technologie findet Anwendung in einer Vielzahl von biologischen Disziplinen Entwicklungsbiologie und Toxikologie. In diesem Video-, Detail haben wir ein Protokoll für die globale Genexpressionsanalyse mit einem umfassenden Oligonukleotid-Microarray-Plattform für die Zebrafisch.
Zusammenfassung
Gene Microarray-Technologie erlaubt die quantitative Messung und Genexpressions-Profiling von Transkripten auf einer genomweiten Basis. Gene Microarray-Technologie wird in zahlreichen biologischen Disziplinen in einer Vielzahl von Anwendungen, darunter globale Genexpressionsanalyse in Bezug auf Entwicklungsstadium, einer Krankheit Zustand verwendet, und in toxischen Reaktionen. Hierin sind wir eine Demonstration der globalen Genexpressionsanalyse mit einem umfassenden Zebrafisch-spezifischen Oligonukleotid-Microarray-Plattform. Der Zebrafisch Ausdruck Microarray-Plattform enthält 385.000 Sonden, 60 Basenpaaren Länge, verhören 37.157 Ziele mit bis zu 12 Sonden pro Target. Für diese Plattform wurden alle cDNA und genomischer Informationen für die Zebrafisch aus verschiedenen genomischen Datenbanken einschließlich Ensembl (http://www.ensembl.org), VEGA (http://vega.sanger.ac.uk), UCSC gesammelt ( http://genome.ucsc.edu) und ZFIN (http://www.zfin.org). Als Ergebnis dieses Ausdrucks-Array bietet eine vollständige Abdeckung des aktuellen Zebrafisch Transkriptom. Der Zebrafisch Ausdruck Microarray wurde von Roche NimbleGen (Madison, WI) gedruckt. Diese technische Demonstration umfasst die Fluoreszenzmarkierung der cDNA-Produkt, die Hybridisierung der markierten cDNA-Produkt an den Microarray-Plattform und Array-Scanning für die Signalerfassung mit dem eine Farbe Analyse-Strategie.
Protokoll
Teil 1: Fluoreszenzmarkierung von cDNA
Der Cy3-Farbstoff in der Microarray-Experiment verwendet wird sensibel auf Foto-Abbau. Das Verfahren soll in einer minimalen Menge an Licht zu nehmen. Die Kennzeichnung Protokoll wurde von der BioPrime Array-CGH Genomic Labeling System Manual 1 angepasst.
- Die Reagenzien für die Kennzeichnung Reaktion bei -20 ° C gelagert Von der BioPrime Array-CGH Genomic Labeling System Tauwetter 2.5X Random Primer Buffer, 10X dCTP-Mix und Nuklease-freies Wasser. Auch auftauen Cy3-dCTP Farbstoff. Nach dem Auftauen kurz zentrifugieren alle Reagenzien vor dem Gebrauch.
- Eine separate Kennzeichnung Reaktion müssen für jede cDNA zu analysierende Probe abgeschlossen sein. In einem 0,6 ml dickwandigen Rohr add 500 ng cDNA, 40 ul 2,5 X Random Primer Buffer und Nuklease-freiem Wasser auf ein Gesamtvolumen von 86 ul zu erreichen.
- Gut mischen und kurz zentrifugieren. Inkubieren Reaktion für 5 Minuten bei 100 ° C auf einem Thermocycler.
- Unmittelbar Transfer-Reaktion in ein Eisbad Gülle für 10 Minuten.
- Zu jedem Röhrchen fügen Sie dann 10 ul 10x dCTP-Mix, 2 ul 1 mM Cy3-dCTP und 2 ul Exo Klenow-DNA-Polymerase. Die Exo Klenow DNA Polymerase sollte in den Gefrierschrank gelassen werden, bis sie benötigt und sollte immer auf Eis gehalten werden.
- Mix Reaktion gut und kurz zentrifugieren.
- Inkubieren Reaktion bei 37 ° C für 16 Stunden in einem Thermocycler.
Teil 2: Fällung von markierten cDNA
- Bevor Sie fortfahren in dem Protokoll wieder auf die NimbleGen Hybridization System und damit das System auf 42 ° C stehen gelassen
- Für jede Reaktion, legen Sie ein Microcon Säule in ein 1,5 ml-Tube. Geben Sie 300 ul 0,1 X SSC, um jede Spalte durch den Inhalt der Kennzeichnung Reaktion (100 ul).
- Zentrifugieren Sie die Röhrchen mit der Säule bei 16.100 xg für 10 Minuten bei 4 ° C.
- Verwerfen Sie den Durchfluss durch, die leicht rosa kann in Farbe, und fügen Sie weitere 300 ul 0,1 X SSC Spalte.
- Wiederholen Sie die Zentrifugation bei 16.100 xg für 10 Minuten bei 4 ° C.
- Verwerfen Sie den Durchfluss durch und füge eine weitere 300 ul 0,1 X SSC Spalte.
- Zentrifugation bei 16.100 xg für 12 Minuten bei 4 ° C.
- Entsorgen Sie durchströmen. Die Durchströmung sollte nach diesem letzten waschen klar, und Sie sollten in der Lage sein, um die rosa markierten cDNA auf die Spalte klicken.
- Apply 100 ul Nuklease-freies Wasser in die Spalte. Entfernen Sie die Spalte aus dem 1,5-ml-Tube und invertieren Säule in ein neues 1,5-ml-Tube.
- Zentrifuge bei 2.300 xg für 2 Minuten bei 4 ° C. Die markierten cDNA sollte in das Wasser, das an der Unterseite des Rohres sammelt rehydriert werden.
- Verwenden Sie einen NanoDrop ND-1000 Spektrophotometer nach den Anweisungen des Herstellers, um DNA-Konzentration berechnen und auswerten Farbstoff Aufnahme durch die Berechnung der Basis-Verhältnis Farbstoff, wo das Nukleotid an Cy3-Verhältnis entspricht dem A 260 / A 550-Verhältnis von 23,15 multipliziert. Die Basis zu färben Verhältnis sollte zwischen 50 und 80 sein.
- Wenn cDNA wenn beschriftet ausreichend, etikettiert aliquoten 6 ug cDNA in ein neues 1,5-ml-Tube.
- Mix in 0,1 × Volumen 3 M NaOAc, pH 5,2 und 1X Volumen Isopropanol. Eine Mischung im Dunkeln für 30 Minuten bei Raumtemperatur inkubieren.
- Next Zentrifuge die Probe bei 16.100 xg für 20 Minuten bei Raumtemperatur.
- Vorsichtig den Überstand verwerfen, ohne die DNA-Pellet, das rosa sein sollte in Farbe.
- Führen Sie eine Ethanol waschen durch Zugabe von 500 ul 70% EtOH zugeben und durch vorsichtiges Schwenken.
- Centrifuge Probe bei 16.100 xg für 5 Minuten bei Raumtemperatur.
- Entfernen Sie den Überstand, das Röhrchen und lassen Sie das Pellet für 5 Minuten trocknen lassen. Bei diesem Schritt das Pellet kann bei -20 ° C gelagert werden, wenn gewünscht oder kann das Protokoll auf der Hybridisierung fortgesetzt werden.
Teil 3: Hybridisierung
Die Hybridisierung Verfahren ist von der Roche NimbleGen Arrays Benutzerhandbuch für Genexpressionsanalyse 2 angepasst.
- Lösen Sie das Pellet in 5 ul der Nuklease-freies Wasser.
- Sobald das Pellet gelöst ist hinzuzufügen Hybridisierung Komponenten in Tabelle 1 aufgeführt. Hybridisierung Komponenten werden von der Roche NimbleGen Hybridization Kit. Nachdem alle Komponenten hinzugefügt werden, das Gesamtvolumen sollte nun insgesamt 18 ul. Gut mischen und kurz drehen Sie das Rohr, um den Inhalt am Boden sammeln.
- Der nächste Schritt ist, um die markierte cDNA, indem sie auf einem Heizblock bei 95 ° C für 5 Minuten denaturiert.
- Sofortüberweisung das Rohr auf die NimbleGen Hybridization System während der Arrays in den folgenden Schritten bereit sind.
- Microarrays sollte immer sorgfältig behandelt und gelagert werden gemäß den Anweisungen des Herstellers. Roche NimbleGen empfiehlt die Speicherung ihrer Expressions-Arrays in der Dunkelheit mit Trockenmittel bei Raumtemperatur.
- Laden Sie das Array in der Präzisions-Mixer Alignment Tool (PMAT) in die NimbleGen Array-Signalverarbeitung Zubehör zur Verfügung gestellt.
- Laden Sie ein X1-Mixer auf den PMAT und entfernen Sie die Klebestreifen mit einer Pinzette. Schließen Sie die PMAT sicher. Drücken Sie auf den Mixer durch das Loch in der PMAT und langsam geöffnet, so dass das Array und Mixer aus dem PMAT lösen.
- Fest drücken Sie mit der Brayer um die Außenseite des Mischers, um die Dichtung auf das Array zu gewährleisten ist vollständig und zu jeder Blasen zu entfernen. Legen Sie die Array-Mischer Komplexes auf die Hybridisierung System warm.
- Mit einer Pipette langsam zugeben 15 ul der markierten Probe in die Einfüllöffnung. Dies ist das Loch in der Mitte des Mixers. Wenden Sie langsam und stetig Druck auf die Pipette, so dass die Probe gleichmäßig verteilt über den Mixer ohne Luftblasen. Wischen Sie überschüssiges Probe aus der Bullaugen und Dichtung Port Löcher mit Klebestreifen.
- Schließen Sie die Verriegelung und starten Vermischung der Hybridisierungslösung mit dem Programm B auf der NimbleGen Hybridization System. Lassen Microarrays für 16-20 Stunden hybridisieren.
Teil 4: Post-Hybridisierung Wash und Scannen von Microarrays
Die Wasch-und Scan-Vorgang wird von der Roche NimbleGen Arrays Benutzerhandbuch für Genexpressionsanalyse 2 angepasst.
- Nach Hybridisierung ist kurz vor dem Abschluss vorzubereiten Waschpuffer wie in Tabelle 2 dargestellt. Pre-warmen Waschpuffer IA bis 42 ° C in einem Wasserbad.
- Schalten Sie GenePix 4000B Array-Scanner und lassen Sie den Scanner für etwa 15 Minuten vor dem Gebrauch warm.
- Gießen Sie den vorgewärmten Waschpuffer IA in eine Schale.
- Entfernen Sie die Array-Mischer Komplex aus der NimbleGen Hybridisierung und gleiten in die Schablone (im Lieferumfang enthalten in der NimbleGen Array-Signalverarbeitung Zubehör). Tauchen Montage in vorgewärmten Waschpuffer IA und entfernen Sie vorsichtig Mischer durch Abziehen von Folie.
- Remove Array aus Vorrichtungs-und agitieren für 15 Sekunden in Waschpuffer IA.
- Schnelle Übertragung von Array in eine Folie Rack in die Raumtemperatur Waschpuffer IB. Man schüttelt die Dia-Rack 2 Minuten kräftig.
- Die Objektträger-Rack in Waschpuffer II und agitieren für 1 Minute.
- Die Objektträger-Rack in den Waschpuffer III und agitieren für 15 Sekunden.
- Übertragen Sie die Array in die Folie Trocknungszentrifuge und Spin für 2 Minuten, um alle Feuchtigkeit zu entfernen. Gehen Sie zum Scannen.
- Es wird empfohlen, um das Array zu scannen, da der Farbstoff ist empfindlich gegen Licht und Ozon.
- Laden Sie das Array, Barcode nach unten in die GenePix 4000B Array-Scanner.
- Öffnen Sie die GenePix Pro-Software und fahren Sie mit Scannen von Array. Die Roche NimbleGen Genexpressionsarrays nutzen eine Farbe Hybridisierung Strategie. Als Ergebnis nur eine Wellenlänge ist für das Scannen erforderlich. Scan Cy3 Fluoreszenz-Signal mit der 532 nm-Laser. Öffnen Sie die Hardware-Einstellung im Dialogfeld und stellen PMT Wert der 532 nm-Laser auf einen Wert von 500.
- In der Hardware-Einstellung im Dialogfeld eingestellten Leistung bis 100%, Pixelgröße bis 5 Mikron, Linien, um durchschnittlich 1, und Fokuslage auf 0 Mikron.
- Führen Sie eine Vorschau-Scan und Scan-Bereich gesetzt.
- Führen Sie zu scannen.
- Sobald das Scannen abgeschlossen ist überprüfen Sie die Bild-Histogramm. Idealerweise 1e-5 normalisiert zählt sollten bei 65.000 Sättigungsgrenze dh ~ 1-2% der Features sind gesättigt werden. Wenn die normierten Werte von weniger als 1e-5 sind, erhöhen Sie die PMT-Verstärkung und scannen Sie das Bild. Wenn die normierte zählt mehr als 1e-5 sind, verringern Sie die PMT-Verstärkung und scannen Sie das Bild.
- Sobald das Scannen abgeschlossen ist das Bild speichern. Es wird empfohlen, eine Standard-Namenskonvention für Bilder für Leichtigkeit in Bezug zu wählen. Das Bild kann nun in die Datenanalyse-Programm der Vorliebe für Daten-Normalisierung und Berechnung der Intensität Werte geladen werden.
Repräsentative Ergebnisse
Sie können erst beginnen, um die Effizienz des Cy3 Fluoreszenz-Markierung Reaktion der cDNA zu beurteilen, wenn zu Beginn der Waschungen mit 0,1 × SSC. Bei jedem weiteren waschen die Durchströmung sollte weniger rosa in der Farbe mit dem letzten Durchströmung klar. Darüber hinaus sollten die Cy3-markierten cDNA-Produkt auf der Säule sichtbar in diesen Waschschritten. Wenn der Durchfluss durch ist hell rosa in der Farbe oder es ist kein rosa Produkt auf der Säule, ist dies ein guter Hinweis darauf, dass die Kennzeichnung Reaktion nicht richtig gehen. Die Kennzeichnung Reaktion kann beurteilt quantitativ mit dem NanoDrop ND-1000-Spektralphotometer zur Konzentration der markierten DNA und Basis zu färben Verhältnis (Abbildung 1) zu bestimmen. Die Kennzeichnung Reaktion routinemäßig ergibt mindestens 10 pg markierter DNA in unserem Labor. Dye Einarbeitung kann mit einem einfachen base / Farbstoff-Verhältnis, wo das Nukleotid an Cy3-Verhältnis entspricht dem A 260 / A 550-Verhältnis von 23,15 multipliziert. Werte im Bereich von 50 bis 80 zeigen, ausreichend Farbstoff Einarbeitung in die Kennzeichnung Reaktion. Wenn die Kennzeichnung Reaktion ergibt sich eine niedrige Konzentration von DNA oder schlechte Farbstoff Gründung, Hybridisierung der Probe auf die microarray wird nicht empfohlen und Kennzeichnung Reaktion sollte wiederholt werden.
Beurteilung der Qualität der Hybridisierung kann durch die Auswertung der Bild-Histogramm und das gescannte Bild des Mikroarrays (Abbildung 2) durchgeführt werden. Acceptable Hybridisierungen haben 1-2% der Funktionen gesättigt (dh, wodurch 1e-5 normalisierte Zählungen an die 65.000 Sättigungsgrenze) mit der PMT Wert nahe 500 gesetzt. Wenn die PMT Wert drastisch von 500 eingestellt werden, um 1e-5 normalisierte Zählungen an die 65.000 Sättigungsgrenze erreichen, dies in der Regel deutet auf eine schlechte Hybridisierung der markierten cDNA auf den Mikroarray. Die Datenqualität wird zusammen, wenn diese Daten durch die anschließende Analyse fortgesetzt wird. Hybridisierung Qualität kann auch qualitativ, indem Sie Bilder der gescannten Array (Abbildung 3) beurteilt werden. Die Roche NimbleGen Hybridization Kit enthält eine Ausrichtung Oligo Mischung aus Cy3-und Cy5-markierten 48mer-Oligonukleotide, die Ausrichtung Features auf den Arrays hybridisiert. Qualitative Beurteilung der Intensität der Ausrichtung Oligos auf andere Funktionen auf dem Array ermöglicht einen Qualitäts-Check auf der Hybridisierung Effizienz der fluoreszenzmarkierten cDNA auf dem Array. Nach weiteren Untersuchungen kann ein Streudiagramm der analysierten Daten verwendet werden, um die relativen Niveaus der Genexpression zu bestimmen und Gene, die zwischen Ihrem Proben (Abbildung 4) zum Ausdruck zu identifizieren.
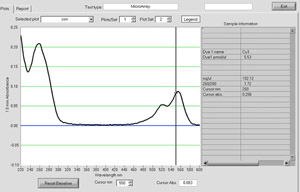
Abbildung 1. Ein NanoDrop Spektrum von cDNA markiert mit Cy3. Markierten cDNA-Ausbeute und Farbstoff Einarbeitung kann anhand einer NanoDrop ND-1000-Spektrophotometer werden. Bitte klicken Sie hier, um eine größere Version der Abbildung 1 zu sehen.
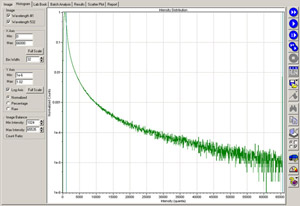
Abbildung 2. Ein GenePix Histogramm für Microarray-Scan. Qualität der Hybridisierung kann durch die Auswertung der Bild-Histogramm beurteilen. Acceptable Hybridisierungen haben 1-2% der Funktionen mit 1e-5 normalisierte Zählungen an die 65.000 Sättigungsgrenze mit der PMT Wert nahe 500 gesetzt gesättigt. Bitte klicken Sie hier, um eine größere Version der Abbildung 2 zu sehen.

Abbildung 3. Eine eingescannte Microarray Bild. Hybridisierung Qualität kann durch Anzeigen der gescannten Bild des Mikroarrays beurteilt werden. Das Bild sollte auf das Vorhandensein von Staub oder andere Störfaktoren Verbot Visualisierung der einzelnen Funktionen untersucht werden und somit zu verhindern genaue Berechnung der Fluoreszenz-Intensität des Merkmals. Die Intensität der Ausrichtung Oligos können die anderen Funktionen auf dem Array zu Hybridisierungseffizienz beurteilen verglichen werden.

Abbildung 4. Ein Streudiagramm einer analysierten Mikroarray-Experiment. Das Ziel dieses Verfahrens ist es, Gene, die in Ihrer Proben zum Ausdruck zu identifizieren. Microarray-Daten können weiter analysiert und betrachtet in einem Streudiagramm, um Gene, die differentiell exprimiert sind zu visualisieren.
Tabelle 1. Komponenten von Roche NimbleGen Hybridization Kit für Hybridisierung Schritt 2 hinzugefügt werden.
| Komponente | Volumen |
| Sample (gelöst in Wasser) | 5 pl |
| 2X Hybridisierungspuffer | 9 ul |
| Hyb A | 3,6 ul |
| Alignment Oligo | 0,4 ul |
| Gesamtvolumen | 18 ul |
Tabelle 2. Wash Puffer zum Waschen der Arrays nach der Hybridisierung vorbereitet werden.
| Komponente | Waschpuffer IA * | Waschpuffer IB | Waschpuffer II | Waschpuffer III |
| Wasser | 225 ml | 225 ml | 225 ml | 225 ml |
| 10X Waschpuffer I, II oder III | 25 ml | 25 ml | 25 ml | 25 ml |
| 1 M DTT | 25 ul | 25 ul | 25 ul | 25 ul |
| Gesamtvolumen | 250 ml | 250 ml | 250 ml | 250 ml |
* Pre-warm bis 42 ° C
Diskussion
Viele Microarray-Protokolle, darunter auch die hier detailliert, verwenden fluoreszierende Farbstoffe als ein Mittel zur Messung und Quantifizierung Hybridisierung. Die Cy3 Fluoreszenzfarbstoff ist empfindlich auf ein Foto, und Ozonabbau. Es wird empfohlen, dass das Verfahren in minimal-Beleuchtung durchgeführt werden. Darüber hinaus können kleine Staubpartikel mit Hybridisierung und Scannen stören. Besondere Aufmerksamkeit sollte sicherstellen, dass die Arbeit ist sauber und frei von Staub sein.
Materialien
| Name | Company | Catalog Number | Comments |
| Array Microcentrifuge Dryer | ISC Bioexpress | C-1303-T | |
| BioPrime Array CGH Genomic Labeling System | Invitrogen | 18095-011 | |
| Cyanine 3-dCTP | PerkinElmer, Inc. | NEL576 | |
| GenePix 4000B | Molecular Devices | GENEPIX 4000B | |
| Microcon YM-30 | EMD Millipore | 42411 | |
| NimbleGen Array Processing Accessories | Roche Group | 5223539001 | |
| NimbleGen Hybridization Kit | Roche Group | 5223474001 | |
| NimbleGen Hybridization System 4 | Roche Group | 5223652001 | |
| NimbleGen Wash Buffer Kit | Roche Group | 5223504001 | |
| X1 Mixers | Roche Group | 5223725001 |
Referenzen
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten