È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi di espressione genica globale Utilizzando un Zebrafish piattaforma Microarray oligonucleotidi
In questo articolo
Riepilogo
Microarray gene sono strumenti potenti in profili di espressione genica a livello di genoma. Questa tecnologia ha applicazioni in una varietà di discipline biologiche tra cui la biologia dello sviluppo e tossicologia. In questo video, abbiamo dettaglio un protocollo per l'analisi globale dell'espressione genica utilizzando una completa piattaforma microarray di oligonucleotidi per il pesce zebra.
Abstract
Gene microarray tecnologia permette la misurazione quantitativa e profili di espressione genica dei livelli di trascrizione su un genoma base. Gene microarray tecnologia è utilizzata in numerose discipline biologiche in una varietà di applicazioni, tra cui analisi globale dell'espressione genica in relazione alla fase di sviluppo, ad uno stato di malattia, e nelle risposte tossici. Qui, ci sono una dimostrazione di analisi globale dell'espressione genica utilizzando una completa zebrafish specifica piattaforma microarray di oligonucleotidi. La piattaforma microarray di espressione zebrafish contiene 385.000 sonde, 60 paia di basi di lunghezza, interrogando 37.157 con target fino a 12 sonde per ogni destinazione. Per questa piattaforma, tutte le informazioni cDNA e genomiche disponibili per il pesce zebra è stato raccolto da banche dati genomiche diverse tra cui Ensembl (http://www.ensembl.org), VEGA (http://vega.sanger.ac.uk), UCSC ( http://genome.ucsc.edu), e ZFIN (http://www.zfin.org). Di conseguenza questo array espressione fornisce una copertura completa del trascrittoma zebrafish corrente. I microarray di espressione zebrafish è stato stampato da Roche NimbleGen (Madison, WI). Questa dimostrazione tecnica comprende la marcatura fluorescente di un prodotto di cDNA, l'ibridazione del prodotto etichettato cDNA alla piattaforma microarray e la scansione di array per l'acquisizione del segnale utilizzando la strategia di analisi del colore.
Protocollo
Parte 1: marcatura fluorescente di cDNA
Il Cy3 colorante utilizzato nell'esperimento microarray è sensibile alla fotodegradazione. La procedura dovrebbe svolgersi in una minima quantità di luce. Il protocollo di etichettatura è stato adattato dalla BioPrime sistema di etichettatura CGH Array Genomic manuale 1.
- I reagenti per la reazione di marcatura sono conservati a -20 ° C. Dal BioPrime CGH Array Buffer Genomic sistema di etichettatura Primer disgelo 2.5X casuale, mescolare dCTP 10X e acqua priva di nucleasi. Anche il disgelo Cy3-dCTP colorante. Dopo lo scongelamento, centrifugare brevemente tutti i reagenti prima dell'uso.
- Una reazione etichettatura separata dovrà essere compilato per ogni campione del DNA da analizzare. In un ml 0,6 pareti spesse tubo aggiungere 500 ng di cDNA, 40 microlitri Buffer 2.5X Primer casuale e nucleasi acqua priva di raggiungere un volume totale di 86 microlitri.
- Mescolare bene e centrifugare brevemente. Incubare reazione per 5 minuti a 100 ° C su un termociclatore.
- Trasferire immediatamente la reazione in un bagno di liquami ghiaccio per 10 minuti.
- Per ogni tubo poi aggiungere 10 microlitri mix dCTP 10X, 2 microlitri 1 mM Cy3-dCTP e 2 microlitri Exo DNA polimerasi Klenow. La DNA polimerasi Exo Klenow dovrebbero essere lasciati nel congelatore fino al momento e deve essere sempre tenuto in ghiaccio.
- Mescolare bene e per breve tempo di reazione centrifuga.
- Incubare la reazione a 37 ° C per 16 ore in un termociclatore.
Parte 2: Precipitazioni di cDNA con etichetta
- Prima di proseguire nel turno protocollo sul sistema di ibridazione NimbleGen e permettere al sistema di equilibrare a 42 ° C.
- Per ogni reazione, inserire una colonna microcontrollore in una provetta da 1,5 ml. Aggiungere 300 ml 0,1 X. SSC per ogni colonna seguito dal contenuto della reazione di marcatura (100 l).
- Centrifugare la provetta contenente la colonna a 16.100 xg per 10 minuti a 4 ° C.
- Gettare il flusso attraverso, che può essere leggermente di colore rosa, e aggiungere un altro 300 ml 0,1 X. SSC alla colonna.
- Ripetere centrifugazione a 16.100 xg per 10 minuti a 4 ° C.
- Gettare il flusso attraverso e aggiungere un ulteriore 300 ml 0,1 X. SSC alla colonna.
- Centrifugare a 16100 xg per 12 minuti a 4 ° C.
- Gettare flusso attraverso. Il flusso attraverso dovrebbe essere chiaro dopo questo ultimo lavaggio e si dovrebbe essere in grado di vedere il cDNA rosa etichetta sulla colonna.
- Applicare 100 ul nucleasi acqua priva di colonna. Rimuovere colonna dal tubo da 1,5 ml e capovolgere colonna in un nuovo tubo di 1,5 ml.
- Centrifugare a 2300 xg per 2 minuti a 4 ° C. Il cDNA marcato dovrebbero essere reidratati in acqua che si raccoglie sul fondo della provetta.
- Utilizzare un NanoDrop ND-1000 spettrofotometro secondo le istruzioni del produttore per calcolare la concentrazione del DNA e valutare l'incorporazione colorante calcolando la base per tingere rapporto in cui il nucleotide a Cy3 rapporto è uguale alla A 260 / A 550 rapporto moltiplicato per 23,15. La base di colorante rapporto dovrebbe essere tra 50 e 80.
- Se cDNA se adeguatamente etichettati, aliquota 6 mg di cDNA marcato in un nuovo tubo di 1,5 ml.
- Mescolare in volume di 0,1 X. NaOAc 3 M, pH 5.2 e 1X isopropanolo. Lasciare miscela incubare al buio per 30 minuti a temperatura ambiente.
- Avanti centrifuga il campione a 16.100 xg per 20 minuti a temperatura ambiente.
- Eliminare attentamente il supernatante senza disturbare il pellet di DNA, che dovrebbe essere di colore rosa.
- Eseguire un lavaggio con l'aggiunta di etanolo 500 microlitri EtOH 70% e mescolare delicatamente per inversione.
- Centrifugare campione a 16.100 xg per 5 minuti a temperatura ambiente.
- Eliminare il surnatante, invertire il tubo, e consentire il pellet asciugare per 5 minuti. A questo punto il pellet possono essere conservati a -20 ° C se lo si desidera o il protocollo può essere continuato per l'ibridazione.
Parte 3: ibridazione
La procedura di ibridazione è tratto dal Manuale Roche NimbleGen utente Array per 2 analisi di espressione genica.
- Sciogliere il pellet in 5 ml di acqua priva di nucleasi.
- Una volta che il pellet è sciolto aggiungere componenti ibridazione elencati nella Tabella 1. Componenti sono ibridazione del kit Roche ibridazione NimbleGen. Dopo che tutti i componenti sono aggiunti il volume totale dovrebbe totale di 18 microlitri. Mescolare bene e brevemente girare il tubo per raccogliere il contenuto in basso.
- Il passo successivo è quello di snaturare il cDNA marcato ponendolo su un blocco di calore a 95 ° C per 5 minuti.
- Trasferire immediatamente il tubo al sistema di ibridazione NimbleGen mentre gli array vengono preparati i seguenti passaggi.
- Microarray dovrebbe sempre essere maneggiati con cura e conservati secondo le istruzioni del produttore. Roche NimbleGen consiglia di conservare le loro matrici di espressione al buio con essiccante a temperatura ambiente.
- Caricare l'array in strumento di allineamento di precisione Mixer (PMAT) fornito tra gli accessori serie elaborazione NimbleGen.
- Caricare un mixer X1 sul PMAT e togliere il nastro adesivo con una pinza. Chiudere il PMAT in modo sicuro. Applicare una pressione al mixer attraverso il buco nel PMAT e aprire lentamente in modo che l'array e mixer svincolarsi dal PMAT.
- Fermamente applicare la pressione con la Brayer intorno alla parte esterna del mixer per assicurare la tenuta alla matrice è completo e per rimuovere eventuali bolle. Posizionare l'array-mixer complesso sul sistema di ibridazione per riscaldarsi.
- Usando una pipetta a spostamento, lentamente, aggiungere 15 ml di campione etichettato nella porta di riempimento. Questo è il foro situato al centro del mixer. Applicare una pressione lenta e costante per la pipetta in modo che il campione si diffonde in modo uniforme attraverso il mixer senza bolle d'aria. Togliere qualsiasi campione in eccesso dai fori porta e la guarnizione oblò con strisce adesive.
- Chiudere il fermo e iniziare a mescolare la soluzione di ibridazione con B programma sul sistema di ibridazione NimbleGen. Microarray permettono di ibridare per 16-20 ore.
Parte 4: post-ibridazione Lava e scansione di microarray
La procedura di lavaggio e di scansione è tratto dal Manuale Roche NimbleGen utente Array per le analisi di espressione genica 2.
- Una volta che l'ibridazione è quasi completata la preparazione tamponi di lavaggio come indicato nella Tabella 2. Preriscaldare IA tampone di lavaggio a 42 ° C in un bagno d'acqua.
- Accendere lo scanner GenePix serie 4000B e consentire lo scanner al caldo per circa 15 minuti prima dell'uso.
- Versare il pre-riscaldato IA tampone di lavaggio in un piatto.
- Rimuovere l'array-mixer complesso del sistema di ibridazione NimbleGen e far scorrere in dima (fornita negli accessori serie elaborazione NimbleGen). Assemblaggio Immergere in pre-riscaldato IA tampone di lavaggio e rimuovere con attenzione mixer peeling lontano da diapositive.
- Rimuovere array da jig e agitare per 15 secondi nel tampone IA lavaggio.
- Trasferire rapidamente array in un rack di scorrimento nella IB Wash Buffer temperatura ambiente. Agitare il rack scivolo vigorosamente per 2 minuti.
- Trasferire il rack di scivolare in Wash Buffer II e agitare per 1 minuto.
- Trasferire il rack di scivolare nel tampone di lavaggio III e agitare per 15 secondi.
- Trasferire l'array nella diapositiva asciugatura centrifuga e gira per 2 minuti per rimuovere tutta l'umidità. Procedere alla scansione.
- Si raccomanda di eseguire la scansione dell'array immediatamente in quanto il colorante è sensibile alla luce e all'ozono.
- Caricare il, codice a barre serie in giù, nello scanner GenePix serie 4000B.
- Aprire il software GenePix Pro e procedere alla scansione di array. La Roche NimbleGen array di espressione genica utilizza una strategia di ibridazione colore. Di conseguenza solo una lunghezza d'onda è necessario per la scansione. Scansione Cy3 segnale di fluorescenza utilizzando il laser a 532 nm. Aprire la finestra di dialogo di impostazione hardware e impostare il valore PMT del laser 532 nm per un valore di 500.
- Nella finestra di dialogo di impostazione hardware, potenza impostata al 100%, dimensione del pixel a 5 micron, le linee di media a 1, e mettere a fuoco la posizione a 0 micron.
- Eseguire una scansione di anteprima e impostare area di scansione.
- Eseguire la scansione.
- Una volta completata la scansione controllare l'istogramma dell'immagine. Idealmente 1e-5 punti normalizzata dovrebbe essere di 65.000 senso che il limite di saturazione ~ 1-2% delle caratteristiche sono saturi. Se i conteggi normalizzati sono meno di 1e-5, aumentare il guadagno PMT e ripetere la scansione della diapositiva. Se i conteggi normalizzati sono maggiori di 1e-5, ridurre il guadagno PMT e ripetere la scansione della diapositiva.
- Una volta completata la scansione salvare l'immagine. Si consiglia di scegliere una convenzione di denominazione standard per le immagini per la facilità di riferimento. L'immagine può essere caricato nel programma di analisi dei dati di preferenza per la normalizzazione dei dati e calcolo dei valori di intensità.
Rappresentante Risultati
È possibile innanzitutto cominciare a valutare l'efficienza della reazione Cy3 etichettatura fluorescenza del cDNA quando si inizia la lava con 0,1 X. SSC. Durante ogni lavaggio successivo il flusso attraverso dovrebbe diventare meno colore rosa con l'ultimo flusso attraverso chiare. Inoltre, il marcato con Cy3 cDNA prodotto dovrebbe essere visibile sulla colonna durante queste fasi di lavaggio. Se il flusso attraverso è rosa brillante a colori o in mancanza di un medicinale rosa sulla colonna, questa è una buona indicazione che la reazione di marcatura non procedere correttamente. La reazione di marcatura può essere valutata quantitativamente con il NanoDrop ND-1000 spettrofotometro per determinare la concentrazione del DNA marcato e la base al rapporto di colorante (Figura 1). La reazione di marcatura produce regolarmente almeno 10 mg di DNA marcato nel nostro laboratorio. Incorporazione colorante può essere calcolato usando una semplice base / colorante rapporto in cui il nucleotide a Cy3 rapporto è uguale alla A 260 / A 550 rapporto moltiplicato per 23,15. Valori compresi tra 50-80 indicano sufficiente incorporazione colorante nella reazione di marcatura. Se la reazione di marcatura produce una bassa concentrazione di DNA o di incorporazione tinta poveri, ibridazione del campione sul microfonoroarray non è raccomandato e reazione di marcatura deve essere ripetuta.
Valutazione della qualità della ibridazione può essere condotto valutando l'istogramma dell'immagine e l'immagine digitalizzata del microarray (Figura 2). Ibridazioni accettabile avrà 1-2% delle caratteristiche saturi (cioè, ottenendo 1e-5 punti normalizzato al limite 65.000 saturazione) con il valore PMT nei pressi 500. Se il valore PMT deve essere regolato drasticamente da 500 a raggiungere 1e-5 punti normalizzato al limite 65.000 saturazione, questo indica generalmente scarsa ibridazione del cDNA marcato al microarray. La qualità dei dati sarà composto, se tale dato è proseguito attraverso l'analisi successive. Qualità ibridazione può anche essere valutata qualitativamente la visualizzazione di immagini dell'array digitalizzata (Figura 3). L'ibridazione Roche NimbleGen Il kit contiene una miscela di oligo allineamento Cy3 e Cy5-etichettati oligonucleotidi 48mer che ibridano le caratteristiche di allineamento sul array. Valutazione qualitativa dell'intensità del oligos allineamento ad altre funzioni sulla matrice permette un controllo di qualità sull'efficienza ibridazione del cDNA fluorescente sul array. A seguito di ulteriori analisi, un grafico a dispersione dei dati analizzati possono essere utilizzati per determinare i livelli relativi di espressione genica e per identificare i geni che sono espressi in modo differenziale tra i campioni (Figura 4).
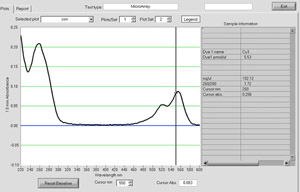
Figura 1. Uno spettro NanoDrop da cDNA marcato con Cy3. Resa cDNA marcato e l'incorporazione colorante può essere valutato utilizzando un NanoDrop ND-1000 spettrofotometro. Si prega di cliccare qui per vedere una versione più grande della figura 1.
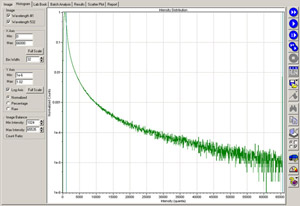
Figura 2. Un istogramma GenePix microarray per la scansione. Qualità del ibridazione può essere valutata analizzando l'istogramma dell'immagine. Ibridazioni accettabile avrà 1-2% delle caratteristiche saturo di 1e-5 punti normalizzato al limite di saturazione 65.000 con il valore PMT nei pressi 500. Si prega di cliccare qui per vedere una versione più grande della figura 2.

Figura 3. Un'immagine microarray digitalizzata. Ibridazione qualità può essere valutata osservando l'immagine digitalizzata del microarray. L'immagine deve essere esaminato per la presenza di polvere o di altri fattori di disturbo che vietano la visualizzazione di ogni funzione e, quindi, impedendo calcolo preciso delle intensità di fluorescenza della funzione. L'intensità del oligo allineamento può essere paragonato alle altre funzioni sulla matrice per valutare l'efficienza di ibridazione.

Figura 4. Un grafico a dispersione di un esperimento microarray analizzati. L'obiettivo di questa procedura è quello di identificare geni che sono differenzialmente espressi in campioni. Dati di microarray possono essere ulteriormente analizzati e visualizzati in un grafico a dispersione per visualizzare geni che sono differenzialmente espressi.
Tabella 1. Componenti da Kit Roche ibridazione NimbleGen da aggiungere per passo ibridazione 2.
| Componente | Volume |
| Campione (sciolto in acqua) | 5 microlitri |
| Ibridazione Buffer 2X | 9 microlitri |
| Ibr A | 3,6 microlitri |
| Allineamento Oligo | 0,4 microlitri |
| Volume totale | 18 microlitri |
Tabella 2. Wash buffer di essere preparati per il lavaggio degli array seguenti ibridazione.
| Componente | Tampone di lavaggio IA * | Tampone di lavaggio IB | Tampone di lavaggio II | Tampone di lavaggio III |
| Acqua | 225 ml | 225 ml | 225 ml | 225 ml |
| Tampone di lavaggio 10X I, II o III | 25 ml | 25 ml | 25 ml | 25 ml |
| 1 M DTT | 25 microlitri | 25 microlitri | 25 microlitri | 25 microlitri |
| Volume totale | 250 ml | 250 ml | 250 ml | 250 ml |
* Pre-riscaldamento a 42 ° C
Discussione
Protocolli di microarray molti, compreso quello dettagliato qui, usare tinture fluorescenti come un mezzo per misurare e quantificare l'ibridazione. Il Cy3 colorante fluorescente è sensibile alle foto e degradazione dell'ozono. Si raccomanda che la procedura sia condotta in illuminazione minima. Inoltre, piccole particelle di polvere possono interferire con l'ibridazione e la scansione. Particolare attenzione dovrebbe essere data per assicurarsi che l'area di lavoro è pulito e privo di polvere.
Materiali
| Name | Company | Catalog Number | Comments |
| Array Microcentrifuge Dryer | ISC Bioexpress | C-1303-T | |
| BioPrime Array CGH Genomic Labeling System | Invitrogen | 18095-011 | |
| Cyanine 3-dCTP | PerkinElmer, Inc. | NEL576 | |
| GenePix 4000B | Molecular Devices | GENEPIX 4000B | |
| Microcon YM-30 | EMD Millipore | 42411 | |
| NimbleGen Array Processing Accessories | Roche Group | 5223539001 | |
| NimbleGen Hybridization Kit | Roche Group | 5223474001 | |
| NimbleGen Hybridization System 4 | Roche Group | 5223652001 | |
| NimbleGen Wash Buffer Kit | Roche Group | 5223504001 | |
| X1 Mixers | Roche Group | 5223725001 |
Riferimenti
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon