Method Article
Perfiles de metiltransferasas y Otros S-Adenosil- L-Homocisteína proteínas que se unen mediante espectrometría de masas de captura compuesto (MCP)
En este artículo
Resumen
Los compuestos de captura se trifuncional pequeñas moléculas para reducir la complejidad del proteoma de funcionamiento reversible de moléculas pequeñas, la interacción de proteínas seguido por foto-reticulación y la purificación. Aquí se utiliza un compuesto de captura con S-Adenosil- L-Homocisteína vinculante como función de selectividad para aislar metiltransferasas de un Escherichia coli toda lisado e identificarlos por los Estados miembros.
Resumen
Hay una variedad de enfoques para reducir la complejidad del proteoma de la base de la pequeña molécula de proteína funcional interacciones, tales como la cromatografía de afinidad o de una actividad de generación de perfiles basados en las proteínas 2. Los compuestos trifuncionales Captura (CC, Figura 1) 3 son la base para un enfoque genérico, en el que el equilibrio inicial impulsada por la interacción entre una sonda pequeña molécula (la función de selectividad, aquí S-adenosil-L-homocisteína, HSA, Figura 1 A) y proteínas de la meta es irreversible fija en la foto-entrecruzamiento entre una función de reacción independiente de foto-activable (en este caso un phenylazide) de la CC y la superficie de las proteínas diana. La función de clasificación (en este caso biotina) sirve para aislar la CC - conjugados de mezclas complejas biológicas con la ayuda de una fase sólida (en este caso cuentas estreptavidina magnética). Dos configuraciones de los experimentos son posibles: "en bolas" 4 o el descrito en la actualidad "en bolas" de configuración (fig. 1B). La función de selectividad puede ser prácticamente cualquier molécula pequeña de interés (sustratos, inhibidores de moléculas de la droga).
S-adenosil-L-metionina (SAM, Figura 1) es, probablemente, el segundo de la ATP, el cofactor más utilizado en la naturaleza 5, 6. Se utiliza como donante metilo de los grupos principales en todos los organismos vivos con la reacción química catalizada por ser SAM-dependiente metiltransferasas (MTases), que metilato ADN 7, 8 ARN, proteínas 9, o pequeñas moléculas 10. Dado el papel crucial de las reacciones de metilación en diversos escenarios fisiológicos (regulación génica, epigenética, el metabolismo), los perfiles de MTases se puede esperar a ser de importancia similar en la proteómica funcional como el perfil de las quinasas. Herramientas analíticas para su perfil, sin embargo, no han estado disponibles. Recientemente hemos introducido un CC con HSA como grupo de selectividad para llenar este vacío tecnológico (Figura 1).
SAH, el producto de la malnutrición aguda después de la transferencia de metilo, es un conocido inhibidor general de los productos MTase 11. Por esta razón y debido a que el cofactor natural de SAM es utilizada por las enzimas más la transferencia de otras partes del cofactor o iniciar reacciones de los radicales, así como debido a su inestabilidad química 12, HSA es una función de selectividad ideal para una CC a MTases objetivo. En este sentido, el informe de la utilidad de la HSA-CC y los MCP por MTases perfiles y otras proteínas de unión a HSA de la DH5a cepa de Escherichia coli (E. coli), una de las mejor caracterizadas de los procariotas, que ha servido como el modelo preferido organismo en un sinnúmero de estudios bioquímicos, biológicos y biotecnológicos. Foto activado reticulación mejora el rendimiento y la sensibilidad del experimento, y la especificidad se puede probar fácilmente en experimentos de competición con un exceso de libertad HSA.
Protocolo
1) Preparación de E. coli lisado celular DH5a
- Inocular un cultivo 2 ml (medio LB en un tubo de ensayo) con el E. coli cepa DH5a directamente de un depósito de glicerina y se incuba a 37 ° C y 250 rpm durante 8 h. Uso de los medios autoclave LB (10 g / l Bacto-triptona, 5 g / L de extracto de levadura, 10 g / L NaCl, pH 7,5).
- Inocular los medios de comunicación LB 250 ml en un matraz de 1 L coctelera con deflectores con la cultura de 2 ml y se incuba durante la noche a 37 ° C y 166 rpm en una incubadora con agitador orbital.
- Inocular cuatro frascos de 5 L agitador, con refuerzos cada uno con 2,5 l con medio LB de la cultura 250 ml (50 ml por cada frasco) y se incuban los cultivos a 37 ° C y 166 rpm hasta un 600 OD de 0,8 que se alcance.
- Recoger las células por centrifugación a 4 ° C g, 3000x durante 20 minutos. Realizar la manipulación posterior a 0-4 ° C o en hielo.
- Vuelva a suspender el material de las células cosechadas en agua Milli-Q, se combinan en un cubo de centrífuga y centrifugar durante 30 minutos a 6000x g y 4 ° C.
- Almacenar el resultado material de la célula 20 g a -20 ° C o -78 º C.
- Vuelva a suspender las células en 100 ml de hielo frío buffer celular apertura (6,7 mM MES, 6,7 mM NaOAc, 6,7 mM de HEPES, 1 mM EDTA, 10 mM β-mercaptoetanol, 200 mM NaCl, pH 7,5, 10% (w / v) glicerol, 0,2 mM PMSF) y ultrasonidos en tres ocasiones durante 1 minuto en cuatro porciones de 25 ml en hielo usando un Sonifier (por ejemplo, Sonopuls HD 2070 de BANDELIN electronic GmbH & Co. KG, amplitud máxima, salida continua).
- Centrifugar el lisado durante la noche a 2370x g y 2 ° C.
- Concentrar el sobrenadante a 14 ml por ultrafiltración (por ejemplo usando el icono de concentradores, 7 mL/9K, de Pierce) a 2370x g y 2 ° C.
- Agotan la viscosos a partir de moléculas pequeñas como el SAM o HSA por filtración en gel a 2 ° C (por ejemplo, Zeba columnas de centrifugado desala, 10 ml, de Pierce; cuatro columnas; buffer de almacenamiento eliminado a 1000x g durante 2 minutos, cuatro veces el equilibrio con 5 ml helada buffer apertura celular y el tampón eliminado en 1000x g durante 2 min, respectivamente, 3,5 ml concentrado aplicado a cada columna; 45 minutos de centrifugación a g 24x, y luego dos veces 2 minutos a 1000x g)
- Suplemento del 13 lisado resultante ml con 13 ml de glicerina de hielo frío y Roche mini-tabletas inhibidor de la proteasa cóctel, EDTA libre. Mezclar y disolver las pastillas.
- Tienda el lisado a -20 ° C
- Determinar la concentración de proteínas totales por el ensayo de Bradford (21 mg / ml en el caso que nos ocupa). Reactivo de Bradford: 100 mg de Coomassie Brilliant Blue G-250 en 50 ml de etanol al 95%, añadir 100 ml de 85% (w / v) de ácido fosfórico, diluir a 1 L cuando el medio se haya disuelto completamente y filtrar a través de papel Whatman # 1 justo antes de su uso.
2) Captura de ensayo (A), Control de la Competencia (C), Jalón (PD), Control de la Competencia de Jalón (CPD), y la prueba de captura combinada de más Pulldown (A + PD)
- Para los experimentos de captura, el caproKit SAH (caprotec bioanalítica GmbH) fue utilizado, que incluye la HSA-CC, sin HSA como competidor, streptavidina revestida de piedras magnéticas con m de diámetro 1 (Dynabeads MyOne estreptavidina C1, Invitrogen Dynal), búfer de captura 5 veces (5X CB, que contiene 100 mM de HEPES, 250 mM de acetato de potasio, 50 mM de acetato de magnesio y un 50% de glicerol) y tampón de lavado 5 veces (5 veces el Banco Mundial, que contiene 250 mM Tris-HCl, pH 7,5, 5 mM EDTA, 5 M NaCl, el 42,5 M octil-β-D-glucopiranósido).
- Para varios experimentos paralelos, se recomienda preparar una mezcla maestra de agua, la captura de búfer y E. coli lisado y para llevar a cabo las reacciones en diferentes tubos de una franja de 200 l tubo-PCR (se recomienda 0,2 mL Thermo-Strip, Thermo Scientific, AB-1114). En este caso, las cantidades para un tubo de reacción se dan. Los resultados de cinco experimentos diferentes se presentan, que son prueba de captura (A), el control de la competencia (C), desplegables (PD), el control de la competencia de telecine (CPD), y la prueba de captura combinada más el menú desplegable (A + PD).
- Para cada reacción, preparar 1,5 ml 1X Banco Mundial mediante la adición de 0,3 ml 5 veces el Banco Mundial a 1,2 ml de agua Milli-Q.
- Prepare SAH-CC cargado streptavidina revestida de piedras magnéticas (caproBeads) en 200 tiras l tubo de PCR. Por lo tanto, mezcle 25 l de 100 mM SAH-CC con 50 l de 10 mg / ml streptavidina revestida de piedras magnéticas para cada alícuota, agite vigorosamente la suspensión resultante a temperatura ambiente durante 2 minutos para permitir la unión de la porción de la biotina de la HSA- CC a la estreptavidina en la superficie de las bolas magnéticas, y recoger las bolas con un imán fuerte (por ejemplo, en las tapas de las tiras de tubo de PCR utilizando el dispositivo caproMagTM magnético, caprotec bioanalítica GmbH). Descartar el sobrenadante, resuspender las caproBeads que resulta en 200 l Banco Mundial, magnéticamente recoger el caproBeads (en las tapas de las tiras de tubos PCR), y desechar el BM supernating. Cerrar los tubos para evitar el secado de los granos.
- Preparar alícuotas de E. coli entero DH5alisado celular en los nuevos tubos de PCR a 0-4 ° C con una mezcla maestra (ver 2.2). Para una reacción, el suplemento de un volumen de agua Milli-Q para un volumen de mezcla para la reacción final de 100 l con 20 l de 5X CB. Mezclar, añadir 0,26 mg E. coli lisado y mezcle suavemente por inversión. Sólo para C y CPD, se añaden 20 l 10 mM solución de competidor HSA y mezcle suavemente por inversión (agregar agua Milli-Q en vez de la solución de HSA de un EP, y un EP +). Sacar una muestra de 1 l de A para su posterior análisis (ver más abajo).
- Suspender la caproBeads en el lisado respectivos y se incuba durante 3 horas a 4 ° C mantener las cuentas en suspensión por la rotación para permitir la unión reversible de las proteínas de unión a HSA la función de selectividad de la SAH SAH-CC.
- Coloque las suspensiones A, C y A + PD en el caproBoxTM (dispositivo para la irradiación de muestras bioquímicas con la luz UV y al mismo tiempo de enfriamiento, caprotec bioanalítica GmbH) e irradiar por un tiempo total de 30 minutos en los tubos cerrados entre 0-4 º C para formar un entrecruzamiento covalente entre la función de reacción de la SAH-CC a las proteínas de unión HSA. Por lo tanto, eliminar las suspensiones después de intervalos de irradiación de 2,5 min, respectivamente, de la caproBoxTM, enfriar en agua con hielo durante unos 15 s (especialmente los párpados), mezclar varias veces por inversión, muy poco tiempo (~ 2 s) centrífuga para eliminar resto de la suspensión en los párpados, y el lugar de nuevo en el caproBoxTM para el intervalo de la radiación que viene.
- Añadir 20 l 10 mM solución de HSA a A, o 20 l de agua Milli-Q de C, PD, CPD, y A + PD y se incuba la suspensión durante 10 min a 4 ° C para desplazar, en A, proteínas de unión HSA no reticulado a la SAH-CC. Mantener las cuentas en suspensión por la rotación o manual intermitente re-suspensión.
- Recoger el caproBeads de las suspensiones, con un fuerte campo magnético (por ejemplo, el caproMagTM), descartar el sobrenadante y lavar los granos en seis ocasiones - por la resuspensión y la recogida - con 200 l Banco Mundial 1X y una vez con 200 l de agua Milli-Q.
- Las cuentas se pueden almacenar en agua Milli-Q durante varias semanas a 4 ° C. Protocolos alternativos existen para su posterior procesamiento de las proteínas de captura y su identificación (véase la discusión).
- Lavar las perlas de tres veces con 200 l de acetonitrilo al 60% (ACN) y liberar las proteínas capturadas de las cuentas por 10 minutos de incubación a temperatura ambiente bajo agitación vigorosa con 200 l 60% ACN/0.2% de ácido trifluoroacético (TFA) (preparación fresca) . El uso de LC-MS de reactivos de grado y el agua.
- Magnéticamente recoger los granos, por separado y se evapora el líquido sobrenadante a sequedad utilizando un evaporador centrífugo (por ejemplo, concentrador de ADN MiVac de Genevac, Inc., Reino Unido). Deseche las cuentas.
3) SDS-PAGE de las proteínas capturadas
- Por SDS-PAGE, se disuelven las proteínas capturadas liberado de la perlas magnéticas (evaporada ACN / TFA soluciones desde el paso 2.12) en 20 l de la muestra buffer SDS (50 mM Tris-HCl, 320 mM β-mercaptoetanol, 2,5% SDS, 0,05% azul de bromofenol , 10% de glicerol, pH 6,8). Mezclar la muestra 1 l procedentes de A (véase el paso 2.5) con 19 l de la muestra buffer SDS, el uso de 5 l de esta solución para el análisis SDS-PAGE (0,25% del ensayo). El calor de las muestras en SDS muestra de amortiguación de 10 min a 95 ° C y dejar que se deja enfriar a temperatura ambiente.
- Analizar por SDS-PAGE (genérico de configuración: MCO ® ProPage 4-20% Tris / glicina prefabricados de geles, gel OLS OmniPage mini sistema de electroforesis SDS funcionamiento de amortiguación: 25 mM Tris base, 200 mM glicina, 0,1% SDS, pH 8,3 , tiempo de ejecución 90 min a una tensión constante de 180 V bajo enfriamiento con hielo del tampón de SDS).
- Tinción de plata del gel usando un método de tinción de MS plata compatible (por ejemplo, ProteoSilver Kit de tinción de plata de Sigma). Un resultado representativo se muestra en la Figura 2.
4) En-gel tríptico resumen de las proteínas y péptidos de extracción de bandas de gel
- Lavar el gel teñido con plata de por lo menos tres veces con 100 ml de agua Milli-Q durante 10 minutos después de dejar la solución de plata mancha se quita.
- Cortar las bandas de gel (por ejemplo, utilizando un bisturí limpio y transferir a un tubo de 0,5 ml Eppendorf) y se almacena a -20 ° C o el proceso directamente. Lave las bandas de gel durante 15 minutos, respectivamente, con 100 l de agua, 100 l de etanol 50%, 100 l de agua, 100 l de etanol al 50%, y durante 5 min con etanol puro. Repita este procedimiento de lavado, una vez más.
- Re-hidratar la banda de gel con 10 l de solución de digestión-gel (12,5 ng / mL de tripsina secuencia de grado en bicarbonato amónico 50 mM, preparan mediante la adición de 7,5 l 0,5 mg / l de solución de tripsina en HCl 1 mM a 292,5 l bicarbonato amónico 50 mM ) por 45 min a 4 ° C. Eliminar el sobrenadante y reemplazar en un 20 l bicarbonato amónico 50 mM (sin tripsina), seguido de incubación a 37 ° C durante la noche mientras se agita.
- Recoger el sobrenadante. Para la extracción del péptido, se incuba la banda de gel con 20 l de ácido fórmico al 5% (FA) durante 15 minutos mientras se agita, añadirACN l 20 y se incubaron durante otros 15 minutos con agitación. Combine el sobrenadante con el sobrenadante anterior y repita el procedimiento de extracción de péptidos, una vez más.
- Se evapora el combinado tres sobrenadantes a sequedad, se disuelve en 10 FA l 5%, mientras temblaba y la aplicación de ultrasonidos (baño de ultrasonidos, por ejemplo, SONOREX de Bandelin, Alemania) y proceder a la desalación (paso 5.2 y posteriores).
5) tríptico resumen de las proteínas capturadas y preparación de péptidos para LC-MS/MS
- Disolver las proteínas capturadas liberado de la perlas magnéticas (evaporada ACN / TFA soluciones desde el paso 2.12) en 10 l bicarbonato amónico 50 mM con un baño de ultrasonidos y agitación, añadir 1 l 0,5 mg / l de tripsina en HCl 1 mM e incubar la solución a 37 ° C durante la noche.
- Desalar la solución que contiene los péptidos trípticos de las proteínas capturadas utilizando material de C18 (por ejemplo 20-10 StageTips l, 20 l punta, Proxeon Biosystems A / S, Odense, Dinamarca, el procedimiento del fabricante: condición previa con 10 l 50% de metanol / 5 FA%, se equilibran con 10 FA l 5%, la carga de péptidos de las proteínas capturadas, lavar con 10 FA l 5%, eluir dos veces con metanol l 10 50% / 5% de FA).
- Se evaporan los péptidos desalada en metanol al 50% / 5% de FA a sequedad, se disuelve en 5,5 l FA 0,1% con agitación y la aplicación de la ecografía (ultrasonido baño) y analizar la muestra por nanoLC-MS/MS.
6) Análisis NanoLC-MS/MS
- Verter la muestra en la placa de la muestra y la placa de lugar en la cromatografía líquida de nano-flow (nanoLC) sistema (por ejemplo, de fácil nLC sistema de cromatografía líquida; Proxeon Biosystems A / S, Dinamarca).
- Use un 0,1% FA en el agua como fase móvil A y 0,1% en FA ACN como fase móvil B. Sólo use disolventes LC-MS de grado.
- Carga de 5 l de la solución de péptidos directamente en una pre-columna (por ejemplo, de la Biosfera nanoflujo C18, 5 m, 120 A, 20 x 0,1 mm; NanoSeparations, Países Bajos), junto a una columna analítica (por ejemplo, de la Biosfera nanoflujo C18, 5 m, 120 A , 100 x 0.075 mm; NanoSeparations, Países Bajos) con FA 5% ACN/0.1%.
- En LC, péptidos durante un gradiente de elución de 80 minutos lineal del 5% ACN/0.1% de FA FA 40% ACN/0.1%, seguido de un adicional de 2 minutos al 100% ACN/0.1% FA y el resto al 100% ACN/0.1% FA para otro 8 minutos con un flujo controlado de 400 nL / min.
- Realizar la espectrometría de masas (MS) el análisis de péptidos eluidos en una gran precisión el estado de la técnica de espectrometría de masas (por ejemplo, LTQ Orbitrap XL espectrómetro de masas, Thermo FisherScientific, Alemania, equipado con una fuente de iones Nanoelectrospray para la ionización por electrospray (ESI); Proxeon Biosystems A / S, Dinamarca).
- Realizar el análisis de espectrometría de masas en el modo dependiente de los datos para cambiar automáticamente entre Orbitrap-MS (modo de perfil) y LTQ-MS/MS (modo de centro de gravedad) de adquisición.
- Controlar el espectrómetro de masas de ciclo de trabajo mediante el establecimiento del control de tiempo de inyección automático de ganancia.
- Adquirir la encuesta de exploración completa de MS espectros (de m / z 300 a 2000) en el Orbitrap con la resolución R = 60000 a m / z 400 (después de la acumulación de un valor objetivo de 500.000 cargos en la trampa de iones lineal).
- Coloque el instrumento para aislar de forma secuencial los iones más intensos (hasta cinco, dependiendo de la intensidad de la señal) de la fragmentación en la trampa de iones lineal utilizando disociación inducida por colisión (CID) en un valor objetivo de 10.000 cargos. Los fragmentos de iones resultantes se registran en el LTQ.
- Para las mediciones de la masa exacta en el modo MS, el uso con una sola carga polydimethylcyclosiloxane fondo de iones (Si (CH 3) 2 O) 6 H + (m / z 445.120025) generados durante el proceso de electrospray de aire ambiente, la masa de bloqueo de tiempo real interno recalibración.
- Dinámica excluir iones de destino ya la masa-seleccionados para CID por la duración de 60 s.
- Set de detección de carga del Estado y el rechazo de los iones de CID con cargo asignado.
- Más ajustes de espectrometría de masas son los siguientes: ajustar el voltaje del spray a 1,6 kV, la temperatura del conjunto de la transferencia capilar calentado a 200 ° C, y la energía de colisión normalizado es de 35% de MS 2. La señal de mínimo requerido para MS 2 es de 500 que cuenta. Aplicar una activación q = 0,25 y un tiempo de activación de 30 ms para MS 2 adquisiciones.
- Para limpiar el sistema LC, realice una prueba en blanco entre dos mediciones consecutivas de CCMS.
7) La identificación de péptidos y proteínas a través de la búsqueda automatizada de bases de datos de secuencia
- Utilizar un algoritmo de identificación de proteínas para analizar los datos MS / MS (en este caso almacena en archivos RAW), por ejemplo, SEQUEST implementado en BioworksBrowser 3.3.1 SP1 (FisherScientific Thermo, Alemania) y X! Tandem (La Organización Mundial de la máquina proteoma; versión 2007.01.01.1) implementado en el Scaffold 3 software (versión Scaffold_3_00_03, Proteoma Software Inc., EE.UU.).
- Realizar base de datos automatizada en contra de la búsqueda más reciente UniProtKB / Swiss-Prot www.expasy.org base de datos de liberación del organismo de investigación (base de datos utilizada para el presente estudio: Escherichia coli, cepa K12, la liberación 57-11).
- Utilice los siguientes valores para la base de datos automatizada de la búsqueda dentro de SEQUEST: 5 ppm tolerancia precursor, un fragmento de la tolerancia uma iones, y la especificidad de la tripsina completa que permite un máximo de dos divisiones perdidas. Permitir que la fosforilación de las modificaciones variable en serina, treonina y la tirosina, la oxidación de metioninas; desamidación en asparagines y la glutamina, la acetilación de la lisina y la serina; formilación en la lisina y la metilación en arginina, lisina, serina, treonina y asparagina. No utilice las modificaciones fijadas en la búsqueda de bases de datos.
- Carga SRF o dta y de los archivos generados por SEQUEST en el andamio 3, que lleva a cabo la evaluación de la probabilidad de asignaciones de péptidos y proteínas mediante la combinación de las identificaciones y SEQUEST X! Tandem búsquedas en las bases. Andamiaje es útil para comparar fácilmente y visualizar listas de proteína a partir de varias muestras (en este caso A, C, PD, CPD, y un EP +).
- Establecer los parámetros dentro de los andamios 3 software que considerar sólo los péptidos con una probabilidad ≥ 95% según lo especificado por el algoritmo Profeta péptido (ref.13). Establecer probabilidades identificación de proteínas para las asignaciones de péptidos múltiples ≥ 95% según el algoritmo Profeta Proteínas 13. Para un solo péptido identificaciones de proteínas, arbitrariamente probabilidad de proteínas a ≥ 50% y inspeccionar manualmente el péptido correspondiente MS / MS espectros. Las proteínas que forman los péptidos similares y no podía ser diferenciado, basado en MS / MS análisis solo se agrupan por el software para satisfacer los principios de parsimonia. La tasa estimada de falso descubrimiento de péptido identificaciones puede ser determinado utilizando el método de base de datos de proteínas invertido y debe ser <1%.
- Los resultados representativos de los experimentos de CCMS, figuran en los cuadros 1, 2, y complementa el cuadro S1 (cuenta que la base de datos de proteínas no está al corriente de algunas proteínas, por ejemplo, PrmB (también conocido como YfcB) o RSMH (también conocido como MraW)), así como en la figura 3.
8) Los resultados representativos
Tabla 1:
| Proteína | ORF | MW / kDa | Descripción | Sustrato | A | C | PD | CPD | Un PD + |
| DCM | b1961 | 53.5 | ADN citosina MTase | ADN (M5C) | 1 | 0 | 0 | 0 | 1 |
| RlmI | b0967 | 44.4 | 23S ARNr m5C1962 MTase | rRNA (M5C) | 17 | 0 | 17 | 0 | 20 |
| RlmL | b0948 | 78.9 | 23S ARNr m2G2445 MTase | rRNA (m2g) | 12 | 0 | 0 | 0 | 10 |
| TRMB | b2960 | 27.3 | tRNA (guanina-N (7) -)-MTase | tRNA (M7G) | 11 | 0 | 0 | 0 | 13 |
| CmoA | b1870 | 27.8 | tRNA (cmo5U34)-MTase | tRNA (mcmo5U) | 7 | 0 | 0 | 0 | 4 |
| RsmG | b3740 | 23.4 | ARNr 16S M7G MTase | rRNA (M7G) | 6 | 0 | 1 | 0 | 5 |
| RSMH | b0082 | 34.9 | ARNr 16S m4C1402 MTase | rRNA (M4C) | 5 | 0 | 0 | 0 | 7 |
| RSMD | b3465 | 21.7 | ARNr 16S m2G966 MTase | rRNA (m2g) | 2 | 0 | 0 | 0 | 2 |
| RSMB | b3289 | 48.3 | ARNr 16S m5C967 MTase | rRNA (M5C) | 1 | 0 | 0 | 0 | 0 |
| MnmC | b2324 | 74.4 | Proteína bifuncional incluye tRNA (MNM (5) s (2) U34)-MTase | tRNA (mnm5s2U) | 1 | 0 | 0 | 0 | 0 |
| PrmB | b2330 | 35.0 | 50S ribosomal proteínas L3 Gln150 MTase | proteína (Gln) | 13 | 0 | 0 | 0 | 15 |
| Cher | b1884 | 32.8 | Quimiotaxis MTase proteínas | proteínas (Glu) | 0 | 0 | 0 | 0 | 1 |
| Comité de Libertad Sindical | b1661 | 44.9 | Ciclopropano-graso-acil-sintetasa fosfolípidos | molécula pequeña | 15 | 0 | 0 | 0 | 14 |
| Tam | b1519 | 29.0 | Trans-2-aconitato MTase | molécula pequeña | 2 | 0 | 0 | 0 | 3 |
| CysG | b3368 | 50.0 | Sintasa Siroheme incluye uroporfirinógeno III-C-MTase | molécula pequeña | 1 | 0 | 0 | 0 | 2 |
| ANTM | b0921 | 29.8 | Proteínas ANTM | (? A) | 7 | 1 | 0 | 0 | 8 |
| MtnN | b0159 | 24.4 | 5'-metiltioadenosina / SAH nucleosidase | b molécula pequeña | 36 | 0 | 0 | 0 | 39 |
| GlnA | b3870 | 51.9 | Glutamina sintetasa | pequeñas moléculas c | 90 | 0 | 0 | 0 | 97 |
| RplK | b3983 | 14.9 | 50S ribosomal proteína L11 | de proteína MTase PRMA d | 2 | 0 | 0 | 0 | 2 |
No es una (total) que se caracteriza
b No metilación, pero la escisión del enlace glucosídico de la HSA
c No metilación pero la unión de la HSA en el sitio de unión a ATP como se muestra por medio de experimentos CCMS con ATP como competidor (datos no mostrados)
d sustrato del 50S ribosomal proteína L11 MTase PRMA, la identificación específica reproducible por el MCP (datos no mostrados)
Tabla 1: MTases y otras proteínas seleccionadas identificadas por los experimentos de CCMS. Los números dados indican el número de péptidos no ponderada del espectro por las proteínas. Las muestras son duplicados de los analizados por SDS-PAGE/silver mancha en la Figura 2. MTases mucho más y otras proteínas de unión a HSA se identifican en el ensayo de los MCPs (A) en comparación con el menú desplegable (PD) y la especificidad de la HSA se muestra por la casi total ausencia de estas proteínas en el control de la competencia (C).
Tabla 2:
| A | C | PD | CPD | Un PD + | |
| A | 111 (64) | ||||
| C | 65 (41) | 107 (46) | |||
| PD | 25 (15) | 23 (13) | 61 (17) | ||
| CPD | 23 (13) | 22 (12) | 20 (14) | 47 (14) | |
| Un PD + | 87 (61) | 64 (41) | 23 (14) | 22 (12) | 124 (67) |
Tabla 2: Número total de las proteínas identificadas en las pistas de SAQ y la superposición de proteína entre las pistas. El número de proteínas identificadas con al menos dos péptidos se indican entre paréntesis. La alta reproducibilidad del método se puede deducir de la coincidencia de alto valor proteico (principalmente de proteínas inespecíficas) entre los experimentos comparables (A vs C y, especialmente, frente a un A + PD sino también PD vs CPD), especialmente con las proteínas fuertemente identificado con por lo menos dos péptidos. Véase también la Figura 3 para los diagramas de Venn y S1 que complementa el cuadro de una lista de todas las proteínas identificadas.
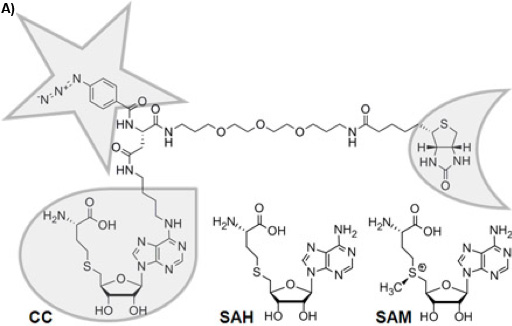
Figura 1 A: Estructura química de los compuestos de captura trifuncional (CC). La función de selectividad se enmarca con una gota, la función de reactividad con una estrella, y la función de clasificación con una media luna. La estabilidad química S-adenosil-L-homocisteína (SAH) es el producto del cofactor S-adenosil-L-metionina (SAM) después de la transferencia del grupo metilo por parte de SAM-dependiente MTases, por lo que la HSA actúa como un inhibidor del producto.
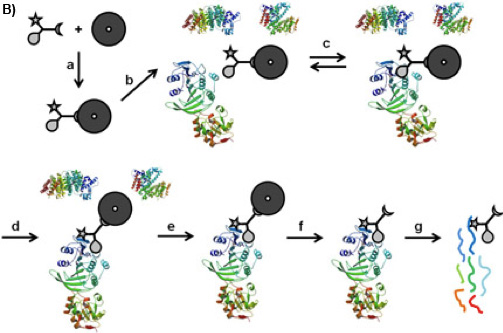
Figura 1B: CCMS, "en-cuentas "de flujo de trabajo. El CC está obligado en los granos magnéticos por su función de clasificación (a), el caproBeads así formado se incubaron con la mezcla compleja de proteínas (b), donde se establece un equilibrio reversible vinculante (c) entre la función de selectividad de la CC y las proteínas diana. tras la irradiación UV (d), la función de reactividad forma covalente reticular. Después de lavar las perlas magnéticas con las proteínas capturadas (e), la escisión de la proteína reticulada CC-complejos de partículas magnéticas (f) y tríptico resumen (g), las proteínas capturadas pueden ser identificados por análisis de MS de los péptidos trípticos.
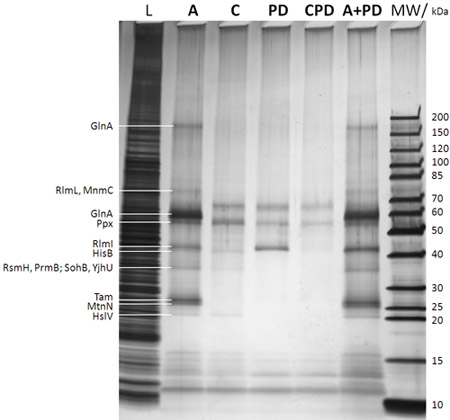
Figura 2: SDS-PAGE/silver análisis de manchas de proteínas capturado (después de la etapa f en la Figura 1B). La descripción del carril se da en la parte superior del gel (MW: marcador de peso molecular con el peso molecular correspondiente de las bandas marcador dado que el propio derecho, L: 0,25% muestra tomada de la coli lisado de E. DH5a célula entera antes de añadir el caproBeads en el paso b en la Figura 1B, A: Ensayo con la adición de un exceso de hemorragia subaracnoidea libre después de la irradiación d UV paso en la Figura 1B, C: control de ensayo incluyendo un exceso de hemorragia subaracnoidea libre competencia en los pasos C y D en la Figura 1B (esencial para determinar las proteínas no específicamente capturado); PD: significado desplegable no d irradiación UV paso en la Figura 1B y sin adición de libre HSA; CPD: el control del menú desplegable con HSA como competidor; PD A +: ensayo combinado desplegable, además significa que no hay otra de HSA libre durante el flujo de trabajo). Las proteínas identificadas por los Estados miembros de la corte después de las bandas de proteínas en gel en gel tríptico resumen se dan a los leftt muy. Es evidente que la foto-reticulación mejora el rendimiento y la sensibilidad del experimento, y la especificidad se puede probar fácilmente en experimentos de competición con un exceso de libertad HSA. Ver Tabla 1 para MTases y otras proteínas seleccionadas identificadas por los experimentos de MCP de los duplicados de las muestras de las que se muestran en la figura de la actualidad.

Figura 3: diagramas de Venn explicativo de la superposición de las proteínas identificadas en el ensayo de los MCPs (A), el control de la competencia (C), y desplegables (PD). Izquierda: Número de MTases y nucleosidase SAH, sólo, en referencia a la Tabla 1. Derecha: Número de todas las proteínas identificadas se refiere a la Tabla 2 y Tabla S1. El número de proteínas identificadas con al menos dos péptidos se indican entre paréntesis.
Discusión
Las siguientes precauciones y comentarios pueden ser útiles cuando se sigue el protocolo descrito: a) Una de las principales ventajas de los países candidatos se encuentra en la formación de un enlace covalente entre la CC y la MTase, ya que esto permite posteriores condiciones de lavado rigurosas. El entrecruzamiento covalente se logra mediante un fotorreacción provocado por la luz UV (310 nm máx.). Luz del techo normal contiene sólo una pequeña fracción de la radiación UV, sin embargo, la protección del SAH-CC de una mayor exposición a la luz de arriba o incluso el sol hasta la irradiación controlada y se enfría en el caproBox. b) Las muestras biológicas de las que el MTases deben ser aisladas pueden contener proteínas propensas a la desnaturalización, por lo tanto es obligatorio para mantener las muestras frescas y evitar la formación de espuma en todo momento. c) El caproBox enfría las muestras a 0-4 º C, las lámparas que emiten la luz UV, sin embargo, también emiten calor. Por lo tanto, es necesario centrifugar brevemente los viales antes de la irradiación, por lo que las proteínas de adhesión a las tapas o paredes del vial no puede formar semillas precipitación. d) Si la resuspensión de las partículas magnéticas (por ejemplo, en las soluciones de lavado), no es posible con la mano, poco se aplican ultrasonidos mediante la colocación de las muestras en un baño de ultrasonidos. e) Recién preparar el 0,2% de solución TFA/60% ACN. Se encontró que de otra manera las proteínas capturados no pueden ser escindidos de las cuentas. f) El análisis final de las proteínas de captura se lleva a cabo por LC-MS/MS. Espectrometría de masas es un método altamente sensible. Es necesario el uso de reactivos exclusivamente LC-MS de grado en los últimos pasos (paso 2.11 y más). Evitar la contaminación de los experimentos de las fuentes de proteínas externas, por ejemplo, la queratina de origen del polvo o del experimentador. Especialmente durante las etapas de la digestión final, se recomienda prestar atención a un espacio de trabajo limpio, usar guantes y una bata de laboratorio y, posiblemente, una redecilla para el pelo o, idealmente, realizar los pasos finales en un banco limpio. Prepare los 50 mM bicarbonato de amonio utilizado para digerir tríptico en LC MS agua grado, el filtro a través de filtro de 0,22 micras, alícuota, almacenar a -20 ° C, y el uso cada alícuota sólo una vez para evitar la contaminación. La solución de tripsina (secuenciación de grado, Roche, preparar un 0,5 mg / l solución mediante la adición de HCl 1 mM de liofilizado tripsina) se puede almacenar a 4 ° C durante varias semanas. g) Para obtener los espectros de masas confiable es esencial contar con un aerosol estable en el análisis de ESI-MS/MS. h) Para los sistemas de LC-MS/MS que no sea la utilizada en el presente estudio, los parámetros de medición y los algoritmos de identificación de péptidos debe ajustarse individualmente.
Las siguientes modificaciones son posibles con respecto al protocolo descrito: a) Como alternativa a la liberación de las proteínas de los granos por TFA 60% ACN/0.2% (paso 2,11), las proteínas pueden ser digeridos directamente tryptically dentro de una suspensión de bolas (mismos volúmenes que en el paso 5.1) o, por SDS-PAGE, las proteínas pueden ser liberados por la suspensión y calefacción las cuentas recogidas después del paso desde 2,9 hasta 95 ° C durante 10 minutos en tampón de muestra SDS (tanto, la suspensión total o sólo el sobrenadante se puede cargar en la bolsa de gel). Se encontró que las cuentas lentamente liberan pequeñas cantidades de polímero en la solución acuosa tríptico resumen en el tríptico resumen de cuentas-incluso después de varias etapas de lavado acuoso. La contaminación interfiere con polímero MS identificación de péptidos y se puede lavar las cuentas usando el 80% ACN (por lo menos tres veces). Después de las etapas de lavado del 80% de ACN, los granos deben ser lavados una vez con agua antes de que el tríptico resumen de perlas. b) Las transferencias Western utilizando estreptavidina-peroxidasa y sustrato horseraddish ECL también se puede utilizar para visualizar la reticulación éxito de la CC contiene biotina a las proteínas. Por lo tanto, ya sea el gel obtenido al paso 3.2 se puede borrar o, debido a que la sensibilidad es de aproximadamente 10 veces mayor que la tinción de plata de los geles, las muestras de 10 l después del paso 2.7 también pueden ser analizados por Western blot. Cuenta que en este último caso las proteínas endógenamente biotina también se detectó, además de las proteínas con biotina artificialmente por la SAH-CC. c) Se encontró que depende del régimen especial (lisado, se dirigió a proteínas diana, la selectividad y la función de la reactividad de que CC), si el "off-talón" de configuración, donde la reacción de reticulación se lleva a cabo entre el CC y libre de proteínas en solución al 4 o el que actualmente se describe "en bolas" de configuración (fig. 1B) tiene un mejor rendimiento.
En general, el método debe ser compatible con cualquier estado de la proteína de isótopos estables de arte o la tecnología de etiquetado de péptido, o la evaluación de la muestra de la captura por electroforesis en gel 2D. En el "off-talón" de configuración, también es posible la captura de las proteínas dentro de células enteras (resultados no publicados). Además, la droga o en el sitio cofactor unión de una proteína puede ser descrito mediante la determinación de la posición de entrecruzamiento de cerca por el de la CC en la secuencia de la proteína por los Estados miembros la secuenciación de péptidos. El modo de unión de una molécula pequeña que una proteína puede ser explorado por el uso de diferentes cposiciones hemical apego a la función de selectividad y las diferentes longitudes de enlace. Como se muestra en el presente estudio, también se asocia la proteína de unión de las proteínas dirigidas por la función de selectividad (RplK como sustrato de PRMA) o desconocidas interacciones pequeña molécula de proteína (HSA a glnA) se pueden identificar. Resumen, la característica adicional de los países candidatos, la reactividad de la foto-reticulación, permite el aislamiento e identificación de proteínas de bajo abundantes o familias funcionales de proteínas a partir de mezclas complejas de proteínas con alta sensibilidad y proporciona a los científicos una herramienta adicional para el estudio de moléculas pequeñas - las interacciones proteína .
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por la organización del programa Human Frontier Science (Premio HFSP 2007, RGP0058/2007-C). Damos las gracias al profesor Richard Roberts para iniciar el proyecto y para un debate fructífero.
Materiales
| Name | Company | Catalog Number | Comments |
| SAH caproKit™ | caprotec bioanalytics GmbH | 1-1010-050 (50 reactions) 1-1010-010 (10 reactions) | Includes the SAH-CC, SAH competitor, streptavidin coated magnetic beads, capture buffer, and wash buffer |
| caproBox™ | caprotec bioanalytics GmbH | 1-5010-003 (110 V) 1-5010-004 (230 V) | For reproducible photo-activation while cooling the samples |
| caproMag™ | caprotec bioanalytics GmbH | 1-5100-001 | For easy handling of magnetic particles without pipetting |
Referencias
- Katayama, H., Oda, Y. Chemical proteomics for drug discovery based on compound-immobilized affinity chromatography. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 855, 21-27 (2007).
- Barglow, K. T., Cravatt, B. F. Activity-based protein profiling for the functional annotation of enzymes. Nat. Methods. 4, 822-827 (2007).
- Koster, H., Little, D. P., Luan, P., Muller, R., Siddiqi, S. M., Marappan, S., Yip, P. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev. Technol. 5, 381-390 (2007).
- Dalhoff, C., Hueben, M., Lenz, T., Poot, P., Nordhoff, E., Koster, H., Weinhold, E. Synthesis of S-Adenosyl-L-homocysteine Capture Compounds for Selective Photoinduced Isolation of Methyltransferases. ChemBioChem. 11, 256-265 (2010).
- Lu, S. C. S-Adenosylmethionine. Int. J. Biochem. Cell. Biol. 32, 391-3952 (2000).
- Cantoni, G. L. Biological methylation: selected aspects. Annu. Rev. Biochem. 44, 435-451 (1975).
- Jeltsch, A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. ChemBioChem. 3, 274-293 (2002).
- Chow, C. S., Lamichhane, T. N., Mahto, S. K. Expanding the nucleotide repertoire of the ribosome with post-transcriptional modifications. ACS Chem. Biol. 2, 610-619 (2007).
- Grillo, M. A., Colombatto, S. S-Adenosylmethionine and protein methylation. Amino Acids. 28, 357-362 (2005).
- Fujioka, M. Mammalian small molecule methyltransferases: their structural and functional features. Int. J. Biochem. 24, 1917-1924 (1992).
- Borchardt, R. T., Salvatore, F., Borek, E., Zappia, V., Williams-Ashman, H. G., Schlenk, F. . The Biochemistry of S-Adenosylmethionine. , 151-171 (1977).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S-adenosyl-l-methionine. Biochemistry. 25, 4444-4449 (1986).
- Keller, A., Nesvizhskii, A. I., Kolker, E., Aebersold, R. Empirical statistical model to estimate the accuracy of peptide identifications made by MS/MS and database search. Anal. Chem. 74, 5383-5392 (2002).
- Nesvizhskii, A. I., Keller, A., Kolker, E., Aebersold, R. A statistical model for identifying proteins by tandem mass spectrometry. Anal. Chem. 75, 4646-4658 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados