Method Article
Profilatura di metiltransferasi e altri S-Adenosil- L-Omocisteina-binding proteine mediante spettrometria di massa Compound Capture (CCMS)
In questo articolo
Riepilogo
Composti cattura sono trifunzionale piccole molecole per ridurre la complessità del proteoma funzionale reversibile da piccola molecola-proteina seguiti da foto-reticolazione e purificazione. Qui si usa un composto di cattura con S-Adenosil- L-Omocisteina vincolante in quanto la funzione di isolare la selettività metiltransferasi da un Escherichia coli intero lisato e identificarli da MS.
Abstract
C'è una varietà di approcci per ridurre la complessità del proteoma sulla base di piccole molecole funzionali-proteina interazioni come cromatografia di affinità 1 o Activity Based Protein Profiling 2. Composti Capture trifunzionale (CC, Figura 1A) 3 sono la base per un approccio generico, in cui l'iniziale equilibrio basato sull'interazione tra una sonda piccola molecola (la funzione di selettività, qui S-adenosil-L-omocisteina, SAH, Figura 1A) e proteine bersaglio è irreversibilmente fissi su foto-reticolazione tra un foto-indipendente attivabile funzione reattività (ecco un phenylazide) del CC e la superficie delle proteine bersaglio. La funzione di ordinamento (qui biotina) serve per isolare il CC - coniugati proteine complesse miscele biologiche con l'aiuto di una fase solida (qui perline streptavidina magnetica). Due configurazioni degli esperimenti sono possibili: "off-perla" 4 o le persone che sono descritti di configurazione "a-perla" (Figura 1B). La funzione di selettività può essere virtualmente qualsiasi piccole molecole di interesse (substrati, inibitori, molecole di farmaco).
S-adenosil-L-metionina (SAM, Figura 1A) è probabilmente, secondo ATP, il cofattore più usato in natura 5, 6. Viene utilizzato come il principale donatore del gruppo metile in tutti gli organismi viventi con la reazione chimica viene catalizzata da SAM-dipendente metiltransferasi (MTases), che il DNA metilato di 7, 8 RNA, proteine 9, o piccole molecole 10. Considerato il ruolo fondamentale delle reazioni di metilazione nelle diverse situazioni fisiologiche (regolazione genica, l'epigenetica, metabolismo), la profilazione dei MTases si può aspettare di diventare di importanza simile in proteomica funzionale come la profilazione delle chinasi. Strumenti analitici per il loro profilo, tuttavia, non sono stati disponibili. Abbiamo recentemente introdotto un CC con emorragia subaracnoidea, come gruppo di selettività di colmare questa lacuna tecnologica (Figura 1A).
SAH, il prodotto di SAM dopo il trasferimento di metile, è un noto inibitore generale del prodotto MTase 11. Per questo motivo e per la naturale cofattore SAM viene utilizzato da enzimi ulteriormente il trasferimento in altre parti del cofattore o innescando reazioni radicali come pure a causa della sua instabilità chimica 12, SAH è una funzione di selettività ideale per un CC a bersaglio MTases. Qui riportiamo l'utilità del SAH-CC e CCMS da MTases profiling e altri SAH-binding proteins dal DH5α ceppo di Escherichia coli (E. coli), uno dei più caratterizzato i procarioti, che ha servito come modello preferito organismo in innumerevoli studi biochimici, biologici e biotecnologici. Fotoattivate reticolazione migliora la resa e la sensibilità dell'esperimento, e la specificità può essere facilmente verificato in esperimenti di competizione con un eccesso di libero SAH.
Protocollo
1) Preparazione di E. coli lisato cellulare DH5α
- Inoculare una cultura 2 ml (media LB in una provetta) con la E. coli DH5α ceppo direttamente da uno stock di glicerolo e incubare a 37 ° C e 250 rpm per 8 ore Utilizzare supporti LB sterilizzati in autoclave (10 g / l Bacto-triptone, 5 g / l estratto di lievito, 10 g / L di NaCl, pH 7,5).
- Inoculare 250 mezzi LB ml in un pallone da 1 L shaker con deflettori con la cultura 2 ml e incubare per una notte a 37 ° C e 166 rpm in un incubatore con shaker orbitale.
- Inoculare quattro 5 L fiaschi shaker con deflettori ogni supporto contenente 2,5 L LB con la cultura da 250 ml (50 ml per ogni cilindro) e incubare le colture a 37 ° C e 166 giri al minuto fino a quando un OD 600 di 0,8 viene raggiunto.
- Raccogliere le cellule per centrifugazione a 4 ° C, 3000x g per 20 minuti. Effettuare ulteriori manipolazioni a 0-4 ° C o in ghiaccio.
- Risospendere il materiale delle cellule raccolte in acqua Milli-Q, si combinano in un secchio da centrifuga e centrifugare per altri 30 minuti a 6000x g e 4 ° C.
- Conservare il materiale risultante 20 g di cellule a -20 ° C o -78 ° C.
- Risospendere le cellule in 100 buffer di ghiaccio apertura fredda cella di ml (6,7 mM MES, NaOAc 6,7 mm, 6,7 HEPES mM, 1 mM EDTA, 10 mM β-mercaptoetanolo, 200 mM NaCl, pH 7,5, 10% (w / v) glicerolo; PMSF 0,2 mm) e sonicare tre volte per 1 minuto in quattro 25 ml su ghiaccio con un sonifier (ad esempio Sonopuls HD 2070 da Bandelin electronic GmbH & Co. KG, ampiezza massima, potenza continua).
- Centrifugare il lisato per tutta la notte a 2370x g e 2 ° C.
- Concentrare il surnatante a 14 mL di ultrafiltrazione (per esempio usando l'icona concentratori, 7 mL/9K, da Pierce) a 2370x g e 2 ° C.
- Riducono il concentrato viscoso da piccole molecole come SAM o emorragia subaracnoidea da gel filtrazione a 2 ° C (es. Zeba Colonne Spin Desalt, 10 ml, da Pierce, quattro colonne; soluzione per la conservazione rimosso a 1000x g per 2 minuti, quattro volte equilibrio con 5 ml ghiacciata del buffer di apertura delle cellule e del buffer rimossi a 1000x g per 2 minuti, rispettivamente, 3,5 ml concentrato applicato a ogni colonna, 45 g di centrifugazione min a 24x, poi due volte 2 min a 1000x g)
- Integrare il conseguente lisato 13 ml con 13 ml di glicerolo ghiacciate e Roche mini tavolette cocktail inibitore della proteasi, EDTA libero. Mescolare e sciogliere le compresse.
- Conservare il lisato a -20 ° C
- Determinare la concentrazione totale di proteine dal saggio Bradford (21 mg / ml nel caso di specie). Reagente di Bradford: 100 mg Coomassie Brilliant Blue G-250 in 50 mL di etanolo al 95%, aggiungere 100 ml di acido fosforico 85% (w / v) e portare a 1 L quando il colorante è completamente sciolta e filtrare su carta Whatman n. 1 solo prima dell'uso.
2) test di cattura (A), il controllo della concorrenza (C), Pulldown (PD), Controllo di concorrenza Pulldown (CPD), e test di cattura combinato più Pulldown (A + PD)
- Per gli esperimenti di cattura, il caproKit SAH (caprotec bioanalytics GmbH) è stato utilizzato, che include il SAH-CC, libero SAH come concorrente, sfere magnetiche streptavidina rivestite con diametro di 1 micron (Dynabeads MyOne Streptavidina C1, Invitrogen Dynal), buffer di cattura 5X (5X CB, contenente 100 mM HEPES, 250 mM acetato di potassio, 50 mM acetato di magnesio e 50% glicerolo), e tampone di lavaggio 5 volte (5X WB, contenente 250 mM Tris HCl, pH 7,5, 5 mM EDTA, 5 M di NaCl, il 42,5 mM ottil-β-D-glucopiranoside).
- Per diversi esperimenti in parallelo, si raccomanda di preparare un master mix di acqua, cattura tampone ed E. coli lisato e per eseguire le reazioni all'interno di tubi di diverso di un 200 ul-PCR bande per tubi (consigliato 0,2 mL Thermo-Strip, Thermo Scientific, AB-1114). Qui, per una quantità di tubo di reazione sono dati. Risultati per cinque diversi esperimenti verranno presentate, che sono test di cattura (A), controllo della concorrenza (C), a tendina (PD), controllo della concorrenza di discesa (CPD), e test di cattura combinato più a tendina (A + PD).
- Per ogni reazione, preparare 1,5 ml 1X WB con l'aggiunta di 0,3 mL WB 5X a 1,2 ml di acqua Milli-Q.
- Preparare SAH-CC caricato sfere magnetiche streptavidina rivestita (caproBeads) in 200 microlitri strisce di provette PCR. Pertanto, mescolare 25 ml di 100 mM SAH-CC con 50 ml di 10 mg / ml sfere magnetiche rivestite di streptavidina per ogni aliquota, scuotere vigorosamente le sospensioni conseguenti a temperatura ambiente per 2 minuti per permettere vincolante della frazione biotina del SAH- CC alla streptavidina sulla superficie magnetica tallone, e raccogliere le perle utilizzando un magnete forte (ad esempio nelle calotte di strisce tubo della PCR utilizzando il dispositivo magnetico caproMagTM, caprotec bioanalytics GmbH). Eliminare il sovranatante, risospendere il caproBeads conseguente 200 l WB, magneticamente raccogliere i caproBeads (nelle calotte di strisce tubo della PCR), ed eliminare il WB supernating. Tubi vicino per evitare l'essiccamento delle perline.
- Preparare aliquote di tutto E.coli DH5αlisato cellulare in nuove provette PCR a 0-4 ° C con una master mix (vedi 2.2). Per una reazione, un supplemento al volume di acqua Milli-Q per un volume finale miscela di reazione di 100 l con 20 l CB 5X. Mescolare, aggiungere 0,26 mg E. coli lisato e mescolare delicatamente per inversione. Solo per C e CPD, aggiungere 20 l 10 mM soluzione concorrente SAH e mescolare delicatamente per inversione (aggiungere Milli-Q acqua invece della soluzione di SAH ad A, PD, e A + PD). Disegna un 1 campione microlitri dalla A per ulteriori analisi (vedi sotto).
- Sospendere il caproBeads nel lisato rispettivi e incubare per 3 ore a 4 ° C mantenendo le perle in sospensione dalla rotazione per consentire legame reversibile delle proteine SAH legame con la funzione di selettività SAH del SAH-CC.
- Posizionare le sospensioni A, C, e A + PD nel caproBoxTM (dispositivo per irradiare campioni biochimici con la luce UV e allo stesso tempo di raffreddamento, caprotec bioanalytics GmbH) e irradiare per un tempo totale di 30 min in tubi chiusi tra 0-4 ° C per formare un crosslink covalenti tra la funzione reattività del SAH-CC per le proteine SAH vincolanti. Quindi, rimuovere le sospensioni dopo intervalli irradiazione di 2,5 min, rispettivamente, dal caproBoxTM, fresco d'acqua ghiacciata per ~ 15 s (in particolare le palpebre), mescolare più volte per inversione, molto poco (~ 2 s) centrifuga per rimuovere sospensione rimanente nei coperchi, e il luogo di nuovo nel caproBoxTM per l'intervallo di irradiazione successiva.
- Aggiungere 20 l soluzione da 10 mM SAH ad A, o 20 l di acqua Milli-Q a C, PD, CPD, e A + PD e incubare la sospensione per 10 minuti a 4 ° C per spostare, in A, proteine SAH vincolanti non reticolato alla SAH-CC. Mantenere le microsfere in sospensione dalla rotazione manuale o intermittente risospensione.
- Raccogliere il caproBeads dal sospensioni utilizzando un magnete forte (ad esempio il caproMagTM), scartare il sovranatante, e lavare le perline sei volte - dalla risospensione e raccolta - con 200 ul 1X WB e una volta con 200 l di acqua Milli-Q.
- Le sfere possono essere memorizzati in acqua Milli-Q per diverse settimane a 4 ° C. Esistono protocolli alternativi per l'ulteriore elaborazione delle proteine catturate e la loro identificazione (vedi discussione).
- Lavare le perle tre volte con 200 microlitri 60% acetonitrile (ACN) e rilasciare le proteine catturate dalle perle di incubazione di 10 minuti a temperatura ambiente sotto vigorosa agitazione con 200 l acido 60% ACN/0.2% trifluoroacetico (TFA) (preparare fresco) . Usa LC-MS-grade reagenti e acqua.
- Magneticamente raccogliere le perline, separare il surnatante ed evaporare sino ad essiccazione, un evaporatore centrifugo (per esempio concentratore DNA MiVac da GeneVac, Inc., UK). Gettare le perle.
3) SDS-PAGE delle proteine Captured
- Per SDS-PAGE, sciogliere le proteine catturate rilasciato dal sfere magnetiche (evaporato ACN / TFA soluzioni dal punto 2.12) in 20 microlitri di buffer campione SDS (50 mM Tris HCl, 320 mM β-mercaptoetanolo, SDS 2,5%, 0,05% blu di bromofenolo , 10% glicerolo, pH 6,8). Mescolare il 1 campione microlitri tratto da A (vedi punto 2.5) con 19 microlitri di buffer campione SDS; usare 5 ml di questa soluzione per l'analisi SDS-PAGE (0,25% del saggio). Riscaldare il campione in campione tampone SDS 10 minuti a 95 ° C e lasciate lasciare raffreddare a temperatura ambiente.
- Analizzare mediante SDS-PAGE (generica configurazione: OLS ® ProPage 4-20% Tris / glicina prefabbricati gel; OLS OmniPage sistema gel elettroforesi mini; tampone SDS esecuzione: 25 mM tris di base, 200 mM glicina, 0,1% SDS, pH 8.3 ; il tempo di esecuzione 90 min a una tensione costante di 180 V con raffreddamento a ghiaccio del tampone di corsa SDS).
- Argento macchia il gel utilizzando un metodo compatibile MS macchia d'argento (per esempio ProteoSilver Kit Argento Stain da Sigma). Un risultato rappresentativo è mostrato nella Figura 2.
4) In-gel Digest Trittico di proteine e peptidi di estrazione Bands Gel
- Lavare il gel d'argento colorato almeno tre volte con 100 ml di acqua Milli-Q per 10 minuti dopo la macchia soluzione bloccante d'argento è stato rimosso.
- Tagliare le bande gel (ad esempio utilizzando un bisturi pulito e il trasferimento in un tubo Eppendorf 0,5 ml) e conservare a -20 ° C o direttamente processo. Lavare le bande gel per 15 minuti, rispettivamente, con 100 microlitri di acqua, 100 microlitri di etanolo al 50%, 100 microlitri di acqua, 100 microlitri di etanolo al 50%, e per 5 minuti con etanolo puro. Ripetere questa procedura di lavaggio, ancora una volta.
- Reidratare la band gel con 10 L in-gel soluzione di digestione (12,5 ng / mL tripsina sequenza di grado in 50 mM di bicarbonato di ammonio, preparare con l'aggiunta di 7,5 microlitri 0,5 mg / soluzione tripsina microlitri si preleva 1 mM HCl a 292,5 microlitri 50 mM di bicarbonato di ammonio ) per 45 minuti a 4 ° C. Rimuovere il surnatante e sostituirlo con 20 l 50 mM di bicarbonato di ammonio (senza tripsina), seguito da incubazione a 37 ° C durante la notte agitando.
- Raccogliere il surnatante. Per l'estrazione del peptide, incubare la band gel con 20 microlitri di acido formico al 5% (FA) per 15 minuti agitando, aggiungere20 ACN microlitri e incubate per altri 15 min agitando. Combina il surnatante con il supernatante precedente e ripetere la procedura di estrazione del peptide ancora una volta.
- Evaporare il combinato tre surnatanti a secco, sciogliere in 10 microlitri FA 5% mentre stringe e l'applicazione di ultrasuoni (bagno a ultrasuoni, ad esempio Sonorex da Bandelin, Germania) e procedere con la dissalazione (punto 5.2 e oltre).
5) Digest Trittico di proteine catturate e preparazione di peptidi per LC-MS/MS
- Sciogliere le proteine catturate rilasciato dal sfere magnetiche (evaporato ACN / TFA soluzioni dal punto 2.12) a 10 L 50 mM di bicarbonato di ammonio utilizzando un bagno ad ultrasuoni e vortex, aggiungere 1 ml 0,5 mg / tripsina microlitri si preleva 1 mM HCl e incubare la soluzione a 37 ° C durante la notte.
- Desalificare la soluzione contenente i peptidi trittico delle proteine catturate con materiale C18 (per esempio 2-10 StageTips microlitri, 20 microlitri punta, Proxeon Biosystems A / S, Odense, Danimarca, procedura del produttore: pre-condizione con 10 L metanolo al 50% / 5 FA%, equilibrato con 10 microlitri FA 5%, carico con peptidi di proteine catturate, lavare con 10 microlitri FA 5%, due volte eluire con 10 microlitri metanolo 50% / 5% FA).
- Evaporare i peptidi dissalate in metanolo al 50% / 5% FA a secco, sciogliere in 5,5 microlitri FA 0,1% mentre stringe e l'applicazione di ultrasuoni (ecografia bagno) e analizzare il campione nanoLC-MS/MS.
6) Analisi NanoLC-MS/MS
- Versare il campione in piastra campione e conservare nel nano-flusso cromatografia liquida (nanoLC) sistema (ad esempio facile-nLC sistema di cromatografia liquida; Proxeon Biosystems A / S, Danimarca).
- Usa 0,1% FA in acqua come fase mobile A e 0,1% in FA ACN come fase mobile B. Utilizzare solo LC-MS solventi grado.
- Di carico 5 microlitri della soluzione del peptide direttamente su una pre-colonna (Biosfera nanoflussi ad esempio C18, 5 micron, 120 A, 20 x 0,1 mm; NanoSeparations, Paesi Bassi) accoppiato ad una colonna analitica (Biosfera nanoflussi ad esempio C18, 5 micron, 120 Å , 100 x 0,075 millimetri; NanoSeparations, Paesi Bassi) con FA 5% ACN/0.1%.
- Durante LC, peptidi eluire nel corso di un gradiente di 80 min lineare ACN/0.1 dal 5% al 40% FA FA% ACN/0.1% seguito da un ulteriore 2 min a 100% ACN/0.1% FA e rimanere al 100% ACN/0.1% FA per un altro min 8 con un flusso controllato di 400 nL / min.
- Eseguire spettrometria di massa (MS) analisi dei peptidi eluiti su un elevata precisione di dichiarare-of-the-art spettrometro di massa (ad esempio LTQ Orbitrap XL spettrometro di massa; Thermo FisherScientific, Germania, equipaggiati con una sorgente di ioni per nanoelectrospray ionizzazione elettrospray (ESI); Proxeon Biosystems A / S, Danimarca).
- Eseguire l'analisi di spettrometria di massa nei dati-dipendente modo per passare automaticamente da Orbitrap-MS (modalità profilo) e LTQ-MS/MS (modalità centroide) di acquisizione.
- Controllare il ciclo di spettrometro di massa dovere impostando il tempo di iniezione di controllo automatico del guadagno.
- Acquisire sondaggio scansione completa MS spettri (da m / z 300-2000) nel Orbitrap con risoluzione r = 60.000 a m / z 400 (dopo l'accumulo di un valore obiettivo di 500.000 oneri del trappola ionica lineare).
- Impostare lo strumento per isolare in modo sequenziale gli ioni più intensi (fino a cinque, a seconda dell'intensità del segnale) per la frammentazione in trappola ionica lineare utilizzando collisione indotta dissociazione (CID) ad un valore target di 10.000 cariche. Gli ioni frammento risultanti sono registrati nel LTQ.
- Per misure accurate di massa in modalità MS, utilizzare il carica singolarmente polydimethylcyclosiloxane sfondo ioni (Si (CH 3) 2 O) 6 H + (m / z 445,120025) generati durante il processo electrospray da aria ambiente come la massa per bloccare in tempo reale interna ricalibrazione.
- Dinamicamente escludere ioni di massa-bersaglio già selezionati per CID, per la durata di 60 s.
- Impostare uno screening dello stato di carica e il rifiuto di ioni per la CID a pagamento non assegnato.
- Ulteriormente le impostazioni di spettrometria di massa sono i seguenti: impostare tensione a spruzzo a 1,6 kV, temperatura impostata del trasferimento capillare riscaldato a 200 ° C, e l'energia di collisione normalizzato è del 35% per MS 2. Il segnale minimo richiesto per MS 2 è di 500 conteggi. Applicare una attivazione q = 0,25 e un tempo di attivazione di 30 ms per MS 2 acquisizioni.
- Per pulire il sistema LC, effettuare una corsa vuota tra due misurazioni consecutive CCMS.
7) Identificazione di Peptidi e Proteine tramite ricerca automatica Sequence Database
- Utilizza un algoritmo di identificazione delle proteine per analizzare la MS / MS dati (nel caso di specie memorizzati nei file raw), SEQUEST esempio implementato in BioworksBrowser 3.3.1 SP1 (FisherScientific Thermo, Germania) e X! Tandem (L'Organizzazione mondiale della macchina Proteome; versione 2007.01.01.1) implementato nella Scaffold 3 software (versione Scaffold_3_00_03, Proteome Software Inc., USA).
- Eseguire banca dati automatizzata ricerca contro le più recenti UniProtKB / Svizzera-Prot www.expasy.org rilascio del database dell'organismo indagato (database utilizzato per questo studio: Escherichia coli, ceppo K12, rilasciare 57-11).
- Utilizzare le seguenti impostazioni per banca dati automatizzata ricerca all'interno SEQUEST: 5 tolleranza precursore ppm, 1 tolleranza frammento amu di ioni, e la specificità tripsina completa che permette fino a due spaccature perdere. Consentire come variabile fosforilazione modifiche a serina, treonina e tirosina, ossidazione del metionine; deamidation a asparagina e glutammina; acetilazione in lisina e serina; formylation a lisina; e la metilazione in arginina, lisina, serina, treonina e asparagina. Non usare le modifiche fisso nella ricerca database.
- Carico SRF o dta ed i file generati dal SEQUEST nel impalcatura 3, che effettua la valutazione probabilità di incarichi di peptidi e proteine identificazioni combinando SEQUEST e X! ricerche nelle banche dati Tandem. Ponteggio è utile per confrontare facilmente e la visualizzazione di liste di proteine da campioni diversi (nel caso di specie A, C, PD, CPD, e A + PD).
- Impostare i parametri all'interno del software 3 impalcatura di prendere in considerazione solo peptidi con probabilità ≥ 95% come specificato dall'algoritmo Profeta Peptide (ref.13). Impostare probabilità di identificazione di proteine per le assegnazioni peptide multipli per ≥ 95% secondo l'algoritmo Profeta Protein 13. Per le identificazioni singola proteina peptidi, proteine probabilità arbitrariamente fissato a ≥ 50% e ispezionare manualmente il corrispondente peptide spettri MS / MS. Le proteine che compongono peptidi simili e non poteva essere differenziato sulla base di analisi MS / MS da soli sono raggruppati in base al software di soddisfare i principi della parsimonia. Il tasso stimato scoperta falsa identificazioni peptide può essere determinato utilizzando il invertito approccio database di proteine e dovrebbero essere <1%.
- Risultati rappresentativi di esperimenti CCMS sono riportati nelle tabelle 1, 2, e supplementare S1 tabella (la mente che il database di proteine non è up-to-date per alcune proteine, ad esempio PrmB (alias YfcB) o RsmH (aka MraW)), come pure come in Figura 3.
8) Risultati Rappresentante
Tabella 1:
| Proteina | ORF | MW / kDa | Descrizione | Substrato | A | C | PD | CPD | A + PD |
| DCM | b1961 | 53,5 | DNA-citosina MTase | DNA (M5C) | 1 | 0 | 0 | 0 | 1 |
| RlmI | b0967 | 44,4 | RRNA 23S m5C1962 MTase | rRNA (M5C) | 17 | 0 | 17 | 0 | 20 |
| RlmL | b0948 | 78,9 | RRNA 23S m2G2445 MTase | rRNA (m2G) | 12 | 0 | 0 | 0 | 10 |
| TRMB | b2960 | 27,3 | tRNA (guanina-N (7) -)-MTase | tRNA (m7G) | 11 | 0 | 0 | 0 | 13 |
| CmoA | b1870 | 27,8 | tRNA (cmo5U34)-MTase | tRNA (mcmo5U) | 7 | 0 | 0 | 0 | 4 |
| RsmG | b3740 | 23,4 | RRNA 16S m7G MTase | rRNA (m7G) | 6 | 0 | 1 | 0 | 5 |
| RsmH | b0082 | 34,9 | RRNA 16S m4C1402 MTase | rRNA (M4C) | 5 | 0 | 0 | 0 | 7 |
| RsmD | b3465 | 21,7 | RRNA 16S m2G966 MTase | rRNA (m2G) | 2 | 0 | 0 | 0 | 2 |
| RsmB | b3289 | 48,3 | RRNA 16S m5C967 MTase | rRNA (M5C) | 1 | 0 | 0 | 0 | 0 |
| MnmC | b2324 | 74,4 | Proteina bifunzionale comprende tRNA (MNM (5) s (2) U34)-MTase | tRNA (mnm5s2U) | 1 | 0 | 0 | 0 | 0 |
| PrmB | b2330 | 35,0 | 50S proteina ribosomiale L3 Gln150 MTase | proteine (Gln) | 13 | 0 | 0 | 0 | 15 |
| Cher | b1884 | 32,8 | Chemiotassi proteina MTase | proteine (Glu) | 0 | 0 | 0 | 0 | 1 |
| Cfa | b1661 | 44,9 | Ciclopropano-grassi-acil-fosfolipidi sintasi | piccola molecola | 15 | 0 | 0 | 0 | 14 |
| Tam | b1519 | 29,0 | Trans-2-aconitate MTase | piccola molecola | 2 | 0 | 0 | 0 | 3 |
| CysG | b3368 | 50,0 | Sintasi Siroheme include uroporfirinogeno-III C-MTase | piccola molecola | 1 | 0 | 0 | 0 | 2 |
| SmtA | b0921 | 29,8 | Proteine smtA | (? A) | 7 | 1 | 0 | 0 | 8 |
| MtnN | b0159 | 24,4 | 5'-Methylthioadenosine / SAH nucleosidase | b piccola molecola | 36 | 0 | 0 | 0 | 39 |
| GlnA | b3870 | 51,9 | Glutammina sintetasi | piccola molecola c | 90 | 0 | 0 | 0 | 97 |
| RplK | b3983 | 14,9 | 50S proteina ribosomiale L11 | di proteine MTase PRMA d | 2 | 0 | 0 | 0 | 2 |
Non uno (completamente) caratterizzato
b Non metilazione ma scissione del legame glicosidico di SAH
c Nessuna metilazione ma vincolante del SAH nel sito legame dell'ATP come dimostrato da esperimenti CCMS con ATP come concorrente (dati non riportati)
d substrato della proteina ribosomiale 50S L11 MTase PRMA; riproducibile identificazione specifica CCMS (dati non riportati)
Tabella 1: MTases e di altre proteine selezionati identificati da esperimenti CCMS. I numeri dati indicano il numero di peptide pesato spettrali per proteica. I campioni sono duplicati di quelli analizzati da SDS-PAGE/silver macchia nella Figura 2. MTases molto di più e altre proteine SAH vincolanti sono identificate nel test CCMS (A) rispetto alla discesa (PD) e specificità SAH è dimostrato dalla quasi totale assenza di queste proteine nel controllo della concorrenza (C).
Tabella 2:
| A | C | PD | CPD | A + PD | |
| A | 111 (64) | ||||
| C | 65 (41) | 107 (46) | |||
| PD | 25 (15) | 23 (13) | 61 (17) | ||
| CPD | 23 (13) | 22 (12) | 20 (14) | 47 (14) | |
| A + PD | 87 (61) | 64 (41) | 23 (14) | 22 (12) | 124 (67) |
Tabella 2: Numero totale di proteine identificate nelle corse CCMS e la sovrapposizione di proteine tra le piste. Il numero di proteine identificate con almeno 2 peptidi sono dati in parentesi. L'alta riproducibilità del metodo può essere dedotta dalla sovrapposizione di proteine (soprattutto di proteine aspecifici) tra esperimenti simili (A vs C e, soprattutto A vs A + PD, ma anche PD vs CPD), soprattutto con le proteine identificate con robusta almeno 2 peptidi. Si veda anche Figura 3 per i diagrammi di Venn e S1 tabella supplementare per un elenco di tutte le proteine identificate.
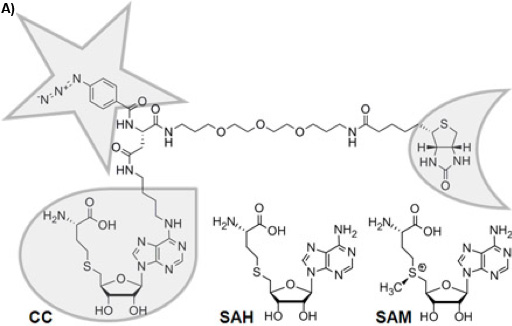
Figura 1A: struttura chimica del composto Capture trifunzionale (CC). La funzione di selettività è incorniciata con una goccia, la funzione di reattività con una stella, e la funzione di ordinamento con una mezza luna. Il chimicamente stabile S-adenosil-L-omocisteina (SAH) è il prodotto cofattore di S-adenosil-L-metionina (SAM) dopo il trasferimento del gruppo metilico da SAM-dipendente MTases, per i quali SAH agisce come inibitore del prodotto.
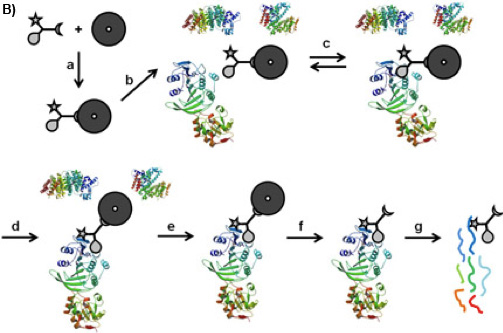
Figura 1B: CCMS "on-perlina "flusso di lavoro. Il CC è legato al sfere magnetiche per la sua funzione di ordinamento (a), il caproBeads così formato vengono incubati con la miscela proteica complessa (b), in cui è stabilito un equilibrio reversibile vincolante (c) tra la funzione di selettività il CC e le proteine bersaglio. Upon irradiazione UV (d), la funzione di reattività forma un crosslink covalenti. Dopo aver lavato le sfere magnetiche recanti le proteine catturate (e), scissione del reticolato CC-proteina complessi dal sfere magnetiche (f) , e triptici digerire (g), le proteine catturate possono essere identificati mediante l'analisi MS dei peptidi trittico.
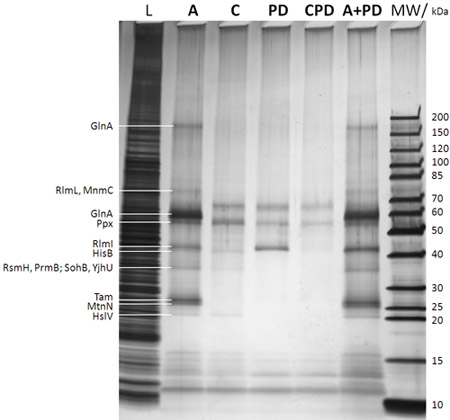
Figura 2: analisi SDS-PAGE/silver macchia di proteine catturate (f dopo passo nella Figura 1B). La descrizione corsia viene dato sulla parte superiore del gel (MW: marcatore di peso molecolare con i pesi molecolari corrispondenti delle bande marker dato al diritto stesso; L: 0,25% del campione tratto dal lisato di cellule E. coli DH5a tutto prima di aggiungere il caproBeads in b passo nella Figura 1B; A: test con l'aggiunta di un eccesso di SAH libero dopo d irradiazione UV passo in Figura 1B; C: il controllo di test tra cui un eccesso di SAH libero come concorrente durante le fasi C e D in Figura 1B (essenziale stabilire gli eventuali non specificamente proteine catturate); PD: significato a tendina non UV d passo irradiazione in Figura 1B e nessuna aggiunta di libero SAH; CPD: il controllo della discesa con SAH come concorrente; A + PD: test combinato più tendina significato non oltre di SAH gratuito durante il flusso di lavoro). Proteine identificate da MS da bande proteiche cut-out dopo il gel in gel trittico digerire sono dati ai più leftt. E 'evidente che la foto-reticolazione migliora la resa e la sensibilità dell'esperimento, e la specificità può essere facilmente verificato in esperimenti di competizione con un eccesso di libero SAH. Vedi Tabella 1 per MTases e di altre proteine selezionati identificati da esperimenti CCMS di campioni duplicato di quelli mostrati nella figura presente.

Figura 3: diagrammi di Venn explaning la sovrapposizione delle proteine identificate nel test del CCMS (A), controllo della concorrenza (C), ed a tendina (PD). A sinistra: Numero di MTases e nucleosidase SAH, solo, riferendosi alla Tabella 1. A destra: Numero di tutte le proteine identificate riferimento alla Tabella 2 e Tabella S1 supplementare. Il numero di proteine identificate con almeno 2 peptidi sono dati in parentesi.
Discussione
Le seguenti precauzioni e osservazioni possono essere utili quando si segue il protocollo descritto: a) Uno dei principali vantaggi di CC sta nella formazione di un legame covalente tra la CC e la MTase, in quanto ciò permette di successive condizioni di lavaggio stringenti. Il covalente reticolazione è ottenuta mediante un fotoreazione innescata dalla luce UV (310 nm max.). Plafoniera normale contiene solo una piccola frazione di raggi UV, invece, proteggere la SAH-CC da lunga esposizione alla luce anche in testa o sole fino al irraggiamento controllato e raffreddato in caproBox. b) I campioni biologici da cui il MTases devono essere isolati possono contenere proteine incline alla denaturazione, di conseguenza è obbligatorio mantenere i campioni freschi ed evitare la formazione di schiuma in ogni momento. c) Il caproBox raffredda i campioni a 0-4 ° C, le lampade che emettono la luce UV, tuttavia, anche emettono calore. Pertanto è necessario centrifugare brevemente le provette prima di irradiazione, così le proteine che aderiscono al tappi o pareti flaconcino non possono formare semi di precipitazione. d) Se risospensione delle sfere magnetiche (ad esempio nelle soluzioni di lavaggio) non è possibile a mano, a breve si applicano gli ultrasuoni, ponendo i campioni in un bagno a ultrasuoni. e) fresco preparare la soluzione allo 0,2% ACN TFA/60%. Abbiamo scoperto che in caso contrario le proteine catturate non può essere aperto dal perline. f) L'analisi finale delle proteine catturate è svolta da LC-MS/MS. Spettrometria di massa è un metodo altamente sensibile. E 'necessario utilizzare i reagenti esclusivamente LC-MS grado nelle fasi finali (punto 2.11 e oltre). Evitare la contaminazione degli esperimenti da fonti di proteine esterne, ad esempio, cheratina proveniente dalla polvere o dal sperimentatore. Soprattutto durante le fasi di digestione finale, si raccomanda di prestare attenzione a uno spazio di lavoro pulito, di indossare guanti ed un camice da laboratorio e, eventualmente, una rete di capelli o idealmente eseguire le fasi finali sotto una panchina pulita. Preparare il tampone di ammonio 50 mM di bicarbonato usato per digerire il trittico in acqua LC MS grado, filtrare attraverso filtro 0,22 micron, aliquota, conservare a -20 ° C, e usare ogni aliquota una sola volta per evitare contaminazioni. La soluzione di tripsina (sequenziamento grado, Roche, preparare un 0,5 ug / ul di soluzione con l'aggiunta di 1 mM HCl al liofilizzato tripsina) può essere conservato a 4 ° C per diverse settimane. g) Per ottenere spettri di massa affidabile è essenziale per avere un getto stabile in analisi ESI-MS/MS. h) Per gli altri sistemi LC-MS/MS rispetto a quello utilizzato nel presente studio, i parametri di misura e gli algoritmi di identificazione del peptide deve essere adattato individualmente.
Le seguenti modifiche sono possibili riguardo al protocollo descritto: a) In alternativa al rilascio delle proteine da perle di TFA 60% ACN/0.2% (punto 2.11), le proteine possono essere direttamente tryptically digerito all'interno di una sospensione tallone (stessi volumi al punto 5.1) o, per SDS-PAGE, le proteine possono essere rilasciati sospendendo il riscaldamento e le perle raccolte dopo passo 2,9-95 ° C per 10 min in tampone campione SDS (entrambi, la sospensione o solo il supernatante può essere caricato nella tasca di gel). Abbiamo scoperto che le perle rilasciare lentamente piccole quantità di polimeri in soluzione acquosa trittico digerire durante il trittico-tallone digerire anche dopo molti lavaggi acquosi. La contaminazione polimero interferisce con l'identificazione MS peptide e può essere lavato via le perline con ACN 80% (almeno tre volte). Dopo i passi ACN 80% di lavaggio, le perline devono essere lavato con acqua prima sul tallone digerire trittico. b) Western blot utilizzando streptavidina-perossidasi horseraddish e substrato ECL può essere utilizzato anche per visualizzare reticolazione successo del CC che contiene biotina alle proteine. Allora, o il gel ottenuto dopo la fase 3.2 possono essere cancellati o, perché la sensibilità è circa 10 volte superiore a quella colorazione argento di gel, 10 campioni di 2,7 microlitri dopo passo può essere analizzata anche da western blot. Ricordate che in quest'ultimo caso le proteine endogeneously biotinilato sarà anche rilevato oltre le proteine artificialmente biotinilato dal SAH-CC. c) Abbiamo scoperto che dipende dal particolare sistema (lisato, indirizzata proteine bersaglio, la selettività e la funzione della reattività di lui CC), se la configurazione "off-perla", dove la reazione di reticolazione avviene tra CC liberi e proteine in soluzione 4 o le persone che sono descritti di configurazione "a-perla" (Figura 1B) funziona meglio.
In generale, il metodo deve essere compatibile con qualsiasi state-of-the proteina isotopo stabile l'arte o la tecnologia di etichettatura peptide, o la valutazione del campione di cattura da parte elettroforesi su gel 2D. Nella configurazione "off-perla", è anche possibile catturare le proteine all'interno delle cellule intere (risultati non pubblicati). Inoltre, il farmaco o sito cofattore legame di una proteina può essere delineato determinando il primo da posizione reticolazione del CC all'interno della sequenza della proteina mediante sequenziamento MS peptide. La modalità di legame di una piccola molecola ad una proteina può essere esplorata utilizzando diversi cposizioni di fissaggio chimici ed alla funzione di selettività e di diverse lunghezze linker. Come mostrato nel presente studio, anche partner legame proteico delle proteine indirizzata dalla funzione di selettività (RplK come substrato di PRMA) o sconosciuto piccola proteina molecola (SAH a GlnA) può essere identificato. Sintesi, la caratteristica aggiuntiva del CC, la foto-reticolazione reattività, consente l'isolamento e l'identificazione di proteine a basso abbondanti o famiglie di proteine funzionali da miscele complesse di proteine ad alta sensibilità e fornisce agli scienziati un ulteriore strumento per lo studio di piccole molecole - interazioni proteina .
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto dal umane Frontier Science Program Organization (HFSP Award 2007, RGP0058/2007-C). Ringraziamo il Prof. Richard Roberts per l'avvio del progetto e per le discussioni fruttuose.
Materiali
| Name | Company | Catalog Number | Comments |
| SAH caproKit™ | caprotec bioanalytics GmbH | 1-1010-050 (50 reactions) 1-1010-010 (10 reactions) | Includes the SAH-CC, SAH competitor, streptavidin coated magnetic beads, capture buffer, and wash buffer |
| caproBox™ | caprotec bioanalytics GmbH | 1-5010-003 (110 V) 1-5010-004 (230 V) | For reproducible photo-activation while cooling the samples |
| caproMag™ | caprotec bioanalytics GmbH | 1-5100-001 | For easy handling of magnetic particles without pipetting |
Riferimenti
- Katayama, H., Oda, Y. Chemical proteomics for drug discovery based on compound-immobilized affinity chromatography. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 855, 21-27 (2007).
- Barglow, K. T., Cravatt, B. F. Activity-based protein profiling for the functional annotation of enzymes. Nat. Methods. 4, 822-827 (2007).
- Koster, H., Little, D. P., Luan, P., Muller, R., Siddiqi, S. M., Marappan, S., Yip, P. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev. Technol. 5, 381-390 (2007).
- Dalhoff, C., Hueben, M., Lenz, T., Poot, P., Nordhoff, E., Koster, H., Weinhold, E. Synthesis of S-Adenosyl-L-homocysteine Capture Compounds for Selective Photoinduced Isolation of Methyltransferases. ChemBioChem. 11, 256-265 (2010).
- Lu, S. C. S-Adenosylmethionine. Int. J. Biochem. Cell. Biol. 32, 391-3952 (2000).
- Cantoni, G. L. Biological methylation: selected aspects. Annu. Rev. Biochem. 44, 435-451 (1975).
- Jeltsch, A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. ChemBioChem. 3, 274-293 (2002).
- Chow, C. S., Lamichhane, T. N., Mahto, S. K. Expanding the nucleotide repertoire of the ribosome with post-transcriptional modifications. ACS Chem. Biol. 2, 610-619 (2007).
- Grillo, M. A., Colombatto, S. S-Adenosylmethionine and protein methylation. Amino Acids. 28, 357-362 (2005).
- Fujioka, M. Mammalian small molecule methyltransferases: their structural and functional features. Int. J. Biochem. 24, 1917-1924 (1992).
- Borchardt, R. T., Salvatore, F., Borek, E., Zappia, V., Williams-Ashman, H. G., Schlenk, F. . The Biochemistry of S-Adenosylmethionine. , 151-171 (1977).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S-adenosyl-l-methionine. Biochemistry. 25, 4444-4449 (1986).
- Keller, A., Nesvizhskii, A. I., Kolker, E., Aebersold, R. Empirical statistical model to estimate the accuracy of peptide identifications made by MS/MS and database search. Anal. Chem. 74, 5383-5392 (2002).
- Nesvizhskii, A. I., Keller, A., Kolker, E., Aebersold, R. A statistical model for identifying proteins by tandem mass spectrometry. Anal. Chem. 75, 4646-4658 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon