Method Article
Methyltransferases의 프로 파일링 및 기타 S - adenosyl - L - 호모 시스테인 결합 단백질
요약
캡처 화합물 사진 crosslinking 및 정화하여 다음 기능을 가역 작은 분자 - 단백질 상호 작용에 의한 프로테옴의 복잡도를 줄이기 위해 trifunctional 작은 분자 수 있습니다. 여기있는 캡처 컴파 운드를 사용하여 S - adenosyl - L - 호모 시스테인 바인딩 대장균 전체 세포는 lysate와 MS에 의해 그들을 식별합니다.
초록
이러한 친화도 크로마 토그래피 1 또는 활동 기반 단백질 프로파일이 같은 기능을 작은 분자 - 단백질 상호 작용에 기초하여 프로테옴의 복잡도를 줄이기 위해 접근의 다양한 방법이 있습니다. Trifunctional 캡처 화합물 (CCS, 그림 1A) 3은 일반적인 접근 방식을 바탕으로하는 최초의 평형 중심의 작은 분자 프로브 (여기 선택성 기능, S - adenosyl - L - 호모 시스테인, SAH, 그림 1A) 사이의 상호 작용 및 대상 단백질은 irreversibly CC의 독립적인 사진 activable 반응 기능 (여기 phenylazide)와 대상 단백질의 표면 사이에 사진 crosslinking에 고정됩니다. 고체 상 (여기서 streptavidin 자성 구슬)의 도움으로 복잡한 생물학 혼합물에서 단백질 conjugates - 정렬 기능 (여기 비오틴)는 CC를 분리하는 역할을합니다. 실험의 두 가지 구성이 가능합니다 : "오프 구슬"4 또는 현재 설명 "에 - 구슬 '구성 (그림 1B). 택 기능은 관심의 거의 모든 작은 분자 (기판, 억제제, 약물 분자) 수 있습니다.
(SAM, 그림 1A) S - Adenosyl - L - 메티오닌은 아마도 ATP, 자연 5, 6에 가장 널리 사용 cofactor의 두번째입니다. 이것은 화학 반응은 DNA 7, RNA 8, 단백질 9, 또는 작은 분자 10 methylate SAM - 종속 methyltransferases (MTases)에 의해 촉매되는 모든 살아있는 유기체의 주요 메틸 그룹 기증자로 사용됩니다. 다양한 생리 시나리오에서 methylation 반응의 중요한 역할 (유전자 조절, epigenetics, 신진 대사)을 감안할 때, MTases의 프로파일 링은 kinases의 프로 파일링 기능으로 proteomics의 유사한 중요성 될 것으로 기대하실 수 있습니다. 자신의 프로파일에 대한 분석 도구는 그러나, 사용할 수 없습니다. 우리는 최근이 기술 격차 (그림 1A)을 채우기 위해 선택성 그룹으로 SAH과 CC를 소개했다.
SAH, 메틸이 완료된 후 SAM의 제품은 알려진 일반 MTase 제품 억제제 11. 이런 이유 때문에 자연 cofactor SAM은 그 화학적 불안정성 12 때문에 SAH가 MTases을 타겟으로 CC를위한 이상적인 선택도 기능입니다 cofactor의 다른 부분을 전송하거나 극단적인 반응뿐만 아니라를 시작 더 효소에 의해 사용됩니다. 여기, 우리는 기본 모델로 하였다 대장균 (E. 대장균), 가장 특징 prokaryotes 중 하나의 변형 DH5α에서 프로 파일링 MTases 및 기타 SAH 결합 단백질에 의해 SAH - CC 및 CCMS의 유틸리티를보고 수많은, 생화학 생물 학적 및 biotechnological 연구 유기체. 사진 활성 crosslinking는 실험의 수율과 감도를 강화하고, 특이성이 쉽게 무료로 SAH을 초과 사용하여 경쟁 실험에 대해 테스트할 수 있습니다.
프로토콜
E. 1) 준비 대장균 DH5α 세포 Lysate
- E.와 2mL 문화 (LB 미디어 테스트 튜브)를 예방 대장균 37 글리세롤 주식과 부화에서 직접 DH5α 변형 ° C, 8 H. 250 RPM autoclaved LB 미디어 (10g / L Bacto - tryptone, 5 G / L 효모 추출물, 10g / L NaCl, 산도 7.5)를 사용합니다.
- 37 박 이상 2 ML 문화와 부화와 baffles와 1 L 쉐이크 플라스크 250 ML의 LB 미디어를 예방 ° C와 궤도 쉐이크로 인큐베이터에서 166 RPM.
- baffles없이 네 다섯 L 쉐이크 flasks 250 ML 문화 (각 플라스크 50 ML)과 ° C와 0.8의 OD 600까지 166 RPM에 도달 37 문화를 부화와 2.5 L의 LB 미디어를 포함하는 각.의 예방
- 20 분 4 ° C, 3000x g에서 원심 분리하여 세포를 수확. 0-4 ° C 또는 얼음에 대한 자세한 처리를 수행합니다.
- 밀리 Q 물의 수확 세포 물질이 다시 중단, 추가로 30 6000x g에서 분 및 4 ° C. 한 원심 분리기 버켓과 원심 분리기에 결합
- -20 ° C 또는 -78 ° C.에 결과 20g 전지 재료를 저장
- 100 ML 아이스 차가운 셀 개방 버퍼 (6.7 MM MES, 6.7 MM NaOAc, 6.7 MM HEPES, 1 MM EDTA (에틸렌 다이아 민 테트라 초산), 10 MM의 β - 메르 캅 토 에탄올, 200 MM NaCl, pH를 7.5, 10 % (W / V)에 세포를 다시 정지 글리세롤, 0.2 MM PMSF)와 sonifier (BANDELIN 전자 GmbH를 & (주) KG, 최대한의 진폭, 지속적인 출력에서 예 SONOPULS HD 2070)을 사용하여 얼음 세 개 25 ML 부분 1 분 세 번 sonicate.
- 2370x g 2 ° C. 밤 동안 lysate를 원심 분리기
- 2370x g 2 ° C.에 ultrafiltration (예 : 피어스의 아이콘 Concentrators, 7 mL/9K를 사용)에 의해 14 ML에 뜨는을 집중
- 2 겔 여과하여 SAM 또는 SAH 같은 작은 분자의 점성 집중을 고갈 ° C (피어스의 예 Zeba 탈염하다 스핀 열, 10 ML,, 4 열, 5 ML과 네번의 평균, 저장 버퍼 2 분 1000x g에서 제거 얼음 차가운 세포 여는 버퍼와 버퍼는 각각 2 분 1000x g에서 삭제, 3.5 ML 각 컬럼에 적용되는 집중, 24x의 g에서 45 분 원심 분리, 1000x g에서 다음 두 번 2 분)
- 13 ML 얼음 차가운 글리세롤과 로체 미니 테아제 억제제의 칵테일 알약, EDTA (에틸렌 다이아 민 테트라 초산) 무료로 결과 13 ML의 lysate을 보완. 믹스 및 정제를 해산.
- -20 ° C에서 lysate를 저장
- 브래드 포드 분석 (21 현재 경우에 MG / ML)로 총 단백질 농도를 결정합니다. 브래드 포드 시약 : 100 MG 쿠매시 브릴리언트 블루 ML 95 % 에탄올 50 G - 250 왓먼 # 1 종이를 통해 100 ML 85% (W / V) 인산, 염료가 완전히 해소했다 1 L로 희석하고, 필터를 추가 단지 사용하기 전에.
2) 캡처 분석 (A), 경쟁 제어 (C), 풀다운 (PD), 풀다운 (CPD)의 경쟁 제어 및 결합 캡처 분석 풀다운 플러스 (A + PD)
- 캡처 실험 들어, SAH caproKit (caprotec bioanalytics GmbH의)은 SAH - CC, 경쟁자로 무료 SAH, 1 μm의 직경 (Dynabeads MyOne Streptavidin C1, Invitrogen Dynal), 배 캡처 버퍼와 streptavidin - 코팅 자기 구슬을 포함하는, 사용했습니다 (5 배 CB, 100 MM의 HEPES, 250 MM 칼륨 아세테이트, 50 MM 마그네슘 아세테이트 및 50 % 글리세롤을 포함하는), 그리고 배 세척 버퍼 (5 배 WB, 250 MM 트리스 HCL, pH를 7.5, 5 MM EDTA (에틸렌 다이아 민 테트라 초산), 5 M NaCl, 42.5를 포함하는 μm의 octyl - β - D - glucopyranoside).
- 그것이 물의 마스터 믹스를 준비하는 것이 좋습니다 여러 병렬 실험, 버퍼와 E.을 캡처 콜리 lysate와 200 μL - PCR 튜브 스트립의 다른 튜브 내에서 반응을 수행하기 위해 (권장 0.2 ML 온습 스트립, 써모 사이 언티픽, AB - 1114). 여기 하나의 반응 관에 대한 수량이 주어집니다. 다섯 가지 실험에 대한 결과는 캡쳐 분석 (A), 경쟁 제어 (C), 풀다운 (PD), 풀다운 (CPD)의 경쟁 제어 및 결합 캡처 분석 플러스 풀다운 (A + PD)하는, 표시됩니다.
- 각 반응 들어, 밀리 Q 물 1.2 ML 0.3 ML 배 WB를 추가하여 1.5 ML 1X WB를 준비합니다.
- 200 μL PCR 튜브에 부착된에 SAH - CC 로드된 streptavidin - 코팅 자석 구슬 (caproBeads)를 준비합니다. 따라서, 각 나누어지는 10 MG / ML streptavidin - 코팅 자석 구슬 50 μL 100 μm의 SAH - CC 중 25 μL를 혼합, 적극적으로의 비오틴 잔기의 바인딩을 허용 2 분 상온에서 결과 suspensions를 흔들 SAH - 자성 비드 표면에 streptavidin, 그리고 (caproMagTM 자기 장치 caprotec bioanalytics GmbH를 사용하여 PCR 튜브에 부착된의 대문자로 등) 강력한 자석을 사용하여 비즈를 수집 CC. supernatants를 폐기, 200 μL WB의 결과 caproBeads을 다시 중단 자기 (磁 气) caproBeads를 (PCR 튜브에 부착된의 대문자로) 수집 및 supernating WB 폐기하십시오. 닫기 튜브는 구슬의 건조되지 않도록합니다.
- E.coli DH5α의 전체의 aliquots 준비0-4에서 새로운 PCR 튜브에 세포 lysate ° C 마스터 믹스를 사용하여 (2.2 참조). 한 반응의 경우, 20 μL 배 CB 100 μL의 최종 반응 믹스 볼륨에 밀리 Q 물의 볼륨을 보충. 믹스, 0.26 MG E.을 추가 콜리 lysate, 그리고 부드럽게 역전으로 섞는다. 오직 C와 CPD를 위해, 20 μL 10 MM SAH의 경쟁 솔루션을 추가하고 부드럽게 (A, PD, 그리고 A + PD에게 밀리 Q 물 대신 SAH 솔루션 추가) 역전하여 섞는다. 자세한 분석 (아래 참조)에서 1 μL 샘플을 그립니다.
- 4 3 H에 대한 각 lysate와 부화에 caproBeads을 중지 ° C SAH - CC의 SAH의 선택도 함수에 SAH 바인딩 단백질의 가역 바인딩을 허용하는 회전에 의해 정지 구슬을 유지.
- suspensions에게 A, C 및 A + PD caproBoxTM에서 (자외선과 동시에 냉각 caprotec bioanalytics GmbH를 함께 생화 학적 샘플을 irradiating에 대한 장치)와 0-4 사이의 닫힌 튜브 30 분 총 시간을 비추다를 배치 ° C SAH 바인딩 단백질 SAH - CC의 반응 함수 사이에 공유 결합 crosslink를 형성합니다. 따라서, (~ 2 S) 매우 빠른 시일 내에, 나머지 정지를 제거하는 원심 분리기를 ~ 15 S (특히 뚜껑)에 얼음물에서 caproBoxTM에서 각각, 2.5 분 방사선 간격 후에 시원한를 suspensions를 제거 역전하여 여러 번 믹스 뚜껑, 그리고 다음 방사선 간격 다시 caproBoxTM로 두십시오.
- C, PD, CPD, 그리고 A + PD로, 20 μL 밀리 Q 물 20 μL 10 MM SAH 솔루션을 추가하고 4 10 분에 대한 중지를 품어 ° C가, A에서 가교하지 SAH 바인딩 단백질을 치환하는 SAH - CC 있습니다. 회전 또는 간헐 수동 다시 정지로 정지 비즈 보관하십시오.
- 강력한 자석을 (caproMagTM 예 :)를 사용하여 suspensions에서 caproBeads를 수집, supernatants를 삭제하고, 구슬 6 번이나 워시 - 다시 서스펜션과 컬렉션 - 200 μL 1X WB 한번 200 μL 밀리 Q 물과 함께.
- 구슬 4 ° C.에 몇 주 동안 밀리 Q 물에 저장할 수 있습니다 대체 프로토콜 (토론 참조) 캡처 단백질과 신분증의 추가 처리를 위해 존재합니다.
- 200 μL 60 % acetonitrile (ACN)과 비즈 세 번 씻으 200 μL 60% ACN/0.2 % trifluoroacetic 산성 (TFA)와 흔들림 활발한 이하 실온에서 10 분 배양하여 비즈에서 캡처한 단백질을 릴리스 (갓 준비) . LC - MS - 등급 시약 및 물을 사용하십시오.
- 자기 (磁 气), 비즈를 수집 분리하여 원심 증발기를 (GeneVac, 주식 회사, 영국의 예 MiVac의 DNA 농축기)를 사용하여 건조에 뜨는을 증발. 비즈 폐기하십시오.
캡처된 단백질의 3) SDS - PAGE
- SDS - PAGE의 경우, 20 μL SDS 샘플 버퍼 (50 MM 트리스 HCL, 320 MM β - 메르 캅 토 에탄올, 2.5 % SDS, 0.05 % bromophenol 파란색으로 자기 비즈 (단계 2.12에서 ACN / TFA 솔루션을 증발)에서 발표 캡처한 단백질 분해 10 % 글리세롤, 산도 6.8). (단계 2.5 참조) 19 μL SDS 샘플 버퍼에서 그려진 1 μL 시료를 혼합, (분석의 0.25 %) SDS - PAGE 분석을 위해이 솔루션의 5 μL를 사용합니다. 95 ° C로 SDS 샘플 버퍼 10 분에 샘플을 가열하고 실온으로 냉각하도록하자.
- 25 MM 트리스베이스, 200 MM 글리신, 0.1 % SDS, pH를 8.3;, OLS omniPAGE 미니 겔 전기 영동 시스템 SDS 실행 버퍼 OLS ® ProPage 4퍼센트부터 20퍼센트까지 트리스 / 글리신 사전 주조 젤 : SDS - PAGE (일반 설치 프로그램에 의해 분석 ;) SDS의 실행 버퍼의 얼음 냉각 아래에 180 V의 정전압에서 시간 90 분을 실행합니다.
- 실버 MS 호환 실버 얼룩 방법 (시그마에서 예 ProteoSilver 실버 스테인 키트)를 사용하여 젤 얼룩. 대표 결과는 그림 2에 표시됩니다.
젤 밴드에서 단백질 및 펩타이드 추출 4) 인 겔 Tryptic 다이제스트
- 실버 얼룩 중지 솔루션 제거 후 10 분, 100 ML 밀리 Q 물과 은색의 스테인드 젤 적어도 세 번 씻으십시오.
- 겔 밴드 (예 : 0.5 ML Eppendorf 튜브에 깨끗한 메스 및 전송을 사용하여)와 -20 ° C에서 저장하거나 직접 프로세스를 잘라 버릴거야. 100 μL 물 100 μL 50 % 에탄올 100 μL 물 100 μL 50 % 에탄올과 순수 에탄올 5 분으로 각각 15 분 겔 밴드 씻으십시오. 다시 한번이 세척 절차를 반복합니다.
- 소화 솔루션 - 겔 10 μL로 다시 수화물 겔 밴드 (12.5은 μL / NG 50 MM의 탄산수 소 암모늄에서 등급 트립신을 시퀀싱, 292.5 μL 50 MM의 탄산수 소 암모늄에 1 ㎜ HCL의 7.5 μL 0.5 μg / μL 트립신 솔루션을 추가하여 준비 ) 4 ° C. 45 분 뜨는을 제거하고 흔들림 동안 ° C 이상의 밤에 37 인큐베이션 다음 20 μL 50 MM의 탄산수 소 암모늄 (트립신 제외)으로 대체합니다.
- 뜨는를 수집합니다. 떨고있는 동안 펩타이드 추출의 경우, 추가, 15 분 20 μL 5 % 개미의 산성 (FA)로 겔 밴드를 품어20 μL ACN과 흔들림 동안에 다른 15 분 incubated. 이전 뜨는로 뜨는을 결합하고 다시 한번 펩타이드 추출 절차를 반복합니다.
- 초음파를 떨고 적용하는 동안 10 μL 5 % FA에 용해 (초음파 목욕 Bandelin에서 예 Sonorex, 독일)과 (5.2 단계 및 추가) 탈염 진행, 건조에 결합하여 세 supernatants을 증발.
5) Tryptic 캡처된 단백질의 다이제스트 및 LC-MS/MS에 대한 펩티드의 작성
- 초음파 욕조, vortexing을 사용하여 10 μL 50 MM의 탄산수 소 암모늄에서 자기 비즈 (단계 2.12에서 증발 ACN / TFA 솔루션)에서 발표 캡처한 단백질을 분해, 1 ㎜ HCL 1 μL 0.5 μg / μL 트립신을 추가하고에서 솔루션을 품어 37 ° C 밤이여.
- 10 μL 50 % 메탄올 / 5 사전 조건 : C18 소재를 사용하여 캡처한 단백질의 tryptic 펩티드 (예 2-10 μL StageTips, 20 μL 팁, Proxeon Biosystems A / S, 오덴세, 덴마크, 제조 업체의 절차를 포함하고있는 솔루션을 탈염하다 %의 FA는 캡처 단백질의 펩티드와 10 μL 5 % FA로 부하 평형 10 μL 5 % FA로 세척, 10 μL의 50 % 메탄올 / 5 % FA)로 두 번 elute.
- 50 % 메탄올 / 건조 5 %의 FA에 desalted 펩티드를 증발, 초음파 (초음파 목욕)을 떨고 적용하는 동안 5.5 μL 0.1 % FA의 용해와 nanoLC-MS/MS하여 샘플을 분석할 수 있습니다.
6) NanoLC-MS/MS 분석
- 나노 흐름 액체 크로마 토그래피 (nanoLC) 시스템 (; Proxeon Biosystems A / S, 덴마크 예 쉬운 NLC 액체 크로마 토그래피 시스템)에 샘플 판과 장소 플레이트에 샘플을 전송합니다.
- 모바일 단계로 물에 0.1 %의 FA를 사용하여 모바일 단계 B. 전용 LC - MS 등급 용제를 사용하는 등 ACN에서 0.1 %의 FA.
- 직접 사전 컬럼에 펩타이드 솔루션 5 μL를 부하 (C18 예 nanoflow의 생물권 보전, 5 μm의 120 X 0.1 mm Å, 20, NanoSeparations, 네덜란드)가 분석 열 (C18 예 nanoflow의 생물권 보전, 5 μm의 120 Å에 결합 100 X 0.075 mm, NanoSeparations, 네덜란드) 5% ACN/0.1 %의 FA를 사용합니다.
- 5% ACN/0.1 %의 FA에서 백퍼센트 ACN/0.1 %에서 추가로 2 분 100 % ACN/0.1 %의 FA와 나머지 다음 40% ACN/0.1 %의 FA로 80 분 선형 그라데이션 동안 LC, elute 펩티드 동안 400 NL / 분 제어 유량 다른 8 분을위한 FA.
- 써모 FisherScientific, 독일, 전기 분무 이온화 (ESI)에 대한 nanoelectrospray 이온 소스를 갖춘,, Proxeon 고정밀 첨단 질량 분석기 (예 : LTQ Orbitrap XL 질량 분석계에 eluted 펩티드의 대량 spectrometric (MS) 분석을 수행 Biosystems A / S, 덴마크).
- 자동 orbitrap - MS (프로파일 모드)와 LTQ-MS/MS (중심 모드) 인수 사이를 전환할 수있는 데이터 종속 모드에서 대량 spectrometric 분석을 수행합니다.
- 분사 시간 자동 이득 제어를 설정하여 질량 분석계의 듀티 사이클을 제어합니다.
- 설문 조사를 취득 풀 스캔 MS 스펙트럼 (M / Z에서 300 2000) M / Z 400 해상도 R = 60,000 (선형 이온 트랩에서 500000 청구의 대상 값을 누적 이후)로 orbitrap 인치
- 순차적으로 10000 요금의 목표 가치 충돌 유발 분해를 (CID)를 사용하여 선형 이온 트랩에 조각에 대한 가장 강렬한 이온 (5까지, 신호 강도에 따라 다름) 분리 수있는 계기를 설정합니다. 그 결과 조각 이온은 LTQ에 기록됩니다.
- MS 모드에서 정확한 질량 측정, 내부 실시간에 대한 잠금 대량으로 주변 공기의 전기 분무 과정에서 발생하는 단독 청구 polydimethylcyclosiloxane 배경 이온 (SI (CH 3) 2 O) 6 H +를 (M / Z 445.120025)를 사용 recalibration.
- 동적으로 이미 대상 이온을 제외 60 기간 S. 대한 CID에 대한 대량 선택
- 충전 상태 검사 및 할당되지 않은 요금과 CID에 대한 이온의 거부를 설정합니다.
- 또한 대량 spectrometric 설정은 다음과 같습니다. 1.6 KV, 200 모세관 가열 이전의 설정 온도 ° C 및 표준 충돌 에너지는 MS 2 35%입니다 스프레이 전압을 설정 MS 2 필요한 최소한의 신호 500 중요합니다. 활성화 Q = 0.25 및 MS이 인수 30 MS의 활성화 시간을 적용합니다.
- LC 시스템을 청소하려면, 연속 2 CCMS 측정 사이의 한 빈 실행을 수행합니다.
자동 시퀀스 데이터베이스 검색을 통해 7) 펩타이드 및 단백질 식별
- 버전, MS / MS 데이터 (RAW 파일에 저장되어있는 현재의 경우), BioworksBrowser 3.3.1 SP1 (FisherScientific 써모, 독일)와 엑스 탄뎀 (글로벌 프로테옴 기계기구에서 구현 예 SEQUEST을 분석하는 단백질 식별 알고리즘을 사용하여 SC에서 2007.01.01.1)을 구현affold 3 소프트웨어 (버전 Scaffold_3_00_03, 프로테옴 소프트웨어 주식 회사, 미국).
- (대장균, 스트레인 K12, 릴리스 57-11 현재 연구에 사용되는 데이터베이스) 조사 유기체의 가장 최근 UniProtKB / 스위스 보호해주는 데이터베이스 릴리스에 대한 검색 www.expasy.org 자동으로 데이터베이스를 수행합니다.
- 5 PPM 전구체 허용, 1 AMU의 조각 이온 허용하고, 최대 두 놓친 cleavages 수 있도록 허용하는 전체 트립신의 특이성 : SEQUEST 내에서 검색 자동 데이터베이스에 대한 다음과 같은 설정을 사용합니다. methionines의 산화,, asparagines과 글루타민에 deamidation, 라이신과 세린의 아세틸화, 라이신에 formylation하며, 아르기닌, 라이신, 세린, 트레오닌에서 methylation, 그리고 아스파라긴 변수 수정의 세린, 트레오닌, 그리고 티로신에서 인산화으로 허용합니다. 데이터베이스 검색에서 해결된 수정을 사용하지 마십시오.
- SRF 또는 DTA 및 SEQUEST 및 엑스 탄뎀 데이터베이스 검색을 결합하여 펩티드 과제 및 단백질 식별의 확률 평가를 수행 스캐폴드 3으로 SEQUEST에 의해 생성된 밖으로 파일을로드합니다. 발판은 쉽게 몇 가지 샘플에서 단백질 목록을 비교하고 시각화하는 데 유용합니다 (현재 경우 A, C, PD, CPD, 그리고 A + PD).
- 로 펩타이드 예언자 알고리즘 (ref.13)에 의해 지정 ≥ 95 % 확률만을 펩티드를 고려하는 스캐폴드 3 소프트웨어 내에서 매개 변수를 설정합니다. 단백질 예언자 알고리즘 13에 따라 ≥ 95 %까지 여러 펩타이드 배정을위한 단백질 식별 확률을 설정합니다. 단일 펩티드 단백질 식별을 위해, 임의의 ≥ 50 %를 단백질 확률을 설정하고 수동으로 해당 펩타이드 MS / MS 스펙트럼을 검사. 유사한 펩티드를 구성하고 혼자 MS / MS 분석을 바탕으로 차별화된 수 없습니다 단백질은 간결의 원칙을 충족하는 소프트웨어에 의해 그룹화됩니다. 펩티드 식별의 예상 거짓 발견 율은 반대로 단백질 데이터베이스 접근법을 사용하고 <1 %되어야 확인할 수 있습니다.
- CCMS 실험의 대표적인 결과는 표 1, 2에 주어진, 그리고 보충 테이블 S1 (단백질 데이터베이스가 어떤 단백질에 대한 최신 없다는 마음 예 PrmB (일명 YfcB) 또는 RsmH (일명 MraW))뿐만 아니라, 그림 3에서와 같이.
8) 대표 결과
표 1 :
| 단백질 | ORF | MW는 / KDA | 설명 | 기판 | C | PD | CPD | + PD | |
| DCM | b1961 | 53.5 | DNA - 사이 토신 MTase | DNA (m5C) | 1 | 0 | 0 | 0 | 1 |
| RlmI | b0967 | 44.4 | 23S rRNA m5C1962 MTase | rRNA (m5C) | 17 | 0 | 17 | 0 | 20 |
| RlmL | b0948 | 78.9 | 23S rRNA m2G2445 MTase | rRNA (m2G) | 12 | 0 | 0 | 0 | 10 |
| TrmB | b2960 | 27.3 | tRNA (구아닌 - N (7) -) - MTase | tRNA (677) | 11 | 0 | 0 | 0 | 13 |
| CmoA | b1870 | 27.8 | tRNA (cmo5U34) MTase | tRNA (mcmo5U) | 7 | 0 | 0 | 0 | 4 |
| RsmG | b3740 | 23.4 | 16 rRNA 677 MTase | rRNA (677) | 6 | 0 | 1 | 0 | 5 |
| RsmH | b0082 | 34.9 | 16 rRNA m4C1402 MTase | rRNA (m4C) | 5 | 0 | 0 | 0 | 7 |
| RsmD | b3465 | 21.7 | 16 rRNA m2G966 MTase | rRNA (m2G) | 2 | 0 | 0 | 0 | 2 |
| RsmB | b3289 | 48.3 | 16 rRNA m5C967 MTase | rRNA (m5C) | 1 | 0 | 0 | 0 | 0 |
| MnmC | b2324 | 74.4 | Bifunctional 단백질 tRNA (mnm (5) S (2) U34) MTase을 포함 | tRNA (mnm5s2U) | 1 | 0 | 0 | 0 | 0 |
| PrmB | b2330 | 35.0 | 50 ribosomal 단백질 L3 Gln150 MTase | 단백질 (Gln) | 13 | 0 | 0 | 0 | 15 |
| 쉐어 | b1884 | 32.8 | Chemotaxis 단백질 MTase | 단백질 (GLU) | 0 | 0 | 0 | 0 | 1 |
| CFA | b1661 | 44.9 | Cyclopropane - 지방산 - 아실 - 인지질의 synthase | 작은 분자 | 15 | 0 | 0 | 0 | 14 |
| 탐 | b1519 | 29.0 | 트랜스 - aconitate 2 MTase | 작은 분자 | 2 | 0 | 0 | 0 | 3 |
| CysG | b3368 | 50.0 | Siroheme의 synthase는 uroporphyrinogen - III C - MTase을 포함 | 작은 분자 | 1 | 0 | 0 | 0 | 2 |
| SmtA | b0921 | 29.8 | 단백질 smtA | (?) | 7 | 1 | 0 | 0 | 8 |
| MtnN | b0159 | 24.4 | 5' - Methylthioadenosine / SAH nucleosidase | 작은 분자 B | 36 | 0 | 0 | 0 | 39 |
| GlnA | b3870 | 51.9 | 글루타민 synthetase | 작은 분자 C | 90 | 0 | 0 | 0 | 97 |
| RplK | b3983 | 14.9 | 50 ribosomal 단백질 L11 | 단백질 MTase PrmA D의 | 2 | 0 | 0 | 0 | 2 |
(완전히) 특징되지 않음
B SAH의 glycosidic 결합의 대한 methylation 아니지만 절단
C 대한 methylation 아니지만 경쟁자로 ATP와 CCMS 실험 (데이터가 표시되지 않음)에 의해 그림과 같이 ATP 바인딩 사이트에 SAH의 결합
50 ribosomal 단백질 L11 MTase PrmA의 D 기판, CCMS (데이터가 표시되지 않음)으로 재현할 특정 식별
표 1 : MTases 및 CCMS 실험에 의해 확인된 다른 선택 단백질. 주어진 숫자는 단백질 당 unweighted 펩타이드 스펙트럼 계산을 나타냅니다. 샘플 SDS-PAGE/silver에 의해 분석 이들의 중복 그림 2에 얼룩이 있습니다. 더 많은 MTases 및 기타 SAH 바인딩 단백질은 풀다운 (PD)와 SAH의 특이성이 경쟁 컨트롤에서이 단백질의 거의 완전한 부재 (C)로 표시됩니다에 비해 CCMS 분석 (A)로 식별됩니다.
표 2 :
| C | PD | CPD | + PD | ||
| 111 (64) | |||||
| C | 65 (41) | 107 (46) | |||
| PD | 25 (15) | 23 (13) | 61 (17) | ||
| CPD | 23 (13) | 22 (12) | 20 (14) | 47 (14) | |
| + PD | 87 (61) | 64 (41) | 23 (14) | 22 (12) | 124 (67) |
표 2 : 실행 사이의 CCMS 실행 및 단백질 중복에서 확인된 단백질의 총 수입니다. 최소한 2 펩티드와 단백질의 식별 번호는 괄호에 부여됩니다. 방법의 높은 재현성을 비교 실험 (A 대 C와 특히 대 + PD뿐만 아니라 PD 대 CPD) 특히 견고하게 식별 단백질과 함께 사이에 (주로 unspecific 단백질)이 높은 단백질 중복에서 유추 수 있습니다 최소한 2 펩티드. 또한 모든 확인된 단백질의 목록에 대한 벤 다이어그램과 보조 테이블 S1 3 그림을 참조하십시오.
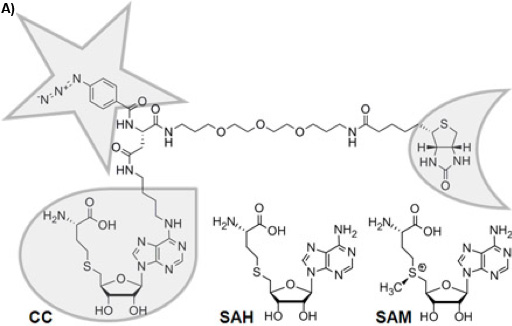
그림 1A : trifunctional 캡처 컴파 운드 (CC)의 화학 구조. 선택도 함수는 비말, 스타와 반응 기능 및 반달과 정렬 기능 프레임입니다. 화학적으로 안정적인 S - adenosyl - L - 호모 시스테인 (SAH)는 SAH가 제품 억제제 역할을하는 SAM - 의존 MTases에 의해 메틸 그룹이 완료된 후 S - adenosyl - L - 메티오닌의 cofactor 제품 (SAM)입니다.
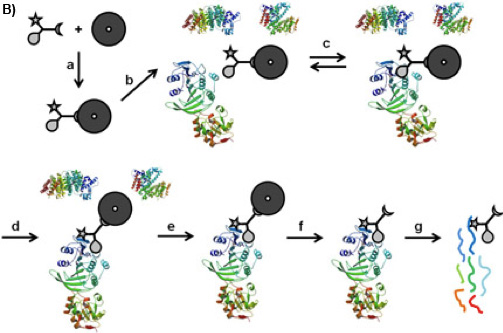
그림 1B : CCMS "에 -비드 "워크플로우. CC는 자사의 정렬 기능 (A)에 의해 자성 비즈의 바인딩은, 그렇게 형성된 caproBeads은 가역 바인딩 평형 (C가)의 선택도 기능 사이의 설립의 복잡한 단백질 혼합물 (B)와 incubated 아르 CC와 대상 단백질. UV 조사시 (D), 반응 함수는 공유 결합 crosslink를 형성합니다. 캡처한 단백질 (E), 자석 구슬의 가교 CC - 단백질 단지의 절단 (F)을 베어링 자성 비즈를 세척 후 그리고 tryptic 다이제스트 (G)는 캡처한 단백질은 tryptic 펩티드의 MS 분석에 의해 확인할 수 있습니다.
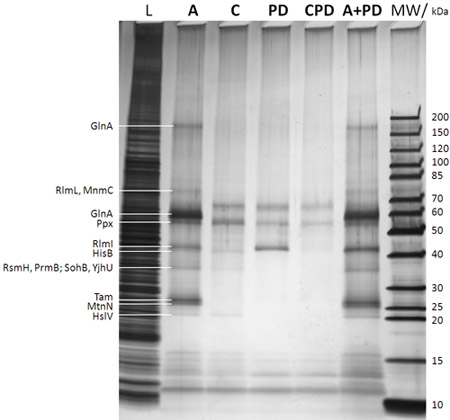
그림 2 : SDS-PAGE/silver 캡처 단백질 얼룩 분석 (그림 1B의 단계 F 이후). 0.25 % 샘플 caproBeads를 추가하기 전에 E. 대장균 DH5a 전체 세포 lysate에서 그려; L 매우 오른쪽으로 지정된 마커 밴드의 해당 분자 무게와 분자량 마커 : 레인 설명은 젤 맨 (MW에 주어집니다 그림 1B에서 UV 방사선 단계 D 이후 무료 SAH의 초과를 추가로 분석, C : 분석 제어 그림 1B에 단계 c와 d 중 경쟁과 같은 자유 SAH의 초과 (필수를 포함하여, 그림 1B에 단계 B에서 이외 구체적으로 캡처 단백질을 결정하기 위해); PD : 풀다운 의미 그림 1B에 없음 UV 방사선 단계 D 무료 SAH없이 또한, CPD : 경쟁자로 SAH를 사용하여 풀다운 메뉴의 제어, A + PD : 더 추가 의미가없는 조합 분석 플러스 풀다운 워크플로우에는 공짜 SAH의). 컷 아웃 단백질 겔 밴드에서 - 겔 tryptic 소화 후 MS에 의해 식별 단백질은 매우 leftt에 부여됩니다. 이 사진 crosslinking이 실험의 수율과 감도를 강화하고, 특이성이 쉽게 무료 SAH의 초과를 사용하여 경쟁 실험에 대한 검사 수 있습니다 분명하다. MTases와 현재 그림에 표시된 사람들의 중복 샘플 CCMS 실험에 의해 확인된 다른 선택된 단백질에 대한 표 1을 참조하십시오.

그림 3 : (A) CCMS 분석 경쟁 제어 (C), 그리고 풀다운 (PD)를 식별 단백질의 중복을 explaning 벤 다이어그램. 왼쪽 MTases 및 SAH의 nucleosidase, 단 수, 표 1을 참조. 오른쪽 : 표 2와 보조 테이블 S1를 참조 모든 확인된 단백질의 개수입니다. 최소한 2 펩티드와 단백질의 식별 번호는 괄호에 부여됩니다.
토론
설명 프로토콜을 다음과 같은 경우 다음과 같은주의 사항 및 의견은 유용할 수 있습니다 :이 이후 엄격한 세척 조건을 허용하는 등 CCS의) 가장 큰 장점은 CC와 MTase 사이에 공유 결합의 형성에 자리잡고 있습니다. 공유 결합 crosslink는 자외선 (310 nm의 최대.)에 의해 실행 photoreaction에 의해 이루어진다. 일반 오버헤드 빛이 자외선의 작은 파편만이 포함되어 있습니다 단, caproBox의 제어와 냉각 조사까지 오버헤드에 더 이상 노출 또는 일 빛으로부터 SAH - CC를 보호합니다. B) MTases가 격리해야하는에서 생물 학적 샘플이 변성하는 경향이 단백질을 포함할 수 있습니다, 따라서 그것은 샘플이 시원한 유지하고 항상 얼굴이 볼만 피하기 위해 필수입니다. C) caproBox는 0-4로 샘플을 냉각 ° C, UV 빛을 발하는 램프가, 그러나 또한 열을 방출. 따라서 그것은 간략하게 조사하기 전에 튜브를 원심 필요하므로 대문자 또는 유리 벽면을 준수하는 단백질 침전의 씨앗을 형성 수 없습니다. 자석 구슬을 다시 중단이 (세척 솔루션의 예) 손으로 불가 능할 경우 D), 곧 초음파 욕조에 샘플을 배치하여 초음파를 적용합니다. E) 갓 0.2 % TFA/60 %의 ACN 솔루션을 준비합니다. 우리는 달리 캡처된 단백질이 구슬에서 죽습되지 않을 수 있습니다는 사실을 발견했습니다. F) 캡처 단백질의 최종 분석 LC-MS/MS에 의해 수행됩니다. 질량 분석법은 매우 민감한 방법입니다. 이것은 마지막 단계 (단계 더 나아가 2.11)에서 독점적으로 LC - MS 등급 시약을 사용해야합니다. 외부 단백질 소스, 먼지 또는 실험자에서 발생하는 등 각질에 의해 실험의 오염을 피하십시오. 특히 최종 소화 단계 동안, 그것은 장갑과 실험 복과 가능한 고정시켜 또는 이상 깨끗한 벤치에서 최종 단계를 수행하기 위해, 깨끗한 작업 공간에 대한 관심을 지불하는 것이 좋습니다. LC MS 학년 물에 tryptic 소화에 사용되는 50 MM의 탄산수 소 암모늄 버퍼, -20 ° C에서 0.22 μm의 필터, 나누어지는, 저장을 통해 필터를 준비하고, contaminations가 발생하지 않도록 한 번만 각 나누어지는을 사용합니다. 트립신 용액 (시퀀싱 학년, 로체, 동결 건조된 트립신 1 MM HCL을 추가하여 0.5 μg / μl 솔루션을 준비)를 4 ° C 몇 주 동안 저장할 수 있습니다. G) 신뢰할 수있는 질량 스펙트럼을 얻으려면 그것은 ESI-MS/MS 분석에서 안정 스프레이를하는 것이 필수적입니다. H) 현재 연구에 사용되는 이외의 LC-MS/MS 시스템의 경우, 측정 매개 변수와 펩티드 식별 알고리즘은 개별적으로 조정해야합니다.
다음 수정은 설명 프로토콜과 관련하여 가능합니다 :)이 또는 60% ACN/0.2 %의 TFA (단계 2.11)에 의해 비즈에서 단백질을 발표하기 위해 단백질 직접 tryptically 구슬 중지 (이내 소화 수있는 동일한 볼륨으로 단계 5.1)이나, SDS - PAGE에 대해, 단백질은 95 단계로 2.9 이후 수집한 비즈를 보류하고 가열에 의해 발표 수 있습니다 ° C SDS 샘플 버퍼에 10 분 (모두 전체 정지 또는 유일한 뜨는 로드할 수에 대한 겔 주머니에). 우리는 비즈 천천히에도 여러 수성 세척 단계 후 온 - 비드 tryptic 소화하는 동안 수성 tryptic 다이제스트 솔루션에 폴리머 소량의 릴리스는 사실을 발견했습니다. 폴리머 오염 MS 펩티드 식별을 방해하고 80 % ACN을 (적어도 세 번)를 사용하여 비즈 씻어 수 있습니다. 80 % ACN 세척 단계 후, 비즈 전에 온 - 비드 tryptic 소화 물에 한번 세탁해야합니다. B) streptavidin - horseraddish 퍼옥시데이즈 및 ECL 기판을 사용하여 서양 blots 또한 단백질에 비오틴이 포함된 CC의 성공 crosslinking을 시각화하는 데 사용할 수 있습니다. 감도가 젤의 은색 얼룩보다 약 10 배 이상이기 때문에 따라서, 단계 3.2 이후 얻은 젤이, blotted 또는 수 있습니다 중, 10 μl 샘플 단계 2.7 이후에도 서양 얼룩으로 분석 수 있습니다. 후자의 경우에는 endogeneously biotinylated 단백질도 인위적으로 SAH - CC로 biotinylated 단백질 이외에 감지됩니다 잊었습니다. C) 우리는, 특별한 시스템 (lysate, 대상 단백질, 선택성 그리고 CC의 반응 기능을 주소)에 따라 사실을 발견 여부 crosslinking 반응은 무료 CC 및 솔루션 4 단백질 사이에 일어난 일입니다 "오프 구슬"구성, 또는 현재 설명 "에 - 구슬 '구성 (그림 1B)은 더 나은 수행합니다.
일반적으로 메서드는 또한와 호환되어야하는 국가 수준의 예술 안정 동위 원소 단백질이나 펩타이드의 라벨 기술, 또는 2D 겔 전기 영동에 의해 캡처 샘플 평가. "오프 구슬"구성에서 전체 셀 (게시되지 않은 결과) 내에 단백질을 포착하는 것도 가능합니다. 또한, 약물이나 단백질의 cofactor 구속력이 사이트는 MS 펩타이드 배열에 의해 단백질 시퀀스 내에서 CC의 가까이별로 crosslinking 위치를 결정하여 설명하실 수 있습니다. 단백질을 작은 분자의 바인딩 모드는 다른 사용자를 사용하여 탐험 수 있습니다택 기능과 다양한 링커 길이에 hemical 첨부 파일 위치. 으로 현재 연구에 나타난 단백질 또한 단백질 바인딩 파트너는 선택도 기능 (PrmA의 기판으로 RplK) 또는 알려지지 않은 작은 분자의 단백질 상호 작용 (GlnA에 SAH) 확인할 수 있습니다하여 해결. 요약, CCS, 사진 crosslinking의 반응의 추가 기능, 고감도와 복잡한 단백질 혼합물에서 낮은 풍부한 단백질이나 기능성 단백질 가족의 격리 및 식별 가능하며 작은 분자를 연구를위한 추가 도구를 사용하여 과학자를 제공 - 단백질 상호 작용 .
공개
감사의 말
이 작품은 인간 프론티어 과학 프로그램기구 (HFSP 수상 2007 년 RGP0058/2007-C)에 의해 지원되었다. 우리는 프로젝트를 시작을위한 유익한 토론을위한 교수 리차드 로버츠 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| SAH caproKit™ | caprotec bioanalytics GmbH | 1-1010-050 (50 reactions) 1-1010-010 (10 reactions) | Includes the SAH-CC, SAH competitor, streptavidin coated magnetic beads, capture buffer, and wash buffer |
| caproBox™ | caprotec bioanalytics GmbH | 1-5010-003 (110 V) 1-5010-004 (230 V) | For reproducible photo-activation while cooling the samples |
| caproMag™ | caprotec bioanalytics GmbH | 1-5100-001 | For easy handling of magnetic particles without pipetting |
참고문헌
- Katayama, H., Oda, Y. Chemical proteomics for drug discovery based on compound-immobilized affinity chromatography. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 855, 21-27 (2007).
- Barglow, K. T., Cravatt, B. F. Activity-based protein profiling for the functional annotation of enzymes. Nat. Methods. 4, 822-827 (2007).
- Koster, H., Little, D. P., Luan, P., Muller, R., Siddiqi, S. M., Marappan, S., Yip, P. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev. Technol. 5, 381-390 (2007).
- Dalhoff, C., Hueben, M., Lenz, T., Poot, P., Nordhoff, E., Koster, H., Weinhold, E. Synthesis of S-Adenosyl-L-homocysteine Capture Compounds for Selective Photoinduced Isolation of Methyltransferases. ChemBioChem. 11, 256-265 (2010).
- Lu, S. C. S-Adenosylmethionine. Int. J. Biochem. Cell. Biol. 32, 391-3952 (2000).
- Cantoni, G. L. Biological methylation: selected aspects. Annu. Rev. Biochem. 44, 435-451 (1975).
- Jeltsch, A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. ChemBioChem. 3, 274-293 (2002).
- Chow, C. S., Lamichhane, T. N., Mahto, S. K. Expanding the nucleotide repertoire of the ribosome with post-transcriptional modifications. ACS Chem. Biol. 2, 610-619 (2007).
- Grillo, M. A., Colombatto, S. S-Adenosylmethionine and protein methylation. Amino Acids. 28, 357-362 (2005).
- Fujioka, M. Mammalian small molecule methyltransferases: their structural and functional features. Int. J. Biochem. 24, 1917-1924 (1992).
- Borchardt, R. T., Salvatore, F., Borek, E., Zappia, V., Williams-Ashman, H. G., Schlenk, F. . The Biochemistry of S-Adenosylmethionine. , 151-171 (1977).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S-adenosyl-l-methionine. Biochemistry. 25, 4444-4449 (1986).
- Keller, A., Nesvizhskii, A. I., Kolker, E., Aebersold, R. Empirical statistical model to estimate the accuracy of peptide identifications made by MS/MS and database search. Anal. Chem. 74, 5383-5392 (2002).
- Nesvizhskii, A. I., Keller, A., Kolker, E., Aebersold, R. A statistical model for identifying proteins by tandem mass spectrometry. Anal. Chem. 75, 4646-4658 (2003).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유