Method Article
Profiling von Methyltransferasen und andere S-Adenosyl- L-Homocystein-bindende Proteine von Capture Compound Mass Spectrometry (CCMS)
In diesem Artikel
Zusammenfassung
Capture Compounds sind trifunktionelle kleinen Molekülen, um die Komplexität des Proteoms durch funktionelle reversible kleines Molekül-Protein-Wechselwirkung durch Photo-Vernetzung und Reinigung gefolgt reduzieren. Hier verwenden wir ein Capture Compound mit S-Adenosyl- L-Homocystein-Bindung, wie Selektivität Funktion Methyltransferasen aus einem isolieren Escherichia coli Ganzzelllysat und weisen sie von MS.
Zusammenfassung
Es gibt eine Vielzahl von Ansätzen, um die Komplexität des Proteoms auf der Basis von funktionalen kleines Molekül-Protein-Interaktionen, wie Affinitätschromatographie 1 oder Activity Based Protein Profiling 2 zu reduzieren. Trifunktionale Capture Compounds (CCs, Abbildung 1A) 3 sind die Basis für einen generischen Ansatz, in der die ursprüngliche Gleichgewicht-driven Interaktion zwischen einem kleinen Molekül-Sonde (die Selektivität Funktion, hier S-Adenosyl-L-Homocystein, SAH, Abbildung 1A) und Target-Proteine irreversibel auf Foto-Vernetzung zwischen einer unabhängigen Foto-aktivierbare Reaktivität Funktion (hier ein Phenylazid) der CC und die Oberfläche der Zielproteine fixiert. Die Sortierfunktion (hier Biotin) dient dazu, den CC zu isolieren - Protein-Konjugate aus komplexen biologischen Mischungen mit Hilfe einer festen Phase (hier Streptavidin Magnetpartikel). Zwei Konfigurationen der Experimente sind möglich: "off-Perle" 4 oder der hier beschriebenen "on-bead"-Konfiguration (Abbildung 1B). Die Selektivität Funktion kann praktisch jedes kleines Molekül von Interesse (Substrate, Inhibitoren, Wirkstoffmoleküle) werden.
S-Adenosyl-L-Methionin (SAM, Abbildung 1A) ist wahrscheinlich, Sekunde, um ATP, die am häufigsten verwendete Cofaktor in der Natur 5, 6. Es ist, als die großen Methylgruppendonator in allen lebenden Organismen mit der chemischen Reaktion durch SAM-abhängigen Methyltransferasen (MTasen), die DNA 7, RNA 8, Proteine 9 oder kleine Moleküle 10 Methylat katalysiert werden. Angesichts der entscheidenden Rolle der Methylierung in verschiedenen physiologischen Szenarien (Genregulation, Epigenetik, Stoffwechsel), kann die Profilierung der MTasen erwartet, dass eine ähnliche Bedeutung in der funktionellen Proteomik, wie die Profilierung der Kinasen zu werden. Analytische Werkzeuge für ihre Profilierung, aber bisher nicht zur Verfügung. Haben wir kürzlich eine CC mit SAH als Selektivität Gruppe auf diese technologische Lücke (Abbildung 1A) zu füllen.
SAH, das Produkt von SAM nach Methyl-Transfer, ist eine bekannte allgemeine MTase Produkt-Inhibitor 11. Aus diesem Grund und weil die natürliche Kofaktor SAM wird durch weitere Enzyme der Übertragung in anderen Teilen der Cofaktor oder den Beginn der Radikalreaktionen sowie gebrauchte aufgrund seiner chemischen Instabilität 12, SAH eine ideale Selektivität Funktion für eine CC zu MTasen Ziel ist. Hier berichten wir über die Nützlichkeit der SAH-CC und CCMS durch Profilierung MTasen und andere SAH-bindende Proteine aus dem Stamm DH5 von Escherichia coli (E. coli), einer der am besten charakterisierten Prokaryoten, die als das bevorzugte Modell gedient hat Organismus in zahllosen biochemischen, biologischen und biotechnologischen Studien. Photo-aktivierten Vernetzung erhöht die Ausbeute und die Empfindlichkeit des Experiments und die Spezifität kann leicht in Konkurrenz Experimente mit einem Überschuss an freien SAH getestet werden.
Protokoll
1) Herstellung von E. coli DH5 Zelllysat
- Impfen ein 2-ml-Kultur (LB-Medium in einem Reagenzglas) mit dem E. coli-Stamm DH5 direkt aus einem Glycerin Lager und bei 37 ° C und 250 rpm für 8 h. Verwenden Sie autoklaviert LB-Medien (10 g / l Bacto-Trypton, 5 g / L Hefeextrakt, 10 g / L NaCl, pH 7,5).
- Inoculate 250 mL LB-Medium in einem 1 L Schüttelkolben mit Schikanen mit den 2 mL Kultur und Inkubation über Nacht bei 37 ° C und 166 rpm in einem Inkubator mit Schüttler.
- Impfen vier 5 L Schüttelkolben mit Schikanen mit je 2,5 L LB-Medien mit dem 250 mL Kultur (50 ml pro Kolben) und inkubieren Sie die Kulturen bei 37 ° C und 166 rpm bis zu einer OD 600 von 0,8 erreicht ist.
- Ernten Sie die Zellen durch Zentrifugation bei 4 ° C, 3000x g für 20 min. Führen Sie die weitere Handhabung bei 0-4 ° C oder auf Eis.
- Re-suspend das geerntete Zellmaterial in Milli-Q Wasser, in einem Zentrifugenbecher und Zentrifuge kombinieren für weitere 30 min bei 6000X g und 4 ° C.
- Bewahren Sie die daraus resultierenden 20 g Zellmaterial bei -20 ° C oder -78 ° C.
- Re-suspend die Zellen in 100 ml eiskaltem Zellöffnung Puffer (6,7 mM MES, 6,7 mM NaOAc, 6,7 mM HEPES, 1 mM EDTA, 10 mM β-Mercaptoethanol, 200 mM NaCl, pH 7,5, 10% (w / v) Glycerin, 0,2 mM PMSF) und beschallen dreimal für 1 min in vier 25 ml Portionen auf Eis mit einem sonifier (zB SONOPULS HD 2070 von BANDELIN electronic GmbH & Co. KG, maximale Amplitude, Dauerleistung).
- Centrifuge das Lysat über Nacht bei 2370x g und 2 ° C.
- Konzentrieren Sie den Überstand in 14 ml durch Ultrafiltration (z. B. mit iCON Konzentratoren, 7 mL/9K von Pierce) bei 2370x g und 2 ° C.
- Einem Abbau der viskosen Konzentrat aus kleinen Molekülen wie SAM oder SAH durch Gelfiltration bei 2 ° C (zB Zeba Desalt Spin Columns, 10 ml, von Pierce, vier Säulen, vier Mal Äquilibrierung mit 5 ml; Pufferspeicher bei 1000x g für 2 min entfernt eiskalt Zellöffnung Puffer und Puffer bei 1000x g für 2 min entfernt, bzw., 3,5 mL, um jede Spalte angewendet konzentrieren; 45 min Zentrifugation bei 24x g, dann zweimal 2 min bei 1000x g)
- Supplement die daraus resultierenden 13 ml Lysat mit 13 ml eiskaltem Glycerin und Roche mini Protease Inhibitor Cocktail Tabletten, EDTA frei. Mix und lösen sich die Tabletten.
- Bewahren Sie das Lysat bei -20 ° C
- Bestimmen Sie die Gesamt-Protein-Konzentration von der Bradford-Assay (21 mg / ml im vorliegenden Fall). Bradford-Reagenz: 100 mg Coomassie Brilliant Blue G-250 in 50 mL 95% Ethanol, 100 ml 85% (w / v) Phosphorsäure, verdünne auf 1 l, wenn der Farbstoff vollständig gelöst hat, und durch Whatman Nr. 1 Papier nur vor dem Gebrauch.
2)-Capture-Assay (A), Wettbewerb Control (C), Pulldown (PD), Competition Kontrolle der Pulldown (CPD) und Combined-Capture-Assay und Pulldown (A + PD)
- Für die Erfassung Experimenten wurde das SAH caproKit (caprotec Bioanalytik GmbH) verwendet, der auch die SAH-CC, frei SAH als Konkurrent, Streptavidin-beschichteten magnetischen Kügelchen mit 1 Mikrometer Durchmesser (Dynabeads MyOne Streptavidin C1, Invitrogen Dynal), 5x Pufferspeicher (5X CB, die 100 mM HEPES, 250 mM Kaliumacetat, 50 mM Magnesium-Acetat und 50% Glycerin) und 5fach-Waschpuffer (5X WB, mit 250 mM Tris HCl, pH 7,5, 5 mM EDTA, 5 M NaCl, 42,5 uM Octyl-β-D-glucopyranosid).
- Für mehrere parallele Versuche empfiehlt es sich, ein Master-Mix aus Wasser herzustellen ist, Pufferspeicher und E. coli-Lysat und Reaktionen in den verschiedenen Rohren von einem 200 ul-PCR-Röhrchen Streifen durchführen (empfohlen 0,2 ml Thermo-Strip, Thermo Scientific, AB-1114). Hier sind die Mengen für einen Reaktionsgefäß gegeben. Ergebnisse für fünf verschiedene Experimente vorgestellt, die Capture-Assay (A), Wettbewerb Kontrolle (C), Pulldown (PD), Wettbewerb Kontrolle von Pulldown (CPD) und kombiniert Capture-Assay und Pull-Down-(A + PD) sind.
- Für jede Reaktion vorzubereiten 1,5 ml 1X WB durch Zugabe von 0,3 mL 5X WB zu 1,2 ml Milli-Q Wasser.
- Bereiten SAH-CC geladen Streptavidin-beschichteten magnetischen Kügelchen (caproBeads) in 200 ul PCR-Röhrchen-Streifen. Deshalb mischen 25 ul 100 uM SAH-CC mit 50 ul von 10 mg / ml Streptavidin-beschichteten magnetischen Kügelchen für jedes Aliquot, kräftig schütteln die resultierenden Suspensionen bei Raumtemperatur für 2 min, damit die Bindung des Biotin-Einheit des SAH- CC an die Streptavidin auf der Magnetic-Bead-Oberfläche und sammeln die Perlen mit einem starken Magneten (z. B. in die Kappen der PCR-Röhrchen-Streifen mit dem caproMagTM magnetische Vorrichtung, caprotec Bioanalytik GmbH). Die Überstände verwerfen, erneut zu suspendieren die daraus resultierenden caproBeads in 200 ul WB, magnetisch sammeln caproBeads (in die Kappen der PCR-Röhrchen-Streifen), und entsorgen Sie die supernating WB. Close Rohre zu vermeiden, Trocknung der Kugeln.
- Bereiten Aliquots von E.coli DH5 ganzeZelllysat in neue PCR-Röhrchen bei 0-4 ° C über einen Master-Mix (siehe 2.2). Für eine Reaktion, zu ergänzen, ein Volumen von Milli-Q Wasser für eine endgültige Reaktion Mix Volumen von 100 ul mit 20 ul 5X CB. Mix, fügen 0,26 mg E. coli-Lysat, und vorsichtig durch Umdrehen mischen. Nur für C-und CPD, mit 20 ul 10 mM SAH Wettbewerber Lösung und vorsichtig durch Umdrehen mischen (add Milli-Q Wasser anstelle von SAH Lösung A, PD, und A + PD). Zeichnen Sie eine 1 ul Probe aus einem für die weitere Analyse (siehe unten).
- Hängen Sie das caproBeads in den jeweiligen Lysats und Inkubation für 3 h bei 4 ° C zu halten die Perlen in Suspension durch eine Drehung um reversible Bindung von SAH-bindende Proteine, die SAH Selektivität in Abhängigkeit von der SAH-CC zu ermöglichen.
- Legen Sie die Suspensionen A, C und A + PD in der caproBoxTM (Gerät zur Bestrahlung von biochemischen Proben mit UV-Licht und gleichzeitig gekühlt wird, caprotec Bioanalytik GmbH) und bestrahlen für eine Gesamtzeit von 30 min in der geschlossenen Röhren zwischen 0-4 ° C zu einer kovalenten Vernetzung zwischen der Reaktivität in Abhängigkeit von der SAH-CC der SAH-bindende Proteine zu bilden. Deshalb entfernen die Suspensionen nach der Bestrahlung Abständen von 2,5 min, jeweils von den caproBoxTM, kühlen in Eiswasser für ~ 15 s (vor allem die Deckel), mischen einige Male durch Inversion, sehr kurz (~ 2 s) Zentrifuge zur Aussetzung verbleibenden entfernen in den Deckeln verschließen und zurück in die caproBoxTM für die nächste Bestrahlung Intervall.
- Add 20 ul 10 mM SAH Lösung A, oder 20 ul Milli-Q Wasser C, PD, CPD, und A + PD und inkubieren der Suspension für 10 min bei 4 ° C zu verdrängen, in A, SAH-bindende Proteine nicht vernetzt die SAH-CC. Halten Sie die Perlen in Suspension durch Rotation oder durch intermittierende manuelle Resuspension.
- Sammeln Sie die caproBeads aus den Suspensionen mit einem starken Magneten (zB die caproMagTM), entsorgen Sie die Überstände und Waschen der Kügelchen sechsmal - durch Resuspension und Sammlung - mit 200 ul 1X WB und einmal mit 200 ul Milli-Q Wasser.
- Die Perlen können in Milli-Q Wasser für mehrere Wochen bei 4 ° C gelagert Alternative Protokolle existieren für die weitere Verarbeitung der aufgenommenen Proteine und ihre Identifikation (siehe Diskussion).
- Waschen Sie die Beads dreimal mit 200 ul 60% Acetonitril (ACN) und lassen Sie die aufgenommenen Proteine von den Beads durch 10 min Inkubation bei Raumtemperatur unter kräftigem Schütteln mit 200 ul 60% ACN/0.2% Trifluoressigsäure (TFA) (frisch angesetzt) . Verwenden LC-MS-grade Reagenzien und Wasser.
- Magnetisch sammeln die Perlen, separate und verdampfen der Überstand zur Trockene mit einem Zentrifugalverdampfer (zB MiVac DNA-Konzentrator aus GeneVac, Inc., UK). Entsorgen Sie die Perlen.
3) SDS-PAGE von Captured Proteine
- Für SDS-PAGE, lösen sich die aufgenommenen Proteine aus dem magnetischen Kügelchen (verdampft ACN / TFA Lösungen aus Schritt 2,12) in 20 ul SDS-Probenpuffer (50 mM Tris HCl, 320 mM β-Mercaptoethanol, 2,5% SDS, 0,05% Bromphenolblau veröffentlicht , 10% Glycerin, pH 6,8). Mischen Sie die 1 ul Probe A (siehe Schritt 2,5) mit 19 ul SDS-Probenpuffer gezogen; verwenden Sie 5 ul dieser Lösung für die SDS-PAGE-Analyse (0,25% des Assay). Erhitzen Sie die Proben in SDS-Probenpuffer 10 min bis 95 ° C und lassen können, um auf Raumtemperatur abkühlen.
- Analysieren von SDS-PAGE (generic-Setup: OLS ® ProPage 4-20% Tris / Glycin Fertiggele; OLS omniPage Mini-Gel-Elektrophorese-System; SDS Laufpuffer: 25 mM Tris-Base, 200 mM Glycin, 0,1% SDS, pH 8,3 ; Laufzeit 90 min bei einer konstanten Spannung von 180 V unter Eiskühlung der SDS-Laufpuffer).
- Silberfärbung des Gels mit einem MS-kompatible Silberfärbung Methode (zB ProteoSilver Silver Stain Kit von Sigma). Ein repräsentatives Ergebnis ist in Abbildung 2 dargestellt.
4) In-Gel-Trypsin-Verdau von Proteinen und Peptiden Extraktion aus Gel Bands
- Waschen Sie die mit Silber gefärbten Gel mindestens dreimal mit 100 ml Milli-Q Wasser für 10 min nach der Silberfärbung Stop-Lösung wurde entfernt.
- Schneiden Sie das Gel Bands (zB mit einem sauberen Skalpell und Transfer in ein 0,5 ml Eppendorf-Röhrchen) und bei -20 ° C oder direkt verarbeiten. Waschen Sie die Gel-Bands für 15 min, jeweils mit 100 ul Wasser, 100 ul 50% Ethanol, 100 ul Wasser, 100 ul 50% Ethanol und 5 min mit reinem Ethanol. Wiederholen Sie diesen Waschvorgang wieder.
- Re-Hydrat das Gel Band mit 10 ul in-Gel-Verdau-Lösung (12,5 ng / ul Sequenzierung grade Trypsin in 50 mM Ammoniumhydrogencarbonat, durch Zugabe von 7,5 ul 0,5 ug / ul Trypsin-Lösung in 1 mM HCl auf 292,5 ul 50 mM Ammoniumhydrogencarbonat vorbereiten ) für 45 min bei 4 ° C. Entfernen Sie den Überstand und ersetzen durch 20 ul 50 mM Ammoniumhydrogencarbonat (ohne Trypsin) durch Inkubation bei 37 ° C über Nacht unter Schütteln.
- Sammeln Sie den Überstand. Für Peptid-Extraktion, inkubieren das Gel Band mit 20 ul 5% Ameisensäure (FA) für 15 min unter Schütteln hinzu20 ul ACN und inkubiert für weitere 15 min unter Schütteln. Kombinieren Sie den Überstand mit dem vorherigen Überstand und wiederholen Sie den Peptid-Extraktionsverfahren noch einmal.
- Man dampft die kombinierten drei Überstände bis zur Trockne, löst in 10 ul 5% FA unter Schütteln und Anwendung von Ultraschall (Ultraschall-Bad, zB Sonorex von Bandelin, Deutschland) und fahren Sie mit Entsalzung (Schritt 5.2 und weitere).
5) tryptischen Verdaus von Captured Proteine und Herstellung von Peptiden für die LC-MS/MS
- Lösen Sie die aufgenommenen Proteine aus dem magnetischen Kügelchen (verdampft ACN / TFA Lösungen aus Schritt 2,12) in 10 ul 50 mM Ammoniumhydrogencarbonat mit einem Ultraschallbad und Vortex veröffentlicht werden, fügen 1 ul 0,5 ug / ul Trypsin in 1 mM HCl und inkubieren Sie die Lösung bei 37 ° C über Nacht.
- Desalt die Lösung mit den tryptischen Peptide des aufgenommenen Proteinen unter Verwendung von C18-Material (z. B. 2-10 ul StageTips, 20 uL Spitze, Proxeon Biosystems A / S, Odense, Dänemark, Das Verfahren des Herstellers: Voraussetzung mit 10 ul 50% Methanol / 5 % FA, mit 10 ul 5% FA äquilibriert, laden mit Peptiden der aufgenommenen Proteine, waschen mit 10 ul 5% FA, eluieren zweimal mit je 10 ul 50% Methanol / 5% FA).
- Man dampft die entsalzt Peptide in 50% Methanol / 5% FA zur Trockne in 5,5 ul 0,1% FA löst unter leichtem Schütteln und Anwendung von Ultraschall (Ultraschallbad) und Analyse der Probe durch nanoLC-MS/MS.
6) NanoLC-MS/MS Analysis
- Transfer-Probe in Probenplatte und Platzteller in den Nano-flow-Flüssigchromatographie (nanoLC) System (zB Easy-nLC Flüssigchromatographiesystem; Proxeon Biosystems A / S, Dänemark).
- Verwenden Sie 0,1% FA in Wasser als mobile Phase A und 0,1% FA in ACN als mobiler Phase B. Nur LC-MS grade Lösungsmitteln.
- Legen Sie 5 ul der Peptid-Lösung direkt auf eine Vorsäule (zB nanoflow Biosphere C18, 5 um, 120 Å, 20 x 0,1 mm; NanoSeparations, Niederlande) zu einer analytischen Säule (zB nanoflow Biosphere C18, 5 um, 120 Å gekoppelt , 100 x 0.075 mm; NanoSeparations, Niederlande) mit 5% ACN/0.1% FA.
- Während LC, eluieren Peptide während einer 80 min linearer Gradient von 5% ACN/0.1% FA zu 40% ACN/0.1% FA um weitere 2 min auf 100% ACN/0.1% FA und die verbleibenden 100% ACN/0.1%, gefolgt FA für weitere 8 min mit einer kontrollierten Durchfluss von 400 Nl / min.
- Führen Massenspektrometrie (MS)-Analyse der eluierten Peptide auf eine hohe Genauigkeit state-of-the-art-Massenspektrometer (zB LTQ Orbitrap XL-Massenspektrometer; Thermo FisherScientific, Deutschland, mit einem Nanoelektrospray Ionenquelle für Elektrospray-Ionisation (ESI) ausgestattet; Proxeon Biosystems A / S, Dänemark).
- Führen Sie die massenspektrometrische Analyse in den Daten-abhängigen Modus automatisch zwischen Orbitrap-MS (Profil-Modus) und LTQ-MS/MS (Schwerpunkt-Modus) Erwerb wechseln.
- Steuerung des Massenspektrometers duty cycle, indem Sie die Einspritzzeit Automatic Gain Control.
- Erwerben Umfrage Full-Scan-MS-Spektren (von m / z 300 bis 2000) in der Orbitrap mit einer Auflösung von r = 60.000 m / z 400 (nach Anreicherung auf einen Zielwert von 500.000 Ladungen in der linearen Ionenfalle).
- Stellen Sie das Instrument, um nacheinander isolieren die intensivsten Ionen (bis zu fünf, je nach Signalstärke) für die Fragmentierung in der linearen Ionenfalle mit Kollisions-induzierte Dissoziation (CID) auf einem Sollwert von 10.000 Ladungen. Das erhaltene Fragment-Ionen sind in der LTQ aufgezeichnet.
- Für eine genaue Masse-Messungen in der MS-Modus, mit dem einfach geladenen Polydimethylcyclosiloxan Hintergrund Ion (Si (CH 3) 2 O) 6 H + (m / z 445,120025) während der Elektrospray-Prozess aus der Umgebungsluft als das Schloss Masse erzeugt für Echtzeit-internen Rekalibrierung.
- Dynamisch auszuschließen Ziel-Ionen bereits für CID für die Dauer von 60 s. massenselektierten
- Set Ladezustand Screening und die Ablehnung von Ionen für CID mit unassigned kostenlos.
- Weitere massenspektrometrische Einstellungen sind wie folgt: set spray Spannung von 1,6 kV, eingestellte Temperatur der beheizten Kapillare Transfer bis 200 ° C, und normalisiert Aufprallenergie bei 35% für MS 2. Die minimale Signal für MS 2 erforderlich ist 500 zählt. Übernehmen einer Aktivierung q = 0,25 und einer Aktivierungszeit von 30 ms für MS 2 Akquisitionen.
- Zur Reinigung des LC-Systems, führen Sie eine leere laufen zwischen zwei aufeinander folgenden CCMS-Messungen.
7) Peptid-und Protein Identification via Automated Sequence Database Search
- Verwenden Sie ein Protein-Identifizierung Algorithmus, um die MS / MS-Daten (im vorliegenden Fall in Raw-Dateien gespeichert sind), zB SEQUEST in BioworksBrowser 3.3.1 SP1 (Thermo FisherScientific, Deutschland) und X! Tandem (The Global Proteome Organization Maschine umgesetzt zu analysieren; Version 2007.01.01.1) in der Sc umgesetztaffold 3-Software (Version Scaffold_3_00_03, Proteome Software Inc., USA).
- Automatisierte Recherche in Datenbanken gegen die jüngsten UniProtKB / Swiss-Prot-Datenbank Release www.expasy.org des Organismus untersucht (Datenbank für vorliegende Studie verwendet: Escherichia coli, Stamm K12, Release 57 bis 11).
- Verwenden Sie die folgenden Einstellungen für die automatische Recherche in Datenbanken innerhalb SEQUEST: 5 ppm Vorläufer Toleranz, 1 amu-Fragment-Ionen-Toleranz und volle Trypsin Spezifität erlauben bis zu zwei verpasste Spaltungen. Erlauben Sie als variable Modifikationen Phosphorylierung an Serin, Threonin und Tyrosin; Oxidation von Methionin; Desamidierung an Asparagin und Glutamin; Acetylierung an Lysin und Serin; Formylierung an Lysin und Methylierung an Arginin, Lysin, Serin, Threonin und Asparagin. Verwenden Sie keine festen Änderungen in der Datenbank zu suchen.
- Laden srf oder dta und aus Dateien, die von SEQUEST in das Gerüst 3, die Wahrscheinlichkeit Beurteilung von Peptid-und Protein-Identifikationen Zuweisungen durch die Kombination SEQUEST und X! Tandem-Datenbank durchsucht führt generiert. Gerüst für leicht zu vergleichen und zu visualisieren Protein Listen aus verschiedenen Proben nützlich (im vorliegenden Fall A, C, PD, CPD, und A + PD).
- Stellen Sie die Parameter innerhalb der Scaffold-3-Software nur Peptide mit ≥ 95% Wahrscheinlichkeit durch die Peptide Prophet Algorithmus (ref.13) angegeben zu betrachten. Set Proteinidentifizierung Wahrscheinlichkeiten für verschiedene Peptid-Zuordnungen zu ≥ 95% nach dem Protein Prophet Algorithmus 13. Für einzelnes Peptid-Protein-Identifikationen, willkürlich Protein Aufenthaltswahrscheinlichkeit auf ≥ 50% und manuell inspizieren dem entsprechenden Peptid MS / MS-Spektren. Proteine, die ähnliche Peptide umfassen und nicht basierend auf MS / MS-Analyse allein unterschieden werden von der Software zu den Grundsätzen der Sparsamkeit erfüllen gruppiert. Die geschätzte False Discovery Rate von Peptid-Identifikationen kann anhand der umgekehrte Protein-Datenbank-Ansatz und sollte <1% sein.
- Repräsentative Ergebnisse der CCMS Experimente sind in den Tabellen 1, 2 gegeben, und ergänzende Tabelle S1 (daran, dass die Protein-Datenbank ist nicht up-to-date für einige Proteine, zB PrmB (aka YfcB) oder RsmH (aka MraW)), sowie wie in Abbildung 3.
8) repräsentative Ergebnisse
Tabelle 1:
| Protein | ORF | MW / kDa | Beschreibung | Substrate | A | C | PD | CPD | A + PD |
| Dcm | b1961 | 53,5 | DNA-Cytosin MTase | DNA (M5C) | 1 | 0 | 0 | 0 | 1 |
| RlmI | b0967 | 44,4 | 23S rRNA m5C1962 MTase | rRNA (M5C) | 17 | 0 | 17 | 0 | 20 |
| RlmL | b0948 | 78,9 | 23S rRNA m2G2445 MTase | rRNA (M2G) | 12 | 0 | 0 | 0 | 10 |
| TRMB | b2960 | 27,3 | tRNA (Guanin-N (7) -)-MTase | tRNA (m7G) | 11 | 0 | 0 | 0 | 13 |
| CmoA | b1870 | 27,8 | tRNA (cmo5U34)-MTase | tRNA (mcmo5U) | 7 | 0 | 0 | 0 | 4 |
| RsmG | b3740 | 23,4 | 16S rRNA m7G MTase | rRNA (m7G) | 6 | 0 | 1 | 0 | 5 |
| RsmH | b0082 | 34,9 | 16S rRNA m4C1402 MTase | rRNA (M4C) | 5 | 0 | 0 | 0 | 7 |
| RSMD | b3465 | 21,7 | 16S rRNA m2G966 MTase | rRNA (M2G) | 2 | 0 | 0 | 0 | 2 |
| RsmB | b3289 | 48,3 | 16S rRNA m5C967 MTase | rRNA (M5C) | 1 | 0 | 0 | 0 | 0 |
| MnmC | b2324 | 74,4 | Bifunktionelle Protein enthält tRNA (mnm (5) s (2) U34)-MTase | tRNA (mnm5s2U) | 1 | 0 | 0 | 0 | 0 |
| PrmB | b2330 | 35,0 | 50S ribosomalen Protein L3 Gln150 MTase | Protein (Gln) | 13 | 0 | 0 | 0 | 15 |
| CheR | b1884 | 32,8 | Chemotaxis-Protein MTase | Protein (Glu) | 0 | 0 | 0 | 0 | 1 |
| Cfa | b1661 | 44,9 | Cyclopropan-Fettsäuren-Acyl-Phospholipid-Synthase | kleines Molekül | 15 | 0 | 0 | 0 | 14 |
| Tam | b1519 | 29,0 | Trans-Aconitat 2-MTase | kleines Molekül | 2 | 0 | 0 | 0 | 3 |
| CysG | b3368 | 50,0 | Siroheme Synthase schließt Uroporphyrinogen-III C-MTase | kleines Molekül | 1 | 0 | 0 | 0 | 2 |
| SMTA | b0921 | 29,8 | Protein SMTA | (? A) | 7 | 1 | 0 | 0 | 8 |
| MtnN | b0159 | 24,4 | 5'-Methylthioadenosin / SAH Nukleosidase | kleines Molekül b | 36 | 0 | 0 | 0 | 39 |
| GlnA | b3870 | 51,9 | Glutaminsynthetase | kleines Molekül c | 90 | 0 | 0 | 0 | 97 |
| RplK | b3983 | 14,9 | 50S ribosomalen Protein L11 | von Protein MTase PRMA d | 2 | 0 | 0 | 0 | 2 |
a Nicht (voll) gekennzeichnet
b Keine Methylierung, sondern die Spaltung der glykosidischen Bindung des SAH
c Keine Methylierung die Bindung von SAH in die ATP-Bindungsstelle durch CCMS Experimente mit ATP als Konkurrent (Daten nicht gezeigt) gezeigt
d Substrat des 50S ribosomalen Protein L11 MTase PRMA; reproduzierbar spezifische Identifizierung von CCMS (Daten nicht gezeigt)
Tabelle 1: MTasen und andere ausgewählte Proteine durch CCMS Experimenten identifiziert. Die angegebenen Zahlen bezeichnen den ungewichteten Peptid spektrale count pro Protein. Die Proben werden Duplikate der von SDS-PAGE/silver analysiert in Abbildung 2 zu färben. Viel mehr MTasen und andere SAH-bindende Proteine sind in der CCMS-Assay (A) im Vergleich zu den Pulldown (PD) und SAH Spezifität wird durch das fast völlige Fehlen dieser Proteine in den Wettbewerb Kontrolle (C) identifiziert werden.
Tabelle 2:
| A | C | PD | CPD | A + PD | |
| A | 111 (64) | ||||
| C | 65 (41) | 107 (46) | |||
| PD | 25 (15) | 23 (13) | 61 (17) | ||
| CPD | 23 (13) | 22 (12) | 20 (14) | 47 (14) | |
| A + PD | 87 (61) | 64 (41) | 23 (14) | 22 (12) | 124 (67) |
Tabelle 2: Gesamtzahl der identifizierten Proteine in der CCMS läuft und Protein Überschneidungen zwischen den Läufen. Die Zahl der Proteine mit mindestens 2-Peptide identifiziert werden, in Klammern angegeben. Die hohe Reproduzierbarkeit der Methode kann von der hohen Protein-Überlappung geschlossen werden (hauptsächlich unspezifische Proteine) zwischen vergleichbaren Experimenten (A vs C und vor allem A vs A + PD, sondern auch PD vs CPD) vor allem mit den Proteinen robust identifizierten sich mit mindestens 2 Peptide. Siehe auch Abb. 3 für Venn-Diagramme und ergänzende Tabelle S1 für eine Liste aller identifizierten Proteine.
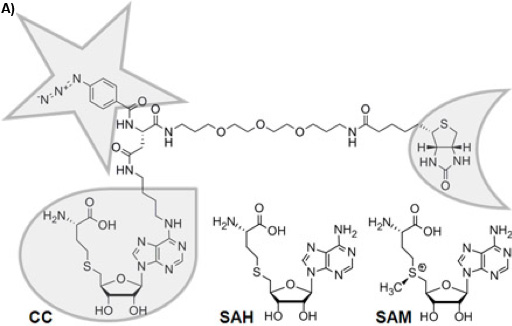
Abbildung 1A: Chemische Struktur des trifunktionalen Capture-Compound (CC). Die Selektivität Funktion wird mit einem Tropfen, die Reaktivität Funktion mit einem Stern, und die Sortier-Funktion mit einem Halbmond umrahmt. Die chemisch stabil S-Adenosyl-L-Homocystein (SAH) ist der Cofaktor Produkt der S-Adenosyl-L-Methionin (SAM) nach Methylgruppentransfer von SAM-abhängige MTasen, für die SAH wirkt wie ein Produkt-Hemmer.
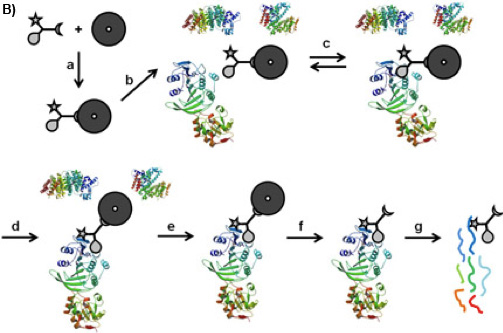
Abbildung 1B: CCMS "on-bead "-Workflow. Der CC basiert auf der magnetischen Beads durch ihre Sortier-Funktion (a) gebunden, die so gebildet caproBeads mit den komplexen Protein-Mischung (b), wo eine reversible Bindung Gleichgewicht (c) zwischen der Selektivität in Abhängigkeit von etablierten inkubiert der CC und die Target-Proteine. Nach UV-Bestrahlung (d), bildet die Reaktivität Funktion einer kovalenten vernetzen. Nach dem Waschen der Magnetpartikel mit dem aufgenommenen Proteine (e), Spaltung der vernetzten CC-Protein-Komplexe aus dem magnetischen Kügelchen (f) und tryptischen Verdaus (g), können die aufgenommenen Proteine durch MS-Analyse der tryptischen Peptide identifiziert werden.
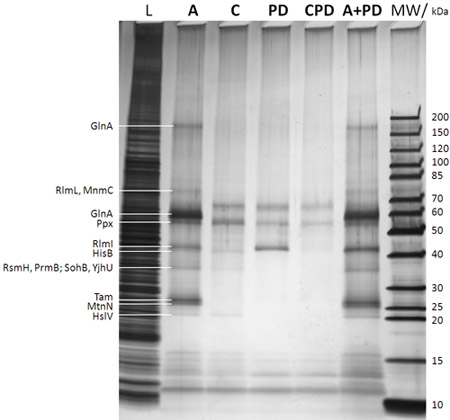
Abbildung 2: SDS-PAGE/silver Fleck Analyse der aufgenommenen Proteine (nach Schritt f in Abbildung 1B). 0,25% Probe aus dem E. coli DH5a Ganzzelllysat vor der Zugabe der caproBeads gezogen:; L Molekulargewichtsmarker mit den entsprechenden Molekulargewichte der Markerbanden gegeben, die ganz rechts: Die Spur Beschreibung ist auf der Oberseite des Gels (MW gegeben in Schritt b in Abbildung 1B; A: Assay mit Zugabe eines Überschusses an freiem SAH nach UV-Bestrahlung Schritt d in Abbildung 1B, C: Kontrolle der Assay auch ein Übermaß an freien SAH als Konkurrent während der Schritte c und d in Abbildung 1B (essentielle um festzustellen, jede nicht-spezifisch erfasst Proteine); PD: Pulldown bedeutet, dass keine UV-Bestrahlung Schritt d in Abbildung 1B und keine Zugabe von freiem SAH; CPD: Kontrolle der Pulldown mit SAH als Wettbewerber; A + PD: kombinierte Test und Pulldown Sinn kein Zusatz freier SAH während des Workflows). Proteine, die von MS aus cut-out-Protein-Gel-Bands nach In-Gel-Trypsin-Verdau identifiziert werden, die sehr leftt gegeben. Es ist offensichtlich, dass Foto-Vernetzung ergeben und die Empfindlichkeit des Experiments erhöht, und die Spezifität kann leicht in Konkurrenz Experimente mit einem Überschuss an freien SAH getestet werden. Siehe Tabelle 1 für MTasen und andere ausgewählte Proteine durch CCMS Experimente Duplikatproben die in der vorliegenden Abbildung gekennzeichnet.

Abbildung 3: Venn-Diagramme Erklären, die Überlappung der identifizierten Proteine in CCMS-Assay (A), Wettbewerb Kontrolle (C), und Pulldown (PD). Links: Anzahl der MTasen und SAH Nukleosidase, nur mit Bezug auf Tabelle 1. Rechts: Anzahl aller identifizierten Proteine, die sich auf Tabelle 2 und Tabelle Ergänzende S1. Die Zahl der Proteine mit mindestens 2-Peptide identifiziert werden, in Klammern angegeben.
Diskussion
Die folgenden Vorsichtsmaßnahmen und Anmerkungen kann nützlich sein, wenn nach dem beschriebenen Protokoll: a) Ein großer Vorteil der CCs liegt in der Bildung einer kovalenten Bindung zwischen dem CC und dem MTase, da dies eine nachträgliche stringentes Waschen Bedingungen. Die kovalente Vernetzung wird durch eine Photoreaktion durch UV-Licht (310 nm max.) Ausgelöst erreicht. Normale Oberlicht enthält nur einen kleinen Bruchteil der UV jedoch zum Schutz der SAH-CC von längeren Belichtungszeiten, um den Overhead oder sogar Sonnenlicht bis in die gesteuerte und gekühlte Bestrahlung im caproBox. b) Die biologischen Proben, aus denen die MTasen isoliert werden sollen können Proteine neigen zur Denaturierung enthalten, folglich ist es zwingend erforderlich, damit die Proben zu kühlen und zu vermeiden Schaumbildung zu allen Zeiten. c) Die caproBox kühlt die Proben auf 0-4 ° C, die Lampen emittieren UV-Licht, aber auch Wärme abgeben. Deshalb ist es notwendig, kurz zentrifugieren die Fläschchen vor der Bestrahlung, so Proteine Einhaltung der Kappen oder Fläschchen Wände können nicht Niederschlag bilden Samen. d) Falls eine erneute Aussetzung der magnetischen Kügelchen (z. B. in den Waschlösungen) ist nicht von Hand möglich ist, in Kürze gelten Ultraschall, indem die Proben in einem Ultraschallbad. e) Frisch bereiten die 0,2% TFA/60% ACN-Lösung. Wir fanden, dass sonst die aufgenommenen Proteine nicht kann von den Beads abgespalten werden. f) Die abschließende Analyse der aufgenommenen Proteine erfolgt durch LC-MS/MS durchgeführt. Die Massenspektrometrie ist eine hochempfindliche Methode. Es ist notwendig, ausschließlich LC-MS reinen Reagenzien in den letzten Schritten (Schritt 2.11 und weitere) zu verwenden. Vermeiden Sie die Kontamination der Experimente durch externe Protein-Quellen, z. B. Keratin aus Staub oder vom Experimentator. Besonders während der letzten Schritte der Verdauung, ist es empfehlenswert, die Aufmerksamkeit auf eine saubere Arbeitsfläche zu bezahlen, Handschuhe und einen Laborkittel und gegebenenfalls ein Haarnetz tragen oder im Idealfall führen Sie die letzten Schritte in einer sauberen Bank. Bereiten Sie die 50 mM Ammoniumhydrogencarbonat-Puffer für tryptischen Verdaus in LC MS-Wasser verwendet, Filter, durch 0,22 um Filter, Aliquot, Lagerung bei -20 ° C, und die Verwendung jedes Aliquot nur einmal zu Kontaminationen zu vermeiden. Die Trypsin-Lösung (Sequenzierung grade, Roche, Vorbereitung einer 0,5 ug / ul Lösung durch Zugabe von 1 mM HCl lyophilisiert Trypsin) kann bei 4 ° C gelagert werden für mehrere Wochen. g) Um eine zuverlässige Massenspektren zu erhalten ist es wichtig, einen stabilen Spray in ESI-MS/MS Analyse haben. h) Für andere LC-MS/MS Systeme als die in der vorliegenden Studie verwendet wird, muss Messparameter und Peptid Identifikationsalgorithmen individuell eingestellt werden.
Die folgenden Änderungen werden mit Bezug auf die beschriebenen Protokoll möglich: a) Alternativ zur Freigabe der Proteine von den Beads um 60% ACN/0.2% TFA (Schritt 2,11), können die Proteine direkt tryptisch innerhalb eines Bead-Suspension (verdaut werden gleiche Volumen wie in Schritt 5.1) oder, für die SDS-PAGE können die Proteine durch die Aussetzung und Heizung die Perlen nach dem Schritt von 2,9 bis 95 gesammelt veröffentlicht ° C für 10 min in SDS-Probenpuffer (beide, die ganze Aussetzung oder nur den Überstand geladen werden kann in das Gel Tasche). Wir fanden, dass die Perlen langsam loslassen kleine Mengen an Polymer in der wässrigen tryptischen Verdaus Lösung während on-bead tryptischen Verdaus auch nach mehreren wässrigen Waschschritte. Das Polymer Verschmutzung stört MS Peptid Identifikation und lässt sich die Perlen mit 80% ACN (mindestens dreimal) gewaschen werden. Nach dem 80% ACN Waschschritte, sollten die Perlen einmal mit Wasser gewaschen werden, bevor sie on-bead tryptischen Verdaus. b) Western Blots mit Streptavidin-Meerrettich-Peroxidase und ECL Substrat kann auch verwendet werden, um erfolgreiche Vernetzung der Biotin enthält, CC, um die Proteine sichtbar zu machen. Also entweder das Gel nach Schritt 3.2 erhalten werden können abgetupft oder, weil die Empfindlichkeit ist etwa 10-fach höher als Silberfärbung von Gelen, 10. Mai ul-Proben nach Schritt 2.7 auch mittels Western Blot analysiert werden. Daran, dass im letzteren Fall endogen biotinylierte Proteine werden auch neben der Proteine künstlich durch die SAH-CC biotinylierten erkannt werden. c) Wir fanden, dass es auf dem speziellen System (Lysat, adressiert Zielproteine, Selektivität und Reaktivität Funktion er CC) abhängt, ob die "off-Bead"-Konfiguration, wo die Vernetzungsreaktion findet zwischen freien CC-und Protein in Lösung 4 oder die hier beschriebenen "on-bead"-Konfiguration (Abbildung 1B) zu besseren Ergebnissen führt.
Im Allgemeinen sollte die Methode auch kompatibel mit allen state-of-the art stabile Isotop Protein oder Peptid Etikettiertechnik oder Beurteilung der Capture Probe durch 2D-Gelelektrophorese. In der "off-Bead"-Konfiguration ist es auch möglich, Proteine innerhalb ganzer Zellen (unveröffentlichte Ergebnisse) zu erfassen. Darüber hinaus kann das Medikament oder Cofaktor-Bindungsstelle eines Proteins durch die Bestimmung der nahe Vernetzung Position des CC in der Proteinsequenz von MS Peptidsequenzierung skizziert werden. Die Art der Bindung eines kleinen Moleküls an ein Protein kann durch die Verwendung unterschiedlicher c erkundet werdenhemische Befestigung Positionen bei der Selektivität Funktion und verschiedene Linker Längen. Wie in der vorliegenden Studie gezeigt, adressiert auch Protein Bindungspartner der Proteine durch die Selektivität Funktion (RplK als Substrat PRMA) oder unbekannte kleines Molekül Protein-Interaktionen (SAH zu glnA) identifiziert werden können. Zusammengefasst, die zusätzliche Funktion des CCs, Foto-Vernetzungsreaktivität, ermöglicht die Isolierung und Identifizierung von niedrigen reichlich Proteine oder funktionelle Protein-Familien aus komplexen Protein-Gemischen mit hoher Empfindlichkeit und liefert den Wissenschaftlern ein zusätzliches Werkzeug für die Untersuchung kleiner Moleküle - Protein-Interaktionen .
Offenlegungen
Danksagungen
Diese Arbeit wurde von der Human Frontier Science Program Organization (HFSP Award 2007, RGP0058/2007-C) unterstützt. Wir danken Prof. Richard Roberts für die Initiierung des Projektes und für fruchtbare Diskussionen.
Materialien
| Name | Company | Catalog Number | Comments |
| SAH caproKit™ | caprotec bioanalytics GmbH | 1-1010-050 (50 reactions) 1-1010-010 (10 reactions) | Includes the SAH-CC, SAH competitor, streptavidin coated magnetic beads, capture buffer, and wash buffer |
| caproBox™ | caprotec bioanalytics GmbH | 1-5010-003 (110 V) 1-5010-004 (230 V) | For reproducible photo-activation while cooling the samples |
| caproMag™ | caprotec bioanalytics GmbH | 1-5100-001 | For easy handling of magnetic particles without pipetting |
Referenzen
- Katayama, H., Oda, Y. Chemical proteomics for drug discovery based on compound-immobilized affinity chromatography. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 855, 21-27 (2007).
- Barglow, K. T., Cravatt, B. F. Activity-based protein profiling for the functional annotation of enzymes. Nat. Methods. 4, 822-827 (2007).
- Koster, H., Little, D. P., Luan, P., Muller, R., Siddiqi, S. M., Marappan, S., Yip, P. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev. Technol. 5, 381-390 (2007).
- Dalhoff, C., Hueben, M., Lenz, T., Poot, P., Nordhoff, E., Koster, H., Weinhold, E. Synthesis of S-Adenosyl-L-homocysteine Capture Compounds for Selective Photoinduced Isolation of Methyltransferases. ChemBioChem. 11, 256-265 (2010).
- Lu, S. C. S-Adenosylmethionine. Int. J. Biochem. Cell. Biol. 32, 391-3952 (2000).
- Cantoni, G. L. Biological methylation: selected aspects. Annu. Rev. Biochem. 44, 435-451 (1975).
- Jeltsch, A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. ChemBioChem. 3, 274-293 (2002).
- Chow, C. S., Lamichhane, T. N., Mahto, S. K. Expanding the nucleotide repertoire of the ribosome with post-transcriptional modifications. ACS Chem. Biol. 2, 610-619 (2007).
- Grillo, M. A., Colombatto, S. S-Adenosylmethionine and protein methylation. Amino Acids. 28, 357-362 (2005).
- Fujioka, M. Mammalian small molecule methyltransferases: their structural and functional features. Int. J. Biochem. 24, 1917-1924 (1992).
- Borchardt, R. T., Salvatore, F., Borek, E., Zappia, V., Williams-Ashman, H. G., Schlenk, F. . The Biochemistry of S-Adenosylmethionine. , 151-171 (1977).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S-adenosyl-l-methionine. Biochemistry. 25, 4444-4449 (1986).
- Keller, A., Nesvizhskii, A. I., Kolker, E., Aebersold, R. Empirical statistical model to estimate the accuracy of peptide identifications made by MS/MS and database search. Anal. Chem. 74, 5383-5392 (2002).
- Nesvizhskii, A. I., Keller, A., Kolker, E., Aebersold, R. A statistical model for identifying proteins by tandem mass spectrometry. Anal. Chem. 75, 4646-4658 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten