Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Electron Microscopy sola partícula Reconstrucción del Complejo exosoma utilizando el método aleatorio inclinación cónica
En este artículo
Resumen
En este artículo se describe un método estándar para obtener una imagen tridimensional (3D) de la reconstrucción de macromoléculas biológicas mediante microscopía electrónica de tinción negativa (EM). En este protocolo, se explica cómo conseguir la estructura 3D del complejo exosoma Saccharomyces cerevisiae con una resolución media, sirviéndose del método al azar cónica reconstrucción de inclinación (ECA).
Resumen
Microscopía electrónica de una sola partícula (EM) de reconstrucción se ha convertido recientemente en una herramienta popular para conseguir las tres dimensiones (3D), estructura de grandes complejos macromoleculares. En comparación con cristalografía de rayos X, que tiene algunas ventajas únicas. En primer lugar, una sola partícula de EM reconstrucción no es necesario para cristalizar la muestra de proteína, que es el cuello de botella en la cristalografía de rayos X, especialmente para los grandes complejos macromoleculares. En segundo lugar, que no necesita grandes cantidades de muestras de proteínas. En comparación con los miligramos de proteínas necesarias para la cristalización, una sola partícula de reconstrucción EM sólo necesita varios micro-litros de solución de proteína en nano-molar concentraciones, utilizando el método de tinción negativa EM. Sin embargo, a pesar de algunas asambleas macromoleculares con alta simetría, sola partícula EM se limita a una resolución relativamente baja (inferior a una resolución de 1 nm) de muchos ejemplares, especialmente los que no tienen simetría. Esta técnica también es limitada por el tamaño de las moléculas bajo estudio, es decir, 100 kDa para los especímenes con tinción negativa y 300 kDa para congelados-hidratados muestras en general.
Para una nueva muestra de estructura desconocida, por lo general utilizan una solución de metales pesados para incorporar las moléculas de tinción negativa. La muestra se examina en un microscopio electrónico de transmisión para llevar dos dimensiones (2D) micrografías de las moléculas. Lo ideal sería que las moléculas de proteína tienen una estructura homogénea en 3D pero presentan diferentes orientaciones en las micrografías. Estas micrografías son digitalizadas y procesadas en los ordenadores como "partículas individuales". El uso de dos dimensiones y la alineación de las técnicas de clasificación, las moléculas homogéneas en el mismo punto de vista se agrupan en clases. Su promedio de mejorar la señal de formas 2D de la molécula. Después de asignar las partículas con la orientación adecuada relativa (ángulos de Euler), seremos capaces de reconstruir las imágenes de partículas en 2D en un volumen virtual en 3D.
En una sola partícula de reconstrucción en 3D, un paso esencial es el de asignar correctamente la orientación adecuada de cada partícula. Existen varios métodos para asignar el punto de vista de cada partícula, incluyendo la reconstitución angular y una inclinación al azar cónica (ECA) el método 2. En este protocolo, que describe nuestras prácticas para conseguir la reconstrucción en 3D del complejo de levadura exosoma con tinción negativa EM y ECA. Cabe señalar que el protocolo de la microscopía electrónica y procesamiento de imágenes sigue el principio básico de la ECA, pero no es la única manera de realizar el método. En primer lugar, describir la manera de integrar la muestra de proteína en una capa de uranilo-Formate con un espesor comparable al tamaño de la proteína, usando una rejilla de carbono perforado cubierto con una capa de película continua de carbono fino. A continuación, la muestra se inserta en un microscopio electrónico de transmisión para recoger Untilted (0 grados) e inclinada (55 grados) pares de micrografías que se utilizará más tarde para el procesamiento y la obtención de un modelo inicial en 3D de la exosoma levadura. Con este fin, llevamos a cabo ensayos clínicos aleatorios y luego refinar el modelo 3D inicial mediante el uso de la proyección correspondiente método de refinamiento 3.
Protocolo
1. Principio del método de inclinación cónica azar
- El principio del método de inclinación cónica requiere tomar al azar un par de micrografías de la misma región de la muestra dentro del microscopio electrónico. Se toma una fotografía de la muestra en una posición Untilted (fig. 1A) y la otra foto se toma de la muestra inclinada en un ángulo de entre 50 y 70 grados (en nuestro caso, utilizamos 55 grados). (Fig. 1B)
- Usar la computadora, la pareja micrografía digitalizados se pone al lado del otro y las imágenes de las mismas partículas que son seleccionados. (Figura 1 C)
- En coordenadas tridimensionales, las imágenes de partículas Untilted y sus socios inclinada se correlacionan entre sí por la dirección del eje de inclinación y el ángulo de inclinación. (Figura 1 D)
- La alineación de las imágenes de partículas Untilted trae las imágenes de las partículas de inclinar a su ubicación azimutal correspondiente. (Figura 1)
- El uso de imágenes múltiples de las partículas inclinada llenando el espacio azimutal, la estructura tridimensional de la molécula puede ser reconstruido utilizando un algoritmo de retroproyección. (Figura 1 F)
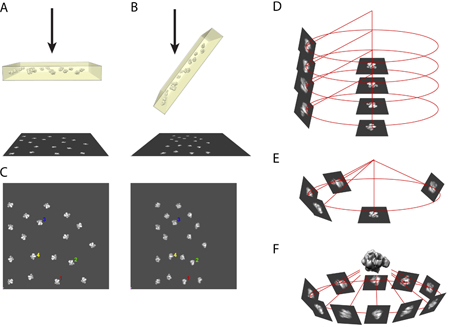
Figura 1. Una ilustración del principio de ECA reconstrucción.
2. Prepare las Rejillas de Holey de carbono cubierto con carbón fino
Justificación: Se utiliza el método de tinción negativa para fijar la muestra de proteína para la reconstrucción de azar inclinación cónica. A fin de preservar las macromoléculas sin demasiado aplanamiento durante el secado, tratamos de incorporar las moléculas de proteína en una mancha profunda con un espesor de la dimensión de las proteínas 4. En general, continua de carbono se utiliza en la toma de muestras con tinción negativa. Este tipo de carbono, sin embargo, es difícil de controlar el espesor de la mancha alrededor de las partículas de proteínas. De esta manera el uso casero rejillas perforadas de carbono cubierto con una fina capa de película de carbono (~ 5 nm de espesor) para hacer muestras con tinción negativa. Pequeños pozos formados por los agujeros permiten mantener la solución de proteína y la solución de mancha en la red por lo que es mucho más fácil de integrar la proteína en un espesor de la mancha óptima. Además, la fina capa de carbono sobre el agujero se reduce el ruido de fondo en gran medida.
- Prepare solución al 0,5% Formvar. En la vitrina, añadir 0,45 g de resina de polivinilo formal y 90 ml de cloroformo en un vaso de vidrio de 100 ml. Use papel de aluminio para cubrir el vaso, y el uso de una barra de agitación pequeños para ayudar a disolver la resina formal sobre un agitador magnético. Se tarda unos 15 minutos para disolver la resina.
- Durante la disolución de la Formvar, vidrio limpio microscopía diapositivas en metanol y secar con Kimwipes.
- Después de la resina Formvar se ha disuelto en cloroformo, se añade 1 ml de glicerol al 50% de la superficie de la solución. Ajustar el volumen del glicerol añadido afecta a la densidad de los agujeros en el carbono perforadas. Moje la punta de un ultrasonicador en la solución a una profundidad de alrededor de 1 pulgada y el uso de ultrasonidos de potencia máxima a 1 minuto para que la emulsión de gotas de glicerol en una solución de Formvar. La solución se vuelve lechosa después de este paso. Ya sonicación causas de menor tamaño de los agujeros en el carbono perforadas.
- Inmediatamente después del tratamiento con ultrasonidos, sumerja el portaobjetos de vidrio limpia verticalmente en la emulsión de 1 segundo, se los saca, y borra el fondo de las diapositivas utilizando papel de filtro para formar una película delgada de plástico sobre la superficie de las láminas. Después se evapora el cloroformo, comprobar la densidad y el tamaño de los orificios de la película bajo un microscopio de luz. Ajustar la condición de preparación de acuerdo con las necesidades. Con la condición descrita aquí, por lo general obtener los agujeros con un diámetro de 3 a 4 micro-metros y 10 a 20 agujeros en cada cuadrado de una malla 400.
- Después de que el portaobjetos de vidrio se seca, cortar el borde de la lámina de plástico sobre la superficie de deslizamiento. Flotar la película fuera en la superficie de agua destilada. La película delgada sobre la superficie del agua se puede observar en un ángulo inclinado en contra de la reflexión de la luz. Lugar 400 de malla de las redes de cobre en la película, uno por uno, con la superficie de las redes de 'suave hacia abajo.
- Recoger la lámina de plástico con rejillas en él con un trozo de papel. Da la vuelta al papel y deje que se seque en una placa de Petri. Empapar el papel en metanol para eliminar el glicerol residual en los agujeros y que el papel se seque al aire.
- Escudo de las rejillas con una capa de carbono con un espesor de aproximadamente 20 nanómetros en un evaporador de carbono. El espesor puede ser determinado por el color gris del carbono.
- Remoje las redes con el carbono en cloroformo durante media hora para eliminar el Formvar. Después de las rejillas se secan, se han obtenido de fabricación casera rejillas perforadas de carbono.
- Evaporar fina capa de carbono, con unos 5 nanómetros de espesor sobre una superficie de mica recién cortada.
- Con cuidado, poner la malla de carbono perforado por debajo de agua destilada. Flotar el carbón fino de la superficie de la mica en la superficie del agua y depositarlo en el carbono perforadasredes poco a poco. Seco de las rejillas en una campana extractora.
3. La tinción negativa del Complejo exosoma
Justificación: Hay muy pocas soluciones de metales pesados manchas que se pueden utilizar para EM tinción negativa, como acetato de uranilo, formato de uranilo, ácido fosfotúngstico, molibdato de amonio, entre otros. Solución de tinción diferentes tiene diferentes propiedades únicas. Por ejemplo, el acetato de uranilo y ofrece un alto contraste de la partícula, pero se puede bloquear complejos de proteínas que no les gusta el ambiente ácido. Para aquellas muestras que, el ácido phosphotungestic a pH neutro puede ser una solución buena mancha. Elegimos formiato de uranilo saturado (UF) solución debido a su granularidad fina y de alta capacidad de penetración en las moléculas.
- Hervir el agua destilada durante 1 min. Enfriar lentamente a temperatura ambiente. Este paso es para eliminar el oxígeno disuelto en el agua.
- Hacer nuevas formiato de uranilo 2% (UF) solución. Mezclar 1 ml de agua y 20 UF mg en un tubo de 1,5 ml. Vortex durante 10 minutos.
- Ajustar el valor de pH de 5,0 mediante la adición de 2 microlitros de 10 M de hidróxido de potasio. Mezclar inmediatamente. El color de la solución debe ser más amarillo. El pH de la solución no debe ser demasiado alto, de lo contrario la mancha precipitados.
- Coloque el tubo de vórtice durante otros 10 minutos.
- Haga girar la solución en una centrífuga de sobremesa a la máxima velocidad durante 10 minutos.
- Filtrar la solución a través de una membrana de 0,2 micrómetros PVDF. Esta es la solución UF fresco. Cubra el tubo de la solución en un pedazo de papel de aluminio para evitar que la luz. La solución tiene que ser utilizado en el mismo día.
- Descarga luminiscente una fina de carbono-en-red llena de agujeros de carbono usando un aparato de luz de descarga de 30 segundos a 25 mA.
- Ponga un pedazo de parafina limpia en el banquillo. Ponga 3 gotas de 50 microlitros de solución UF mancha en la parte superior de la parafina.
- Diluir el complejo exosoma a una concentración de 50 ~ 100 nm utilizando el tampón de dilución (25 mM Tris-HCl pH 7,5, 100 mM NaCl, 2 mM DTT). Poner 4 microlitros de la proteína diluida en el que brillan alta red. Deje que la muestra de estancia en la parrilla de un minuto. (Nota:. Tal concentración final de las moléculas en general, da una densidad óptima de bien de partículas dispersas en la parrilla de tinción negativa para formato de uranilo o solución de uranilo acecate mancha, el fosfato o alta concentración de sal (más de 0,5 M) en general es no es bueno para obtener buenos resultados manchas. Nuestra experiencia sugiere que Hepes o canalizaciones funcionan bien con las soluciones de uranilo mancha.)
- Use un pedazo de papel de filtro para secar la solución residual desde el borde de la parrilla y voltear la red inmediatamente en la parte superior de las gotas de la mancha y lave la parrilla durante unos 10 segundos en cada gota.
- Después del último aclarado, que la estancia mancha en la red de otro 1 min y luego borrar la mancha de distancia por un pedazo de papel de filtro. Mantenga una capa delgada de solución de mancha en la superficie de la rejilla con el fin de conseguir un buen resultados de la tinción de profundidad. Deje que la parrilla se secan rápidamente en una campana extractora.
4. Microscopía Electrónica del Complejo exosoma
Justificación: Cualquier microscopio electrónico de transmisión con una etapa de inclinación puede ser usado para recolectar pares de inclinación de la muestra para el ensayo clínico de reconstrucción. En teoría, el ángulo superior de la muestra se puede inclinar para recopilar los datos, mejor. En la práctica, debido al diseño del soporte de la muestra y la geometría de la red, el ángulo máximo es operable limitada de 50 a 70 grados. En este protocolo, sólo describimos nuestro procedimiento a través de un FEI Tecnai-12 microscopio electrónico. Para los otros modelos de microscopios, las operaciones deben ser ajustados de acuerdo a los requisitos del proyecto y la propiedad del instrumento.
- Coloque la rejilla de la muestra en el portamuestras y luego poner el soporte en un microscopio de electrones de FEI Tecnai -12. El microscopio es operado a 120 kV. Usamos Gatan Ultrascan4000 cámara CCD para tomar fotos. Asegúrese de que el 'Flip alrededor del eje vertical "en el diálogo' cámara de configuración" de la interfaz digital Micrografía está marcada para asegurar la imparcialidad determinación correcta. (Nota:. Esto es importante especialmente si el lector se basa en SPIDER ECA procedimientos de reconstrucción de un modelo 3D)
- Compruebe la red muestra a bajo aumento para encontrar las mejores plazas de colores. Este tipo de plazas debe tener una docena de agujeros con una dimensión de entre 1 y 2 micrómetros y áreas oscuras manchas en ellos. (Figura 2)

Figura 2. Una micrografía de bajo aumento de una cuadrícula que muestra cuadros con manchas de buenos y malos. - Activar el modo de baja dosis de la interfaz de usuario FEI y alinear la búsqueda, el enfoque y la posición en el modo de exponer a dosis bajas. Usamos aumentos de 150.000 para el enfoque, la exposición de 52.000 y 1,5 metros de longitud en la cámara de difracción para la búsqueda. El tiempo de exposición se ajusta a 1 segundo. Establecer la posición de enfoque 2 micrómetros de distancia desde el punto de venta la exposiciónition a lo largo del eje de inclinación.
- Buscar los orificios con manchas bien en modo de búsqueda, y guardar las ubicaciones. Los agujeros suelen tener un buen gradiente de mancha oscura en la que se observa bajo el modo de búsqueda. Tome una fotografía CCD de la plaza. Inclinación de la muestra a 55 grados, tome otra foto. Compare las dos imágenes, identificar los agujeros en los dos pares de microfotografías. (Figura 3)
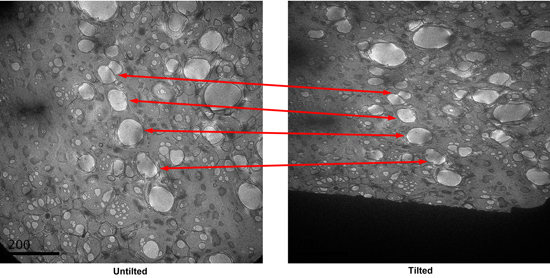
Figura 3. Un par de inclinación de micrografías de una plaza en el modo de búsqueda. Correspondientes agujeros vinculados se indican. - La inclinación de la fase de vuelta a 0 grados. Utilizando el kit de baja dosis para tomar imágenes de cada agujero identificado en el modo de búsqueda a gran aumento. El desenfoque es del orden del -0,7 micrómetros.
- Después de todos los agujeros se han tomado fotos de, la inclinación de la etapa a 55 grados. Tomar microfotografías de la muestra inclinada con el mismo aumento que los Untilted con un desenfoque de alrededor de -1,2 micrómetros. Identificar los pares correspondientes inclinada de microfotografías sobre la base de los patrones en las micrografías de bajo aumento. El patrón irregular de las redes de fabricación casera llena de agujeros de carbono ayuda a la correlación. Examinar pares de inclinación de micrografías y eliminar las micrografías con manchas mal como superficiales las zonas manchadas (las partículas parecen tener un halo alrededor de ellos en condiciones inclinada). (Figura 4)
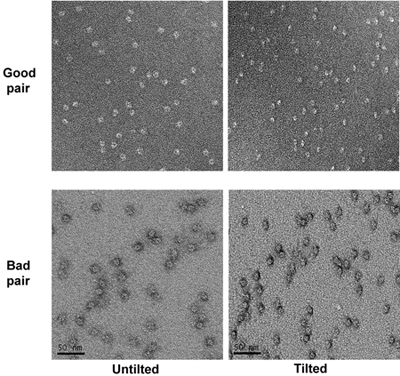
Figura 4. Dos pares de inclinación de micrografías de la muestra a gran aumento. Las micrografías bueno y lo malo están marcados.
5. Procesamiento de imágenes de los Datos
Justificación: Existen diferentes opciones y paquetes de software para llevar a cabo la reconstrucción de ECA en el ordenador. El más comúnmente utilizado es SPIDER 5. Un protocolo básico para realizar RCT en SPIDER se puede encontrar en la página web http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Un protocolo detallado para realizar RCT en SPIDER se describe en el artículo de Shaikh et al. 6 En nuestro protocolo, se utiliza una combinación de Imagic-5 7 y SPIDER en la versión en video del protocolo. También ofrecemos un procedimiento alternativo para el uso exclusivo SPIDER en la versión de texto del protocolo.
- Configuración de los programas. Usamos la proc2d en el paquete de 8 EMAN para cambiar el formato de imagen de Gatan imagen digital al formato de imagen de la araña. Imagic-5 se utiliza para hacer la alineación de 2D. SPIDER se utiliza para hacer la reconstrucción 3D y refinamiento.
Subsección 1: Escoger pares de inclinación de las partículas.
- Convertir los archivos *. dm3 Gatan imagen digital de formato de imagen de SPIDER con el comando proc2d en EMAN. Los pares inclinados y Untilted se nombran en el patrón como *** *** t.spi y u.spi, respectivamente.
- Elige los pares de partículas mediante el programa 'WEB' distribuidos en el paquete del programa SPIDER. Siga las instrucciones en el Las coordenadas son automaticamente guardados en dcu ***. spi y dct ***. spi de partículas Untilted e inclinado respectivamente. Otro documento dcb ***. spi contiene la información de ángulo de inclinación entre las partículas inclinada y Untilted (Nota:. Los tres ángulos durante el ajuste en la web entre los pares de inclinación no garantizan la imparcialidad correcto de la reconstrucción final, ya que theta no tiene la signo (valor positivo) y phi gamma y se basan en los valores iniciales establecidos para ellos. Examen de la dirección del gradiente de desenfoque en la micrografía inclinada podrían ayudar a establecer los valores correctos inicial de la phi y la gamma antes de la instalación con el fin de obtener una correcta imparcialidad de la figura de reconstrucción. 5 ilustra la convención correcta.)
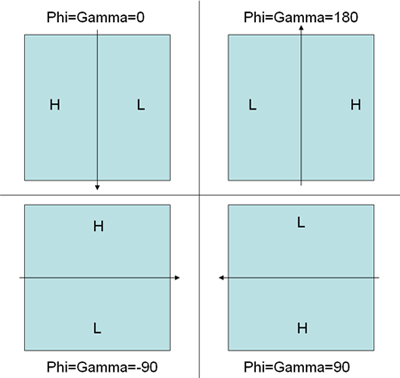
Figura 5. Un ejemplo para explicar la convención de la determinación del ángulo de inclinación WEB pares de partículas elegir el modo de ajuste determinado por el gradiente de desenfoque. H representa gran desenfoque zona, mientras que L representa bajo desenfoque área. La flecha representa el eje de inclinación. Los correspondientes ángulos correctos inicial de PHI y GAMMA se indican para cada esquema. - Caja de todas las partículas recogido utilizando una versión modificada del script SPIDER como se muestra en la página web http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Guión ahorra las pilas de partículas Untilted e inclinada como u.spi y t.spi. El número de micrografía donde las partículas son de debe guardarse en particle_list.spi. (Nota: Esto es muy importante para la generación de los archivos correcto ángulo de EulerECA).
Subsección 2: en dos dimensiones y la alineación de la clasificación de imágenes de partículas Untilted.
- Convertir las partículas Untilted en Imagic-5 formato mediante el programa em2em en Imagic-5 paquetes. Alinear y clasificar a las partículas en clases homogéneas iterativa utilizando el Imagic-5 programas (Anexo A). Con el comando msa-nombres de su clase en Imagic-5 para generar las clases búsqueda en la tabla de las partículas, lo que nos ahorramos como imagic_classes.lis. (Nota:. La clasificación y la alineación es aumentar el número de partículas con la misma forma, así como reducir la variación en una clase de mapa de la varianza de cada clase puede proporcionar información sobre la calidad de la clase.)
- Generar un archivo de trazado (ali_50.plt en el video de demostración) para los valores de traslación y rotación de la alineación de cada partícula con el comando encabezado en Imagic-5.
- Convertir las clases de tabla de consulta en los archivos de SPIDER documento base_file ***. spi usando un script en perl lis2spi.pl distribuidos en http://cryoem.berkeley.edu .
- Convertir los valores de traslación y rotación de la alineación de cada partícula del archivo de trazado generado para la traducción y los valores de la rotación en el paso 5.6 en ali_50.spi SPIDER archivo de documentos mediante un guión plt2spi.pl distribuidos en http://cryoem.berkeley.edu .
Usamos Imagic-5 para la alineación de 2D y la clasificación, ya que da el mejor rendimiento en esta tarea en nuestras manos. Estrategia alternativa de SPIDER para la alineación de dos dimensiones y la clasificación se puede encontrar en http://www.wadsworth.org/spider_doc/spider/docs/align.html . También hemos utilizado SPIDER para realizar el análisis en 2D en las partículas de la Untilted exosoma. A continuación se presenta un procedimiento simple.
Alternativa 5.5) Use la alineación de referencia libre tal como se describe en el alinear las imágenes. Dos guiones simples se pueden encontrar Guardar la rotación y cambio de todas las partículas en un angular_file.spi archivo del documento.
Alternativa 5.6) Clasificar las partículas alineadas en grupos con el mismo punto de vista como se describe en Hemos utilizado el método K-means para la clasificación. Generar base_file ***. spi sobre la base de la clasificación.
Subsección 3: Reconstrucción tridimensional usando las imágenes de partículas inclinada.
- Filtro pasa banda, la máscara y el centro de las partículas inclinada al igual que para las partículas en Untilted Imagic-5. Generar un nuevo conjunto de datos para las partículas título. (Nota: Este paso es opcional que se puede hacer en SPIDER..)
- Crear archivos anglular documento de las clases look-up table documentos ali_50.spi, el DCB ***. archivos spi generado por web en el paso 5.3 y la lista de partículas partile_list.spi archivo desde el paso 5.4 mediante el script de araña como en el Apéndice B.
- Usar secuencias de comandos de reconstrucción de SPIDER para hacer la reconstrucción de cada una de ellas como se describe en http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Cada clase de partículas contribuye a un volumen de reconstrucción en 3D. Los modelos en 3D pueden ser examinados en la UCSF Chimera-9. Una proyección en dos dimensiones del modelo 3D en ángulo de Euler (0,0,0) se puede comparar con el promedio de la clase correspondiente en 2D de las partículas Untilted para verificar la calidad de la reconstrucción. Buscar volúmenes similares, alinear y combinar para generar volúmenes iniciales siguiendo el procedimiento http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Subsección 4: Perfeccionamiento de la reconstrucción en 3D con imágenes Untilted de partículas.
- ¿Es el refinamiento de proyección correspondiente del volumen combinado inicial en contra de todas las partículas Untilted para obtener una mayor resolución volume cono sin falta y el artefacto aplanar el uso de scripts SPIDER como se describe en http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Los resultados representativos:
Utilizando el método de ensayo clínico, se han obtenido alrededor de 50 reconstrucciones de la exosoma de un total de 5.000 pares de inclinación (Figura 6). De los 50 modelos 3D, podemos ver las distintas orientaciones de la sesión compleja en la parrilla con principalmente dos vistas ortogonales. Un artefacto de aplastamiento es detectable en muchos de los volúmenes en la dirección perpendicular a la superficie del carbón. Se realizó la alineación y fusión de los volúmenes 3D para generar dos primeros volúmenes de las vistas ortogonales. Usando el 5000 imágenes de partículas Untilted, hemos obtenido el mismo final de la reconstrucción en 3D de la exosoma a unos 18 Angstrom de resolución de los dos modelos iniciales (Figura 7). La estructura revela la arquitectura de la exosoma levadura y proporcionó información sobre la vía de la contratación de ARN sustrato 10.

Figura 6. 50 modelos 3D del complejo exosoma por ECA reconstrucción.
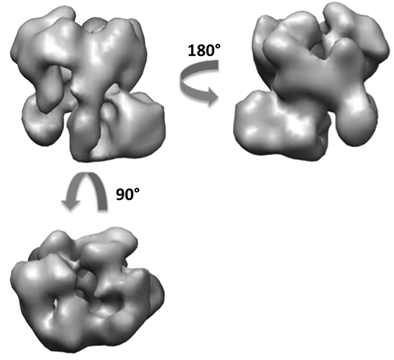
Figura 7. Reconstrucción en 3D del complejo exosoma después de refinamiento.
Apéndice:
Apéndice A. El archivo de comandos para la alineación de 2D y la clasificación en Imagic-5.
Archivo: auto_align_i.sh
Haga clic aquí para archivo
Apéndice B. El archivo de comandos para generar archivos angular para la reconstrucción 3D de SPIDER.
Archivo: generate_angular_file.spi
Haga clic aquí para archivo
Discusión
En este artículo se presenta un protocolo detallado de preparación de muestras y reconstrucción tridimensional del complejo exosoma mediante microscopía electrónica de tinción negativa. Usando este método, se obtuvo la reconstrucción en 3D utilizando el método aleatorio inclinación cónica sin ningún conocimiento previo de la estructura. Método aleatorio inclinación cónica no requiere necesariamente de una muestra homogénea, pero la proyección a juego siguientes refinamiento paso necesitaría una muestra...
Divulgaciones
Agradecimientos
Los autores desean agradecer a los miembros de Nogales laboratorio de la Universidad de California-Berkeley en ayudar a establecer los protocolos iniciales y los miembros del laboratorio de Wang en la Universidad de Yale en su ayuda para establecer los protocolos completos. Reconocemos también el personal de la crio-ME de instalaciones y de Alto Rendimiento Centro de Cómputos de la Facultad de Medicina de Yale por su apoyo. HW Smith es una familia Adjudicatario.
Materiales
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Referencias
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados