Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Simple reconstruction microscopie électronique de particules du complexe Exosome Utilisation de la méthode aléatoire Tilt conique
Dans cet article
Résumé
Cet article décrit une méthode standard pour obtenir un en trois dimensions (3D) la reconstruction des macromolécules biologiques en utilisant la microscopie électronique négative coloration (EM). Dans ce protocole, nous expliquons comment obtenir la structure 3D du complexe Saccharomyces cerevisiae exosomes à moyenne résolution utilisant la méthode aléatoire coniques de reconstruction d'inclinaison (ECR).
Résumé
Simple microscopie électronique de particules (EM) la reconstruction est récemment devenu un outil populaire pour obtenir les trois dimensions (3D) la structure des complexes macromoléculaires grande. Comparé à cristallographie aux rayons X, il a quelques avantages uniques. Tout d'abord, seule la reconstruction de particules EM n'a pas besoin de cristalliser l'échantillon de protéine, qui est le goulot d'étranglement dans cristallographie aux rayons X, notamment pour les grands complexes macromoléculaires. Deuxièmement, il n'a pas besoin de grandes quantités d'échantillons de protéines. Comparativement aux milligrammes de protéines nécessaires à la cristallisation, la seule particule EM besoins de reconstruction que plusieurs micro-litres de solution de protéine à la nano-concentrations molaires, en utilisant la méthode de coloration négative EM. Cependant, malgré un peu d'assemblages macromoléculaires à symétrie élevée, seule particule EM est limité à une résolution relativement faible (inférieure à une résolution de 1 nm) pour de nombreux spécimens en particulier ceux sans symétrie. Cette technique est également limitée par la taille des molécules à l'étude, soit 100 kDa pour les spécimens colorés négativement et 300 kDa pour frozen-hydraté spécimens en général.
Pour un nouvel échantillon de structure inconnue, on utilise généralement une solution de métal lourd à intégrer les molécules par coloration négative. L'échantillon est ensuite examiné au microscope électronique à transmission à prendre en deux dimensions (2D) micrographies des molécules. Idéalement, les molécules de protéines ont une structure homogène 3D mais présentent des orientations différentes dans les micrographies. Ces micrographies sont numérisées et traitées dans les ordinateurs comme des «particules uniques". En utilisant deux dimensions d'alignement et les techniques de classification, les molécules homogènes dans les mêmes vues sont regroupées en classes. Leurs moyennes améliorer le signal de formes 2D de la molécule. Après nous assignons les particules dans le bon sens relatif (angles d'Euler), nous serons en mesure de reconstruire les images 2D de particules dans un volume virtuel en 3D.
Dans la reconstruction de particules 3D unique, une étape essentielle est d'assigner correctement l'orientation correcte de chaque particule unique. Il existe plusieurs méthodes pour attribuer le point de vue pour chaque particule, dont la reconstitution angulaire 1 et aléatoire d'inclinaison conique (ECR) la méthode 2. Dans ce protocole, nous décrivons notre pratique à obtenir la reconstruction 3D du complexe de la levure à l'aide exosomes coloration négative EM et ECR. Il convient de noter que notre protocole de la microscopie électronique et de traitement de l'image suit le principe de base du RCT, mais n'est pas la seule façon d'exécuter la méthode. Nous décrivons d'abord comment intégrer l'échantillon de protéine dans une couche d'uranyle-Formiate d'une épaisseur comparable à la taille des protéines, en utilisant une grille de carbone troué recouvert d'une couche de film continu de carbone mince. Puis l'échantillon est inséré dans un microscope électronique à transmission à recueillir Untilted (0 degré) et incliné (55 degrés) paires de micrographies qui sera utilisé plus tard pour le traitement et l'obtention d'un premier modèle 3D de la exosomes levure. À cette fin, nous effectuons RCT et ensuite raffiner le modèle initial en 3D en utilisant la projection correspondant 3 la méthode de raffinement.
Protocole
1. Principe de la méthode aléatoire Tilt conique
- Le principe de la méthode d'inclinaison coniques aléatoires nécessite de prendre une paire de micrographies de la même région de l'échantillon à l'intérieur du microscope électronique. Une photo est prise de l'échantillon dans une position non incliné (figure 1A) et l'autre photo est prise de l'échantillon incliné à un angle de 50 à 70 degrés (dans notre cas, nous utilisons 55 degrés). (Figure 1B)
- Utilisation de l'ordinateur, la paire de micrographie numérisée est mis côte à côte et des images à partir des mêmes particules sont sélectionnées. (Figure 1C)
- En coordonnées tridimensionnelles, les images de particules non incliné et leurs partenaires inclinée sont corrélés les uns aux autres par la direction de l'axe d'inclinaison et l'angle d'inclinaison. (Figure 1D)
- L'alignement des images de particules non incliné apporte des images de particules inclinée à leurs correspondants endroits azimutale. (Figure 1E)
- En utilisant plusieurs images de particules inclinée remplissant l'espace azimutale, la structure tridimensionnelle de la molécule peut être reconstruit en utilisant un algorithme de back-projection. (Figure 1F)
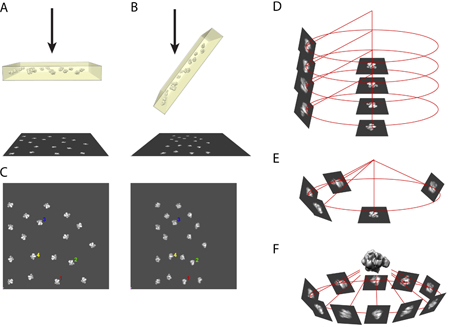
Figure 1. Une illustration du principe d'ECR de reconstruction.
2. Préparer les grilles de trous de carbone recouverte de carbone mince
Justification: Nous utilisons la méthode de coloration négative de fixer l'échantillon de protéines pour la reconstruction d'inclinaison aléatoire conique. Afin de préserver les macromolécules sans trop aplatir pendant le séchage, nous essayons d'intégrer les molécules de protéines dans une tache profonde avec une épaisseur à la dimension des protéines 4. En général, le carbone continue est utilisé dans la fabrication des échantillons colorés négativement. Ce genre de carbone, cependant, il est difficile de contrôler l'épaisseur tache autour des particules de protéines. Nous utilisons donc fait maison grilles de carbone troué recouvert d'une fine couche de film de carbone (épaisseur ~ nm 5) de faire des échantillons colorés négativement. Petits puits formé par les trous permettent à retenir la solution de protéines et la solution tache sur la grille de sorte qu'il est beaucoup plus facile d'intégrer des protéines dans une épaisseur optimale tache. Par ailleurs, la mince couche de carbone sur le trou réduit le bruit de fond considérablement.
- Préparer la solution Formvar 0,5%. Dans la hotte, ajouter 0,45 g de résine de vinyle formelle et 90 ml de chloroforme dans un bécher de verre de 100 ml. Utiliser du papier d'aluminium pour couvrir le bécher, et utiliser un agitateur pour aider les petits dissolution de la résine formelle sur un agitateur magnétique. Il faut environ 15 minutes pour dissoudre la résine.
- Lors de la dissolution de l'Formvar, verre microscopique propres diapositives dans le méthanol et essuyez avec Kimwipes.
- Après la résine Formvar est entièrement dissous dans du chloroforme, ajouter 1 ml de glycérol 50% de la surface de la solution. Réglage du volume de la glycérine ajoutée affecte la densité des trous sur le carbone à trous. Trempez la pointe d'un ultrasonicator dans la solution à environ 1 cm de profondeur et d'utiliser la puissance maximale à soniquer 1 min pour faire émulsion de gouttelettes de glycérol en solution Formvar. La solution devient laiteuse après cette étape. Plus sonication entraîne plus petite taille des trous sur le carbone à trous.
- Immédiatement après la sonication, tremper les lames de verre nettoyé à la verticale dans l'émulsion pendant 1 seconde, les sortir, et éponger les fonds des diapositives en utilisant un papier filtre pour former un mince film plastique sur la surface des lames. Après l'évaporation choloroform, vérifier la densité et la taille des trous dans le film sous un microscope optique. Ajuster la condition de préparation selon les besoins. Avec la condition décrite ici, nous obtenons généralement des trous d'un diamètre de 3 ~ 4 micro-mètres et de 10 ~ 20 trous dans chaque carré d'une grille de 400 mesh.
- Après les lames de verre sont à sec, couper le bord du film plastique sur la surface de la lame. Float le film hors de la surface de l'eau distillée. Le film mince sur la surface de l'eau peut être observé à un angle de Bragg contre la réflexion de la lumière. Placer des grilles en cuivre de 400 mesh sur la pellicule, un par un, avec les grilles "surface lisse vers le bas.
- Ramassez le film plastique avec des grilles sur l'aide d'un morceau de papier. Retournez le papier et laisser sécher dans un boîte de Pétri. Faire tremper le papier dans le méthanol pour enlever la glycérine résiduelle dans les trous et laissez le papier sécher à l'air.
- Enduire les grilles d'une couche de carbone avec l'épaisseur de ~ 20 nanomètres dans un évaporateur de carbone. L'épaisseur peut être déterminée par la couleur grise du carbone.
- Faire tremper les grilles avec du carbone dans le chloroforme pendant une demi-heure pour enlever le Formvar. Après les grilles sont séchées, nous avons obtenu home-made grilles de carbone à trous.
- Evaporer mince couche de carbone avec environ 5 nanomètres d'épaisseur sur la surface de mica fraîchement clivé.
- Soigneusement mis les grilles de carbone holey sous l'eau distillée. Flotteur du carbone mince de la surface de mica à la surface de l'eau et le déposer sur le carbone holeygrilles lentement. Sécher les grilles dans une hotte.
3. Coloration négative du complexe Exosome
Justification: Il existe assez peu de solutions de métaux lourds tache qui peut être utilisé pour EM coloration négative, y compris l'acétate d'uranyle, le formiate d'uranyle, l'acide phosphotungstique, molybdate d'ammonium et d'autres. Différentes solutions tache a différentes propriétés uniques. Par exemple, l'acétate d'uranyle offre un contraste élevé de la particule, mais peut se bloquer complexes protéiques qui n'aiment pas l'environnement acide. Pour ces échantillons, l'acide phosphotungestic à pH neutre peut être une bonne solution tache. Nous choisissons Formiate uranyle saturés (UF) en raison de sa solution de granularité fine et la capacité de pénétration élevé dans les molécules.
- Faites bouillir de l'eau distillée pendant 1 min. Refroidir lentement à température ambiante. Cette étape est d'éliminer l'oxygène dissous dans l'eau.
- Faire frais Formiate uranyle 2% (UF) solution. Mélanger 1 ml d'eau et 20 mg UF dans un tube de 1,5 ml. Vortex pendant 10 min.
- Ajuster le pH à 5,0 par addition de 2 microlitre de 10 M d'hydroxyde de potassium. Mélanger immédiatement. La couleur de la solution devrait être plus jaune. Le pH de la solution ne doit pas être trop élevée, sinon la tache précipités.
- Placer le tube sur le Vortexer pendant 10 minutes supplémentaires.
- Faites tourner la solution sur une centrifugeuse de bureau à la vitesse maximale pendant 10 min.
- Filtrer la solution à travers une membrane de PVDF 0,2 micromètre. C'est la solution UF frais. Couvrir le tube de solution dans un morceau de papier d'aluminium pour empêcher la lumière. La solution doit être utilisée dans la même journée.
- Une décharge luminescente mince de carbone-sur-holey grille de carbone en utilisant un appareil à décharge luminescente pendant 30 secondes à 25 mA de courant.
- Placer un morceau de parafilm nettoyage sur le banc. Mettez 3 gouttes de solution de 50 microlitre UF tache sur le dessus du parafilm.
- Diluer le complexe exosomes à une concentration de 50 ~ 100 nm en utilisant le tampon de dilution (25 mM Tris-HCl pH 7,5, NaCl 100 mM, 2 mM DTT). Mettez 4 microlitre de la protéine diluée sur la lueur déchargée grille. Laisser l'échantillon rester sur la grille pendant une minute. (Remarque:. Une telle concentration finale de molécules donne généralement une densité optimale de bien-particules dispersées sur la grille négativement colorées pour le formiate d'uranyle ou de solution acecate uranyle tache, le phosphate ou une forte concentration de sel (plus de 0,5 M) est en général pas bon pour obtenir des résultats coloration bien. Notre expérience suggère que Hepes ou des tuyaux de bien travailler avec les solutions uranyle tache.)
- Utilisez un morceau de papier filtre pour éponger la solution résiduelle à partir du bord de la grille et retournez le quadrillage immédiatement au-dessus des gouttelettes tache et rincez la grille pendant environ 10 secondes sur chaque gouttelette.
- Après le dernier rinçage, laissez le séjour tache sur la grille pour un autre 1 min puis tamponnez la tache de suite par un morceau de papier filtre. Gardez une fine couche de solution de tache sur la surface de la grille afin d'obtenir une bonne profondeur des résultats tache. Laissez sécher rapidement la grille dans une hotte.
4. Electron Microscopy du Complexe Exosome
Justification: Toute microscope électronique à transmission avec une scène inclinable peut être utilisé pour collecter les paires d'inclinaison de l'échantillon pour la reconstruction RCT. En théorie, l'angle supérieur de l'échantillon peut être incliné pour recueillir des données, le meilleur. Dans la pratique, en raison de la conception du porte-échantillon et la géométrie de la grille, l'angle maximal utilisable est limitée de 50 à 70 degrés. Dans ce protocole, nous décrivons seulement notre procédure en utilisant un microscope FEI Tecnai-12 électrons. Pour les autres modèles de microscopes, les opérations doivent être ajustés en fonction de l'exigence du projet et la propriété de l'instrument.
- Mettez la grille de l'échantillon dans le porte-échantillon et puis mettez le support dans un microscope électronique FEI Tecnai -12. Le microscope est exploitée à 120 kV. Nous utilisons Gatan Ultrascan4000 caméra CCD pour prendre des photos. Assurez-vous que le 'Flip autour de l'axe vertical »dans le dialogue de la« caméra config' du Micrograph Interface numérique n'est pas cochée pour garantir la détermination correcte impartialité. (Remarque:. Ce qui est important surtout si le lecteur s'appuie sur des procédures de reconstruction SPIDER RCT pour obtenir un modèle 3D)
- Vérifiez la grille de l'échantillon à faible grossissement pour trouver la meilleure carrés colorés. Ce genre de carrés devrait avoir une douzaine de trous de dimension d'environ 1 ~ 2 micromètres et les endroits sombres et coloration en eux. (Figure 2)

Figure 2. Une micrographie faible grossissement d'une grille montrant carrés avec des taches de bonnes et mauvaises. - Activez le mode faible dose de l'interface utilisateur FEI et d'aligner la recherche, mise au point et d'exposer la position en mode à faible dose. Nous utilisons des grossissements de 150 000 pour se concentrer, 52000 pour l'exposition et de 1,5 mètre de longueur de la caméra en diffraction pour la recherche. Le temps d'exposition est ajustée à 1 seconde. Réglez la position du foyer 2 micromètres loin du POS d'expositionition long de l'axe d'inclinaison.
- Trouvez les trous avec une bonne coloration en mode de recherche, et enregistrer les emplacements. Les trous ont généralement une bonne nuit de gradient tache dans les observe sous le mode de recherche. Prenez une photo CCD de la place. Inclinez le spécimen à 55 degrés, prendre une autre photo. Comparez les deux photos, d'identifier les trous jumelés dans les deux micrographies. (Figure 3)
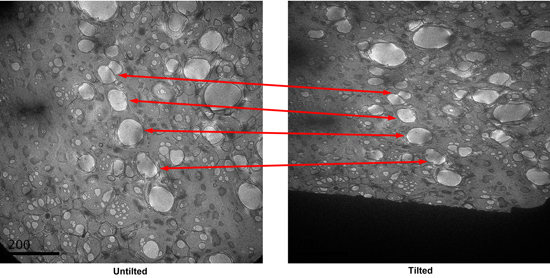
Figure 3. Une paire d'inclinaison du microscope d'un carré en mode de recherche. Correspondant trous jumelés sont indiqués. - Inclinez le stade de retour à 0 degré. En utilisant le kit de faibles doses de prendre des photos de chaque trou identifiés dans le mode de recherche à fort grossissement. La défocalisation utilisée est d'environ -0,7 micromètre.
- Après tous les trous ont été prises des photos de, inclinez le stade à 55 degrés. Prenez micrographies de l'éprouvette inclinée au même grossissement que ceux Untilted avec un flou d'environ -1,2 micromètre. Identifier les paires correspondantes de micrographies inclinée sur la base des tendances dans les micrographies faible grossissement. L'irrégularité des grilles faites maison de carbone holey aide de la corrélation. Examiner les paires d'inclinaison du microscope et de supprimer les micrographies avec des taches de mauvaises telles que les zones peu colorées (particules semblent avoir un halo autour d'eux sous condition incliné). (Figure 4)
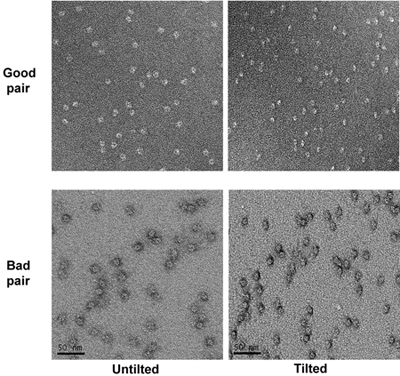
Figure 4. Deux paires d'inclinaison du microscope de l'échantillon à fort grossissement. Les micrographies bons et les mauvais sont marqués.
5. Traitement de l'image des données
Justification: Il existe différentes options et des logiciels pour effectuer la reconstruction RCT dans l'ordinateur. Le plus généralement utilisé est SPIDER 5. Un protocole de base pour effectuer RCT dans SPIDER peut être trouvée dans la page web http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Un protocole détaillé pour effectuer RCT dans SPIDER est décrite dans l'article de Shaikh et al. 6 Dans notre protocole, nous utilisons une combinaison de IMAGIC-5 7 et SPIDER dans la version vidéo de ce protocole. Nous fournissons également une procédure alternative pour utiliser uniquement SPIDER dans la version texte de ce protocole.
- Configuration des programmes. Nous utilisons les proc2d dans le RESE 8 forfait de changer le format d'image numérique Gatan image au format d'image SPIDER. IMAGIC-5 est utilisée pour faire l'alignement 2D. SPIDER est utilisé pour faire de la reconstruction 3D et de raffinement.
Sous-section 1: Cueillette des paires d'inclinaison des particules.
- Convertir les fichiers *. dm3 Gatan image numérique au format d'image en utilisant la commande SPIDER proc2d au RESE. Les paires incliné et non incliné sont nommés en jacquard comme *** et *** t.spi u.spi, respectivement.
- Choisissez les paires de particules en utilisant le programme «WEB» distribués dans le paquet de programme SPIDER. Suivez les instructions dans le Les coordonnées sont enregistrées dans automaticly DCU ***. spi et dct ***. spi pour les particules non incliné et incliné respectivement. Un autre document dcb ***. SPI contient les informations angle d'inclinaison entre les particules incliné et non incliné (Note:. Les trois angles lors du montage dans le Web entre les paires d'inclinaison ne garantissent pas l'impartialité correct de la reconstruction finale parce thêta n'a pas le signe (valeur positive) et phi et gamma s'appuient sur les valeurs initiales mis à leur disposition. L'examen de la direction du gradient de défocalisation sur la micrographie inclinée permettrait de définir les valeurs correctes initiale de la phi et gamma avant la pose afin d'obtenir une bonne impartialité de la figure de la reconstruction. 5 illustre la convention correcte.)
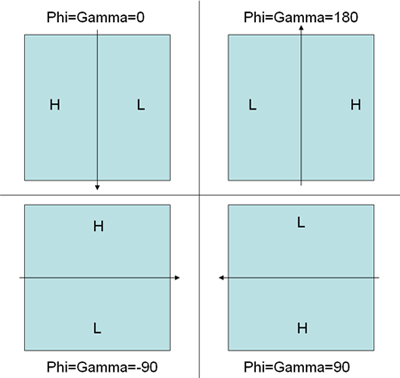
Figure 5. Une illustration pour expliquer la Convention de la détermination de l'angle d'inclinaison dans WEB-paire de particules picking mode de montage déterminée par le gradient de défocalisation. H représente haute défocalisation espace tandis que L représente faible défocalisation région. La flèche représente axe de basculement. La correspondante correcte angles initiaux pour l'AMP et gamma sont indiquées pour chaque régime. - Boîte à toutes les particules repris en utilisant une version modifiée du script SPIDER comme indiqué dans la page web http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Script enregistre les piles de particules non incliné et incliné que u.spi et t.spi. Le nombre de micrographie où les particules sont d'devraient être enregistrées dans particle_list.spi. (Note: Ceci est très important pour la génération des fichiers corrects angle d'Euler pourRCT).
Sous-section 2: Deux dimensions d'alignement et la classification des images de particules non incliné.
- Convertir les particules non incliné en IMAGIC-5 formats en utilisant le programme de em2em Imagic-5 colis. Aligner et classer les particules en classes homogènes itérative utilisant la IMAGIC-5 programmes (Annexe A). Utilisation de la commande MSA-noms en classe dans IMAGIC-5 pour générer des classes recherche dans la table des particules, que nous enregistrer en tant que imagic_classes.lis. (Remarque:. La classification et l'alignement est d'augmenter le nombre de particules ayant la même forme ainsi que de réduire la variation dans une classe Variance carte de chaque classe peuvent fournir des informations sur la qualité de la classe.)
- Générer un fichier de tracé (ali_50.plt dans la vidéo de démonstration) pour les valeurs de translation et de rotation de l'alignement de chaque particule en utilisant la commande-tête dans IMAGIC-5.
- Convertir les classes look-up table dans SPIDER documents fichiers base_file spi ***. utilisant un script perl lis2spi.pl distribués dans http://cryoem.berkeley.edu .
- Convertir les valeurs translation et rotation de l'alignement de chaque particule à partir du fichier graphique généré pour la traduction et les valeurs de rotation à l'étape 5.6 dans ali_50.spi SPIDER fichier du document en utilisant un script de plt2spi.pl distribués dans http://cryoem.berkeley.edu .
Nous utilisons IMAGIC-5 pour l'alignement 2D et de classification, car elle donne la meilleure performance sur ce travail dans nos mains. Stratégie alternative de SPIDER pour l'alignement à deux dimensions et la classification se trouve à l' http://www.wadsworth.org/spider_doc/spider/docs/align.html . Nous avons également utilisé SPIDER pour effectuer l'analyse en 2D sur les particules de l'Untilted exosomes. Suite à une procédure simple.
Alternative 5.5) Utiliser l'alignement de référence sans que décrites dans le aligner les images. Deux scripts simples peuvent être trouvés Enregistrer la rotation et déplacement de toutes les particules dans un angular_file.spi fichier du document.
Alternative 5.6) classer les particules alignées en groupes avec la même vue comme décrit dans Nous avons utilisé la méthode des K-moyennes pour la classification. Générer base_file ***. spi basée sur la classification.
Sous-section 3: Reconstruction tridimensionnelle en utilisant les images de particules incliné.
- Filtre passe-bande, le masque et le centre des particules inclinée comme pour les particules non incliné en IMAGIC-5. Générer un nouveau dataset pour les particules intitulé. (Remarque: Cette étape est facultative Il peut être fait dans SPIDER..)
- Créer des fichiers de documents issus des classes anglular look-up table des documents ali_50.spi, le DCB ***. fichiers générés par SPI Web à l'étape 5.3 et le fichier de particules partile_list.spi la liste de l'étape 5.4 en utilisant le script SPIDER comme dans l'annexe B.
- Utilisez des scripts de reconstruction dans SPIDER à faire de la reconstruction de chaque classe comme décrit dans http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Chaque classe de particules contribue à un volume de reconstruction 3D. Les modèles 3D peuvent être examinés dans l'UCSF Chimera-9. Une projection à deux dimensions du modèle 3D à angles d'Euler (0,0,0) peut être comparée à la moyenne de la classe correspondante en 2D des particules non incliné pour vérifier la qualité de la reconstruction. Trouver des volumes similaires, aligner et de les fusionner pour générer des volumes initiaux suivant la procédure http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Sous-section 4: Raffinement de la reconstruction 3D utilisant des images de particules non incliné.
- Ne le raffinement de projection correspondant du volume fusionnées initiale contre toutes les particules non incliné pour obtenir un volum plus haute résolutione sans cône d'artefact manquant et aplatir à l'aide de scripts SPIDER tel que décrit dans http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Les résultats représentatifs:
En utilisant la méthode ECR, nous avons obtenu environ 50 reconstructions de l'exosome sur un total de 5000 paires d'inclinaison (figure 6). Du 50 modèles en 3D, nous pouvons voir les différentes orientations de la séance complexes sur la grille avec principalement deux vues orthogonales. Un artefact aplatissement est également décelable dans la plupart des volumes dans la direction perpendiculaire à la surface du carbone. Nous avons effectué l'alignement et la fusion des volumes 3D afin de générer deux volumes initiaux des vues orthogonales. Utilisation du 5000 images de particules non incliné, nous avons obtenu les mêmes reconstruction finale en 3D de la exosomes à environ 18 Angstrom la résolution des deux modèles initiaux (figure 7). La structure a révélé l'architecture de la levure et exosomes donné un aperçu sur la voie de recrutement ARN substrat 10.

Figure 6. 50 modèles en 3D du complexe exosomes par les ECR de reconstruction.
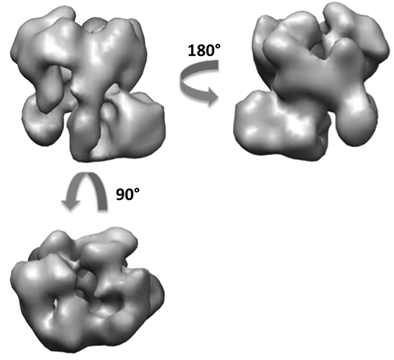
Figure 7. Reconstruction 3D du complexe exosomes après affinement.
Annexe:
Annexe A. Le fichier de script pour l'alignement 2D et la classification dans IMAGIC-5.
Fichier: auto_align_i.sh
Cliquez ici pour le fichier
Annexe B. Le fichier de script pour générer des fichiers angulaire pour la reconstruction 3D dans SPIDER.
Fichier: generate_angular_file.spi
Cliquez ici pour le fichier
Discussion
Dans cet article nous présentons un protocole détaillé de préparation des échantillons et les trois dimensions de reconstruction du complexe exosomes utilisant la microscopie électronique négative coloration. En utilisant cette méthode, nous avons obtenu la reconstruction 3D utilisant la méthode aléatoire d'inclinaison conique sans aucune connaissance préalable de la structure. Aléatoire la méthode d'inclinaison conique n'est pas nécessairement un échantillon homogène, mais l'étape de pr...
Déclarations de divulgation
Remerciements
Les auteurs tiennent à remercier les membres du laboratoire à Nogales UC-Berkeley en aidant à établir des protocoles initiaux et les membres de Wang laboratoire à l'Université de Yale dans leur aider à établir les protocoles plein. Nous reconnaissons aussi le personnel en cryo-EM installations et centre de calcul haute performance à l'école de médecine de Yale pour leur soutien. HW Smith est une famille de boursiers.
matériels
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Références
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon