È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Singola particella Microscopia Elettronica ricostruzione del complesso Exosome Utilizzo del metodo casuale Tilt conico
In questo articolo
Riepilogo
Questo articolo descrive un metodo standard per ottenere un modello tridimensionale (3D) ricostruzione delle macromolecole biologiche mediante microscopia elettronica a colorazione negativa (EM). In questo protocollo, viene spiegato come ottenere la struttura 3D del complesso exosome Saccharomyces cerevisiae a media risoluzione con il metodo casuale conica ricostruzione tilt (RCT).
Abstract
Singolo microscopia elettronica di particelle (EM) la ricostruzione è recentemente diventato uno strumento popolare per ottenere il tridimensionale (3D) struttura di complessi macromolecolari di grandi dimensioni. Rispetto alla cristallografia a raggi X, presenta alcuni vantaggi unici. In primo luogo, singola particella EM ricostruzione non ha bisogno di cristallizzare il campione, che è il collo di bottiglia in cristallografia a raggi X, soprattutto per i grandi complessi macromolecolari. In secondo luogo, non ha bisogno di grandi quantità di campioni di proteine. Rispetto ai milligrammi di proteine necessarie per la cristallizzazione, singola particella ricostruzione EM deve solo alcuni micro-litri di soluzione proteica al nano-molare concentrazioni, utilizzando il metodo di colorazione negativa EM. Tuttavia, nonostante un paio di assemblee macromolecolari con simmetria alto, singola particella EM è limitata ad una risoluzione relativamente bassa (inferiore a 1 nm risoluzione) per molti esemplari specialmente quelli privi di simmetria. Questa tecnica è inoltre limitato dalle dimensioni delle molecole in fase di studio, cioè 100 kDa per i campioni negativamente colorati e 300 kDa per frozen-idratata esemplari in generale.
Per un nuovo campione di struttura sconosciuta, si usano generalmente una soluzione di metalli pesanti di inserire le molecole di colorazione negativa. Il campione viene poi esaminato al microscopio elettronico a trasmissione di prendere due-dimensionali (2D) micrografie delle molecole. Idealmente, le molecole di proteine hanno una struttura omogenea 3D, ma presentano diversi orientamenti nelle micrografie. Queste micrografie sono digitalizzati ed elaborati in computer come "particelle singolo". Utilizzando bidimensionale allineamento e tecniche di classificazione, le molecole omogeneo le stesse idee sono raggruppati in classi. Loro medie potenziare il segnale di forme 2D della molecola. Dopo aver assegnato le particelle con il corretto orientamento relativo (angoli di Eulero), saremo in grado di ricostruire le immagini delle particelle 2D in un volume virtuale in 3D.
Nella ricostruzione di singole particelle 3D, un passo essenziale è quello di assegnare correttamente il corretto orientamento di ogni singola particella. Ci sono diversi metodi per assegnare la visualizzazione per ogni particella, compresa la ricostituzione angolare 1 e casuale conica tilt (RCT) metodo 2. In questo protocollo, si descrive la nostra pratica a ottenere la ricostruzione 3D del complesso lievito exosome con colorazione negativa EM e RCT. Va notato che il nostro protocollo di microscopia elettronica e l'elaborazione delle immagini segue il principio di base della RCT, ma non è l'unico modo per eseguire il metodo. Abbiamo prima descritto come incorporare il campione in uno strato di uranile-formiato con uno spessore paragonabile alle dimensioni delle proteine, utilizzando una griglia di carbonio bucata coperta da uno strato continuo di pellicola sottile di carbonio. Poi il campione viene inserito in un microscopio elettronico a trasmissione per raccogliere Untilted (0-laurea) e inclinata (55 gradi) paia di micrografie che verranno utilizzati in seguito per l'elaborazione e ottenere un primo modello 3D del exosome lievito. A tal fine, noi dobbiamo fare RCT e poi affinare il modello iniziale 3D utilizzando la proiezione di corrispondenza metodo raffinatezza 3.
Protocollo
1. Principio del metodo casuale Tilt conico
- Il principio del metodo casuale inclinazione conica richiede prendendo un paio di micrografie della stessa regione del campione all'interno del microscopio elettronico. Uno scatto del campione in una posizione Untilted (Figura 1A) e l'altra immagine è presa del campione in posizione inclinata tra 50 e 70 gradi (nel nostro caso, utilizziamo 55 gradi). (Figura 1B)
- Utilizzando il computer, la coppia micrografia digitalizzato è messo fianco a fianco e le immagini le stesse particelle sono selezionati. (Figura 1C)
- In coordinate tridimensionali, le immagini di particelle Untilted e dei loro partner inclinato sono correlate tra loro dalla direzione dell'asse di inclinazione e l'angolo di inclinazione. (Figura 1D)
- L'allineamento delle immagini delle particelle Untilted porta le immagini delle particelle inclinato alle loro posizioni corrispondenti azimutale. (Figura 1E)
- L'uso di immagini multiple di particelle inclinato riempire lo spazio azimutale, la struttura tridimensionale della molecola può essere ricostruita utilizzando un algoritmo di retroproiezione. (Figura 1F)
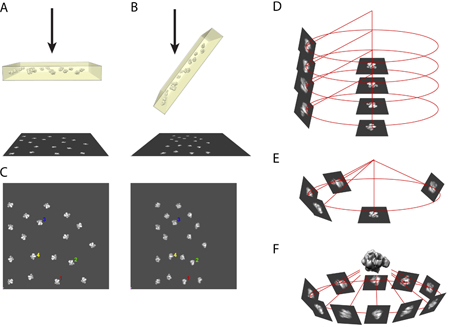
Figura 1. Un esempio del principio di RCT per la ricostruzione.
2. Preparare le Griglie di Holey carbonio coperto con Carbon sottile
Motivazioni: Si utilizza il metodo di colorazione negativa per fissare il campione casuale per la ricostruzione inclinazione conica. Al fine di preservare le macromolecole senza troppo appiattimento durante l'asciugatura, cerchiamo di inserire le molecole proteiche in una macchia profonda con uno spessore di circa la dimensione delle proteine 4. In generale, il carbonio viene utilizzato continua a fare provini negativamente colorati. Tale tipo di carbonio, tuttavia, è difficile da controllare lo spessore macchia intorno al particelle proteiche. Abbiamo così l'uso fatto in casa griglie holey carbonio ricoperto da un sottile strato di pellicola di carbonio (~ spessore 5 nm) per rendere esemplari negativamente colorati. Pozzi poco formata dai fori permettono mantenendo la soluzione proteica e la soluzione macchia sulla griglia in modo da è molto più facile da incorporare proteine in uno spessore ottimale macchia. Inoltre, il sottile strato di carbonio sul foro riduce il rumore di fondo molto.
- Preparare la soluzione Formvar 0,5%. Nella cappa, aggiungere 0,45 g di resina di polivinile formale e 90 mL di cloroformio in un bicchiere da 100 ml di vetro. Utilizzare un foglio di alluminio per coprire il bicchiere, e utilizzare un piccolo bar mescolare per sciogliere resina formali su un agitatore magnetico. Ci vogliono circa 15 minuti per sciogliere la resina.
- Durante la dissoluzione della Formvar, vetro microscopia pulito diapositive in metanolo e asciugare con Kimwipes.
- Dopo la resina Formvar è completamente dissolto in cloroformio, aggiungere 1 ml di glicerolo al 50% alla superficie della soluzione. Regolazione del volume del glicerolo aggiunto influisce sulla densità dei fori sul carbonio bucata. Immergere la punta di un ultrasonicatore nella soluzione a circa 1 cm di profondità e di utilizzare la massima potenza per ultrasuoni da 1 minuto a fare emulsione di goccioline di glicerolo in soluzione Formvar. La soluzione diventa lattiginosa dopo questo passaggio. Sonicazione cause più piccole dimensioni dei fori sul carbonio bucata.
- Subito dopo la sonicazione, immergere le lastre di vetro pulito verticalmente nel emulsione per 1 secondo, portarli fuori, e asciugare il fondo diapositive 'usando carta da filtro per formare una sottile pellicola di plastica sulla superficie delle diapositive. Dopo il cloroformio evapora, controllare la densità e le dimensioni dei fori nel film sotto un microscopio ottico. Regolare la condizione di preparazione in base alle esigenze. Con la condizione descritta qui, in genere ottenere fori del diametro di 3 ~ 4 micro-metri e 10 ~ 20 fori in ogni quadrato di una griglia di 400 mesh.
- Dopo la vetrini sono asciutti, tagliare il bordo della pellicola di plastica sulla superficie del vetrino. Galleggiante del film fuori sulla superficie di acqua distillata. La sottile pellicola sulla superficie dell'acqua può essere osservato con un angolo guardando contro la riflessione della luce. Posto 400-mesh griglie di rame sul film uno dopo l'altro, con superficie liscia delle griglie 'rivolto verso il basso.
- Prendete la pellicola di plastica con griglie su di essa utilizzando un pezzo di carta. Capovolgere la carta e lasciare asciugare in una capsula di Petri. Mettere a bagno la carta in metanolo per rimuovere il glicerolo residuo nei fori e lasciare che la carta asciugare all'aria.
- Cappotto le griglie con uno strato di carbonio dello spessore di circa 20 nanometri in un evaporatore di carbonio. Lo spessore può essere determinato dal colore grigio del carbonio.
- Mettere a bagno le griglie con carbonio in cloroformio per mezz'ora per rimuovere il Formvar. Dopo le griglie sono secchi, abbiamo ottenuto fatto in casa griglie di carbonio bucata.
- Evaporare sottile strato di carbonio con circa 5 nanometri di spessore sulla superficie di mica appena spaccati.
- Accuratamente messo le griglie di carbonio bucata sotto acqua distillata. Float il carbonio sottile dalla superficie mica sulla superficie dell'acqua e depositarlo sul carbonio bucatagriglie lentamente. Asciugare le griglie in una cappa aspirante.
3. La colorazione negativa del Complesso Exosome
Spiegazione: Ci sono alcune soluzioni metallo pesante macchia che può essere utilizzato per EM colorazione negativa, tra acetato di uranile, formiato di uranile, acido fosfotungstico, molibdato di ammonio, e altri. Un'altra soluzione macchia possiede particolari caratteristiche uniche. Per esempio, acetato di uranile offre un elevato contrasto della particella, ma potrebbe bloccarsi complessi proteici che non amano l'ambiente acido. Per tali campioni, acido phosphotungestic a pH neutro può essere una buona soluzione macchia. Abbiamo scelto formiato di uranile saturi (UF) soluzione grazie alla sua granularità fine e ad alta capacità di penetrazione in molecole.
- Bollire l'acqua distillata per 1 minuto. Raffreddarlo lentamente a temperatura ambiente. Questo passo è quello di rimuovere l'ossigeno disciolto dall'acqua.
- Fai fresca 2% formiato di uranile (UF) soluzione. Miscelare 1 ml di acqua e 20 UF mg in una provetta 1,5 ml. Vortex per 10 minuti.
- Regolare il pH a 5,0 con l'aggiunta di 2 microlitri di 10 M di idrossido di potassio. Mescolare immediatamente. Il colore soluzione deve essere più giallo. Il pH della soluzione non deve essere troppo alto, altrimenti la macchia precipitati.
- Mettere il tubo su vortex per altri 10 min.
- Gira la soluzione su un tavolo centrifuga alla massima velocità per 10 min.
- Filtrare la soluzione attraverso una membrana PVDF 0,2 micrometri. Questa è la soluzione UF fresca. Coprire il tubo di soluzione in un foglio di alluminio per impedire alla luce. La soluzione deve essere utilizzato nella stessa giornata.
- Scarica a bagliore uno sottile di carbonio-over-holey griglia di carbonio utilizzando un bagliore-scarico apparecchio per 30 secondi a 25 mA di corrente.
- Mettete un pezzo di parafilm pulita in panchina. Mettere 3 gocce di soluzione di 50 UF microlitro macchia sulla parte superiore del parafilm.
- Diluire il complesso exosome ad una concentrazione di 50 ~ 100 nM utilizzando il tampone di diluizione (25 mM Tris-HCl pH 7,5, 100 mM NaCl, 2 mM DTT). Mettere 4 microlitri della proteina diluito sul bagliore-scarica griglia. Lasciate che il campione soggiorno sulla griglia per un minuto. (Nota:. Una tale concentrazione finale di molecole in genere dà una densità ottimale di ben particelle disperse in griglia negativamente macchiato Per formiato di uranile o acecate soluzione uranile macchia, il fosfato o alta concentrazione di sale (più di 0,5 M) in generale è non va bene per ottenere buoni risultati della colorazione. nostra esperienza suggerisce che Hepes o Pipes lavorare bene con le soluzioni di uranile macchia.)
- Usare un pezzo di carta da filtro per asciugare la soluzione residua dal bordo della griglia e capovolgere la griglia subito sopra delle gocce di macchia e lavare la griglia per circa 10 secondi per ogni goccia.
- Dopo l'ultimo risciacquo, lasciare che il soggiorno macchia sulla griglia di partenza per un altro min 1 e poi asciugare la macchia via da un pezzo di carta da filtro. Mantenere un sottile strato di soluzione macchia sulla superficie della griglia al fine di ottenere un buon risultato profondo macchia. Lasciate asciugare rapidamente la griglia in una cappa aspirante.
4. Microscopia Elettronica del Complesso Exosome
Spiegazione: Ogni microscopio elettronico a trasmissione, con una fase inclinabile può essere usato per raccogliere le coppie inclinazione del campione per RCT ricostruzione. In teoria, più l'angolo il campione può essere inclinato per raccogliere dati, meglio è. In pratica, grazie al design del titolare del campione e la geometria della griglia, l'angolo massimo utilizzabile è limitata 50-70 gradi. In questo protocollo, si limita a descrivere la nostra procedura utilizzando un FEI Tecnai-12 microscopio elettronico. Per gli altri modelli di microscopi, le operazioni devono essere adeguate secondo le esigenze del progetto e la proprietà dello strumento.
- Mettere la griglia del campione nel porta-campione e poi mettere il supporto in un Tecnai FEI -12 microscopio elettronico. Il microscopio è gestito a 120 kV. Noi usiamo Gatan Ultrascan4000 camera CCD per scattare foto. Assicurarsi che il 'Flip intorno all'asse verticale' nella finestra di dialogo 'fotocamera config' di Digital interfaccia Micro-non è selezionata per garantire la corretta determinazione manualità. (Nota:. Questo è importante soprattutto se il lettore si basa su SPIDER RCT procedure di ricostruzione per ottenere un modello 3D)
- Controllare la griglia del campione a basso ingrandimento per trovare la migliore quadrati colorati. Tale tipo di piazze dovrebbe avere una decina di fori con dimensione di circa 1 ~ 2 micron e aree macchia scura in loro. (Figura 2)

Figura 2. Un microscopio a basso ingrandimento di una griglia che mostra piazze con le macchie di buoni e cattivi. - Attivare la modalità a basse dosi di Interfaccia utente FEI e allineare la ricerca, messa a fuoco ed esporre la posizione in modalità a basso dosaggio. Noi usiamo ingrandimenti di 150.000 per il fuoco, 52.000 per l'esposizione e 1,5 metri in lunghezza fotocamera diffrazione per la ricerca. Il tempo di esposizione viene regolata a 1 secondo. Impostare la posizione del fuoco 2 micrometri di distanza dal pos esposizioneition lungo l'asse inclinabile.
- Trova i fori con una buona colorazione in modalità di ricerca e salvare i luoghi. I fori hanno generalmente una buona pendenza macchia scura in esse osservate sotto la modalità di ricerca. Scatta una foto CCD della piazza. Inclinare il campione a 55 gradi, scattare un'altra foto. Confrontare le due immagini, identificare i fori appaiati nei due micrografie. (Figura 3)
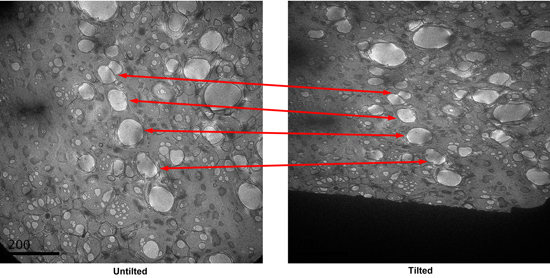
Figura 3. Una coppia di inclinazione micrografie di una piazza in modalità di ricerca. Corrispondenti fori appaiati sono indicati. - Inclinare la fase a 0 gradi. Utilizzando il kit a basso dosaggio per scattare foto di ogni foro individuato nel modo di ricerca a forte ingrandimento. Il defocus utilizzato è di circa -0,7 micrometro.
- Dopo che tutti i fori sono state prese le immagini di, inclinare la fase a 55 gradi. Prendere micrografie del campione inclinato allo stesso ingrandimento, come quelli Untilted con una sfocatura di circa -1,2 micrometro. Identificare le coppie corrispondenti inclinato di micrografie in base agli schemi della micrografie basso ingrandimento. Il modello irregolare del fatto in casa holey griglie di carbonio aiuta la correlazione. Esaminare le coppie inclinazione di micrografie e rimuovere il microscopio con macchie male come superficiale aree colorate (particelle sembrano avere un alone intorno a loro in condizioni inclinato). (Figura 4)
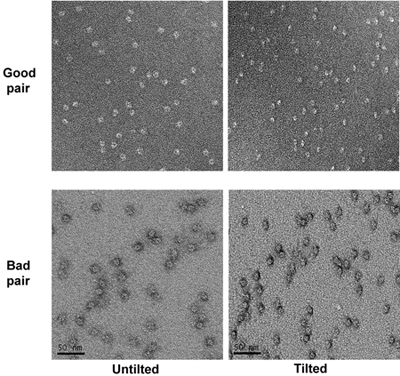
Figura 4. Due coppie di inclinazione di micrografie del campione ad alto ingrandimento. Le micrografie bene e il male sono contrassegnati.
5. Image Processing dei dati
Spiegazione: Ci sono diverse opzioni e pacchetti software per eseguire la ricostruzione RCT in computer. Il più comune è SPIDER 5. Un protocollo di base per eseguire RCT in SPIDER possono trovare nella pagina web http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Un protocollo dettagliato per eseguire RCT in SPIDER è descritto nell'articolo di Shaikh et al. 6 Nel nostro protocollo, utilizziamo una combinazione di IMAGIC-5 7 e SPIDER nella versione video del protocollo. Forniamo anche una procedura alternativa da utilizzare esclusivamente SPIDER nella versione testo del protocollo.
- Installazione dei programmi. Usiamo il proc2d nel pacchetto di 8 EMAN per modificare il formato immagine da Gatan immagine digitale di immagine in formato SPIDER. IMAGIC-5 è usato per fare l'allineamento 2D. SPIDER è usato per fare la ricostruzione 3D e la raffinatezza.
Comma 1: Picking coppie inclinazione delle particelle.
- Convertire i file *. dm3 Gatan immagini digitali a formato immagine SPIDER usando il comando proc2d in EMAN. Le coppie inclinato e Untilted sono nominati nel modello come t.spi *** e *** u.spi, rispettivamente.
- Scegli le coppie di particelle utilizzando il programma 'WEB' distribuito in pacchetto di programmi SPIDER. Seguire le istruzioni del Le coordinate sono automaticly salvati in DCU ***. spi e DCT ***. spi per particelle Untilted e inclinate rispettivamente. Un altro documento DCB ***. spi contiene le informazioni di inclinazione angolo tra particelle inclinato e Untilted (Nota:. I tre angoli durante il montaggio in WEB tra le coppie di inclinazione non garantiscono la corretta manualità della ricostruzione finale perché theta non ha la segno (valore positivo) e phi e gamma si basano sui valori iniziali insieme a loro. Esaminando la direzione del gradiente defocus sul microscopio inclinato aiuterebbe impostare i valori corretti iniziale del phi e gamma prima del montaggio al fine di ottenere una corretta manualità della ricostruzione. Figura 5 illustra la convenzione corretta.)
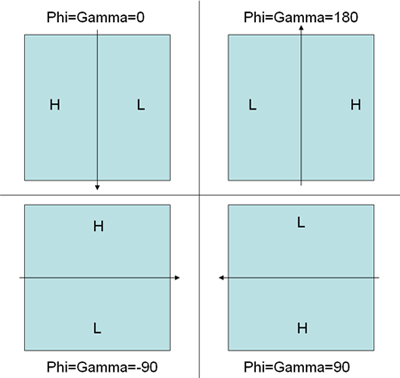
Figura 5. Un esempio per spiegare la convenzione di determinazione dell'angolo di inclinazione in WEB-coppia particella raccolta modalità di raccordo determinato dal gradiente defocus. H rappresenta alta defocus zona mentre L rappresenta bassa defocus zona. La freccia rappresenta l'asse inclinabile. I corrispondenti angoli corretti iniziale per PHI e GAMMA sono indicati per ogni regime. - Box tutte le particelle raccolte utilizzando una versione modificata dello script SPIDER come indicato nella pagina web http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Lo script salva le pile di particelle Untilted e inclinato come u.spi e t.spi. I numeri del microscopio in cui le particelle vengono dalla devono essere salvati in particle_list.spi. (Nota: Questo è molto importante per la generazione del file corretto angolo di Eulero perRCT).
Comma 2: bidimensionale allineamento e la classificazione delle immagini delle particelle Untilted.
- Convertire le particelle in Untilted IMAGIC-5 formato utilizzando il programma em2em in Imagic-5 pacchetto. Allineare e classificare le particelle in classi omogenee iterativamente utilizzando il IMAGIC-5 programmi (Appendice A). Utilizzando la MSA-nomi-in-class comando IMAGIC-5 per generare classi della tabella di ricerca delle particelle, che salva come imagic_classes.lis. (Nota:. La classificazione e l'allineamento è quello di aumentare il numero di particelle con la stessa forma e ridurre la variazione in una classe varianza mappa di ogni classe in grado di fornire informazioni sulla qualità della classe.)
- Generare un file grafico (ali_50.plt nel video dimostrativo) per i valori di traslazione e rotazione di allineamento di ogni particella utilizzando il comando di intestazione IMAGIC-5.
- Convertire le classi look-up table nel documento SPIDER file base_file ***. spi utilizzando un lis2spi.pl script perl distribuito in http://cryoem.berkeley.edu .
- Convertire i valori di traslazione e rotazione di allineamento di ogni particella dal file grafico generato per la traduzione e valori di rotazione al punto in 5,6 ali_50.spi file di documento SPIDER utilizzando un plt2spi.pl copione distribuito in http://cryoem.berkeley.edu .
Noi usiamo IMAGIC-5 per l'allineamento 2D e classificazione perché dà le migliori prestazioni in questo lavoro nelle nostre mani. Strategia alternativa a Spider per il bidimensionale allineamento e la classificazione sono disponibili all'indirizzo http://www.wadsworth.org/spider_doc/spider/docs/align.html . Abbiamo anche usato SPIDER per eseguire l'analisi 2D sulle particelle Untilted del exosome. Che segue è una semplice procedura.
Alternative 5.5) Utilizzare l'allineamento di riferimento esente da come descritto nella allineare le immagini. Due semplici script si possono trovare Salva la rotazione e lo spostamento di tutte le particelle in un angular_file.spi file di documento.
Alternative 5.6) Classificare le particelle allineati in gruppi con la stessa vista come descritto nella Abbiamo usato K-si intende il metodo per la classificazione. Genera base_file ***. spi in base alla classificazione.
Comma 3: Ricostruzione tridimensionale utilizzando le immagini delle particelle inclinato.
- Filtro passa-banda, maschera e il centro le particelle inclinato così come per le particelle in Untilted IMAGIC-5. Generare un nuovo set di dati per le particelle dal titolo. (Nota: questo passaggio è facoltativo e può essere fatto in SPIDER..)
- Creare file di documenti anglular dalle classi look-up table documenti ali_50.spi, i DCB ***. spi file generati dal web al punto 5.3 e la lista delle particelle partile_list.spi file da passo 5,4 usando lo script SPIDER come in Appendice B.
- Utilizzare gli script di ricostruzione in SPIDER a fare la ricostruzione di ogni classe, come descritto nella http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Ogni classe di particelle contribuisce a un volume ricostruzione 3D. I modelli 3D possono essere esaminate in UCSF Chimera-9. Una proiezione bidimensionale del modello 3D in un angolo di Eulero (0,0,0) può essere paragonato alla media corrispondente classe 2D dalle particelle Untilted per verificare la qualità della ricostruzione. Trova volumi simili, allineare e fondere per generare volumi iniziali seguendo la procedura http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Comma 4: Affinamento della ricostruzione 3D utilizzando le immagini delle particelle Untilted.
- Fare raffinatezza proiezione corrispondenza del volume fuse iniziale contro tutte le particelle Untilted per ottenere una risoluzione più elevata volume senza cono mancanti e artefatto appiattimento utilizzando script di Spider, come descritto nella http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Rappresentante dei risultati:
Utilizzando il metodo RCT, abbiamo ottenuto circa 50 ricostruzioni del exosome da un totale di 5.000 coppie di inclinazione (Figura 6). Dal 50 modelli 3D, possiamo vedere diversi orientamenti della seduta complessa sulla griglia di partenza con principalmente due viste ortogonali. Un artefatto appiattimento è rintracciabile in molti dei volumi nella direzione perpendicolare alla superficie di carbonio. Abbiamo eseguito l'allineamento e la fusione dei volumi 3D per generare due volumi iniziali in viste ortogonali. Utilizzando il 5.000 immagini delle particelle Untilted, abbiamo ottenuto lo stesso finale ricostruzione 3D della exosome a circa 18 Angstrom risoluzione da entrambi i modelli iniziale (Figura 7). La struttura ha rivelato l'architettura della exosome lievito e fornito indicazioni sul percorso di reclutamento substrato RNA 10.

Figura 6. 50 modelli 3D del complesso exosome da RCT ricostruzione.
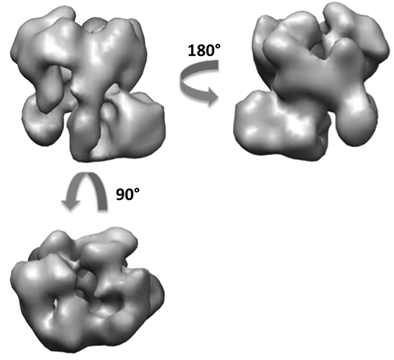
Figura 7. Ricostruzione 3D del complesso exosome dopo raffinatezza.
Appendice:
Appendice A. Il file di script per l'allineamento e la classificazione in 2D IMAGIC-5.
File: auto_align_i.sh
Clicca qui per il file
Appendice B. Il file di script per la generazione di file angolare per la ricostruzione in 3D SPIDER.
File: generate_angular_file.spi
Clicca qui per il file
Discussione
In questo articolo vi presentiamo un protocollo dettagliato di preparazione del campione e la ricostruzione tridimensionale del complesso exosome tramite microscopia elettronica a colorazione negativa. Utilizzando questo metodo, abbiamo ottenuto la ricostruzione 3D utilizzando il metodo casuale conica tilt senza alcuna conoscenza della struttura. Metodo casuale inclinazione conica non richiede necessariamente un campione omogeneo, ma il passo successivo affinamento proiezione di corrispondenza avrebbe bisogno di un camp...
Divulgazioni
Riconoscimenti
Gli autori desiderano ringraziare i membri di Nogales laboratorio alla UC-Berkeley, contribuendo a stabilire i protocolli iniziali ed i membri di Wang laboratorio presso la Yale University nel loro aiuto per stabilire i protocolli pieno. Riconosciamo anche il personale di crio-EM struttura e High-Performance Center di calcolo a Yale School of Medicine per il loro sostegno. HW è una famiglia Smith affidatario.
Materiali
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Riferimenti
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon