Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
De proteínas de membrana de superposición de ensayo: Un protocolo de prueba de interacción entre las proteínas soluble e insoluble In vitro
En este artículo
Resumen
Pruebas de interacción proteína-proteína es indispensable para la disección de la funcionalidad de las proteínas. Aquí, presentamos un In vitro Proteína-proteína ensayo de unión a la sonda una membrana de proteína inmovilizada con una proteína soluble. Este ensayo proporciona un método fiable para comprobar la interacción entre una proteína insoluble y de una proteína en una solución.
Resumen
Las interacciones entre las proteínas de la validación de diferentes es de vital importancia para la investigación de sus funciones biológicas a nivel molecular. Hay varios métodos, tanto in vitro como in vivo, para evaluar la unión a proteínas, y por lo menos dos métodos que complementen las carencias de cada uno debe realizar para obtener conocimientos fiables.
Para un ensayo in vivo, la complementación bimolecular de fluorescencia (BiFC) ensayo representa el enfoque más popular y menos invasivo que permite detectar interacción proteína-proteína en las células vivas, así como identificar la localización intracelular de las proteínas que interactúan 1,2. En este ensayo, no fluorescente mitades N-y C-terminal de la GFP y sus variantes están fusionados a las proteínas de prueba, y cuando las dos proteínas de fusión se unen debido a las interacciones de las proteínas a prueba, la señal fluorescente se reconstituye 3-6 . Debido a que su señal es fácilmente detectable por microscopía confocal de epifluorescencia o, BiFC se ha convertido en una poderosa herramienta de elección entre los biólogos celulares para el estudio de las interacciones proteína-proteína en tres células vivas. Este ensayo, sin embargo, a veces puede producir resultados falsos positivos. Por ejemplo, la señal fluorescente se puede reconstituir dos fragmentos de las buenas prácticas agrarias dispuestas hasta 7 nm de cada uno de otro por cerca de embalaje en un compartimiento subcelular pequeña, más bien que debido a las interacciones específicas 7.
Debido a estas limitaciones, los resultados obtenidos de las tecnologías de células en vivo de imágenes deben ser confirmados por un enfoque independiente sobre la base de un principio diferente para la detección de interacciones entre proteínas. Co-inmunoprecipitación (Co-IP) o el glutatión transferasa (GST) pull-down ensayos representan tales métodos alternativos que se utilizan comúnmente para analizar interacciones proteína-proteína in vitro. Sin embargo, eEn estos ensayos, sin embargo, las proteínas de las pruebas debe ser fácilmente soluble en el búfer que supportsused para la reacción de unión. Por lo tanto, las interacciones específicas que implican una proteína insoluble no puede ser evaluado por estas técnicas.
Aquí, se expone el protocolo para el análisis de la proteína de membrana superpuesta vinculante, lo que evita este problema. En esta técnica, la interacción entre las proteínas solubles e insolubles puede ser fiable a prueba, porque una de las proteínas es inmovilizado en una matriz de membrana. Este método, en combinación con los experimentos in vivo, tales como BiFC, ofrece un método fiable para investigar y caracterizar las interacciones entre las proteínas fielmente soluble e insoluble. En este artículo, la unión entre el virus del mosaico del tabaco (TMV) proteína de movimiento (MP), que ejerce múltiples funciones viral en la célula a célula de transporte 8.14, y una planta de interactor identificado recientemente celular, el tabaco ankyrin repetir que contienen proteínas (ANK ) 15, se demuestra mediante esta técnica.
Protocolo
1. Expresión y la extracción de las proteínas
- Diferencialmente etiqueta de las proteínas de la prueba para su detección. Etiqueta de la proteína a ser inmovilizado en la membrana (ProIM) con una etiqueta de un tamaño más grande (por ejemplo, IVA), y la etiqueta no fusionados se puede utilizar como un control de inmovilizado negativo (ProIMnc). Etiqueta de la proteína a ser utilizado como una sonda soluble (PROSOL), ya sea con una grande o una pequeña etiqueta. Fusible de la misma etiqueta a un control adecuado de proteínas solubles en negativo (ProSOLnc) e incluir en el ensayo de interacción para confirmar la especificidad de la unión detectado.
- Elegir el sistema de expresión de la proteína para expresar proteínas etiquetadas, es decir, E. coli, baculovirus, etc, dependiendo de la necesidad de modificaciones post traduccionales potencial y el rendimiento de las proteínas.
- Extracto ProIM y nc ProIM del organismo de elección (paso 1.2) mediante SDS-PAGE de tampón de carga (20% de glicerol, 4% SDS, 20 mM Tris-HCl, pH6.8), que contiene un cóctel inhibidor de la proteasa. Salir de la suspensión de células a temperatura ambiente durante 15 minutos, añadir un 0,5% de 2-mercaptoetanol y se hierve durante 5 minutos.
- Extracto de PROSOL y nc PROSOL del organismo de elección (paso 1.2) usando un estándar de protocolos 16. Estas proteínas deben ser fácilmente soluble en el tampón de unión (140 mM NaCl, 10 mM Tris-HCl pH 7,4, 2 mM EDTA, 2 mM DTT, 1% de BSA, 0,1% Tween 20) utilizado en el paso 4.1.
- Determinar la concentración de las proteínas recombinantes utilizando un método estándar, como Bio-Rad kit de ensayo de la proteína. como Bio-Rad kit de ensayo de la proteína. Si el extracto crudo, en lugar de la proteína purificada, se utiliza para el ensayo, la estimación de la concentración de la proteína de interés en este extracto por densitometría de barrido de la banda correspondiente a un SDS-poliacrilamida gel teñido con azul brillante de Coomassie R-250 y el uso de conocidos concentraciones de BSA como referencia.
2. Inmovilización de ProIM y ProIMnc en la membrana
- Resolver los dos conjuntos de extractos, cada uno con 1 mg de ProIM y nc ProIM en un gel de poliacrilamida-SDS de acuerdo con el protocolo estándar 16.
- Después de la electroforesis, el lugar de la transferencia que el gel en 100 ml de tampón de transferencia (60 mM Glicina, 10 mM Tris, el 0,0006% SDS, 20% MeOH) y se incuba con agitación suave durante 20 minutos a temperatura ambiente.
- Electrotransferencia las proteínas contenidas en el gel a una membrana de nitrocelulosa de acuerdo con el protocolo estándar 16.
3. Re-plegamiento de las proteínas de membrana
- Después de electrotransferencia, se incuba la membrana durante 15 minutos en 15 ml de un buffer (30 mM Tris-HCl pH 7,4, 0,05% de Tween 20) con agitación suave para eliminar los restos de SDS.
- Después de vaciar con cuidado, la transferencia de la membrana a 25 ml de tampón de desnaturalización (7 M clorhidrato de guanidina, 2 mM EDTA, 50 mM DTT, 50 mM Tris-HCl pH 8,3). y se incuba durante 2 horas a temperatura ambiente con agitación suave. (Nota: La membrana de nitrocelulosa se vuelve opaco cuando se incubaron en el tampón de desnaturalización). y se incuba durante 2 horas a temperatura ambiente con agitación suave.
- La transferencia de la membrana a 25 ml de TBS (10 mM Tris-HCl, 150 mM NaCl, pH 7,4) e incubar con agitación suave durante 5 minutos (Nota: Durante este paso, la membrana recupera su color blanco original).
- La transferencia de la membrana a 25 ml de tampón de unión (véase el paso 1.2) e incubar a 4 ° C con agitación suave durante la noche.
4. La prueba de las ProIM por PROSOL
- Corte la membrana en dos tiras, conteniendo ambos ProIM y nc ProIM.
- Que las soluciones de hibridación PROSOL y ProSOLnc mediante la dilución de la preparación con 1.10 mg de cualquiera de PROSOL o ProSOLnc en 10 ml de tampón de unión libre. La transferencia de cada membrana en la solución de hibridación o PROSOL ProSOLnc e incubar durante 1,5 horas a temperatura ambiente con agitación suave.
- Quitar las membranas de las soluciones de hibridación y enjuáguese la boca tres veces durante 15 minutos en cada una de TBS.
5. Visualización de interacción proteína-proteína mediante inmunotransferencia
- Bloquear la membrana con el 2,5% de leche desnatada en TBST (10 mM Tris-HCl, 140 mM NaCl, 0,05% de Tween 20, pH 7,4) durante 1 hora a temperatura ambiente. Diluir el anticuerpo primario (anti-strepII anticuerpo policlonal de conejo) en el 0,5% de leche desnatada en TBST a la concentración recomendada por el fabricante.
- Coloque las membranas bloqueado en la solución de anticuerpos, y se incubó durante 1 hora a temperatura ambiente o por la noche a 4 ° C con agitación suave.
- Enjuague las membranas en TBST 20 ml durante 15 minutos, y dos veces por 5 min a temperatura ambiente con agitación suave.
- Diluir el anticuerpo secundario (anticuerpo anti-conejo IgG) conjugado con peroxidasa de rábano picante (HRP) en el 0,5% de leche desnatada en TBST a la concentración recomendada por el fabricante. Coloque las membranas en la secundaria de anticuerpossolución y se incuba durante 1 hora a temperatura ambiente con agitación suave.
- Enjuague las membranas en TBST 20 ml durante 15 minutos, y dos veces por 5 min a temperatura ambiente con agitación suave. Después de que el enjuague final en TBS, visualizar la interacción proteína-proteína con un sustrato de quimioluminiscencia HRP (por ejemplo, Millipore Immobilon oeste de quimioluminiscencia sustrato HRP).
- Sonda de las mismas membranas con el anticuerpo primario para la etiqueta fusionado a ProIM, seguido por el anticuerpo secundaria apropiada como se describe en los pasos 5.1 a 5.4. Visualizar ProIM y ProIMnc como se describe en el paso 5.5 para validar la identidad de las bandas obtenidas en el ensayo de proteínas de membrana superpuesta (paso 5.5). En la mayoría de los casos, no es necesario pelar, ya que la señal obtenida por este paso es mucho más fuerte que la señal residual obtenida en la etapa 5.5.
6. Los resultados representativos:
La interacción ANK-MP fue observado por BiFC en las células epidérmicas del tabaco (Figura 1). Debido a que MP es una proteína muy insoluble cuando se expresa en bacterias o en las plantas, la proteína de la membrana superposición de ensayo se adoptó para validar esta interacción in vitro (fig. 1B). Extractos de proteínas que contiene 1 g de GST-MP (ProIM) o GST fusionadas (ProIM nc) se resolvieron por SDS-poliacrilamida electroforesis en gel, seguido por electrotransferencia a una membrana de nitrocelulosa. Cuando estos ProIMs se probaron con solubles ANK-strepII (PROSOL), GST-MP, pero no fusionados GST, exhibió unión (fig. 1B, carriles 1, 2, en comparación con los carriles 5, 6). Por otra parte, cuando el mismo conjunto de ProIMs se probaron con un ProSOLnc relación, es decir, la quinasa de Arabidopsis NADH citoplásmico etiquetados strepII (NADH3-strepII), no vinculante, no se observó, lo que demuestra la especificidad de la interacción ANK-MP (fig. 1B, carriles 3, 4).
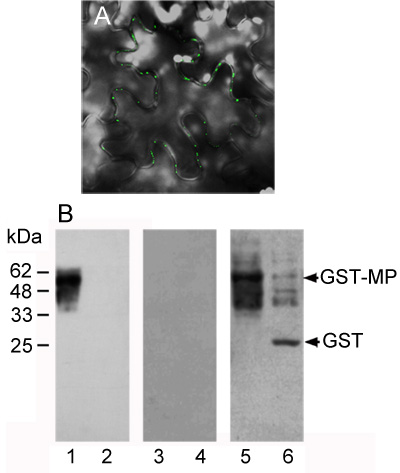
Figura 1. La unión específica de tabaco ANK a TMV MP in vivo e in vitro. (A) ANK-MP interacción de las células vivas del tabaco epidérmica detectada por BiFC. Fuerte señal de YFP fue reconstruido cuando MP y ANK, fusionado a C-terminal y N-terminal mitades de YFP, respectivamente, fueron coexpressed en la epidermis del tabaco siguientes microbombardment de sus genes de codificación. Esta señal BiFC acumulado en puntos lagrimales en la periferia de la célula, que son diagnósticos de plasmodesmos 15. (B) ANK-MP interacción in vitro detectada por superposición de ensayo de proteínas de membrana. Extractos de proteínas que contiene 1 g de GST-MP (ProIM) o no fusionados GST (ProIMnc) se resolvieron en un 15% SDS-gel de poliacrilamida, seguido por electrotransferencia a una membrana de nitrocelulosa. El GST-MPProIM GSTProIMnc y se incubaron con 0,5 mg / ml de ANK-strepII (PROSOL), y ANK vinculante fue detectado por el sondeo de la membrana con el anticuerpo de conejo anti-strepII policlonales, seguido de IgG anti-conejo + anticuerpo secundario conjugado con M HRP (calles 1 y 2). Ni GST-MPProIM GSTProIMnc ni interactuó con una proteína no relacionada, la cinasa citoplasmática Arabidopsis NADH, fusionado con la etiqueta strepII (ProSOLnc, carriles 3 y 4). La identidad de la banda observada en este ensayo fue confirmada por el sondeo de la membrana con el anticuerpo anti-GST (carriles 5 y 6). Cuando la membrana se trata con tampón de desnaturalización sin ser lavadas con buffer A, la unión de la GST-MP para ANK se pierde, mientras que las proteínas identificadas contenidas en el GST y GST-MP contining extractos de proteína reaccionaron con ANK-strepII, lo que demuestra la importancia de la etapa de 3,1 antes del proceso de desnaturalización (líneas 7 y 8).
Discusión
Este enfoque es adecuado para las pruebas de interacciones proteína-proteína entre las combinaciones de las proteínas, cuando al menos uno de los cuales las proteínas se disuelve fácilmente en el tampón de unión, y se aplicó con éxito a otra combinación de proteínas 17,18. El iInteractions entre las proteínas que son insolubles en estas condiciones no pueden ser probados por el presente Protocolo.
Además, el replegamiento de ProIM éxito es crítico para el ensayo. En...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
El trabajo en nuestro laboratorio es apoyado por becas de los NIH, el USDA Instituto Nacional de Alimentación y la Agricultura, NSF, BARD, DOE, y FBS al VC
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| Proteína kit de ensayo | Bio-Rad | 500-0001 | |
| Proteinasa cóctel de inhibidores de | SIGMA | S8820 | |
| Mini-Protean sistema | Bio-Rad | 165-8000 | |
| Semi-seco Western Blot SD electrotransferencia sistema | Bio-Rad | 170-3940 | |
| IgG anti-conejo conjugado con peroxidasa de rábano de anticuerpos | GenScript | A00098 | |
| Anti-GST anticuerpo policlonal de conejo | GenScript | A00097 | |
| Anti-strepII | GenScript | A00626 | |
| Biotrace, nitrocelulosa NT membrana de transferencia | Pall Científico | 27377-000 | |
| Immobilon occidental HRP sustrato quimioluminiscente | Millipore | WBKL S0 050 |
Referencias
- Hu, C. D., Chinenov, Y., Kerppola, T. K. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Mol Cell. 9, 789-798 (2002).
- Citovsky, V. Subcellular localization of interacting proteins by bimolecular fluorescence complementation in planta. J Mol Biol. 362, 1120-1131 (2006).
- Kerppola, T. K. Design and implementation of bimolecular fluorescence complementation (BiFC) assays for the visualization of protein interactions in living cells. Nat Protoc. 1, 1278-1286 (2006).
- Shyu, Y. J. Visualization of protein interactions in living Caenorhabditis elegans using bimolecular fluorescence complementation analysis. Nat Protoc. 3, 588-596 (2008).
- Citovsky, V., Gafni, Y., Tzfira, T. Localizing protein-protein interactions by bimolecular fluorescence complementation in planta. Methods. 45, 196-206 (2008).
- Ohad, N., Shichrur, K., Yalovsky, S. The analysis of protein-protein interactions in plants by bimolecular fluorescence complementation. Plant Physiol. 145, 1090-1099 (2007).
- Zamyatnin, A. A. Assessment of the integral membrane protein topology in living cells. Plant J. 46, 145-154 (2006).
- Citovsky, V., Knorr, D., Schuster, G., Zambryski, P. C. The P30 movement protein of tobacco mosaic virus is a single-strand nucleic acid binding protein. Cell. 60, 637-647 (1990).
- Heinlein, M., Epel, B. L., Padgett, H. S., Beachy, R. N. Interaction of tobamovirus movement proteins with the plant cytoskeleton. Science. 270, 1983-1985 (1995).
- Tomenius, K., Clapham, D., Meshi, T. Localization by immunogold cytochemistry of the virus coded 30 K protein in plasmodesmata of leaves infected with tobacco mosaic virus. Virology. 160, 363-371 (1987).
- Ding, B. Secondary plasmodesmata are specific sites of localization of the tobacco mosaic virus movement protein in transgenic tobacco plants. Plant Cell. 4, 915-928 (1992).
- Meshi, T. Function of the 30 kd protein of tobacco mosaic virus: involvement in cell-to-cell movement and dispensability for replication. EMBO J. 6, 2557-2563 (1987).
- Wolf, S., Deom, C. M., Beachy, R. N., Lucas, W. J. Movement protein of tobacco mosaic virus modifies plasmodesmatal size exclusion limit. Science. 246, 377-379 (1989).
- Waigmann, E., Lucas, W. J., Citovsky, V., Zambryski, P. C. Direct functional assay for tobacco mosaic virus cell-to-cell movement protein and identification of a domain involved in increasing plasmodesmal permeability. Proc. Natl. Acad. Sci. USA. 91, 1433-1437 (1994).
- Ueki, S., Spektor, R., Natale, D. M., Citovsky, V. ANK, a host cytoplasmic receptor for the Tobacco mosaic virus cell-to-cell movement protein, facilitates intercellular transport through plasmodesmata. PLoS Pathog. 6, e1001201-e1001201 (2010).
- Coligan, J. E., Dunn, B. M., Ploegh, H. L., Speicher, D. W., Wingfield, P. T., Taylor, G. . , (2011).
- Tzfira, T., Vaidya, M., Citovsky, V. VIP1, an Arabidopsis protein that interacts with Agrobacterium VirE2, is involved in VirE2 nuclear import and Agrobacterium infectivity. EMBO J. 20, 3596-3607 (2001).
- Chen, M. H., Sheng, J., Hind, G., Handa, A., Citovsky, V. Interaction between the tobacco mosaic virus movement protein and host cell pectin methylesterases is required for viral cell-to-cell movement. EMBO J. 19, 913-920 (2000).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados