Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La evaluación de la hipermutación somática en Ramos células B después de sobreexpresión o derribo de genes específicos
En este artículo
Resumen
Se describe cómo realizar las infecciones por retrovirus o lentivirus de la sobreexpresión o siRNA que contienen las construcciones en la línea de Ramos humanos de células B y la forma de medir la hipermutación somática en estas células.
Resumen
Las células B comienzan su vida con los anticuerpos de baja afinidad generado por V (D) J recombinación. Sin embargo, al detectar un patógeno, la variable (V) la región de una inmunoglobulina (Ig) del gen se encuentra mutado aproximadamente 100.000 veces más que el resto del genoma a través de hipermutación somática (SHM), resultando en una alta afinidad 1,2 anticuerpos. Además, la recombinación de cambio de clase (CSR) produce anticuerpos con diferentes funciones efectoras dependiendo del tipo de respuesta inmune que se necesita para un patógeno en particular. Tanto la RSE y SHM se inician por la citidina deaminasa inducida por activación (AID), que desamina residuos de citosina en el ADN para producir uracilos. Estos uracilos son procesados por las formas propenso a errores de las vías de reparación, con el tiempo provocando mutaciones y recombinación 3.1.
Nuestra actual comprensión de los detalles moleculares de SHM y RSE provienen de una combinación de estudios en ratones, las células primarias, las líneas celulares, y el F-Cellexperimentos de REE. Modelos de ratones siguen siendo el estándar de oro con nocauts genética que muestra un papel crítico para la reparación de muchos factores (por ejemplo, Ung, MSH2, MSH6, Exo1, y de la polimerasa η) 4-10. Sin embargo, no todos los genes se prestan a los estudios de octavos de final. Por ejemplo, golpes de gracia de varios de doble filamento romper las proteínas de reparación son embrionariamente letal o poner en peligro el desarrollo de células B 14/11. Por otra parte, a veces la función específica de una proteína en SHM o CSR pueden estar enmascarados por más defectos global causado por el golpe de gracia. Además, puesto que los experimentos en ratones pueden ser largos, alterando la expresión de genes individuales en las líneas celulares se ha convertido en un primer paso cada vez más popular para la identificación y caracterización de genes candidatos 15-18.
Ramos - un linfoma de Burkitt línea celular que constitutivamente se somete a SHM - ha sido una popular línea celular modelo para estudiar SHM 18-24. Una de las ventajas de las células de Ramos es que tienen una semi-construido en práctica-Cuantitativa medida de SHM. Las células de tipo salvaje expresa IgM y, como recogen las mutaciones, algunas de las mutaciones eliminar la expresión de IgM. Por lo tanto, analizando la pérdida de IgM mediante el escaneo de células activadas por fluorescencia (FACS) proporciona una rápida lectura para el nivel de SHM. Una medición más cuantitativa de la SHM se puede obtener por secuenciación directa de los genes de anticuerpos.
Dado que las células Ramos son difíciles de transfectar, producimos derivados estables que han aumentado o reducido la expresión de un gen individual al infectar las células con las construcciones retrovirales o lentiviral que contienen un casete de sobreexpresión o una corta horquilla de ARN (siRNA), respectivamente. Aquí se describe la forma en que infectan a las células Ramos y luego utilizar estas células para investigar el papel de genes específicos en el SHM (Figura 1).
Protocolo
1. Preparación de muestras y las células
- Realizar una preparación mediana escala de ADN (Midiprep) de la sobre-expresión o siRNA que contienen las construcciones y la retroviral vector pKat2 envases o embalajes de los vectores lentiviral pVSV-G, pMDLg / pRRE, y PRSV-Ap 25. Use 6 mg de ADN para construir cada uno y ya sea de 4 mg de pKat2 (para las infecciones por retrovirus) o 1,5 mg de ADN de cada uno de los pVSV-g, pMDLg / pRRE, y PRSV-Rev vectores de embalaje (para las infecciones por lentivirus).
- Prepare BOSC 23 medios de comunicación. Para la transfección, se usan ambas enmendada Dulbecco modificado de Eagle (DMEM), así como DMEM completo. Para hacer DMEM completo, añadir 1% de tampón HEPES, 1% de penicilina-estreptomicina-glutamina (PGS), y el 10% de suero de ternera recién nacida (NCS) a una botella de DMEM.
- Ramos preparar los medios de comunicación. Para el paso de la infección, que completa RPMI-1640 (RPMI) mediante la adición de HEPES 1%, PGS 1% y el 10% de suero fetal bovino (SFB) a una botella de RPMI. De paso la siembra de células, make "condicionada" por los medios de comunicación el crecimiento de células Ramos en RPMI completo durante 2 días, dando vueltas a las células hacia abajo a 400 X g durante 4 minutos y filtrar el sobrenadante con un filtro de 0,2 micras en un frasco estéril. Al igual que otros medios de comunicación completo, medio condicionado se almacena a 4 º C hasta que sea necesario.
- Split 3 x 10 6 BOSC 23 células en una placa de 100 mm en 10 ml de DMEM completo el día antes de la transfección de modo que las células serán menos 50-60% de confluencia el día de la transfección. Hacer una placa de células para cada vector tiene previsto transfección. Es importante que las células no se han mantenido en la cultura durante demasiado tiempo, el uso de las células bajo el número de pases o de las células descongeladas recientemente, tanto para la transfección y medidas infección para asegurar una buena infección.
- Incluyen controles para sus experimentos. Por transfections y las infecciones, crear un control negativo (células no transfectadas o infectadas) y un control positivo (virus que expresa GFP y el gen de resistencia a la puromicina). Para ARNhc derribo experiments, infectar a un pozo con un revuelto o el control del shRNA irrelevante.
2. Transfección de células BOSC 23
- Caliente las botellas de DMEM sin modificar y completar DMEM a 37 º C.
- Retire el medio de la BOSC 23 células y reemplazar con 5 ml de DMEM completo. Volver a las células a una incubadora a 37 ° C hasta su uso en el paso 2.6.
- Caliente el frasco de FuGENE 6 a temperatura ambiente durante 5 minutos y luego agitar brevemente (<1 segundo).
- Alícuota de 600 l DMEM sin modificaciones en un tubo de microcentrífuga de 1,5 ml.
- Diluir 30 l FuGENE 6 en los 600 l enmendada DMEM, agitar brevemente (<1 segundo), y luego se incuban a temperatura ambiente durante cinco minutos. Tenga cuidado de la pipeta directamente en los medios de comunicación. No deje que el FuGENE 6 toque los lados del tubo.
- Añadir 6 g construcción y, o bien 4 mg pKat2 (infecciones retrovirales) o 1,5 mg de cada uno de pVSV-G, pMDLg / pRRE, y PRSV-Ap (infecciones de lentivirus) para el DMEM / FuGENE 6 mezclar y agitar bri efly (<1 segundo), y luego se incuban a temperatura ambiente durante 25 minutos.
- Añadir el ADN / DMEM / FuGENE 6 de mezcla al BOSC 23 celdas y volver a una incubadora a 37 ° C durante 24 horas.
- Después de 24 horas, añadir 5 ml de DMEM completo a las células transfectadas y volver a una incubadora a 37 ° C durante otras 24 horas.
3. La recolección de partículas virales
- La cosecha de 10 ml de los medios de comunicación de la transfectadas BOSC 23 celdas con una jeringa de 10 ml.
- Adjuntar un 0,45 micras filtro hasta el final de la jeringa y filtro de los medios de comunicación en un tubo de polipropileno limpio 15 ml.
- Utilice los medios de comunicación viral o congelar inmediatamente como partes alícuotas de 1 ml en viales de 2 ml criogénico a -80 ° C.
- Después de la cosecha los medios de comunicación viral, confirman que la transfección eficiente se ha producido en las células de control positivo por FACS. Esto también se puede hacer para todas las muestras si la construcción contiene un fluorescente (GFP, por ejemplo) o un marcador de superficie celular (por ejemplo, Thy1).
- Semillas de 2 ml de 2 x 10 5 células / ml de células Ramos en RPMI completo en una placa de 6 pocillos 24 horas antes de la infección. Una vez más, asegúrese de usar el paso número bajo de células o células recientemente descongelados para asegurar una buena infección.
- Si se usa alícuotas congeladas de virus, descongelar los medios de comunicación viral rápida a 37 ° C.
- Mezclar 1 ml de medio viral con 3 l de 10 mg / ml polibreno. La concentración final de polibreno debe ser de 10 mg / ml en un volumen total de 3 ml.
- Añadir la mezcla de virus / polibreno a las células y se mezcla bien con la pipeta. Como se mencionó anteriormente, un bien debe ser utilizado como un control no infectados, agregar un mL RPMI completo y 3 polibreno l a este pozo en lugar de virus de 1 ml. Asegúrese de infectar a un bien con un control pareado negativo. También infecta un bien con un vector que contiene las buenas prácticas agrarias, para determinar la eficiencia de infección por FACS.
- Se centrifuga la placa de 6 pocillos a 3000 X g durante 90 minutos a temperatura ambiente.
- Volver a la placauna incubadora de 37 ° C durante 48 horas.
- Después de 48 horas, la desaceleración de las células infectadas en un tubo de polipropileno de 15 ml a 400 X g durante 4 minutos a temperatura ambiente. Retire el papel por aspiración, asegurándose de no perturbar el sedimento celular. Resuspender las células en RPMI fresco.
- Confirmar la eficacia de la infección en la muestra de control positivo por FACS. Como antes, usted puede hacer esto para todas sus muestras si la construcción contiene un fluorescente (GFP, por ejemplo) o un marcador de superficie celular (por ejemplo, Thy1).
- Inicio de la selección de las células (si procede). Nuestra contiene shRNA construcciones contienen un casete de resistencia a la puromicina. Seleccionamos ARNhc las células infectadas por Ramos con 0,25 g / ml puromycin. Nos encontramos con que los derivados de las células de Ramos a veces muestran susceptibilidad alterada a la puromicina y, por tanto, llevamos a cabo para optimizar las curvas de matar a la concentración de puromicina para cada derivado. También el tratamiento de las células no infectadas con el control de puromicina para confirmar que la selección sea efectiva.
5. La siembra de células individuales
Siembra solo puede realizarse en una de dos maneras: por FACS de clasificación o de forma manual.
- FACS: Use un clasificador FACS con un adaptador de la placa a la semilla una célula / pocillo en una placa de 96 pocillos pre-llenado con 100 L de acondicionado del 40% RPMI/40% media/20% de SFB. Este método proporciona una gran cantidad de clones de células más simple por placa.
- De forma manual: el Conde de las células. Diluir una alícuota de células a 10 células / ml en el 40% RPMI/40% de SFB acondicionado media/20%. Utilizar 10 mL de células diluidas por cada placa sembrada. Clones diferentes variables muestran una recuperación en esta etapa por lo tanto, es útil la creación de varias placas a diferentes concentraciones (de 3 a 20 células / ml). Utilice el que produce menos crecimientos para evitar los clones derivados de varias celdas). Use una pipeta multicanal a las semillas placas de 96 pocillos con 100 L de células diluidas.
- Colocar la placa en una incubadora a 37 ° C durante 2 semanas.
- Compruebe cada unde los pozosr el microscopio para confirmar la presencia de células de un día después de la siembra. Las células individuales puede ser difícil de encontrar debido a la necesidad de centrarse en la profundidad correcta. Centrándose en los números y estampan en la parte inferior de la placa de 96 pocillos debe ayudar. Las células son más fáciles de encontrar cuando se sembró por el clasificador FACS ya que la mayoría de los pozos tienen un celular.
- Después de 2 semanas, las colonias que crecen pueden ser apenas visibles a simple vista mirando las placas de abajo. Son mucho más fáciles de ver bajo el microscopio. Colonias tienden a crecer a lo largo de los bordes de las colonias más bien y por lo general en una placa de crecer a lo largo del borde mismo en diferentes pozos. Marcar pozos con el crecimiento celular robusto y la transferencia de las células para placas de 24 pocillos en 1 ml de RPMI y volver a una incubadora a 37 ° C.
- Mantener las células en 1 x 10 5 a 1 x 10 6 células / ml. Cambiar los medios de comunicación haciendo girar las células en 400 X g durante 4 minutos a temperatura ambiente y la eliminación de los medios de comunicación, según sea necesario. Reemplazar con una RPMI ml fresco.
- Después de varios days de crecimiento, la transferencia de las células en una placa de 6 pocillos y seguir creciendo en una incubadora a 37 ° C.
- Después de 3 semanas, analizar los clones para la pérdida de IgM (como se describe más abajo).
- Siguen creciendo las células durante 2 semanas más y luego analizar los clones de nuevo en el punto 5 semanas de tiempo.
6. Análisis: la pérdida de IgM (3 semanas y 5 semanas)
- Alícuota de 5 x 10 5 células de cada pocillo en un tubo de microcentrífuga de 1,5 ml y el giro de las células hacia abajo a 400 X g durante 4 minutos a temperatura ambiente. Utilizar las células de cada uno de los pozos de control y shRNA knock-down, así como una muestra de un control sin mancha.
- Lavar las células en 1 ml de PBS y luego girar de nuevo a 400 X g durante 4 minutos a temperatura ambiente.
- Resuspender en 50 l de PBS (control sin tinción) o PBS + dilución 1:20 de la EP-anticuerpo marcado con α-IgM humano. Incubar en hielo durante 1 hora.
- Giro de las células hacia abajo a 400 X g durante 4 minutos a temperatura ambiente.
- Lave la cells dos veces con 1 ml de PBS y el giro de 400 X g durante 4 minutos cada vez a temperatura ambiente.
- Eliminar el sobrenadante y resuspender en 100 l de PBS. La transferencia de las células teñidas resuspendido a un tubo de FACS.
- Analizar las células utilizando una máquina de FACS.
- Comparar el porcentaje de IgM - las células de los controles frente a la shRNA derribado las células. Como se señaló anteriormente, la pérdida de IgM, medido por FACS es un semi-cuantitativos fiables medida de SHM en las células de Ramos.
7. Análisis: Confirmar knock-down por RT-PCR cuantitativa (5 semanas)
- Alícuota de 3 x 10 6 células de cada bien y centrifugar a 400 X g durante 4 minutos a temperatura ambiente.
- Prepare ARN a partir de la alícuota de las células. Utilizamos TRIzol de acuerdo con las recomendaciones del fabricante.
- Preparar cDNA a partir de la preparación de ARN. Usamos 2 mg de ARN y II Superíndice de acuerdo con las recomendaciones del fabricante.
- Realizar una PCR cuantitativa utilizando el cDNA de confirmaciónm knock-down. Utilizamos GAPDH como control de normalización, y el kit de SYBR qPCR rápido en un volumen de 10 l de reacción total.
8. Análisis: la secuenciación genómica (5 semanas)
- Alícuota de 5 x 10 6 células de cada bien y centrifugar a 400 X g durante 4 minutos a temperatura ambiente.
- Aislar el ADN genómico a partir de estas células. Utilizamos el Asistente para SV genómica del ADN kit de purificación de acuerdo a las recomendaciones del fabricante.
- Amplificar por PCR del DNA genómico de los genes de Ig V 19 (Tabla 1) y otros genes de interés utilizando la polimerasa KAPA HiFi ADN. Nosotros usamos esta polimerasa, debido a su baja las tasas de error, pero cualquier polimerasa de alta fidelidad PCR debería ser aceptable.
- Gel purificar los productos de PCR en un 1% en gel de agarosa utilizando el kit de extracción de gel QIAquick.
- Algunas polimerasas añadir una sola desoxiadenosina (A) de residuos de los extremos 3 'de los productos de PCR, mientras que otros no lo hacen. Estos tres 'A las colas son necesarios para la asistencia técnica TOPOetapa de clonación. La polimerasa KAPA HiFi no deja 3 'A las colas. Una cola de los productos de PCR mediante la incubación de 25 l de gel purificado producto de PCR con 3 l de amortiguación 10 veces la reacción de Taq, 1 l 10 mM dNTPs, y 1 unidad de Taq polimerasa a 72 º C durante 30 minutos.
- Clon de los productos purificados gel-PCR utilizando el kit de clonación TOPO TA. Transformar los productos de la reacción en el TOPO siempre E. coli DH5a R-T1 células competentes y la placa en placas de LB / agar que contiene 100 mg / ml ampicilina y se completará con 1,6 mg de X-gal de acuerdo con las recomendaciones del fabricante.
- Enviar colonias individuales para la secuenciación.
- Realizar un análisis de secuencia para determinar los tipos y ubicaciones de las mutaciones.
9. Los resultados representativos:
Como se señaló anteriormente, se utiliza un vector viral de control positivo que se expresa tanto en las buenas prácticas agrarias, así como un gen de resistencia a la puromicina. Vemos típicamente un 50-75% células GFP + dos díasdespués de la transfección. En nuestro infecciones, vemos típicamente un 50-70% son células GFP + dos días después de la infección, pero antes de la selección. Después de la selección está completa,> 95% de las células GFP +.
Como representante de los experimentos, se muestran los efectos de la ayuda que sobreexpresan o derribar un factor de reparación en las células de Ramos. En concreto, transfectadas un vector retroviral para la sobreexpresión de las ayudas vinculadas a través de un sitio a IRES Thy1.1 junto con el vector retroviral embalaje pKat2 BOSC en 23 celdas. Que contiene el virus de medios de comunicación se filtró y luego se utiliza para infectar a las células Ramos. Tanto de tipo salvaje (WT) y ayudar a las células que sobreexpresan (AID HI), sola célula sin semillas y cultivadas durante 3 semanas, momento en el que se analizaron para la expresión de genes de QRT-PCR (Figura 2) y para la pérdida de IgM por FACS (Figura 3 ). Para la tinción FACS, se incubaron las células con ambos diluidos 1:20 PE-etiquetados α-anticuerpos humanos IgM, así como 1:400 diluido FITC α-rata CD90/mouseCD90.1 (Thy1.1) de anticuerpos. En un experimento separado, los lentivirus que contienen una irrelevante shRNA (control) o un siRNA contra un factor de reparación de ADN de alta fidelidad, espera que proteja contra la SHM se hicieron en BOSC 23 células, y luego infectados en un clon de células de alta AID Ramos. Una vez más, la expresión de genes (Figura 4) y la pérdida de IgM (Figura 5) se analizaron en los clones de células solo después de 3 semanas. Ya que el factor de reparación se piensa para proporcionar protección contra las mutaciones durante SHM, vemos un aumento en la pérdida de IgM y SHM, en ausencia del factor. Si hubiéramos derribado un factor involucrado en la generación de SHM mutaciones asociadas, que se ha visto una disminución en la pérdida de IgM en su lugar.
Como era de esperar, nuestro QRT-PCR resultados muestran un marcado incremento en la expresión de AID después de la infección de las células con un vector de sobreexpresión de la AID (Figura 2), mientras que los niveles de la caída de ADN factor de reparar en la presencia de un siRNA dirigido contra ese factor (Figura 4 ). Debido a que cada clones puede variar, puede ver alguna variación en los niveles de expresión génica. Debido a que la expresión de genes puede variar, es necesario para confirmar la expresión de cada clon tiene previsto analizar más a fondo. ShRNAs diferentes contra el mismo gen puede tener diferentes efectos sobre la expresión génica y, por lo que deben investigar varias shRNAs para determinar cuál tiene el mayor impacto. Caída también puede ser confirmado en el nivel de proteínas por inmunotransferencia. Del mismo modo, los clones individuales pueden variar en gran medida en los niveles de pérdida de IgM. Algunos clones podría demostrar un "jackpot" efecto, en una mutación de IgM se produjo en una de las divisiones primera célula - por ejemplo, verá> 50% de pérdida de IgM, simplemente porque una de las celdas se convirtió en IgM - en la etapa de dos células. La influencia de estos clones se reduce al mínimo mediante el cálculo de la media de varios clones de células individuales.

Figura 1. Esquema de la red transeuropeainfección, la infección y el análisis de la pérdida de IgM. Ver texto para más detalles.
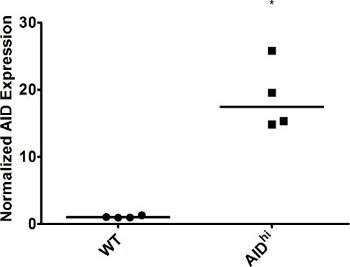
Figura 2. AID sobreexpresión en células de Ramos. WT y ayudar a las células de alta Ramos fueron célula clonada y se cultivan durante tres semanas, en el que se midió la expresión perspectiva de las ayudas en los distintos clones de QRT-PCR. Expresión de GAPDH se utilizó para la normalización. WT se establece en 1. El valor de la mediana se indica. * Indica un valor de p <0,01, calculado como un estudiante de una cola de la t-test.
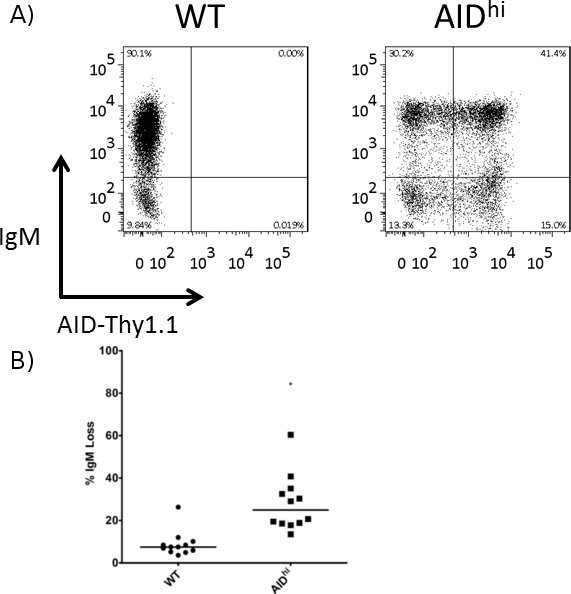
Figura 3. Aumento de la pérdida de IgM en las células de alta AID, en comparación con las células WT. IgM de superficie se midió por FACS en el punto 3 semanas de tiempo, y el porcentaje de pérdida de IgM se calculó para el TE y las células AID alta. (A) Representante FACS trama de un clon de un clon de WT y AID alta. La AID sobreexpresión vHéctor tiene la ayuda vinculada a Thy1.1 través de un sitio IRES, por lo que Thy1.1 expresión se usa como un sustituto de la expresión de la AID. (B) Cuantificación de la pérdida de IgM en WT y varios clones de alta AID. El valor de la mediana se indica. * Indica un valor de p <0,01, calculado como un estudiante de una cola de la t-test.
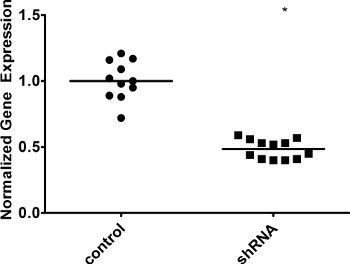
Figura 4. Derribo de un factor de reparación en las células de alta AID. Las células fueron infectadas, ya sea con un irrelevante el control shRNA (control) o nuestra ARNhc de interés (shRNA), y la única célula cabeza de serie. Después de 3 semanas, la expresión génica en clones individuales se midió mediante QRT-PCR. Expresión de GAPDH se utilizó para la normalización. WT se establece en 1. El valor de la mediana se indica. * Indica un valor de p <0,01, calculado como un estudiante de una cola de la t-test.
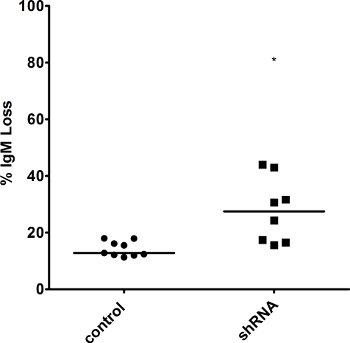
Figura 5. Aumento de la pérdida de IgMen las células con niveles de alta AID derribado de un factor de reparación. IgM de superficie en varios clones se midió por FACS en el punto 3 semanas de tiempo, y el porcentaje de pérdida de IgM se calculó en las células que sobreexpresan sigue siendo una ayuda. El valor de la mediana se indica. * Indica un valor de p <0,01, calculado como un estudiante de una cola de la t-test.

Tabla 1. Primer secuencias de los genes de Ig 19: el Cμ región constante, la cadena pesada de la región V (V H), y la cadena ligera de la región V (V L).
Discusión
Como se mencionó anteriormente, los modelos de líneas celulares para la diversificación de anticuerpos se han convertido en un popular punto de partida para identificar nuevas proteínas que influyen en los diferentes pasos en la diversificación de anticuerpos. En este trabajo presentamos un método para el uso de la infección viral a las proteínas ya sea derribo o sobre-expresan en los Ramos de células B línea y luego examinar el impacto en SHM.
Para estos estudios, que utilizan tan...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
El pMSCV-AID-I-Thy1.1 y pKat2 vectores eran una especie de regalo de la Dirección General de Schatz y el pVSV-G, PRSV Ap-, y pMDLg / pRRE vectores eran una especie de regalo de BR Cullen.
Materiales
Reactivos sugiere - la mayoría de estos pueden ser sustituidos con productos similares de otros fabricantes.
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
|---|---|---|---|
| 6-y claro TC tratados con placas | Corning | 3516 | |
| 10 ml BD Luer-Loksyringes | BD Medical | 309604 | |
| 24-alejado TC-tratados placas | Corning | 3526 | |
| De 96 pocillos de poliestireno claro fondo plano TC-tratados microplacas | Corning | 3596 | |
| 100 mm TC-tratados placas de cultivo | Corning | 430167 | |
| Filtros de jeringa Acrodisc, 0,45 m | Pall Life Sciences | 4604 | |
| Agar | Teknova | A7777 | |
| Agarosa | GeneMate | E-3120-500 | |
| Ampicilina | Sigma | A0166 | 100 mg / ml en agua |
| BD FACSCanto II citómetro de flujo | BD Biosciences | o similar | |
| BD Falcon tubos redondos inferior de poliestireno | BD Biosciences | 352054 | por FACS |
| BOSC 23 celdas | ATCC | CRL-11270 | |
| CO 2 capaz de incubadora a 37 ° C | |||
| DMEM (Dulbecco modificado de Eagle) | Sigma | D6429 | |
| FBS (suero fetal bovino) | Gemini Bio-productos | 100-106 | |
| FITC α-rata CD90/mouse CD90.1 anticuerpos | BioLegend | 202503 | FITCα-Thy1.1 |
| FuGENE 6 Transfección reactivo | Roche | 11814443001 | |
| Solución tampón HEPES | Invitrogen | 15630-080 | |
| KAPA HiFi ADN polimerasa | KAPA Biosystems | KK2101 | |
| LB caldo (caldo de lisogenia - Luria) en polvo | Difco | 240230 | |
| MISIÓN TRC ARNhc acciones glicerol bacteriana | Sigma | ARNhc vectores | |
| NCS (suero de ternera recién nacido) | Gemini Bio-productos | 100-504 | |
| PBS (solución buffer fosfato) | Invitrogen | 70011 | diluye a 1x en el agua |
| PE-α humana de anticuerpos IgM | BioLegend | 314508 | |
| PGS (penicilina-estreptomicina-glutamina solución) | Gemini Bio-Productos | 400-110 | |
| Polybrene (Hexadimetrina bromuro) | Sigma | 107689 | 10 mg / ml en agua |
| PureYield plásmido sistema Midiprep | Promega | A2495 | |
| Puromicina | Sigma | P8833 | 250 pg / mL en el agua |
| QIAquick gel extracción kit | QIAGEN | 28706 | |
| Ramos (AR 1) las células | ATCC | CRL-1596 | |
| RPMI-1640 | Sigma | R8758 | |
| Superíndice II | Invitrogen | 18064-022 | |
| SYBR RÁPIDO qPCR kit | KAPA Biosystems | KK4601 | |
| Taq ADN polimerasa | Invitrogen | 18038-042 | |
| TOPO TA kit de clonación | In vitrogeneración | K4520-01 | |
| TRIzol | Invitrogen | 15596-026 | |
| Asistente de purificación de ADN genómico SV sistema | Promega | A2361 | |
| X-Gal [5-bromo-4-cloro-3-β-indoyl-D-galatopyranoside] | Growcells | C-5687 | 40 mg / ml en DMSO |
Referencias
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annu. Rev. Biochem. 76, 1-22 (2007).
- Peled, J. U. The biochemistry of somatic hypermutation. Annu. Rev. Immunol. 26, 481-511 (2008).
- Longerich, S., Basu, U., Alt, F., Storb, U. AID in somatic hypermutation and class switch recombination. Curr. Opin. Immunol. 18, 164-174 (2006).
- Bardwell, P. D. Altered somatic hypermutation and reduced class-switch recombination in exonuclease 1-mutant mice. Nat. Immunol. 5, 224-229 (2004).
- Martomo, S. A., Yang, W. W., Gearhart, P. J. A role for Msh6 but not Msh3 in somatic hypermutation and class switch recombination. J. Exp. Med. 200, 61-68 (2004).
- Phung, Q. H. Increased hypermutation at G and C nucleotides in immunoglobulin variable genes from mice deficient in the MSH2 mismatch repair protein. J. Exp. Med. 187, 1745-1751 (1998).
- Rada, C., Ehrenstein, M. R., Neuberger, M. S., Milstein, C. Hot spot focusing of somatic hypermutation in MSH2-deficient mice suggests two stages of mutational targeting. Immunity. 9, 135-141 (1998).
- Rada, C. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Curr. Biol. 12, 1748-1755 (2002).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase eta is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. J. Exp. Med. 204, 17-23 (2007).
- Masuda, K. DNA polymerase eta is a limiting factor for A:T mutations in Ig genes and contributes to antibody affinity maturation. Eur. J. Immunol. 38, 2796-2805 (2008).
- Lim, D. S., Hasty, P. A mutation in mouse rad51 results in an early embryonic lethal that is suppressed by a mutation in p53. Mol. Cell. Biol. 16, 7133-7143 (1996).
- Xiao, Y., Weaver, D. T. Conditional gene targeted deletion by Cre recombinase demonstrates the requirement for the double-strand break repair Mre11 protein in murine embryonic stem cells. Nucleic. Acids. Res. 25, 2985-2991 (1997).
- Zhu, J., Petersen, S., Tessarollo, L., Nussenzweig, A. Targeted disruption of the Nijmegen breakage syndrome gene NBS1 leads to early embryonic lethality in mice. Curr. Biol. 11, 105-109 (2001).
- Luo, G. Disruption of mRad50 causes embryonic stem cell lethality, abnormal embryonic development, and sensitivity to ionizing radiation. Proc. Natl. Acad. Sci. U. S. A. 96, 7376-7381 (1999).
- Pavri, R. Activation-induced cytidine deaminase targets DNA at sites of RNA polymerase II stalling by interaction with Spt5. Cell. 143, 122-133 (2010).
- Lee-Theilen, M., Matthews, A. J., Kelly, D., Zheng, S., Chaudhuri, J. CtIP promotes microhomology-mediated alternative end joining during class-switch recombination. Nat. Struct. Mol. Biol. 18, 75-79 (2011).
- Basu, U. The RNA exosome targets the AID cytidine deaminase to both strands of transcribed duplex DNA substrates. Cell. 144, 353-363 (2011).
- Yabuki, M., Fujii, M. M., Maizels, N. The MRE11-RAD50-NBS1 complex accelerates somatic hypermutation and gene conversion of immunoglobulin variable regions. Nat. Immunol. 6, 730-736 (2005).
- Sale, J. E., Neuberger, M. S. TdT-accessible breaks are scattered over the immunoglobulin V domain in a constitutively hypermutating B cell line. Immunity. 9, 859-869 (1998).
- Cumbers, S. J. Generation and iterative affinity maturation of antibodies in vitro using hypermutating B-cell lines. Nat. Biotechnol. 20, 1129-1134 (2002).
- Papavasiliou, F. N., Schatz, D. G. Cell-cycle-regulated DNA double-stranded breaks in somatic hypermutation of immunoglobulin genes. Nature. 408, 216-221 (2000).
- Parsa, J. Y. AID mutates a non-immunoglobulin transgene independent of chromosomal position. Mol. Immunol. 44, 567-575 (2007).
- Zhang, W. Clonal instability of V region hypermutation in the Ramos Burkitt's lymphoma cell line. Int. Immunol. 13, 1175-1184 (2001).
- Ukai, A. Induction of a:T mutations is dependent on cellular environment but independent of mutation frequency and target gene location. J. Immunol. 181, 7835-7842 (2008).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. J. Virol. 72, 8463-8471 (1998).
- Nakamura, M. High frequency class switching of an IgM+ B lymphoma clone CH12F3 to IgA+ cells. Int. Immunol. 8, 193-201 (1996).
- Muramatsu, M. Specific expression of activation-induced cytidine deaminase (AID), a novel member of the RNA-editing deaminase family in germinal center B cells. J. Biol. Chem. 274, 18470-18476 (1999).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados