JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
특정 유전자의 Overexpression 또는 최저 후 라모스의 B 세포에서 체세포 Hypermutation을 평가
요약
우리는 overexpression 또는 shRNA 함유 인간 라모스 B - 세포주에서 어떻게 이러한 세포에 체세포 hypermutation를 측정하는 구성합니다.의 retroviral 또는 lentiviral 감염을 수행하는 방법을 설명합니다
초록
B 세포는 V (D) J 재조합에 의해 생성된 낮은 친화 항체와 그들의 인생을 시작합니다. 그러나 병원체를 감지시, 면역 글로불린 (IG) 유전자의 변수 (V) 지역은 높은 친화 항체 1,2의 결과로, 약 100,000 배 더 체세포 hypermutation (SHM)을 통해 게놈의 나머지 부분보다 변이이다. 또한, 클래스 스위치 재조합 (CSR)은 특정 병원체에 대한 필요한 면역 반응의 종류에 따라 다른 이펙터 기능을 가진 항체를 생산하고 있습니다. CSR과 SHM 모두 uracils 생산 DNA의 사이 토신 잔류물을 deaminates 활성화 유발 cytidine의 deaminase (AID)에 의해 시작됩니다. 이러한 uracils 결국 돌연변이와 재조합 1-3 이어지는, 복구 경로의 오류가 발생하기 쉬운 형태로 처리됩니다.
SHM와 CSR의 분자 자세한 내용은 우리의 현재 이해가 생쥐의 연구 조합에서 온, 기본 셀, 셀 라인, 세포 - F합 실험. 마우스 모델은 많은 수리 요인 (예 : 엉, Msh2, Msh6, Exo1, 그리고 효소 η) 40-10 위해 중요한 역할을 보여주는 유전자 knockouts있는 황금 표준 남아 있습니다. 그러나, 모든 유전자 녹아웃 연구에 대한 의무가 있습니다. 예를 들어, 여러 번 스트랜드 브레이크 수리 단백질의 knockouts는 embryonically 치명거나 B - 세포 개발에 11-14을 손상. 또한, 때로는 SHM 또는 CSR에서 단백질의 특정 기능은 녹아웃으로 인한 더 많은 글로벌 결함에 의해 마스크 수 있습니다. 또한, 마우스의 실험 이후 셀 라인에서 개별 유전자의 표현이 후보 유전자 15-18을 식별하고 특성화하기 위해 점점 더 인기를 첫 번째 단계가되었습니다 변경, 긴 수 있습니다.
라모스는 - constitutively SHM를 겪습 Burkitt의 림프종 세포 라인 - SHM 18-24 공부를하기 위해 인기있는 세포 라인 모델을하고 있습니다. 라모스 세포 중 하나 장점은 그들이 내장된 편리한 준결승있다는 것입니다SHM의 - 양적 측정합니다. 야생 타입 세포들은 돌연변이, IgM 표현을 때려 눕힌 변이의 일부를 데리러으로 IgM을 표현합니다. 따라서, 형광 - 활성 세포 검사 (FACS)의 IgM 손실을 시금 것은 SHM의 수준에 대한 읽기 밖으로 빠르게 제공합니다. SHM보다 양적 측정은 직접 항체 유전자를 시퀀싱하여 얻을 수 있습니다.
라모스의 세포가 transfect하기가 어렵습니다 때문에, 우리는 각각 overexpression 카세트 또는 짧은 머리 핀의 자형으로 RNA (shRNA) 중 하나를 포함하는 retroviral 또는 lentiviral 구조와 세포를 감염 증가 또는 개별 유전자의 표현을 떠난 이후 안정적인 파생 상품을 생산합니다. 여기, 우리가 라모스 세포를 감염 후 SHM에서 특정 유전자 (그림 1)의 역할을 조사하기 위해 이러한 세포를 사용하는 방법에 대해 설명합니다.
프로토콜
1. 준비 샘플 및 세포
- overexpression 또는 shRNA 함유 구성과 retroviral 벡터 pKat2 포장 또는 포장 lentiviral 벡터 pVSV - G, pMDLg / pRRE 및 pRSV - 레브 25.의 DNA (midiprep)의 중간 규모 준비를 수행 각각의 구성 6 μg의 DNA와 pVSV - G, pMDLg / pRRE 각각에 대해 하나 pKat2 4 μg (retroviral 감염의 경우) 또는 1.5 μg의 DNA와 pRSV - 수익 포장 벡터 (lentiviral 감염)을 사용합니다.
- BOSC 23 미디어를 준비합니다. transfection 들어, unamended Dulbecco의 수정 이글 배지 (DMEM)뿐만 아니라 완전한 DMEM을 모두 사용합니다. 완전한 DMEM을 만들려면, DMEM의 병 1 %의 HEPES 버퍼, 1 % 페니실린 - 스트렙토 마이신 - 글루타민 (PGS)와 10 %의 신생아 송아지 혈청 (NCS)를 추가합니다.
- 라모스 미디어를 준비합니다. 감염 단계, RPMI 한병에 1 % HEPES, 1 % PGS,와 10 % 태아 소 혈청 (FBS)을 추가하여 RPMI - 1640 배지 (RPMI)을 완료합니다. 단세포 시딩 단계, M에 대한2 일간 전체 RPMI에서 라모스 세포 성장을 사분 400 X g에서 세포를 아래로 회전하고 멸균 병에 필터 0.2 μm의로 뜨는을 필터링하여 착륙 "시설"미디어. 다른 전체 미디어와 마찬가지로 에어컨 미디어가 필요 ° C까지 4 저장됩니다.
- 세포 50~60%에 transfection의 confluency 날이 될 transfection 전에 날이 10 ML 완전한 DMEM에서 100 mm 판으로 분할 3 X 10 6 BOSC 23 세포. 당신이 transfecting 계획의 각 벡터에 대한 세포 중 하나가 접시를 확인합니다. 세포가 너무 오랫동안 문화에 보관하지 않은 것이 중요하며 transfection하고 좋은 감염을 확보하기 위해 감염 단계에 모두 낮은 통로 번호 세포 또는 최근 해동 세포를 사용합니다.
- 실험에 대한 컨트롤을 포함합니다. transfections 및 감염, 부정적인 제어 (untransfected 또는 감염되지 않은 세포)와 긍정적인 제어 (GFP 및 puromycin - 저항 유전자를 표현 바이러스)를 만들 수 있습니다. shRNA 노크 다운 exper 위해iments은 스크램블이나없는 shRNA 제어와 잘 감염.
2. Transfecting BOSC 23 세포
- 37 unamended DMEM하고 완전한 DMEM의 병 따뜻한 ° C.
- BOSC 23 세포에서 미디어를 제거하고 완전한 DMEM 5 ML로 바꿉니다. 2.6 단계에서 사용할 때까지 37 ° C 배양기에 세포를 반환합니다.
- (<1 초) 후 간단히 와동 5 분 상온에서 FuGENE 6 병을 따뜻한.
- 1.5 ML의 microcentrifuge 튜브로 나누어지는 600 μL unamended DMEM합니다.
- 600 μL unamended DMEM, 와류 간단히 (<1 초)로 30 μL FuGENE 6 희석 후 5 분 동안 상온에서 부화. 미디어에 직접 피펫에주의하십시오. FuGENE 6 튜브의 측면을 만지지 않도록하십시오.
- DMEM / FuGENE 6 믹스, 소용돌이 브리 6 μg 구조 중 4 μg pKat2 (retroviral 감염) 또는 1.5 μg pVSV - G, pMDLg / pRRE 각각의, 그리고 pRSV - 레브 (lentiviral 감염)을 추가 다음 efly (<1 초), 그리고 25 분 동안 상온에서 부화.
- BOSC 23 세포에 대한 DNA / DMEM / FuGENE 6 믹스를 추가하고 24 시간 동안 37 ° C 배양기로 돌아갑니다.
- 24 시간 후에, transfected 세포 5 ML 완전한 DMEM을 추가하고 24 시간 동안 37 ° C 배양기로 돌아갑니다.
3. 바이러스 입자를 수확
- 10 ML의 주사기로 transfected BOSC 23 세포에서 미디어의 10 ML을 수확.
- 주사기의 끝에 필터 0.45 μm의를 부착하고 깨끗한 15 ML의 폴리 프로필렌 튜브에 미디어를 필터링합니다.
- -80에서 즉시 바이러스 미디어를 사용하거나 두 ML 극저온 튜브 1 ML aliquots로 동결 ° C.
- 바이러스 미디어를 수확 후 효율적인 transfection은 FACS에 의해 긍정적인 제어 세포에 발생했음을 확인합니다. 구조는 형광등 (예 : GFP) 또는 세포 - 표면 마커 (예 : Thy1) 중 하나를 포함하는 경우이 또한 모든 샘플을 할 수 있습니다.
- 24시간 감염 전에 6 자 플레이트에 전체 RPMI에서 2 × 10 5 세포 / ML 라모스 세포의 씨앗이 ML. 다시, 좋은 감염을 확보하기 위해 낮은 통로 번호 세포 또는 최근 해동 세포를 사용해야합니다.
- 바이러스의 냉동 aliquots를 사용하는 경우 37 빠르게 입소문 미디어를 녹여 ° C.
- 10 MG / ML polybrene 3 μL 1 개 ML 바이러스성 미디어를 섞는다. polybrene의 최종 농도는 10 μg / 3 ML의 총 볼륨에서 ML해야합니다.
- 세포에 바이러스 / polybrene 믹스를 추가하고 pipetting으로 잘 섞는다. 이 잘 대신 1 ML 바이러스 1 ML 완료 RPMI 3 μL polybrene을 추가, 앞서 하나는 잘 감염되지 않은 컨트롤로 사용해야합니다 언급했다. 일치 대조군 잘 하나를 감염해야합니다. 우리는 또한 FACS에 의한 감염의 효율성을 결정하기 위해, GFP 함유 벡터와 잘 하나를 감염.
- 상온에서 90 분간 3,000 X g에서 6 자 번호판을 원심 분리기.
- 에 번호판을 반환48 시간 동안 37 ° C 배양기.
- 48 시간 후, 실온에서 4 분간 400 X g에서 15 ML의 폴리 프로필렌 튜브에 감염된 세포를 다운 스핀. 세포 펠렛을 방해하지 않도록해야하고, 흡인하여 미디어를 제거합니다. 신선한 RPMI에서 세포를 Resuspend.
- FACS에 의해 긍정적인 컨트롤 샘플에서 감염의 효율성을 확인합니다. 구조는 형광등 (예 : GFP) 또는 세포 - 표면 마커 (예 : Thy1) 중 하나를 포함하는 경우 이전처럼, 당신은 모든 샘플이 작업을 수행할 수 있습니다.
- (적절한 경우) 세포의 선택을 시작합니다. 우리 shRNA 함유 구조는 모든 puromycin 저항 카세트가 포함되어 있습니다. 우리는 0.25 μg / ML puromycin과 shRNA - 라모스 감염 세포를 선택합니다. 우리는 라모스 세포의 파생 상품이 때로는 puromycin에 느끼기 쉬운 상태를 변경 표시하고, 따라서 우리는 각 파생에 대한 puromycin의 농도를 최적화하기 위해 곡선을 죽일 수행할 것을 발견했습니다. 또한 선택이 효과가 확인 puromycin과 감염되지 않은 컨트롤 세포를 치료.
5. 단세포 시딩
단일 시딩는 두 가지 방법 중 하나로 수행할 수 있습니다 : 정렬하거나 수동으로 FACS로.
- FACS : 씨앗 한 셀에 플레이트 어댑터 FACS 분류기를 사용하여 / 잘 96 - 웰 플레이트 40 % RPMI/40 % 시설 media/20 % FBS 100 μL 사전 가득. 이 방법은 많은에게 접시 당 하나의 세포 클론을 제공합니다.
- 수동 : 세포를 세어보세요. 40% RPMI/40 % 시설 media/20 % FBS 10 셀 / ML로 세포의 나누어지는을 희석. 각 씨앗 접시 희석 세포의 10 ML을 사용합니다. 다른 클론 따라서는 서로 다른 농도 (3 ~ 20 세포 / ML에 이르기까지)에 여러 접시를 설정하는 데 유용합니다이 단계에서 회복 변수를 표시합니다. 여러 세포에서 발생하는 클론을 피하기 위해 적은 outgrowths)를 생산 하나를 사용하십시오. 희석 세포 100 μL와 씨앗 96 - 웰 플레이트에 멀티채널 피펫을 사용합니다.
- 2 주 동안 37 ° C 배양기에서 접시를 놓습니다.
- 개인 우물 unde를 확인R 세포의 존재 시딩 후 하루 확인하는 현미경. 단일 세포가 올바른 깊이에 초점을 맞추고 필요에 따라 찾기 위해 까다로운 수 있습니다. 96 - 웰 플레이트의 하단에 인쇄 잘 숫자에 초점을하면 도움이 될 것입니다. 세포는 대부분의 우물이 휴대폰을 가지고부터 FACS의 분류기에서 씨앗을 찾을 때 쉽게됩니다.
- 이주 후, 성장 식민지는 거의 아래에서 접시를보고하여 육안으로 볼 수 있습니다. 그들은 현미경으로 볼 훨씬 쉽게됩니다. 식민지는 다른 우물에서 동일한 가장자리를 따라 성장 접시에 잘 그리고 일반적으로 대부분의 콜로니의 가장자리를 따라 성장하는 경향이있다. 마크 강력한 세포의 성장과 함께 우물 1 ML RPMI 24 - 잘 접시에 세포를 전송 및 37 ° C 배양기로 돌아갑니다.
- 1 X 10월 5일에서 1일까지 X 10 6 세포 / ML에서 세포를 유지합니다. 상온에서 사분 400 X g에서 세포를 회전하고 필요에 따라 미디어를 제거하여 미디어를 변경합니다. 한 ML 신선한 RPMI로 바꿉니다.
- 여러 D 후성장 ays는 6 잘 접시에 세포를 전송 및 37 ° C 배양기에서 성장을 계속합니다.
- 삼주 후, IgM 손실 (아래 설명)의 클론을 분석할 수 있습니다.
- 2 개 주 동안 세포 성장을 계속하고 5 주 시점에서 다시 클론을 분석할 수 있습니다.
6. 분석 : IgM의 손실 (삼주 5 주)
- 나누어지는 5 × 10 5 1.5 ML의 microcentrifuge 튜브에 각 우물에서 세포와 실온에서 4 분간 400 X g에서 세포를 다운 스핀. 흠없는 컨트롤에 대한 컨트롤과 shRNA - 똑 다운 우물의 각 세포뿐만 아니라, 한 샘플을 사용하십시오.
- PBS 1 ML에있는 세포를 씻어 후 실온에서 4 분간 400 X g에서 다시 돌린다.
- PBS (흠없는 제어) 또는 PBS의 50 μL에 Resuspend + PE - 라벨이 α - 인간 IgM 항체의 희석 1시 20분. 1 시간 동안 얼음에 품어.
- 상온에서 사분 400 X g에서 세포를 다운 스핀.
- C 씻으십시오두번 사분 400 X g을위한 PBS와 스핀 1 ML 실온에서 각 시간 ells.
- PBS 100 μL에 뜨는와 resuspend을 제거합니다. FACS 튜브에 resuspended 스테인드 세포를 전송합니다.
- FACS 기계를 사용하여 세포를 분석합니다.
- shRNA 노크 다운 세포 대 컨트롤의 세포 - IgM의 비율을 비교합니다. 마찬가지로 이전에 명시된 바와 같이, FACS로 측정으로 IgM 손실은 라모스 세포의 SHM의 신뢰할 수있는 세미 양적 측정하기위한 한 방법입니다.
7. 분석 : 정량 RT - PCR (5 주)에 의해 똑 다운 확인
- 나누어지는 3 X 10 6 각 우물에서 세포와 실온에서 4 분간 400 X g에서 다운 스핀.
- 세포의 나누어지는에서 RNA를 준비합니다. 우리는 제조 업체의 권장 사항에 따라 TRIzol을 사용합니다.
- RNA 초등학교에서 cDNA 준비합니다. 우리는 제조 업체의 권장 사항에 따라 2 μg RNA와 윗첨자 II를 사용합니다.
- confir로 cDNA를 사용하여 정량 PCR을 수행m 노크 다운. 우리는 10 μL 총 반응 부피의 정상화 제어 및 SYBR FAST qPCR 키트로 GAPDH를 사용합니다.
8. 분석 : 게놈 배열 (5 주)
- 나누어지는 5 X 10 6 각 우물에서 세포와 실온에서 4 분간 400 X g에서 다운 스핀.
- 이러한 세포의 게놈 DNA를 분리. 우리는 제조 업체의 권장 사항에 따라 마법사 SV의 게놈의 DNA 정제 키트를 사용합니다.
- PCR은 IG V 유전자 19 일 (표 1)과 KAPA 고음질의 DNA 효소를 사용하여 관심의 다른 유전자에 대한 게놈 DNA를 증폭. 우리 때문에 낮은 오류 요금이 효소를 사용할 수 있지만 어떤 높은 충실도 PCR 효소가 허용되어야합니다.
- 젤 QIAquick 겔 추출 키트를 사용하여 1 % 아가로 오스 겔에 PCR 제품을 정화.
- 다른하지는 않지만 일부 polymerases는 PCR 제품의 3 '끝을 단일 deoxyadenosine (A) 찌꺼기를 추가합니다. 이러한 3 '꼬리는 TOPO TA 필요합니다복제 단계. KAPA 고음질의 중합 효소 3 '꼬리를 남겨하지 않습니다. A - 꼬리 30 분 72 ° C에서 3 μL 10X DNA 형성 촉매 반응 버퍼, 1 μL 10 MM의 dNTPs, 1 단위 DNA 형성 촉매 효소 25 μL 젤 - 정화 PCR 제품을 잠복기로 PCR 제품.
- TOPO TA 클로닝 키트를 사용하여 겔 - 정화 PCR 제품을 복제. 제공된 E.에 TOPO 반응에서 제품을 변환 대장균 DH5α - T1 100 μg / ML 암피실린 및 제조 업체의 권장 사항에 따라 X - 여자의 1.6 MG로 보충을 포함한 LB / 한천 접시에 R 능력 셀 및 플레이트.
- 시퀀싱 개별 식민지를 보내십시오.
- 유형 및 변이의 위치를 결정하는 시퀀스 분석을 수행합니다.
9. 대표 결과 :
전에 바와 같이, 우리 둘 다 GFP뿐만 아니라 puromycin - 저항 유전자를 표현하는 긍정적인 제어 바이러스 벡터를 사용합니다. 우리는 일반적으로 이일 50-75% GFP + 세포를 참조하십시오transfection 후. 우리의 감염에서, 우리는 일반적으로 50~70% 세포가 + 이일 감염 후 있지만 선택하기 전에 GFP 알 수 있습니다. 선택이 완료되면, 세포의> 95 %는 GFP의 +입니다.
대표 실험으로, 우리는 overexpressing 보조 또는 라모스 세포의 수리 계수를 쓰러뜨린의 효과를 보여줍니다. 특히, 우리는 BOSC 23 세포에 retroviral 벡터 포장 pKat2과 함께 Thy1.1에 IRES 사이트를 통해 연결된 도움을 retroviral overexpression 벡터를 transfected. 바이러스가 포함된 미디어 필터링 후 라모스 세포를 감염하는 데 사용되었다. 모두 야생 유형 (WT)와 AID overexpressing (AID 안녕하세요) 전지는 하나의 세포 (그림 3 그들이 qRT - PCR (그림 2)와 FACS에서 IgM 손실에 대한 유전자 발현에 대한 분석되었던 시점에서 씨앗을 3 주 동안 성장했다 ). FACS의 얼룩은 전지 PE - 레이블 α - 인간 IgM 항체뿐만 아니라 1:400 희석 FITC - 라벨이 α - 쥐 CD90/mouse 희석 모두 1시 20분와 incubated되었습니다CD90.1 (Thy1.1) 항체. 별도의 실험에서, 두없는 shRNA (제어) 또는 SHM 방지하기 위해 기대 높은 충실도 DNA 수리 계수에 대한 shRNA를 포함한 lentiviruses은 BOSC 23 세포에서 만들어진 후 라모스 세포의 AID 안녕 클론에 감염. 다시 유전자 표현 (그림 4)와 IgM의 손실 (그림 5) 3 주 후에 단세포 클론에서 분석했다. 복구 계수가 SHM 동안 돌연변이에 대한 보호를 제공하기 위해 생각되기 때문에, 우리는 요소의 부재에서 IgM 손실 및 SHM 증가를 참조하십시오. 우리가 SHM - 연관된 변이를 생성과 관련된 요인 - 무너뜨렸다이있다면, 우리는 대신 IgM 손실의 감소를 본 것입니다.
예상했던대로, 우리 qRT - PCR 결과, 원조 overexpression 벡터 (그림 2)와 세포의 감염 다음과 같은 보조 표현에 표시된 증가를 보여 동안 요인에 대한 대상 shRNA의 존재의 DNA 복구 계수 드롭의 수준 (그림 4 ). 개인 clon 때문에초밖에는 다를 수 있습니다, 당신은 유전자 발현 수준에서 일부 변화를 볼 수 있습니다. 유전자 발현이 다를 수 있기 때문에, 그것은 당신이 더 이상 분석에 대한 계획을 각 클론의 표현을 확인하는 것이 필요합니다. 동일한 유전자에 대한 다양한 shRNAs는 물론, 그래서 당신이 가장 큰 영향을 결정하기 위해 여러 shRNAs을 스크린해야 유전자 발현에 다른 영향을 미칠 수 있습니다. 최저도 immunoblotting으로 단백질 수준에서 확인할 수 있습니다. 마찬가지로, 각각의 클론 IgM 손실 수준에서 크게 다를 수 있습니다. 일부 클론 IgM 변이는 초기 세포 부문 중 하나에서 발생하는 "잭팟"효과를 입증 수도 있습니다 - 예를 들어, 셀 중 하나가 IgM 된 단순히 때문에> 50 %의 IgM 손실을 볼 수 있습니다 - 두 세포 단계에서. 이 클론의 영향은 여러 단일 세포 클론에 대한 평균을 계산하여 최소화합니다.

그림 1. 트랜스에 대한 도식IgM 분실 fection, 감염 및 분석. 자세한 내용은 텍스트를 참조하십시오.
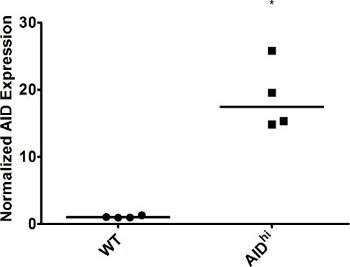
라모스 세포의 그림 2. 처치 overexpression. WT와 AID 안녕 라모스 세포는 각 클론의 포인트 보조 표현이 qRT - PCR로 측정되었다되는 단일 세포 복제와 3 주 동안 성장,했다. GAPDH의 표현은 정상화를 위해 사용되었다. WT는 1로 설정되었습니다. 평균 값이 표시됩니다. * 한 꼬리 학생의 T - 테스트로 계산 <0.01의 AP 가치를 나타냅니다.
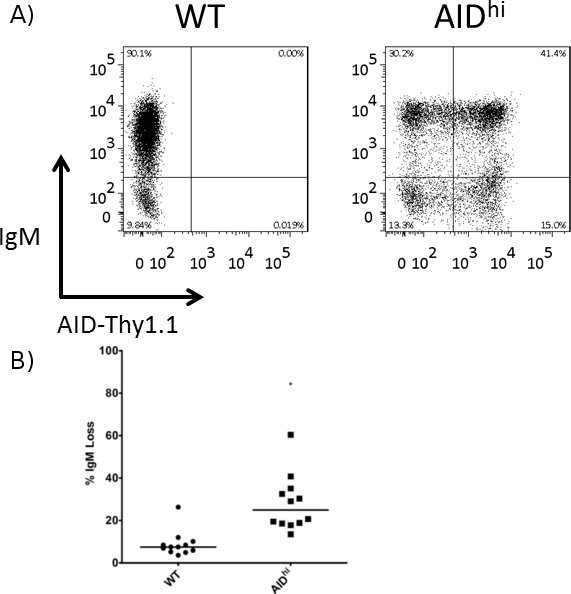
WT 세포에 비해 그림 3. 처치 안녕하세요 전지에서 IgM 손실을 증가시켰습니다. 표면 IgM은 3 주 시점에서 FACS로 측정되었고, IgM 손실의 비율이 WT와 원조 안녕 세포 계산되었다. WT 복제 및 AID 님 클론의 (A) 대표 FACS 계획. 도움 overexpression V경은 IRES 사이트를 통해 Thy1.1에 연결된 AID 정말 Thy1.1 표현식은 보조 표현 대리로 사용됩니다했다. 여러 WT와 AID 님 클론에서 IgM 손실 (B) Quantitation. 평균 값이 표시됩니다. * 한 꼬리 학생의 T - 테스트로 계산 <0.01의 AP 가치를 나타냅니다.
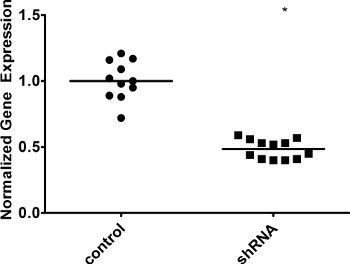
그림 4. 원조 안녕 세포의 수리 요소의 최저. 세포는 어느없는 shRNA 컨트롤 (제어) 또는 관심있는 우리 shRNA (shRNA) 및 단일 셀 씨앗에 감염되었습니다. 삼주 후, 각각의 클론의 유전자 발현은 qRT - PCR로 측정되었다. GAPDH의 표현은 정상화를 위해 사용되었다. WT는 1로 설정되었습니다. 평균 값이 표시됩니다. * 한 꼬리 학생의 T - 테스트로 계산 <0.01의 AP 가치를 나타냅니다.
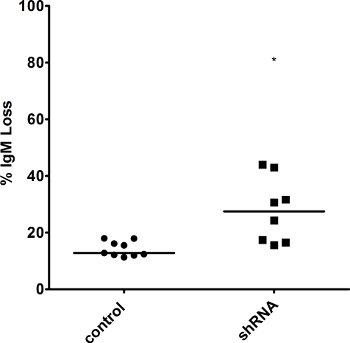
그림 5. 향상 IgM 손실복구 계수의 - 무너뜨렸다 수준 원조 안녕 세포 인치 여러 클론의 표면 IgM은 3 주 시점에서 FACS로 측정되었고, IgM 손실의 비율은 세포가 여전히 overexpressing 보조으로 계산되었다. 평균 값이 표시됩니다. * 한 꼬리 학생의 T - 테스트로 계산 <0.01의 AP 가치를 나타냅니다.

. IG의 유전자 19 표 1 뇌관 시퀀스 : 일정 지역 Cμ, 중쇄 V 영역 (V H)와 경쇄 V 영역 (V L).
Access restricted. Please log in or start a trial to view this content.
토론
마찬가지로 이전에 논의, 항체 다양성을위한 세포주 모델 항체 다변화하는 동안 여러 단계에 영향을 소설 단백질을 식별하는 인기있는 출발점이되고 있습니다. 우리는 여기서 라모스 B - 세포 라인에 노크 다운 또는 overexpress 중 단백질로 바이러스 감염을 사용하는 방법을 제시하고 SHM에 미치는 영향을 검사합니다.
이러한 연구에 대한, 우리는 WT 라모스와 AID 안녕 라...
Access restricted. Please log in or start a trial to view this content.
공개
관심 없음 충돌 선언하지 않습니다.
감사의 말
pMSCV - AID - I - Thy1.1과 pKat2 벡터는 DG Schatz와 pVSV - G, pRSV - 레브의 종류 선물했고, pMDLg / pRRE 벡터는 BR 컬른에서 어떤 선물했다.
Access restricted. Please log in or start a trial to view this content.
자료
추천 시약 - 이들의 대부분은 다른 업체에서 유사한 제품과 대체 수 있습니다.
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 |
|---|---|---|---|
| 6 - 잘 맑은 TC - 대우 접시 | 코닝 | 3516 | |
| 10 ML BD Luer - Loksyringes | BD 의료 | 309,604 | |
| 24 잘 맑은 TC - 대우 접시 | 코닝 | 3526 | |
| 96 - 웰 맑은 평면 바닥 폴리스티렌 TC - 대우 microplates | 코닝 | 3596 | |
| 100mm TC - 대우 문화 요리 | 코닝 | 430,167 | |
| Acrodisc 주사기 필터, 0.45 μm의 | 막 생명 과학 | 4604 | |
| 한천 | Teknova | A7777 | |
| 아가로 오스 | GeneMate | E - 3120-500 | |
| 암피실린 | 시그마 | A0166 | 물 100 밀리그램 / ML |
| BD FACSCanto II 흐름 cytometer | BD Biosciences | 또는 유사 | |
| BD 팔콘 원형 바닥 폴리스티렌 튜브 | BD Biosciences | 352,054 | FACS에 대한 |
| BOSC 23 세포 | ATCC | CRL - 11270 | |
| 37 수 CO 2 배양기 ° C | |||
| DMEM (Dulbecco의 수정 이글 배지) | 시그마 | D6429 | |
| FBS (태아 소 혈청) | 쌍둥이 바이오 제품 | 100-106 | |
| FITC α - 쥐 CD90/mouse CD90.1 항체 | BioLegend | 202,503 | FITCα - Thy1.1 |
| FuGENE 6 Transfection 시약 | 호슈 | 11814443001 | |
| HEPES 버퍼 솔루션 | Invitrogen | 15630-080 | |
| KAPA 고음질의 DNA 효소 | KAPA Biosystems | KK2101 | |
| LB 국물 (국물 lysogeny - Luria) 분말 | Difco | 240,230 | |
| 임무 TRC shRNA 세균 글리세롤 주식 | 시그마 | shRNA 벡터 | |
| NCS (신생아 송아지 혈청) | 쌍둥이 바이오 제품 | 100-504 | |
| PBS (인산염 버퍼 용액) | Invitrogen | 70,011 | 물에 1X로 희석 |
| PE α - 인간 IgM 항체 | BioLegend | 314,508 | |
| PGS (페니실린 - 스트렙토 마이신 - 글루타민 솔루션) | 쌍둥이 바이오 Products | 400-110 | |
| Polybrene (hexadimethrine 브로마이드) | 시그마 | 107,689 | 물 10 밀리그램 / ML |
| PureYield 플라스미드 Midiprep 시스템 | Promega | A2495 | |
| Puromycin | 시그마 | P8833 | 250 μg / 물 속에 ML |
| QIAquick 겔 추출 키트 | QIAGEN | 28,706 | |
| 라모스 (RA 1) 세포 | ATCC | CRL - 1596 | |
| RPMI - 1640 배지 | 시그마 | R8758 | |
| 어깨 II | Invitrogen | 18064-022 | |
| SYBR FAST qPCR 키트 | KAPA Biosystems | KK4601 | |
| DNA 형성 촉매 DNA 중합 효소 | Invitrogen | 18038-042 | |
| TOPO TA 복제 키트 | Invitro세대 | K4520 - 01 | |
| TRIzol | Invitrogen | 15596-026 | |
| 마법사 SV의 게놈 DNA의 정화 시스템 | Promega | A2361 | |
| X - 걸 [5 - 브로모 -4 - 클로로 - 3 - indoyl - β - D - galatopyranoside] | Growcells | C - 5687 | DMSO 40 밀리그램 / ML |
참고문헌
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annu. Rev. Biochem. 76, 1-22 (2007).
- Peled, J. U. The biochemistry of somatic hypermutation. Annu. Rev. Immunol. 26, 481-511 (2008).
- Longerich, S., Basu, U., Alt, F., Storb, U. AID in somatic hypermutation and class switch recombination. Curr. Opin. Immunol. 18, 164-174 (2006).
- Bardwell, P. D. Altered somatic hypermutation and reduced class-switch recombination in exonuclease 1-mutant mice. Nat. Immunol. 5, 224-229 (2004).
- Martomo, S. A., Yang, W. W., Gearhart, P. J. A role for Msh6 but not Msh3 in somatic hypermutation and class switch recombination. J. Exp. Med. 200, 61-68 (2004).
- Phung, Q. H. Increased hypermutation at G and C nucleotides in immunoglobulin variable genes from mice deficient in the MSH2 mismatch repair protein. J. Exp. Med. 187, 1745-1751 (1998).
- Rada, C., Ehrenstein, M. R., Neuberger, M. S., Milstein, C. Hot spot focusing of somatic hypermutation in MSH2-deficient mice suggests two stages of mutational targeting. Immunity. 9, 135-141 (1998).
- Rada, C. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Curr. Biol. 12, 1748-1755 (2002).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase eta is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. J. Exp. Med. 204, 17-23 (2007).
- Masuda, K. DNA polymerase eta is a limiting factor for A:T mutations in Ig genes and contributes to antibody affinity maturation. Eur. J. Immunol. 38, 2796-2805 (2008).
- Lim, D. S., Hasty, P. A mutation in mouse rad51 results in an early embryonic lethal that is suppressed by a mutation in p53. Mol. Cell. Biol. 16, 7133-7143 (1996).
- Xiao, Y., Weaver, D. T. Conditional gene targeted deletion by Cre recombinase demonstrates the requirement for the double-strand break repair Mre11 protein in murine embryonic stem cells. Nucleic. Acids. Res. 25, 2985-2991 (1997).
- Zhu, J., Petersen, S., Tessarollo, L., Nussenzweig, A. Targeted disruption of the Nijmegen breakage syndrome gene NBS1 leads to early embryonic lethality in mice. Curr. Biol. 11, 105-109 (2001).
- Luo, G. Disruption of mRad50 causes embryonic stem cell lethality, abnormal embryonic development, and sensitivity to ionizing radiation. Proc. Natl. Acad. Sci. U. S. A. 96, 7376-7381 (1999).
- Pavri, R. Activation-induced cytidine deaminase targets DNA at sites of RNA polymerase II stalling by interaction with Spt5. Cell. 143, 122-133 (2010).
- Lee-Theilen, M., Matthews, A. J., Kelly, D., Zheng, S., Chaudhuri, J. CtIP promotes microhomology-mediated alternative end joining during class-switch recombination. Nat. Struct. Mol. Biol. 18, 75-79 (2011).
- Basu, U. The RNA exosome targets the AID cytidine deaminase to both strands of transcribed duplex DNA substrates. Cell. 144, 353-363 (2011).
- Yabuki, M., Fujii, M. M., Maizels, N. The MRE11-RAD50-NBS1 complex accelerates somatic hypermutation and gene conversion of immunoglobulin variable regions. Nat. Immunol. 6, 730-736 (2005).
- Sale, J. E., Neuberger, M. S. TdT-accessible breaks are scattered over the immunoglobulin V domain in a constitutively hypermutating B cell line. Immunity. 9, 859-869 (1998).
- Cumbers, S. J. Generation and iterative affinity maturation of antibodies in vitro using hypermutating B-cell lines. Nat. Biotechnol. 20, 1129-1134 (2002).
- Papavasiliou, F. N., Schatz, D. G. Cell-cycle-regulated DNA double-stranded breaks in somatic hypermutation of immunoglobulin genes. Nature. 408, 216-221 (2000).
- Parsa, J. Y. AID mutates a non-immunoglobulin transgene independent of chromosomal position. Mol. Immunol. 44, 567-575 (2007).
- Zhang, W. Clonal instability of V region hypermutation in the Ramos Burkitt's lymphoma cell line. Int. Immunol. 13, 1175-1184 (2001).
- Ukai, A. Induction of a:T mutations is dependent on cellular environment but independent of mutation frequency and target gene location. J. Immunol. 181, 7835-7842 (2008).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. J. Virol. 72, 8463-8471 (1998).
- Nakamura, M. High frequency class switching of an IgM+ B lymphoma clone CH12F3 to IgA+ cells. Int. Immunol. 8, 193-201 (1996).
- Muramatsu, M. Specific expression of activation-induced cytidine deaminase (AID), a novel member of the RNA-editing deaminase family in germinal center B cells. J. Biol. Chem. 274, 18470-18476 (1999).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유