Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluer hypermutation somatique dans les cellules B Ramos, après surexpression ou Knockdown de gènes spécifiques
Dans cet article
Résumé
Nous décrivons comment effectuer des infections rétrovirales ou lentiviraux de la surexpression ou de shRNA contenant des constructions dans les cellules B humaines Ramos en ligne et comment mesurer hypermutation somatique dans ces cellules.
Résumé
Les cellules B commencent leur vie avec des anticorps de faible affinité générés par V (D) J. Toutefois, après la détection d'un pathogène, la variable (V) la région d'une immunoglobuline (Ig) gène est muté environ 100.000 fois plus que le reste du génome grâce à hypermutation somatique (SHM), résultant de l'affinité des anticorps 1,2 de haut. De plus, la commutation isotypique (CSR) produit des anticorps avec les différentes fonctions effectrices selon le type de réponse immunitaire qui est nécessaire pour un agent pathogène particulier. Tant en matière de RSE et la SHM sont initiées par la cytidine désaminase induite par activation (AID), qui désamine résidus cytosine dans l'ADN pour produire uraciles. Ces uraciles sont traitées par des formes d'erreurs des voies de réparation, pour aboutir finalement à des mutations et recombinaisons 1-3.
Notre compréhension actuelle des détails moléculaires de SHM et la RSE proviennent d'une combinaison d'études chez la souris, des cellules primaires, lignées cellulaires et des cellules-fexpériences ree. Des modèles murins reste l'étalon-or avec des KO génétique montrant des rôles essentiels pour de nombreux facteurs de réparation (par exemple Ung, MSH2, MSH6, Exo1, et la polymérase η) 4-10. Cependant, tous les gènes sont susceptibles d'études à élimination directe. Par exemple, KO de plusieurs double-brin de protéines de réparation des cassures sont embryonnaire létal ou altérer développement des cellules B 11-14. D'ailleurs, parfois la fonction spécifique d'une protéine dans SHM ou RSE peuvent être masqués par plusieurs défauts mondial causé par les huitièmes de finale. En outre, depuis les expériences chez la souris peut être long, en modifiant l'expression des gènes individuels dans des lignées cellulaires est devenu une étape plus populaire d'abord identifier et caractériser les gènes candidats 15-18.
Ramos - une ligne de lymphome de Burkitt qui subit constitutivement SHM - a été un populaire lignée cellulaire modèle pour étudier SHM 18-24. Un avantage de cellules Ramos, c'est qu'ils ont un semi intégré pratique-Quantitative mesure de SHM. Cellules de type sauvage d'exprimer des IgM et, comme ils prennent des mutations, certaines mutations assommer l'expression d'IgM. Par conséquent, le dosage de la perte d'IgM par la numérisation des cellules activées par fluorescence (FACS) fournit un moyen rapide de lecture pour le niveau de SHM. Une mesure plus quantitative de la SHM peut être obtenu par séquençage direct des gènes d'anticorps.
Comme les cellules Ramos sont difficiles à transfecter, nous produisons des dérivés stables qui ont augmenté ou abaissé expression d'un gène particulier en infectant les cellules avec des constructions rétrovirales ou lentiviraux qui contiennent soit une cassette surexpression ou une épingle à cheveux courts ARN (shRNA), respectivement. Ici, nous décrivons comment nous infecter les cellules Ramos et ensuite utiliser ces cellules pour étudier le rôle des gènes spécifiques sur SHM (figure 1).
Protocole
1. Préparation des échantillons et les cellules
- Effectuer une préparation à moyenne échelle de l'ADN (Midiprep) de la surexpression ou shRNA contenant des constructions et de l'emballage vecteur rétroviral pKat2 ou les vecteurs lentiviraux emballage pVSV-G, pMDLg / pRRE et pRSV-Rev 25. Utilisez 6 ug d'ADN pour chaque construction et soit 4 pg d'pKat2 (pour infections rétrovirales) ou 1,5 ug d'ADN pour chacun des pVSV-g, pMDLg / pRRE, et les vecteurs d'emballage pRSV-Rev (pour les infections lentiviraux).
- Préparer BOSC 23 médias. Pour la transfection, utilisez les deux moyennes non amendée Eagle modifié par Dulbecco (DMEM), ainsi que du DMEM complet. Pour faire DMEM complet, ajouter 1% de tampon HEPES, 1% de pénicilline-streptomycine-glutamine (PGS), et 10% de sérum de veau nouveau-né (NCS) pour une bouteille de DMEM.
- Préparer Ramos médias. Pour l'étape de l'infection, assurez complète milieu RPMI-1640 (RPMI) en ajoutant 1% d'HEPES, PGS 1%, et 10% de sérum fœtal bovin (FBS) d'une bouteille de RPMI. Pour l'étape d'ensemencement cellulaire unique, mAke "conditionné" par les médias de plus en plus les cellules Ramos dans RPMI complet pour deux jours, le filage bas les cellules vers le bas à 400 X g pendant 4 minutes et filtrer le surnageant avec une filtre de 0,2 um dans un flacon stérile. Comme d'autres milieux complets, des milieux conditionnés sont stockés à 4 ° C jusqu'à ce que nécessaire.
- Split 3 x 10 6 cellules BOSC 23 dans une plaque de 100 mm dans 10 ml DMEM complet le jour avant la transfection afin que les cellules seront à 50-60% de confluence le jour de la transfection. Faire une plaque de cellules pour chaque vecteur de vous projetez sur la transfection. Il est important que les cellules n'ont pas été maintenues en culture depuis trop longtemps; faible utilisation des cellules ou des cellules nombre de passage récemment dégelée à la fois pour la transfection et les étapes d'infection afin d'assurer une infection bon.
- Inclure des contrôles de vos expériences. Pour les transfections et les infections, de créer un contrôle négatif (cellules non transfectées ou non infecté) et un contrôle positif (virus qui exprime la GFP et la puromycine gène de résistance). Pour shRNA le knock-down experriences, infecter un bien avec un brouillés ou le contrôle shRNA pertinent.
2. Transfectant BOSC 23 cellules
- Chaud les bouteilles de non amendée DMEM et DMEM complet à 37 ° C.
- Retirez le support du 23 BOSC et remplacer les cellules avec 5 ml de DMEM complet. Retour aux cellules d'un incubateur à 37 ° C jusqu'à son utilisation dans l'étape 2.6.
- Chauffer la bouteille de 6 FuGENE à température ambiante pendant 5 minutes, puis brièvement au vortex (<1 seconde).
- Aliquoter 600 uL de DMEM non amendée dans un tube de 1,5 ml.
- Diluer 30 uL FuGENE 6 dans les 600 uL de DMEM non amendée, brièvement au vortex (<1 seconde), puis incuber à température ambiante pendant cinq minutes. Faites attention à la pipette directement dans les médias. Ne laissez pas les 6 FuGENE toucher les parois du tube.
- Ajouter 6 g de construire et, soit 4 mg pKat2 (infections rétrovirales) ou 1,5 ug chacun des pVSV-G, pMDLg / pRRE et pRSV-Rev (infections lentiviraux) à l'DMEM / 6 FuGENE mélanger, tourbillons de l'IRB eFly (<1 seconde), puis incuber à température ambiante pendant 25 minutes.
- Ajouter l'ADN / DMEM / 6 FuGENE mélange pour les 23 cellules Bosc et le retour à un incubateur à 37 ° C pendant 24 heures.
- Après 24 heures, ajouter 5 ml DMEM complet pour les cellules transfectées et le retour à un incubateur à 37 ° C pendant 24 heures.
3. La récolte des particules virales
- Récolte les 10 ml de médias de la transfectées BOSC 23 cellules avec une seringue de 10 ml.
- Fixez un filtre à 0,45 um à la fin de la seringue et le filtre des médias dans un tube en polypropylène propre de 15 ml.
- Utiliser les médias virale immédiatement ou congeler que 1 mL dans 2 ml flacons cryogéniques à -80 ° C.
- Après la récolte des médias viraux, confirment que la transfection efficaces s'est produite dans les cellules de contrôle positif par FACS. Cela peut aussi être effectuée pour tous les échantillons si le concept contient soit une lampe fluorescente (GFP, par exemple) ou un marqueur de surface cellulaire (par exemple Thy1).
- Graine 2 ml de 2 x 10 5 cellules / ml dans du RPMI cellules Ramos complète dans une plaque à 6 puits 24 heures avant l'infection. Encore une fois, veillez à utiliser les cellules à faible nombre de passages ou des cellules récemment dégelée pour assurer l'infection bon.
- Si vous utilisez aliquotes congelées de virus, de dégel, les médias virale rapide à 37 ° C.
- Mélanger 1 ml de milieu virale avec 3 ul de 10 mg / mL polybrène. La concentration finale de polybrène devrait être de 10 ug / ml dans un volume total de 3 ml.
- Ajouter le mélange virus / polybrène aux cellules et bien mélanger par pipetage. Comme mentionné précédemment, un puits doit être utilisé comme un contrôle non infectés, ajouter 1 ml de RPMI complet et 3 polybrène uL à ce même lieu de 1 ml de virus. Soyez sûr d'infecter un puits avec un témoin apparié négative. Nous avons également infecter un bien avec un vecteur GFP-contenant, afin de déterminer l'efficacité d'infection par FACS.
- Centrifuger la plaque de 6 puits à 3000 X g pendant 90 minutes à température ambiante.
- Retour à la plaqueà 37 ° C incubateur pendant 48 heures.
- Après 48 heures, ralentit les cellules infectées dans un tube en polypropylène 15 mL à 400 X g pendant 4 minutes à température ambiante. Retirez le support par aspiration, en étant sûr de ne pas perturber le culot cellulaire. Resuspendre les cellules dans RPMI frais.
- Confirmer l'efficacité d'infection dans l'échantillon de contrôle positif par FACS. Comme avant, vous pouvez faire cela pour tous vos échantillons si la construction contient soit une lampe fluorescente (GFP, par exemple) ou un marqueur de surface cellulaire (par exemple Thy1).
- Démarrer la sélection des cellules (le cas échéant). Notre shRNA contenant des constructions contiennent tous une cassette de résistance à la puromycine. Nous sélectionnons shRNA cellules infectées Ramos avec 0,25 pg / ml de puromycine. Nous constatons que les dérivés de cellules Ramos montrent parfois altéré la susceptibilité à la puromycine et, par conséquent, nous procédons à tuer les courbes pour optimiser la concentration de puromycine pour chaque produit dérivé. Traiter aussi les cellules de contrôle non infectés par la puromycine pour confirmer que la sélection est efficace.
5. L'ensemencement de cellules isolées
Simple d'ensemencement peut être effectué dans l'une des deux façons: par FACS de tri ou manuellement.
- FACS: Utilisation d'un trieur FACS avec une plaque d'adaptation aux semences 1 cellule / puits dans une plaque de 96 puits pré-remplie avec 100 ul de 40% RPMI/40% de FBS conditionnée media/20%. Cette méthode donne beaucoup plus simples clones cellulaires par plaque.
- Manuellement: Compter les cellules. Diluer une aliquote de cellules à 10 cellules / ml dans 40% RPMI/40% conditionnée media/20% de FBS. Utilisez 10 ml de cellules diluées pour chaque plaque ensemencés. Différents clones montrera récupération variable à ce stade il est donc utile de mettre en place plusieurs plaques à différentes concentrations (allant de 3 à 20 cellules / ml). Utilisez celle qui produit moins d'excroissances afin d'éviter les clones provenant de plusieurs cellules). Utiliser une pipette multicanaux aux semences plaques à 96 puits avec 100 ul de cellules diluées.
- Placer la plaque dans un incubateur à 37 ° C pendant 2 semaines.
- Vérifiez puits individuels UEDNr le microscope pour confirmer la présence de cellules un jour après le semis. Les cellules individuelles peuvent être difficiles à trouver en raison de la nécessité de se concentrer à la bonne profondeur. En se concentrant sur les chiffres ainsi imprimé au bas de la plaque de 96 puits devraient vous aider. Les cellules sont faciles à trouver quand ensemencé par le trieur FACS puisque la plupart des puits ont une cellule.
- Après 2 semaines, des colonies de culture peuvent être à peine vu par l'oeil nu en regardant les plaques d'en bas. Ils sont beaucoup plus faciles à voir sous le microscope. Colonies ont tendance à croître le long des bords de la plupart des colonies bien et généralement dans une plaque poussent le long du même bord dans différents puits. Mark puits avec la croissance des cellules robustes et de transférer les cellules à plaques à 24 puits dans 1 ml de RPMI et revenir à un incubateur à 37 ° C.
- Maintenir les cellules à 1 x 10 5 à 1 x 10 6 cellules / ml. Changer les médias en faisant tourner les cellules à 400 X g pendant 4 minutes à température ambiante et de retirer le support nécessaire. Remplacer avec 1 ml de RPMI frais.
- Après plusieurs days de croissance, de transférer les cellules d'une plaque à 6 puits et poursuivre sa croissance dans un incubateur à 37 ° C.
- Après 3 semaines, d'analyser les clones pour la perte de l'IgM (comme décrit ci-dessous).
- Continuez à cultiver les cellules pendant 2 semaines et plus, puis analyser les clones de nouveau au point cinq semaines de temps.
6. Analyse: la perte d'IgM (3 semaines et 5 semaines)
- Aliquote de 5 x 10 5 cellules de chaque puits dans un tube de 1,5 ml et de spin des cellules vers le bas à 400 X g pendant 4 minutes à température ambiante. Utiliser les cellules de chacun des contrôle et de shRNA-knock-down de puits, ainsi que d'un échantillon pour un contrôle non colorées.
- Laver les cellules dans 1 ml de PBS, puis tourner à nouveau à 400 X g pendant 4 minutes à température ambiante.
- Remettre en suspension dans 50 pl de PBS (contrôle non coloré) ou PBS + dilution 1:20 de PE-étiquetés anticorps IgM α-humain. Incuber sur la glace pendant 1 heure.
- Faites tourner les cellules vers le bas à 400 X g pendant 4 minutes à température ambiante.
- Laver les caunes deux fois avec 1 mL de PBS et de spin pour 400 X g pendant 4 minutes à chaque fois à température ambiante.
- Enlever le surnageant et remettre en suspension dans 100 pi de PBS. Transférer les cellules resuspendues taché à un tube FACS.
- Analyser les cellules en utilisant une machine FACS.
- Comparez le pourcentage d'IgM - cellules à partir des commandes vs le shRNA knocked-down cellules. Comme noté précédemment, la perte d'IgM telle que mesurée par FACS est un semi-quantitatifs fiables mesure de SHM dans les cellules Ramos.
7. Analyse: Confirmer le knock-down par RT-PCR quantitative (5 semaines)
- Aliquoter 3 x 10 6 cellules de chaque puits et spin bas à 400 X g pendant 4 minutes à température ambiante.
- Préparer l'ARN à partir de la partie aliquote de cellules. Nous utilisons TRIzol selon les recommandations du fabricant.
- Préparer l'ADNc de la préparation de l'ARN. Nous utilisons 2 ug d'ARN et de Superscript II, conformément aux recommandations du fabricant.
- Effectuer une PCR quantitative en utilisant l'ADNc pour confirmationm le knock-down. Nous utilisons la GAPDH comme un contrôle de normalisation, et le kit SYBR qPCR FAST dans un volume de 10 uL de réaction totale.
8. Analyse: génomique séquençage (5 semaines)
- Aliquote de 5 x 10 6 cellules de chaque puits et spin bas à 400 X g pendant 4 minutes à température ambiante.
- Isoler l'ADN génomique à partir de ces cellules. Nous utilisons la génomique Assistant SV DNA kit de purification selon les recommandations du fabricant.
- PCR amplifier l'ADN génomique pour les gènes V d'Ig 19 (tableau 1) et tout autres gènes d'intérêt en utilisant la polymérase ADN KAPA HiFi. Nous utilisons cette polymérase raison de son faible taux d'erreurs, mais toute la polymérase haute fidélité PCR devrait être acceptable.
- Gel de purifier les produits de PCR sur un gel à 1% d'agarose en utilisant le kit d'extraction de gel QIAquick.
- Certains polymérases ajouter un simple désoxyadénosine (A) de résidus aux extrémités 3 'des produits de PCR, tandis que d'autres n'en ont pas. Ces trois «A la queue sont nécessaires pour la TA TOPOétape de clonage. La polymérase KAPA HiFi ne laisse 3 'Une queue. Une queue-produits de la PCR en incubant 25 uL purifié sur gel produit PCR avec 3 pi de tampon de réaction 10x Taq, 1 pi 10 mM de dNTPs, et 1 unité de Taq polymérase à 72 ° C pendant 30 minutes.
- Clone des produits de PCR purifiés sur gel en utilisant le kit TOPO TA Cloning. Transformer les produits de la réaction TOPO dans la condition E. coli DH5a-T1 R cellules compétentes et la plaque sur LB / agar plaques contenant 100 ug / ml d'ampicilline et complété avec 1,6 mg de X-gal selon les recommandations du fabricant.
- Envoyer des colonies individuelles pour le séquençage.
- Effectuer une analyse de séquence pour déterminer les types et emplacements des mutations.
9. Les résultats représentatifs:
Comme indiqué précédemment, nous utilisons un vecteur viral qui contrôle positif GFP exprime à la fois ainsi que d'un gène puromycine-résistance. Nous observons habituellement 50-75% des cellules GFP + deux joursaprès la transfection. Dans notre infections, nous observons habituellement à 50-70% des cellules sont GFP + deux jours après l'infection, mais avant la sélection. Après la sélection est terminée,> 95% des cellules sont GFP +.
Comme expériences représentatives, nous montrent les effets de l'AID surexprimant ou abattre un facteur de réparation dans les cellules Ramos. Plus précisément, nous avons transfecté un vecteur rétroviral pour la surexpression d'aide lié via un site IRES de Thy1.1 avec le vecteur rétroviral dans les emballages pKat2 BOSC 23 cellules. Contenant le virus des médias a été filtré et ensuite utilisé pour infecter les cellules Ramos. Les deux type sauvage (WT) et l'aide surexprimant (AID Salut) cellules ont été semées seule cellule et cultivées pendant 3 semaines, à quel point ils ont été analysés pour l'expression génique par PCR qRT (figure 2) et pour la perte d'IgM par FACS (figure 3 ). Pour la coloration FACS, les cellules ont été incubées avec deux 1:20 diluée PE-étiquetés anticorps IgM α-humain ainsi que 1:400 diluée marqué FITC α-rat CD90/mouseCD90.1 (Thy1.1) anticorps. Dans une expérience séparée, lentivirus contenant soit une pertinents shRNA (contrôle) ou une shRNA contre un facteur de haute-fidélité de réparation d'ADN devraient protéger contre SHM ont été faites dans BOSC 23 cellules, puis infectés dans un clone de cellules SECOURS Salut Ramos. Encore une fois, l'expression des gènes (figure 4) et la perte d'IgM (figure 5) ont été analysés dans des clones de cellules simples après 3 semaines. Depuis le facteur de réparation est pensé pour offrir une protection contre les mutations au cours SHM, nous voyons une augmentation de la perte des IgM et SHM, en l'absence du facteur. Si nous avions knocked-down un facteur impliqué dans la génération SHM mutations associées, nous avons constaté une diminution de la perte d'IgM à la place.
Comme prévu, notre qRT PCR résultats montrent une augmentation marquée de l'expression aides suivantes infection des cellules avec un vecteur d'une surexpression AIDE (figure 2), tandis que les niveaux de la chute de facteur de réparation de l'ADN en présence d'un shRNA dirigé contre ce facteur (figure 4 ). Parce Clon individuelses peut varier, vous pourriez voir une certaine variation dans les niveaux d'expression des gènes. Parce que l'expression des gènes peut varier, il est nécessaire de confirmer l'expression de chaque clone que vous prévoyez sur l'analyse plus loin. ShRNA différentes contre le même gène peut avoir des effets différents sur l'expression génique, ainsi, vous devez donc l'écran shRNA plusieurs méthodes pour déterminer qui a le plus d'impact. Knockdown peut également être confirmée au niveau protéique par immunoblot. De même, des clones individuels peuvent varier considérablement dans les niveaux de perte d'IgM. Certains clones pourraient démontrer un "jackpot" effet, où une mutation IgM eu lieu dans l'une des divisions cellulaires plus tôt - par exemple, vous verrez une perte> 50% des IgM simplement parce que l'une des cellules est devenu IgM - au stade de deux cellules. L'influence de ces clones est minimisé par le calcul de la médiane pour plusieurs clones de cellules simples.

Figure 1. Schéma pour le transperfection, l'infection et l'analyse de la perte d'IgM. Voir le texte pour plus de détails.
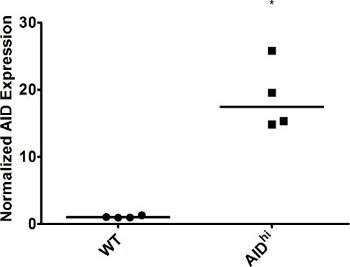
Surexpression SECOURS Figure 2. Dans les cellules Ramos. WT et AID Salut cellules Ramos ont été clonés seule cellule et cultivées pendant 3 semaines, au cours de laquelle l'expression SECOURS point clones individuels a été mesurée par qRT-PCR. L'expression de GAPDH a été utilisée pour la normalisation. WT a été fixé à 1. La valeur médiane est indiquée. * Indique une valeur p de <0,01, calculé comme un étudiant unilatéral t-test.
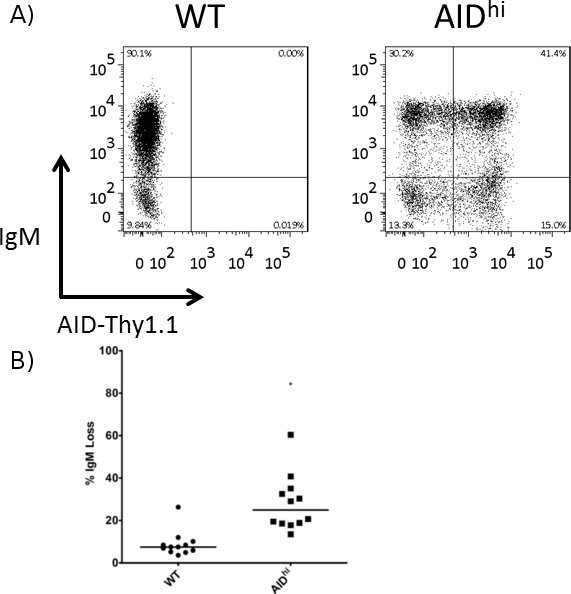
Figure 3. Augmentation de la perte d'IgM dans les cellules Salut aide par rapport aux cellules WT. IgM de surface a été mesurée par FACS au point 3 semaines de temps, et le pourcentage de perte d'IgM a été calculé pour WT et les cellules Salut AID. (A) Représentant FACS intrigue d'un clone WT et un clone SECOURS Salut. La surexpression SECOURS vecteur a aide liée à Thy1.1 via un site IRES, alors Thy1.1 expression est utilisée comme substitut à l'expression de l'AID. (B) La quantification de la perte d'IgM dans WT plusieurs clones et de l'AID Salut. La valeur médiane est indiquée. * Indique une valeur p de <0,01, calculé comme un étudiant unilatéral t-test.
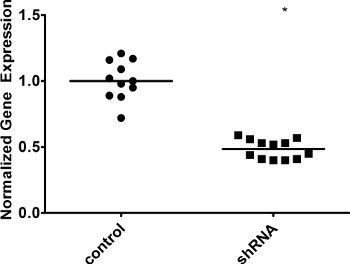
Figure 4. Knockdown d'un facteur de réparation dans les cellules Salut AID. Les cellules ont été infectées soit avec un shRNA de contrôle hors de propos (de contrôle) ou notre shRNA d'intérêt (shRNA), et seule cellule ensemencés. Après 3 semaines, l'expression des gènes dans les clones individuels a été mesurée par qRT-PCR. L'expression de GAPDH a été utilisée pour la normalisation. WT a été fixé à 1. La valeur médiane est indiquée. * Indique une valeur p de <0,01, calculé comme un étudiant unilatéral t-test.
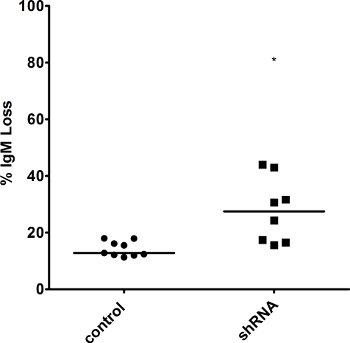
Figure 5. IgM perte accruedans les cellules Salut AID niveaux knocked-down d'un facteur de réparation. IgM de surface dans plusieurs clones a été mesurée par FACS au point 3 semaines de temps, et le pourcentage de perte d'IgM a été calculée dans les cellules surexprimant toujours une aide. La valeur médiane est indiquée. * Indique une valeur p de <0,01, calculé comme un étudiant unilatéral t-test.

Tableau 1 Primer séquences des gènes de l'Ig 19:. Cμ région constante de l', la chaîne lourde de la région V (V H) et la chaîne légère de la région V (V L).
Access restricted. Please log in or start a trial to view this content.
Discussion
Comme indiqué précédemment, les modèles de la lignée cellulaire pour la diversification des anticorps sont devenus un point de départ pour identifier de nouvelles protéines qui influencent les différentes étapes au cours de diversification des anticorps. Nous présentons ici une méthode pour utiliser une infection virale à des protéines soit le knock-down ou une surexpression dans les cellules B Ramos ligne et ensuite d'examiner l'impact sur la SHM.
Pour ces études, nous...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Le pMSCV-AID-I-Thy1.1 et pKat2 vecteurs ont été un cadeau aimable de la DG Schatz et l'pVSV-G, pRSV-Rev, et pMDLg / pRRE vecteurs ont été un cadeau du genre BR Cullen.
Access restricted. Please log in or start a trial to view this content.
matériels
Réactifs suggérée - la plupart d'entre eux peuvent être remplacés par des produits similaires d'autres fournisseurs.
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|
| 6-même évident TC-traitée plaques | Corning | 3516 | |
| 10 ml BD Luer-Loksyringes | BD Medical | 309604 | |
| 24 puits claires TC-traitée plaques | Corning | 3526 | |
| 96 puits à fond plat en polystyrène transparent TC-traitée microplaques | Corning | 3596 | |
| 100 mm boîtes de culture TC-traitée | Corning | 430167 | |
| Filtres pour seringue Acrodisc, 0,45 um | Pall Life Sciences | 4604 | |
| Agar | Teknova | A7777 | |
| Agarose | GeneMate | E-3120-500 | |
| Ampicilline | Sigma | A0166 | 100 mg / mL dans l'eau |
| BD FACSCanto cytomètre en flux II | BD Biosciences | ou similaire | |
| BD Falcon tubes ronds en polystyrène en bas | BD Biosciences | 352054 | pour la FACS |
| BOSC 23 cellules | ATCC | CRL-11270 | |
| CO 2 capable d'incubateur 37 ° C | |||
| DMEM (milieu de Dulbecco modifié Eagle) | Sigma | D6429 | |
| FBS (sérum de veau fœtal) | Gemini Bio-Produits | 100-106 | |
| FITC α-rat CD90/mouse CD90.1 d'anticorps | BioLegend | 202503 | FITCα-Thy1.1 |
| Réactif de transfection FuGENE 6 | Roche | 11814443001 | |
| Solution tampon HEPES | Invitrogen | 15630-080 | |
| Polymérase ADN KAPA HiFi | KAPA Biosystems | KK2101 | |
| LB Broth (bouillon de lysogénie - Luria) Poudre | Difco | 240230 | |
| MISSION TRC shRNA stocks de glycérol bactérienne | Sigma | vecteurs shRNA | |
| NCS (sérum de veau nouveau-né) | Gemini Bio-Produits | 100-504 | |
| PBS (solution tampon phosphate) | Invitrogen | 70011 | diluée dans de l'eau à 1x |
| PE α-humain IgM | BioLegend | 314508 | |
| PGS (pénicilline-streptomycine-glutamine solution) | Gemini Bio-Produits | 400-110 | |
| Polybrène (hexadiméthrine bromure) | Sigma | 107689 | 10 mg / mL dans l'eau |
| PureYield système plasmidique Midiprep | Promega | A2495 | |
| Puromycine | Sigma | P8833 | 250 pg / mL dans l'eau |
| QIAquick gel extraction kit | QIAGEN | 28706 | |
| Ramos (PR 1) des cellules | ATCC | LCR-1596 | |
| RPMI-1640 | Sigma | R8758 | |
| SuperScript II | Invitrogen | 18064-022 | |
| SYBR FAST qPCR kit | KAPA Biosystems | KK4601 | |
| Taq ADN polymérase | Invitrogen | 18038-042 | |
| TOPO TA Cloning kit | InvitroGen | K4520-01 | |
| TRIzol | Invitrogen | 15596-026 | |
| Assistant Genomic DNA SV système de purification | Promega | A2361 | |
| X-Gal [5-bromo-4-chloro-3-β-indoyl-D-galatopyranoside] | Growcells | C-5687 | 40 mg / ml dans le DMSO |
Références
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annu. Rev. Biochem. 76, 1-22 (2007).
- Peled, J. U. The biochemistry of somatic hypermutation. Annu. Rev. Immunol. 26, 481-511 (2008).
- Longerich, S., Basu, U., Alt, F., Storb, U. AID in somatic hypermutation and class switch recombination. Curr. Opin. Immunol. 18, 164-174 (2006).
- Bardwell, P. D. Altered somatic hypermutation and reduced class-switch recombination in exonuclease 1-mutant mice. Nat. Immunol. 5, 224-229 (2004).
- Martomo, S. A., Yang, W. W., Gearhart, P. J. A role for Msh6 but not Msh3 in somatic hypermutation and class switch recombination. J. Exp. Med. 200, 61-68 (2004).
- Phung, Q. H. Increased hypermutation at G and C nucleotides in immunoglobulin variable genes from mice deficient in the MSH2 mismatch repair protein. J. Exp. Med. 187, 1745-1751 (1998).
- Rada, C., Ehrenstein, M. R., Neuberger, M. S., Milstein, C. Hot spot focusing of somatic hypermutation in MSH2-deficient mice suggests two stages of mutational targeting. Immunity. 9, 135-141 (1998).
- Rada, C. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Curr. Biol. 12, 1748-1755 (2002).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase eta is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. J. Exp. Med. 204, 17-23 (2007).
- Masuda, K. DNA polymerase eta is a limiting factor for A:T mutations in Ig genes and contributes to antibody affinity maturation. Eur. J. Immunol. 38, 2796-2805 (2008).
- Lim, D. S., Hasty, P. A mutation in mouse rad51 results in an early embryonic lethal that is suppressed by a mutation in p53. Mol. Cell. Biol. 16, 7133-7143 (1996).
- Xiao, Y., Weaver, D. T. Conditional gene targeted deletion by Cre recombinase demonstrates the requirement for the double-strand break repair Mre11 protein in murine embryonic stem cells. Nucleic. Acids. Res. 25, 2985-2991 (1997).
- Zhu, J., Petersen, S., Tessarollo, L., Nussenzweig, A. Targeted disruption of the Nijmegen breakage syndrome gene NBS1 leads to early embryonic lethality in mice. Curr. Biol. 11, 105-109 (2001).
- Luo, G. Disruption of mRad50 causes embryonic stem cell lethality, abnormal embryonic development, and sensitivity to ionizing radiation. Proc. Natl. Acad. Sci. U. S. A. 96, 7376-7381 (1999).
- Pavri, R. Activation-induced cytidine deaminase targets DNA at sites of RNA polymerase II stalling by interaction with Spt5. Cell. 143, 122-133 (2010).
- Lee-Theilen, M., Matthews, A. J., Kelly, D., Zheng, S., Chaudhuri, J. CtIP promotes microhomology-mediated alternative end joining during class-switch recombination. Nat. Struct. Mol. Biol. 18, 75-79 (2011).
- Basu, U. The RNA exosome targets the AID cytidine deaminase to both strands of transcribed duplex DNA substrates. Cell. 144, 353-363 (2011).
- Yabuki, M., Fujii, M. M., Maizels, N. The MRE11-RAD50-NBS1 complex accelerates somatic hypermutation and gene conversion of immunoglobulin variable regions. Nat. Immunol. 6, 730-736 (2005).
- Sale, J. E., Neuberger, M. S. TdT-accessible breaks are scattered over the immunoglobulin V domain in a constitutively hypermutating B cell line. Immunity. 9, 859-869 (1998).
- Cumbers, S. J. Generation and iterative affinity maturation of antibodies in vitro using hypermutating B-cell lines. Nat. Biotechnol. 20, 1129-1134 (2002).
- Papavasiliou, F. N., Schatz, D. G. Cell-cycle-regulated DNA double-stranded breaks in somatic hypermutation of immunoglobulin genes. Nature. 408, 216-221 (2000).
- Parsa, J. Y. AID mutates a non-immunoglobulin transgene independent of chromosomal position. Mol. Immunol. 44, 567-575 (2007).
- Zhang, W. Clonal instability of V region hypermutation in the Ramos Burkitt's lymphoma cell line. Int. Immunol. 13, 1175-1184 (2001).
- Ukai, A. Induction of a:T mutations is dependent on cellular environment but independent of mutation frequency and target gene location. J. Immunol. 181, 7835-7842 (2008).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. J. Virol. 72, 8463-8471 (1998).
- Nakamura, M. High frequency class switching of an IgM+ B lymphoma clone CH12F3 to IgA+ cells. Int. Immunol. 8, 193-201 (1996).
- Muramatsu, M. Specific expression of activation-induced cytidine deaminase (AID), a novel member of the RNA-editing deaminase family in germinal center B cells. J. Biol. Chem. 274, 18470-18476 (1999).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon