È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutare mutazione somatica nel Ramos cellule B dopo iperespressione o Knockdown di specifici geni
In questo articolo
Riepilogo
Ci descrivono come eseguire infezioni retrovirali o lentivirali di sovraespressione o shRNA contenenti costruisce nell'essere umano a cellule B Ramos line e come misurare mutazione somatica in queste cellule.
Abstract
Le cellule B iniziano la loro vita con gli anticorpi a bassa affinità generato da V (D) J ricombinazione. Tuttavia, al rilevamento un agente patogeno, la (V) regione variabile di un gene immunoglobuline (Ig) è mutato circa 100.000 volte più del resto del genoma attraverso la mutazione somatica (SHM), con conseguente 1,2 anticorpi ad alta affinità. Inoltre, passare ricombinazione di classe (CSR) produce anticorpi con differenti funzioni effettrici a seconda del tipo di risposta immunitaria che è necessario per un particolare agente patogeno. Sia CSR e SHM vengono avviate attraverso l'attivazione indotta citidina deaminasi (AID), che deamina residui di citosina nel DNA per produrre uracils. Queste uracils sono trattati con forme soggette a errori di percorsi di riparazione, alla fine portano a mutazioni e la ricombinazione 1-3.
La nostra attuale comprensione dei dettagli molecolari di SHM e RSI provenire da una combinazione di studi in topi, pile, di linee cellulari e cellule-free esperimenti. Modelli murini rimangono il gold standard con fori genetica che mostrano ruoli critici per la riparazione di molti fattori (ad esempio Ung, MSH2, MSH6, Exo1 e polimerasi η) 4-10. Tuttavia, non tutti i geni sono suscettibili per gli studi di eliminazione diretta. Per esempio, KO di proteine diverse rottura a doppio filamento di riparazione sono embrionalmente letali o mettere in pericolo lo sviluppo delle cellule B 11-14. Inoltre, a volte la funzione specifica di una proteina in SHM o CSR può essere mascherato da più difetti globale causato dalla eliminazione diretta. Inoltre, dal momento che gli esperimenti sui topi possono essere lunghe, alterando l'espressione di singoli geni in linee cellulari è diventata una tappa sempre più popolare primi a identificare e caratterizzare geni candidati 15-18.
Ramos - una linea cellulare di linfoma di Burkitt che subisce costitutivamente SHM - è stata una popolare linea cellulare modello per studiare SHM 18-24. Un vantaggio di cellule Ramos è che hanno un built-in semi conveniente-Quantitativa misura di SHM. Cellule wild-type esprimere IgM e, come raccogliere le mutazioni, alcune delle mutazioni knock out espressione IgM. Pertanto, il saggio perdita IgM di fluorescenza attivata la scansione cellulare (FACS) fornisce una rapida lettura per il livello di SHM. Una misura più quantitativa di SHM possono essere ottenute direttamente sequenziamento dei geni anticorpali.
Dato che le cellule Ramos è difficile trasfezione, produciamo derivati stabili che hanno aumentato o diminuito l'espressione di un singolo gene da infettare le cellule con costrutti retrovirali o lentivirali che contengono una cassetta iperespressione o un breve hairpin RNA (shRNA), rispettivamente. Qui, descriviamo il modo in cui infettare le cellule Ramos e quindi utilizzare queste cellule per studiare il ruolo di specifici geni in SHM (Figura 1).
Protocollo
1. Preparazione dei campioni e le cellule
- Eseguire una preparazione media scala del DNA (midiprep) della sovraespressione o shRNA contenenti costrutti e il vettore retrovirale imballaggio pKat2 o vettori lentivirali imballaggio pVSV-G, pMDLg / pRRE e pRSV-Rev 25. Usare 6 mg per ogni DNA costruire e uno di 4 microgrammi pKat2 (per infezioni retrovirali) o 1,5 mg di DNA per ciascuna delle pVSV-g, pMDLg / pRRE e pRSV-Ap vettori di imballaggio (per le infezioni lentivirali).
- Preparare 23 BOSC media. Per la trasfezione, utilizzare sia emendata Dulbecco modificato Eagle medium (DMEM), così come DMEM completo. Per rendere completo DMEM, aggiungere 1 tampone HEPES%, 1% penicillina-streptomicina-glutamina (PGS), e il 10% di siero di vitello neonato (NCS) per una bottiglia di DMEM.
- Preparare Ramos media. Per la fase di infezione, di dare integrale RPMI-1640 medium (RPMI) con l'aggiunta di 1% HEPES, 1% PGS, e il 10% di siero fetale bovino (FBS) per una bottiglia di RPMI. Per passo la semina delle cellule, mAke "condizionato" dei media da una crescente cellule Ramos in RPMI completo per 2 giorni, riducendo la velocità delle celle verso il basso a 400 X g per 4 minuti e filtrare il surnatante con un filtro 0,2 micron in una bottiglia sterile. Come altri mezzi di comunicazione completo, media condizionata è conservato a 4 ° C fino al momento dell'uso.
- Split 3 x 10 6 23 BOSC cellule in un piatto da 100 mm in 10 ml completo DMEM il giorno prima della trasfezione in modo che le cellule sarà al 50-60% confluenza del giorno di trasfezione. Fai una piastra di cellule per ogni vettore si pensa di trasfezione. E 'importante che le cellule non sono state mantenute in coltura per troppo tempo, l'uso delle cellule a basso numero di passaggio o di cellule recentemente scongelato sia per la trasfezione e passi l'infezione per assicurare una buona infezione.
- Includono i controlli per i vostri esperimenti. Per trasfezioni e infezioni, creare un controllo negativo (cellule non infette o untransfected) e un controllo positivo (virus che esprime la GFP e puromicina gene per la resistenza). Per shRNA knock-down compeiments, infettare un bene con un strapazzate o controllo shRNA irrilevante.
2. 23 BOSC trasfezione delle cellule
- Riscaldare le bottiglie di emendata DMEM e DMEM completo a 37 ° C.
- Rimuovere i supporti dal 23 BOSC cellule e sostituire con 5 ml di DMEM completo. Ritorna le cellule a 37 ° C in incubatore fino al momento dell'uso in fase 2.6.
- Scaldare la bottiglia di FuGENE 6 a temperatura ambiente per 5 minuti poi mescolare brevemente nel vortex (<1 secondo).
- Aliquota 600 microlitri DMEM emendata in una provetta 1,5 ml microcentrifuga.
- Diluire 30 microlitri FuGENE 6 nel 600 microlitri DMEM non emendato, mescolare brevemente nel vortex (<1 secondo), e poi incubare a temperatura ambiente per cinque minuti. Fare attenzione a pipetta direttamente nella media. Non lasciare che la FuGENE 6 toccare i lati del tubo.
- Aggiungere 6 costruire mg o 4 mg e pKat2 (infezioni retrovirali) o 1,5 mg ogni pVSV-G, pMDLg / pRRE e pRSV-Rev (infezioni lentivirale) al DMEM / FuGENE 6 mix, vortice bri Efly (<1 secondo), e poi incubare a temperatura ambiente per 25 minuti.
- Aggiungere il DNA / DMEM / FuGENE 6 mix al 23 BOSC cellule e ritorno a 37 ° C in incubatore per 24 ore.
- Dopo 24 ore, aggiungere 5 ml completo DMEM alle cellule trasfettate e tornare a 37 ° C in incubatore per altre 24 ore.
3. La raccolta particelle virali
- Raccolto il 10 mL di supporto dal transfettate BOSC 23 celle con una siringa da 10 ml.
- Collegare un filtro di 0,45 micron fino alla fine della siringa e il filtro dei media in un tubo di polipropilene pulita da 15 ml.
- Utilizzare i mezzi di comunicazione virale subito o congelare aliquote come 1 ml in 2 fiale ml criogenico a -80 ° C.
- Dopo la raccolta i mezzi di comunicazione virale, confermano che la trasfezione efficiente si è verificato nelle cellule controllo positivo da FACS. Questo può essere fatto anche per tutti i campioni, se il costrutto contiene un fluorescente (GFP per esempio) o di una superficie cellulare marcatore (ad esempio Thy1).
- Semi di 2 ml di 2 x 10 5 cellule / ml cellule Ramos in RPMI completo in un 6-pozzetti 24 ore prima dell'infezione. Anche in questo caso, assicurarsi di utilizzare le celle a basso numero di passaggio o di cellule recentemente scongelati per assicurare una buona infezione.
- Se si utilizza aliquote congelate di virus, il disgelo media virali rapidamente a 37 ° C.
- Mescolare 1 ml mezzi virale con 3 ml di 10 mg / ml polibrene. La concentrazione finale di polibrene dovrebbe essere di 10 mg / ml in un volume totale di 3 ml.
- Aggiungere il virus / polibrene mix per le cellule e mescolare bene pipettando. Come accennato in precedenza, un bene dovrebbe essere utilizzato come controllo non infetto; aggiungere 1 ml di RPMI completo e 3 polibrene microlitri di questo bene, invece di virus 1 ml. Assicurati di infettare un pozzetto con un controllo abbinati negativo. Abbiamo anche infettare un pozzetto con una GFP-vettore contenente, per determinare l'efficienza infezione da FACS.
- Centrifugare il 6-pozzetti a 3000 X g per 90 minuti a temperatura ambiente.
- Riportare la piastra per37 ° C in incubatore per 48 ore.
- Dopo 48 ore, centrifugare le cellule infette in un tubo da 15 ml in polipropilene a 400 X g per 4 minuti a temperatura ambiente. Rimuovere il supporto per aspirazione, facendo attenzione a non disturbare il pellet. Risospendere le cellule in fresco RPMI.
- Confermare l'efficienza di infezione nel campione di controllo positivo da FACS. Come prima, è possibile farlo per tutti i vostri campioni se il costrutto contiene un fluorescente (GFP per esempio) o una superficie cellulare marcatore (ad esempio Thy1).
- Inizio selezione di celle (se del caso). Il nostro shRNA contenenti costruisce contengono tutti una cassetta resistenza puromicina. Selezioniamo shRNA cellule infettate Ramos con 0,25 mg / mL puromicina. Troviamo che i derivati delle cellule Ramos a volte mostrano alterata suscettibilità alle puromicina e, quindi, eseguire uccidere le curve per ottimizzare la concentrazione di puromicina per ogni derivato. Anche il trattamento di cellule di controllo non infettate con puromicina per confermare che la selezione è efficace.
5. Semina singola cellula
Semina singolo può essere eseguita in due modi: da FACS ordinamento o manualmente.
- FACS: Utilizzare un FACS sorter con un adattatore piastra alle sementi 1 cella / e in una piastra a 96 pozzetti pre-riempita con 100 ml di 40% RPMI/40% condizionata media/20% FBS. Questo metodo dà un sacco cloni cellulari più solo per piastra.
- Manualmente: Contare le cellule. Diluire una parte di cellule a 10 cellule / ml nel 40% RPMI/40% condizionata media/20% FBS. Usare 10 ml di cellule diluito per ogni piatto seminato. Diversi cloni mostrerà di recupero variabili in questa fase quindi è utile per creare piatti diversi a diverse concentrazioni (da 3 a 20 cellule / ml). Utilizzare quello che produce meno escrescenze per evitare cloni derivanti da più celle). Utilizzare una pipetta multicanale per seminare piastre a 96 pozzetti con 100 ml di cellule diluita.
- Posizionare la piastra in un incubatore a 37 ° C per 2 settimane.
- Controllare i singoli pozzi under il microscopio per confermare la presenza di cellule un giorno dopo la semina. Singole cellule può essere difficile da trovare a causa della necessità di messa a fuoco la corretta profondità. Concentrandosi sui numeri ben impresso sul fondo della piastra a 96 pozzetti dovrebbe aiutare. Le cellule sono più facili da trovare quando seminato dal selezionatore FACS poiché la maggior parte i pozzi hanno una cella.
- Dopo 2 settimane, colonie di crescita può essere a mala pena visti ad occhio nudo, cercando in piatti dal basso. Essi sono molto più facili da vedere al microscopio. Le colonie tendono a crescere lungo i bordi del pozzo e di solito più colonie in un piatto crescono lungo il bordo stesso in pozzi diversi. Segna i pozzi con la crescita cellulare robusto e trasferire le cellule a 24-pozzetti in 1 ml di RPMI e tornare a 37 ° C incubatore.
- Mantenere le cellule a 1 x 10 5 a 1 x 10 6 cellule / ml. Modificare i media facendo girare le cellule a 400 X g per 4 minuti a temperatura ambiente e di estrarre il supporto, se necessario. Sostituire con 1 ml di RPMI fresca.
- Dopo diversi days di crescita, il trasferimento alle cellule di un 6-pozzetti e continuare a crescere in un incubatore a 37 ° C.
- Dopo 3 settimane, analizzare i cloni per la perdita di IgM (come descritto di seguito).
- Continuano a crescere le cellule per altre 2 settimane e poi analizzare i cloni di nuovo al punto 5 settimane di tempo.
6. Analisi: la perdita di IgM (3 settimane e 5 settimane)
- Aliquota 5 x 10 5 cellule di ogni pozzetto in una provetta da microcentrifuga 1,5 ml e girare le celle in basso a 400 X g per 4 minuti a temperatura ambiente. Utilizzare le cellule da ogni controllo e shRNA-knock-down pozzi, così come un campione per un controllo senza macchia.
- Lavare le cellule in 1 ml di PBS e poi girare di nuovo a 400 X g per 4 minuti a temperatura ambiente.
- Risospendere in 50 ml di PBS (controllo non colorate) o PBS + 1:20 diluizione del PE-marcato α-umano degli anticorpi IgM. Incubare in ghiaccio per 1 ora.
- Spin le cellule giù a 400 X g per 4 minuti a temperatura ambiente.
- Lavare il cells due volte con 1 ml di PBS e spin per 400 X g per 4 minuti ogni volta a temperatura ambiente.
- Rimuovere il surnatante e risospendere in 100 ml di PBS. Trasferire le cellule risospese macchiato di un tubo di FACS.
- Analizzare le cellule utilizzando una macchina FACS.
- Confrontare la percentuale di IgM - le cellule dai controlli contro il shRNA smontati cellule. Come notato prima, la perdita IgM come misurato da FACS è un affidabile semi-quantitativa misura di SHM nelle cellule Ramos.
7. Analisi: Conferma knock-down da RT-PCR quantitativa (5 settimane)
- Aliquota 3 x 10 6 cellule di ogni pozzetto e spin down a 400 X g per 4 minuti a temperatura ambiente.
- Preparare l'RNA dalla aliquota di cellule. Noi usiamo TRIzol secondo le raccomandazioni del fabbricante.
- Preparare cDNA dalla preparazione RNA. Usiamo 2 mg di RNA e Superscript II secondo le raccomandazioni del fabbricante.
- Eseguire una PCR quantitativa utilizzando il cDNA di confermam knock-down. Utilizziamo GAPDH come controllo normalizzazione, e FAST kit qPCR SYBR in un volume di 10 L di reazione totale.
8. Analisi: il sequenziamento genomico (5 settimane)
- Aliquota 5 x 10 6 cellule di ogni pozzetto e spin down a 400 X g per 4 minuti a temperatura ambiente.
- Isolare il DNA genomico da queste cellule. Usiamo il genoma guidata SV kit di purificazione del DNA secondo le raccomandazioni del fabbricante.
- PCR amplifica il DNA genomico per i geni Ig V 19 (Tabella 1) e qualsiasi altri geni di interesse utilizzando la DNA polimerasi KAPA HiFi. Usiamo questa polimerasi a causa del suo basso tasso di errori, ma ogni alta fedeltà polimerasi PCR dovrebbe essere accettabile.
- Gel purificare i prodotti di PCR su un 1% gel di agarosio utilizzando il kit di gel QIAquick estrazione.
- Alcuni polimerasi aggiunge un singolo deossiadenosina (A) residui a fini 3 'dei prodotti di PCR, mentre altri no. Questi 3 'A code sono necessari per il TA TOPOpasso clonazione. La polimerasi KAPA HiFi non lascia 3 'A code. A-coda i prodotti PCR incubando 25 microlitri di gel-purificati prodotto PCR con 3 ml tampone di reazione 10x Taq, 1 ml 10 mM dNTPs, e 1 unità di Taq polimerasi a 72 ° C per 30 minuti.
- Clonare il gel-purificati prodotti PCR utilizzando il TOPO TA Cloning kit. Trasformare i prodotti della reazione TOPO nel fornita E. coli DH5α T1-R cellule competenti e piastra su LB / piastre di agar contenente 100 mg / ml ampicillina ed integrata con 1,6 mg di X-gal in base alle raccomandazioni del fabbricante.
- Invia singole colonie per il sequenziamento.
- Effettuare analisi di sequenza per determinare i tipi e le posizioni di mutazioni.
9. Rappresentante dei risultati:
Come notato in precedenza, usiamo un vettore positivo di controllo virale che esprime sia la GFP e un puromicina gene per la resistenza. Noi di solito vede il 50-75% delle cellule GFP + due giornidopo la trasfezione. Nel nostro infezioni, di solito vede il 50-70% delle cellule sono GFP + due giorni dopo l'infezione, ma prima selezione. Dopo la selezione è terminata,> 95% delle cellule sono + GFP.
Come esperimenti di rappresentanza, mostriamo gli effetti degli aiuti iperespressione o abbattere un fattore di riparazione nelle cellule Ramos. In particolare, abbiamo transfettate un vettore retrovirale sovraespressione per gli aiuti connessi attraverso un sito IRES per Thy1.1 insieme con il vettore retrovirale pKat2 imballaggio in 23 BOSC cellule. Virus contenenti media è stata filtrata e poi utilizzato per infettare le cellule Ramos. Entrambe le wild-type (WT) e AID sovraesprimono (AID hi) le cellule sono state singola cella seminati e coltivati per 3 settimane, ea quel punto sono stati analizzati per l'espressione genica attraverso qRT-PCR (Figura 2) e per la perdita di IgM da FACS (Figura 3 ). Per la colorazione FACS, le cellule sono state incubate con entrambe le 1:20 diluita PE marcate α-umano di anticorpi IgM e diluito 1:400 FITC-marcato α-ratto CD90/mouseCD90.1 (Thy1.1) anticorpi. In un esperimento separato, che contiene sia un lentivirus irrilevante shRNA (controllo) o un shRNA contro un fattore ad alta fedeltà di riparazione del DNA dovrebbe proteggere contro SHM sono state effettuate in 23 BOSC celle e quindi infettati in un clone di cellule SOCCORSO hi Ramos. Anche in questo caso, l'espressione genica (Figura 4) e la perdita di IgM (Figura 5) sono stati analizzati nei cloni singola cella dopo 3 settimane. Dal momento che il fattore di riparazione è pensato per fornire una protezione contro le mutazioni durante SHM, vediamo un aumento della perdita di IgM e SHM, in assenza del fattore. Se avessimo abbattuto un fattore coinvolto nella generazione SHM mutazioni associate, avremmo visto una diminuzione della perdita di IgM, invece.
Come previsto, il nostro qRT-PCR risultati mostrano un marcato aumento dell'espressione SOCCORSO dopo l'infezione di cellule con un vettore iperespressione AIUTO (Figura 2), mentre i livelli del fattore di caduta di riparazione del DNA in presenza di un shRNA mirato contro tale fattore (figura 4 ). Perché clon individualees possono variare, si potrebbe vedere qualche variazione nei livelli di espressione genica. Poiché l'espressione genica può variare, è necessario confermare l'espressione in ogni clone si prevede di analizzare ulteriormente. ShRNAs diverse contro lo stesso gene potrebbe avere effetti diversi sulla espressione genica e, così si dovrebbe schermo shRNAs diversi per determinare quale ha il maggiore impatto. Knockdown può anche essere confermata a livello proteico mediante immunoblotting. Allo stesso modo, cloni individuo può variare notevolmente a livello di perdita di IgM. Alcuni cloni potrebbero dimostrare un effetto "jackpot", dove una mutazione IgM è verificato in una delle prime divisioni cellulari - per esempio, si vedrà la perdita di IgM> 50% semplicemente perché una delle celle divenne IgM - alla stadio di due cellule. L'influenza di questi cloni è ridotto al minimo calcolando la media per diversi cloni singola cella.

Figura 1. Schematica per la trasmissioneperfezione, l'infezione e l'analisi di perdita di IgM. Si veda il testo per i dettagli.
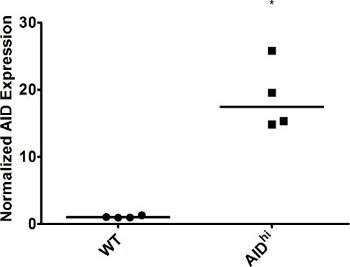
Figura 2. SOCCORSO sovraespressione in cellule Ramos. WT e AID hi cellule Ramos singola cella sono stati clonati e cresciuti per 3 settimane, in cui è stata misurata l'espressione SOCCORSO punto cloni singoli qRT-PCR. Espressione di GAPDH è stato utilizzato per la normalizzazione. WT è stato impostato a 1. Il valore mediano è indicato. * Indica un valore di p <0,01, calcolato come una coda di Student t-test.
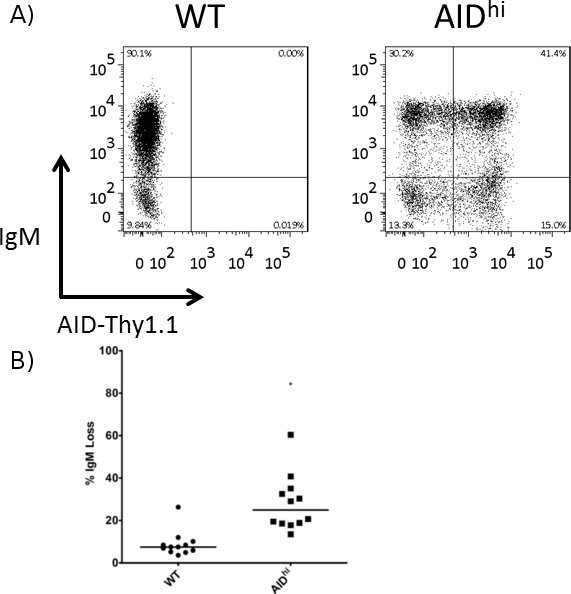
Figura 3. Aumentata perdita di IgM in celle hi AID rispetto alle cellule WT. IgM superficie è stata misurata mediante FACS al punto 3 settimane di tempo, e la percentuale di perdita di IgM è stato calcolato per WT e celle hi AIUTO. (A) Rappresentante trama FACS di un clone WT e un clone AID hi. L'AID sovraespressione vettore ha AID collegato a Thy1.1 tramite un sito IRES, in modo Thy1.1 espressione è utilizzata come un surrogato per l'espressione AIUTO. (B) La quantificazione della perdita di IgM nel WT diversi cloni hi AID. Il valore mediano è indicato. * Indica un valore di p <0,01, calcolato come una coda di Student t-test.
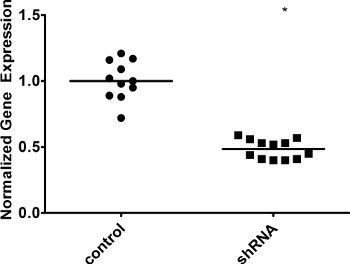
Figura 4. Knockdown di un fattore di riparazione nelle cellule hi AIUTO. Le cellule sono state infettate con un controllo sia irrilevante shRNA (controllo) o il nostro shRNA di interesse (shRNA), e singole cellule seminato. Dopo 3 settimane, l'espressione genica nei cloni individuali è stata misurata mediante qRT-PCR. Espressione di GAPDH è stato utilizzato per la normalizzazione. WT è stato impostato a 1. Il valore mediano è indicato. * Indica un valore di p <0,01, calcolato come una coda di Student t-test.
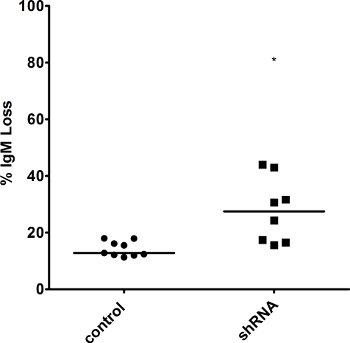
Figura 5. Perdita IgM Maggiorenelle cellule SOCCORSO hi con livelli abbattuto di un fattore di riparazione. IgM di superficie in diversi cloni è stata misurata mediante FACS al punto 3 settimane di tempo, e la percentuale di perdita di IgM è stata calcolata in cellule ancora AID sovraesprimono. Il valore mediano è indicato. * Indica un valore di p <0,01, calcolato come una coda di Student t-test.

. Tabella 1 sequenze primer per i geni Ig 19: il Cμ regione costante, la pesante catena regione V (V H), e la luce regione catena V (V L).
Discussione
Come discusso precedentemente, i modelli di linee cellulari per la diversificazione degli anticorpi sono diventati un punto di partenza per identificare nuove proteine che influenzano le diverse fasi durante la diversificazione degli anticorpi. Vi presentiamo qui un metodo per l'utilizzo di infezione virale alle proteine o knock-down o iperespressione in cellule B Ramos linea e quindi esaminare l'impatto sulla SHM.
Per questi studi, ci avvaliamo sia Ramos WT e AID hi...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Il pMSCV-AID-I-Thy1.1 e pKat2 vettori erano un gentile dono della DG Schatz e pVSV-G, pRSV-Rev, e pMDLg / pRRE vettori erano un gentile dono da BR Cullen.
Materiali
Reagenti suggerito - la maggior parte di questi possono essere sostituiti con prodotti simili di altri vendor.
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
|---|---|---|---|
| 6-ben chiaro TC-trattati piastre | Corning | 3516 | |
| 10 mL BD Luer-Loksyringes | BD Medical | 309604 | |
| 24-ben chiaro TC-trattati piastre | Corning | 3526 | |
| 96-polistirolo ben chiaro fondo piatto TC-trattati micropiastre | Corning | 3596 | |
| TC-100 millimetri trattati cultura piatti | Corning | 430167 | |
| Filtri per siringa Acrodisc, 0,45 micron | Pall Life Sciences | 4604 | |
| Agar | Teknova | A7777 | |
| Agarosio | GeneMate | E-3120-500 | |
| Ampicillina | Sigma | A0166 | 100 mg / mL in acqua |
| BD FACSCanto II citometro a flusso | BD Biosciences | o simili | |
| BD Falcon rotondo fondo polistirolo tubi | BD Biosciences | 352054 | per FACS |
| BOSC 23 celle | ATCC | CRL-11270 | |
| CO 2 incubatore capace di 37 ° C | |||
| DMEM (medio Dulbecco modificato Eagle) | Sigma | D6429 | |
| FBS (siero fetale bovino) | Gemini Bio-Prodotti | 100-106 | |
| FITC α-ratto CD90/mouse CD90.1 anticorpi | BioLegend | 202503 | FITCα-Thy1.1 |
| FuGENE 6 Transfection Reagent | Roche | 11814443001 | |
| HEPES soluzione tampone | Invitrogen | 15630-080 | |
| KAPA HiFi DNA polimerasi | KAPA Biosystems | KK2101 | |
| LB Broth (brodo lisogenia - Luria) Polvere | Difco | 240230 | |
| MISSIONE TRC shRNA magazzino glicerolo batterico | Sigma | shRNA vettori | |
| NCS (siero di vitello neonato) | Gemini Bio-Prodotti | 100-504 | |
| PBS (soluzione tampone fosfato) | Invitrogen | 70011 | diluito a 1x in acqua |
| PE-α umano anticorpi IgM | BioLegend | 314508 | |
| PGS (penicillina-streptomicina-glutamina soluzione) | Gemini Bio-Prodotti | 400-110 | |
| Polibrene (hexadimethrine bromuro) | Sigma | 107689 | 10 mg / ml in acqua |
| PureYield sistema Midiprep plasmidi | Promega | A2495 | |
| Puromicina | Sigma | P8833 | 250 mcg / ml in acqua |
| QIAquick gel kit di estrazione | QIAGEN | 28706 | |
| Ramos (RA 1) le cellule | ATCC | CRL-1596 | |
| RPMI-1640 medium | Sigma | R8758 | |
| SuperScript II | Invitrogen | 18064-022 | |
| SYBR VELOCE qPCR kit | KAPA Biosystems | KK4601 | |
| Taq DNA polimerasi | Invitrogen | 18038-042 | |
| TOPO TA Cloning Kit | Vitrogenerazione | K4520-01 | |
| TRIzol | Invitrogen | 15596-026 | |
| Wizard SV sistema di purificazione del DNA genomico | Promega | A2361 | |
| X-Gal [5-bromo-4-cloro-3-indoyl-β-D-galatopyranoside] | Growcells | C-5687 | 40 mg / ml in DMSO |
Riferimenti
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annu. Rev. Biochem. 76, 1-22 (2007).
- Peled, J. U. The biochemistry of somatic hypermutation. Annu. Rev. Immunol. 26, 481-511 (2008).
- Longerich, S., Basu, U., Alt, F., Storb, U. AID in somatic hypermutation and class switch recombination. Curr. Opin. Immunol. 18, 164-174 (2006).
- Bardwell, P. D. Altered somatic hypermutation and reduced class-switch recombination in exonuclease 1-mutant mice. Nat. Immunol. 5, 224-229 (2004).
- Martomo, S. A., Yang, W. W., Gearhart, P. J. A role for Msh6 but not Msh3 in somatic hypermutation and class switch recombination. J. Exp. Med. 200, 61-68 (2004).
- Phung, Q. H. Increased hypermutation at G and C nucleotides in immunoglobulin variable genes from mice deficient in the MSH2 mismatch repair protein. J. Exp. Med. 187, 1745-1751 (1998).
- Rada, C., Ehrenstein, M. R., Neuberger, M. S., Milstein, C. Hot spot focusing of somatic hypermutation in MSH2-deficient mice suggests two stages of mutational targeting. Immunity. 9, 135-141 (1998).
- Rada, C. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Curr. Biol. 12, 1748-1755 (2002).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase eta is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. J. Exp. Med. 204, 17-23 (2007).
- Masuda, K. DNA polymerase eta is a limiting factor for A:T mutations in Ig genes and contributes to antibody affinity maturation. Eur. J. Immunol. 38, 2796-2805 (2008).
- Lim, D. S., Hasty, P. A mutation in mouse rad51 results in an early embryonic lethal that is suppressed by a mutation in p53. Mol. Cell. Biol. 16, 7133-7143 (1996).
- Xiao, Y., Weaver, D. T. Conditional gene targeted deletion by Cre recombinase demonstrates the requirement for the double-strand break repair Mre11 protein in murine embryonic stem cells. Nucleic. Acids. Res. 25, 2985-2991 (1997).
- Zhu, J., Petersen, S., Tessarollo, L., Nussenzweig, A. Targeted disruption of the Nijmegen breakage syndrome gene NBS1 leads to early embryonic lethality in mice. Curr. Biol. 11, 105-109 (2001).
- Luo, G. Disruption of mRad50 causes embryonic stem cell lethality, abnormal embryonic development, and sensitivity to ionizing radiation. Proc. Natl. Acad. Sci. U. S. A. 96, 7376-7381 (1999).
- Pavri, R. Activation-induced cytidine deaminase targets DNA at sites of RNA polymerase II stalling by interaction with Spt5. Cell. 143, 122-133 (2010).
- Lee-Theilen, M., Matthews, A. J., Kelly, D., Zheng, S., Chaudhuri, J. CtIP promotes microhomology-mediated alternative end joining during class-switch recombination. Nat. Struct. Mol. Biol. 18, 75-79 (2011).
- Basu, U. The RNA exosome targets the AID cytidine deaminase to both strands of transcribed duplex DNA substrates. Cell. 144, 353-363 (2011).
- Yabuki, M., Fujii, M. M., Maizels, N. The MRE11-RAD50-NBS1 complex accelerates somatic hypermutation and gene conversion of immunoglobulin variable regions. Nat. Immunol. 6, 730-736 (2005).
- Sale, J. E., Neuberger, M. S. TdT-accessible breaks are scattered over the immunoglobulin V domain in a constitutively hypermutating B cell line. Immunity. 9, 859-869 (1998).
- Cumbers, S. J. Generation and iterative affinity maturation of antibodies in vitro using hypermutating B-cell lines. Nat. Biotechnol. 20, 1129-1134 (2002).
- Papavasiliou, F. N., Schatz, D. G. Cell-cycle-regulated DNA double-stranded breaks in somatic hypermutation of immunoglobulin genes. Nature. 408, 216-221 (2000).
- Parsa, J. Y. AID mutates a non-immunoglobulin transgene independent of chromosomal position. Mol. Immunol. 44, 567-575 (2007).
- Zhang, W. Clonal instability of V region hypermutation in the Ramos Burkitt's lymphoma cell line. Int. Immunol. 13, 1175-1184 (2001).
- Ukai, A. Induction of a:T mutations is dependent on cellular environment but independent of mutation frequency and target gene location. J. Immunol. 181, 7835-7842 (2008).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. J. Virol. 72, 8463-8471 (1998).
- Nakamura, M. High frequency class switching of an IgM+ B lymphoma clone CH12F3 to IgA+ cells. Int. Immunol. 8, 193-201 (1996).
- Muramatsu, M. Specific expression of activation-induced cytidine deaminase (AID), a novel member of the RNA-editing deaminase family in germinal center B cells. J. Biol. Chem. 274, 18470-18476 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon