Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Purificación por afinidad de los complejos de ribonucleoproteína Influenza Virus de la cromatina de las células infectadas
En este artículo
Resumen
Los virus de influenza replicar su genoma de ARN, en asociación con la cromatina de la célula huésped. A continuación, presentamos un método para purificar intactas ribonucleoprotein complejos virales de la cromatina de las células infectadas. Purificada complejos virales pueden ser analizados por tanto Western blot y extensión del cebador de contenido de proteínas y ARN, respectivamente.
Resumen
Como todos los virus negativo-hebra de ARN, el genoma de los virus de influenza está empaquetado en forma de complejos de ribonucleoproteína (vRNP virales), en los que se encapsidado el genoma de cadena simple por la nucleoproteína (NP), y asociado con el complejo de polimerasa trimérica consistente de la PA, PB1, PB2 y subunidades. Sin embargo, en contraste con la mayoría de los virus ARN, virus de la influenza realizar la síntesis de ARN viral en los núcleos de las células infectadas. Interesantemente, la síntesis de ARNm viral utiliza celulares pre-ARNm como cebadores, y se ha propuesto que este proceso se lleva a cabo en la cromatina 1. Las interacciones entre la polimerasa viral y el huésped ARN polimerasa II, así como entre NP y nucleosomas huésped también se han caracterizado 1,2.
Recientemente, la generación de virus de la influenza recombinante que codifican un Uno-estrep-Tag genéticamente fusionado al extremo C-terminal de la subunidad PB2 de la polimerasa viral (RWSN-PB2-Strep 3) ha seres descrito. Estos virus recombinantes permite la purificación de PB2 que contienen complejos, incluyendo vRNPs, desde las células infectadas. Para obtener purificada vRNPs, cultivos de células infectadas, y vRNPs son purificados por afinidad a partir de lisados derivados de estas células. Sin embargo, los procedimientos de lisis utilizado hasta la fecha se han basado en un paso de lisis detergente, que, a pesar de la presencia de una nucleasa general, a menudo extraer cromatina determinada único material ineficientemente.
Nuestro trabajo preliminar sugiere que una gran parte de vRNPs nucleares no se extrajeron durante la lisis celular tradicional, y por lo tanto no podría ser purificado por afinidad. Para aumentar esta eficiencia de la extracción, y para separar la cromatina se dirigía de no cromatina determinada vRNPs nucleares, hemos adaptado un protocolo de paso a paso la extracción subcelular de la influenza células infectadas por virus. En pocas palabras, este procedimiento primero separa los núcleos de la célula y, a continuación extractos solubles proteínas nucleares (aquí denominado "nucleoplasmic" fracción).El restante material nuclear insoluble se digiere con Benzonase, un ADN inespecífica / ARN nucleasa, seguido de dos pasos de extracción de sal: en primer lugar utilizando 150 mM de NaCl (denominado "CH150"), a continuación, NaCl 500 mM ("ch500") (fig. 1 ). Estos pasos de extracción de sal fueron elegidos en base a nuestra observación de que 500 mM de NaCl fue suficiente para solubilizar el 85% de vRNPs nucleares y aún así permitir la unión de vRNPs etiquetados a la matriz de afinidad.
Después de fraccionamiento subcelular de las células infectadas, es posible purificar por afinidad vRNPs PB2-etiquetados de cada fracción individual y analizar su proteína y componentes de ARN utilizando Western Blot y extensión del cebador, respectivamente. Recientemente, hemos utilizado este método para descubrir que las exportaciones vRNP forma de complejos en los puntos finales después de la infección en la fracción de la cromatina se extrajo con 500 mM de NaCl (ch500) 3.
Protocolo
Un diagrama de flujo esquemático del protocolo se muestra en la fig. 1 y una tabla de los reactivos se presentan a continuación.
1. Infección (16 - 24 h)
- Semilla a las células HeLa en 5 150 mm platos de plástico de cultivo de células en un medio de Dulbecco modificado de Eagle (DMEM)-medio-alto de glucosa, de tal manera que alcanzará aproximadamente el 80% de confluencia al día siguiente (aproximadamente 5 x 10 8 células). Es importante que las células no llegan a 100% de confluencia.
- Al día siguiente, se diluye la Strep-etiquetados stock de virus de la gripe en salina tamponada con fosfato (PBS) que contenía 0,3% de albúmina sérica bovina (BSA) a una multiplicidad de infección de 3.
- Eliminar el medio de crecimiento de las células, se lava una vez con PBS, y se añaden 10 ml de PBS que contenía el virus a las células. Incubar durante 1 hora a temperatura ambiente.
- Sustituir el medio de infección con DMEM-alto de glucosa, y continuar la incubación a 37 ° C.
2. Fraccionamiento subcelular (3 h)
- Lavar las células una vez con PBS frío y luego suspender las células en PBS frío con una espátula de goma y la transferencia a un tubo cónico de 50 ml.
- Las células de pellets en una centrífuga de mesa durante 5 min a 1.000 rpm y luego aspirar el PBS.
- Resuspender pellet de células en 10 ml de tampón de sacarosa frío (10 mM, pH 7,9 HEPES, 10 mM de KCl, 2 mM de acetato de Mg (MgOAc), 3 mM de CaCl 2, 340 mM de sacarosa, 1 mM de ditiotreitol (DTT), 1% de mezcla de inhibidor de la proteasa) . Después de la suspensión, pasar las células a través de una punta de pipeta 200 l unido a un plástico de 10 ml de suero pipeta para interrumpir grupos de células.
- Incubar 10 minutos en hielo. Invierta suavemente periódicamente para volver a suspender las células.
- Añadir Nonidet P-40 (NP-40) a una concentración final de 0,5% y vórtice a alta velocidad durante 15 segundos. Inmediatamente centrifugar durante 10 minutos a 3500 xg a 4 ° C en un centrífugas de mesage.
- Eliminar el sobrenadante y se almacena a 4 º C. Esta es la fracción citoplásmica. El sedimento debe ser más pequeño y más blanca que la célula original de pellets. Todas las fracciones se pueden almacenar por lo menos durante 4 horas a 4 ° C.
- Lavar el precipitado en 5 ml de tampón frío sacarosa, de re-centrífuga como en el paso 2,5 y descartar el sobrenadante.
- Resuspender el sedimento (que contiene núcleos) en 1,5 ml de tampón de extracción en frío nucleoplasmic (HEPES 50 mM, pH 7,9, 150 mM de KOAc, 1,5 mM MgCl 2, 0,1% de NP-40, 1 mM de DTT, 1% de mezcla de inhibidor de la proteasa).
- Pasar a un frío, en cristal, 4 ml Dounce homogeneizadora con una mano apretada y homogeneizar los núcleos con 20 golpes.
Evite la formación de espuma durante la homogeneización. La resistencia debe aumentar después de aproximadamente 10 golpes. Homogeneización eficiente puede ser confirmado con un microscopio de contraste de fase. - Transferir los núcleos homogeneizada a un tubo de microcentrífuga de 1,5 ml y se incuba 20 minutos a una rueda que gira a 4 °C.
- Centrifugar a 16.000 x g en una microcentrífuga a 4 ° C durante 10 min.
- Eliminar el sobrenadante y se almacena a 4 º C. Esta es la fracción nucleoplasmic.
- Resuspender el precipitado en 1,5 ml de tampón de digestión (50 mM HEPES pH 7,9, 10 mM de NaCl, 1,5 mM MgCl2, 1 mM DTT, 1% de mezcla inhibidor de la proteasa, 100 U / ml (Benzonase para el análisis de ARN, sustituto con 20 U / ml de RNasa libre de DNasa I)) precalentado a 37 ° C, y se incuba 10 minutos a 37 ° C.
- Añadir 42 l de NaCl 5 M (para la concentración final de 150 mM de NaCl) y se incuba 20 minutos más a 4 ° C.
- Centrifugar a 16.000 x g en una microcentrífuga a 4 ° C durante 10 min.
- Eliminar el sobrenadante y se almacena a 4 º C. Esta es la fracción CH150.
- Resuspender el pellet en 1,5 ml de frío tampón de alta salinidad (50 mM, pH 7,9 HEPES, 500 mM de NaCl, 1,5 mM de MgCl 2, 0,1% de NP-40, 1 mM de DTT, 1% de mezcla de inhibidor de proteasa) y se incuba 20 minutos en hielo.
- Centrifugar a 16.000 x g </ Em> en una microcentrífuga a 4 ° C durante 10 min.
- Eliminar el sobrenadante y se almacena a 4 º C. Esta es la fracción ch500.
3. Strep-etiqueta Purificación (2 h)
- Añadir 300 l de NaCl 5 M a la fracción citoplásmica (para la concentración final de 150 mM de NaCl).
- Alícuota de 100 l de concentrado de Strep-Tactin Sefarosa por fracción en un tubo cónico de 15 ml.
- Añadir 10 volúmenes de tampón de lavado Strep (20 mM, pH 7,9 HEPES, 150 mM de NaCl (500 mM de NaCl para ch500 muestras), 1 mM EDTA) a perlas, invertir el tubo para mezclar, y centrifugar 5 min a 1.000 x g en una mesa centrifugar.
- Descartar el sobrenadante, repita el paso 3,3 dos veces y luego volver a suspender el pellet de talón en un volumen igual de tampón de lavado Strep.
- Añadir 200 l de lavado Strep-Tactin suspensión cordón a cada fracción (en tubos de 1,5 ml para microcentrífuga nuceloplasmic y CH150, y ch500 fracciones y un tubo cónico de 15 ml para la fracción citoplasmática).
- Girartubos de 1 hora a 4 ° C en una rueda giratoria.
- Centrifugar los tubos de 1 min a 1000 x g a 4 ° C. Desechar el sobrenadante.
- Añadir 1 ml de solución de lavado en frío por estreptococo a cada tubo. Transferencia de cuentas de tubo de 15 ml (fracción citoplasmática) al tubo de 1,5 ml. Lavar todos los tubos de 1 min a 4 ° C, después de centrifugación como en el paso 3,7. Repita este paso de lavado dos veces.
- Después del último lavado, eliminar todos los rastros de tampón de lavado utilizando una pipeta de punta plana. Añadir 100 l de buffer de elución (tampón de lavado que contiene Strep 2,5 mM destiobiotina) y mezclar suavemente.
- Incubar 15 minutos en hielo. Mezclar suavemente perlas de vez en cuando tocando el fondo de los tubos.
- Tubos de centrífuga 1 min a 1000 x g a 4 ° C en microcentrífuga. Recoger el líquido sobrenadante que contienen eluidas Strep-etiquetados vRNPs.
4. Los resultados representativos
La eficacia del fraccionamiento se juzga mejor por análisis de transferencia Western de los lisados utilizando fraccionadas ANTIBodies específicos para los marcadores subcelulares (Fig. 2). Específicamente, el fraccionamiento subnuclear éxito debería muestran poca o ninguna celular ARN polimerasa II (Pol II) en la nucleoplasmic, o fracción soluble nuclear 4, y la mayoría se extrajo con 150 mM de NaCl 5. Una degradación de Pol II es también reproducible observada en las células infectadas por virus de la gripe en comparación con las células no infectadas, en consonancia con la literatura 6.
Purificación de vRNPs se juzga mejor por la plata o tinción de Coomassie, como se muestra para un eluato citoplásmica en la figura. 3. En nuestra experiencia, la mayoría Strep-PB2 se purifica como parte de un vRNP totalmente formado, es decir, soluble polimerasa poco es capturado. Esto se refleja en el PN de alta: PA/PB1/PB2 relación observada por la plata o la tinción de Coomassie. Una proporción menor sugiere contaminación tampón con RNasas, como varios ARNasas ubicuas (como RNasa A) se sabe que el ARN viral escinden de tal manera que la disociaciónse comió la polimerasa de la NP multímeros 7. Curiosamente, el tratamiento con Benzonase solo, mientras que suficiente para digerir ARN viral, no parece tener un efecto en las proporciones estequiométricas de proteínas vRNP. A pesar de que no se puede explicar este fenómeno, se observó alteración de vRNPs después de la digestión con RNasas otros (datos no mostrados), sugiriendo que Benzonase puede dejar algunas regiones importantes de la estructura del ARN intacto. Después de la purificación de vRNPs desde el citoplasma y nucleoplasma (realizado sin digestión con nucleasa), 8 bandas débiles pero discreto en altos pesos moleculares pueden ser observadas por tinción con plata, que corresponden a los pesos moleculares predichos de los 8 segmentos del genoma de la gripe (ver ref. 3 ).
Las cantidades relativas de vRNPs purificadas a partir de cada fracción a las 9 horas después de la infección se muestran en la figura. 4. La distribución de vRNPs varía durante el curso de la infección, con fuerte acumulación en la fracción CH150 en puntos de tiempo primeros, yuna mayor acumulación en el nucleoplasma y el citoplasma en momentos finales.
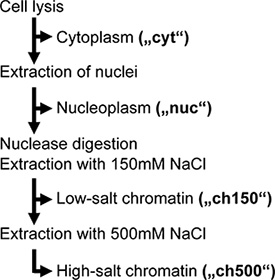
Figura 1. Diagrama de flujo del fraccionamiento subcelular. Adaptado de ref. 3.
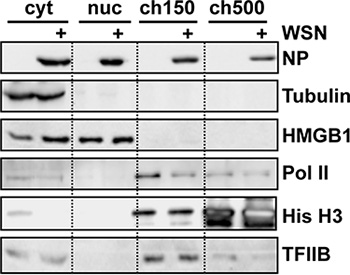
Figura 2. Análisis Western blot de fracciones subcelulares de la influenza células infectadas por virus. 10 9 células HeLa fueron infectados con la gripe WSN cepa del virus durante 9 horas antes del fraccionamiento subcelular. Igual cantidad de proteína total fueron cargados en cada carril y se analizaron con los anticuerpos que aparecen. ARN polimerasa II (Pol II) se detectó utilizando clon 8WG16, que reconoce todas las formas del dominio C-terminal (la banda hipofosforilada es más prominente). Adaptado de ref. 3.

Figura 3.Análisis de tinción de plata de la vRNPs purificada. Se infectaron células HeLa con RWSN (como un control negativo) o RWSN-PB2-Strep durante 9 h, seguido de purificación por estreptococo de la fracción citoplásmica. Asterisco indica que las bandas que no son visibles después de la digestión con RNasa. Adaptado de ref. 3.

Figura 4. Análisis de tinción de plata de la vRNPs purificados a partir de diferentes fracciones celulares. 10 9 células HeLa fueron infectados con RWSN-PB2-estreptococo o RWSN durante 9 horas seguidas por fraccionamiento subcelular y purificación Strep. Cantidades iguales de eluato de cada fracción se cargaron. Adaptado de ref. 3.
Discusión
Aunque muchos estudios han identificado recientemente las proteínas individuales o de redes celulares implicados en la infección por el virus de la influenza 8, el significado funcional de la mayoría de estas interacciones aún no está claro. Dada la dependencia absoluta de la cromatina a base de funciones para la síntesis de ARN del virus de influenza y la compleja naturaleza biofísica y bioquímica del núcleo 9, nuevas técnicas se requieren para dilucidar estas funciones. El fraccionamien...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Los autores desean agradecer a Nada Naffakh y Marie-Anne Rameix-Welti (Instituto Pasteur) para el virus RWSN-PB2-Strep.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| DMEM-alta glucosa | Gibco | 11965-092 | |
| BSA | Sigma | A9418 | |
| Inhibidor de la proteasa mezcla G | Serva | 39101 | |
| Benzonase nucleasa | Novagen | 71206 | 25 U / l |
| DNasa I, libre de RNasa | ThermoScientific | EN0523 | 50 U / l |
| Homogeneizador Dounce | Wheaton | 432-1271 | Utilice el tipo "B" maja |
| Strep-Tactin Sepharose | IBA GmbH | 2-1201-025 | 50% formato de columna suspensión también se puede utilizar |
| Destiobiotina | IBAGmbH | 2-1000-002 |
Referencias
- Engelhardt, O. G., Smith, M., Fodor, E. Association of the influenza A virus RNA-dependent RNA polymerase with cellular RNA polymerase II. J. Virol. 79, 5812-5812 (2005).
- Garcia-Robles, I., Akarsu, H., Müller, C. W., Ruigrok, R., Baudin, F. Interaction of influenza virus proteins with nucleosomes. Virology. 332, 329 (2005).
- Chase, G. P., Rameix-Welti, M. A., Zvirbilene, A., Zvirblis, G., Götz, V., Wolff, T., Naffakh, N., Schwemmle, M. Influenza virus ribonucleoprotein complexes gain preferential access to cellular export machinery through chromatin targeting. PLoS Pathogens. 7, e1002187 (2011).
- Kimura, H., Tao, Y., Roeder, R. G., Cook, P. R. Quantitation of RNA polymerase II and its transcription factors in an HeLa cell: little soluble holoenzyme but significant amounts of polymerases attached to the nuclear substructure. Mol. Cell. Biol. 19, 5383-5383 (1999).
- Henikoff, S., Henikoff, J. G., Sakai, A., Loeb, G. B., Ahmad, K. Genome-wide profiling of salt fractions maps physical properties of chromatin. Genome Res. 19, 460 (2009).
- Rodriguez, A., Perez-Gonzalez, A., Nieto, A. Influenza virus infection causes specific degradation of the largest subunit of cellular RNA polymerase II. J. Virol. 81, 5315-5315 (2007).
- Ye, Z., Liu, T., Offringa, D. P., McInnis, J., Levandowski, R. A. Association of influenza virus matrix protein with ribonucleoproteins. J. Virol. 73, 7467-7467 (1999).
- Watanabe, T., Watanabe, S., Kawaoka, T., Y, . Cellular networks involved in the influenza virus life cycle. Cell Host Microbe. 7, 427 (2010).
- Engelhardt, O. G., Fodor, E. Functional association between viral and cellular transcription during influenza virus infection. Rev. Med. Virol. 16, 329-345 (2006).
- Schwartz, L. B., Sklar, V. E., Jaehning, J. A., Weinmann, R., Roeder, R. G. Isolation and partial characterization of the multiple forms of deoxyribonucleic acid-dependent ribonucleic acid polymerase in the mouse myeloma, MOPC 315. J. Biol. Chem. 249, 5889 (1974).
- Lambert, J. P., Baetz, K., Figeys, D. Of proteins and DNA--proteomic role in the field of chromatin research. Mol. Biosyst. 6, 30 (2010).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J. Virol. 85, 6618 (2011).
- Liao, T. L., Wu, C. Y., Su, W. C., Jeng, K. S., Lai, M. M. Ubiquitination and deubiquitination of NP protein regulates influenza A virus RNA replication. EMBO J. 29, 3879 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados