Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Affinitätsreinigung des Influenza-Virus Ribonukleoproteinkomplexe aus dem Chromatin der infizierten Zellen
In diesem Artikel
Zusammenfassung
Influenza-Viren replizieren ihre RNA-Genom in Verbindung mit Host-Zell-Chromatin. Hier präsentieren wir eine Methode, um intakte virale Ribonukleoproteinkomplexe aus dem Chromatin von infizierten Zellen zu reinigen. Gereinigten viralen Komplexe können sowohl durch Western-Blot und Primer-Extension von Protein-und RNA-Gehalt analysiert werden, beziehungsweise.
Zusammenfassung
Wie alle Negativ-Strang-RNA-Viren, wird das Genom von Influenzaviren in Form von viralen Ribonukleoproteinkomplexe (vRNP), in dem die einzelsträngige Genoms durch das Nukleoprotein (NP) eingekapselt ist, und mit der Polymerase-Komplex aus trimeren verpackt der PA, PB1, PB2 und Untereinheiten. Allerdings, im Gegensatz zu den meisten RNA-Viren, Influenza-Viren führen virale RNA-Synthese in den Kernen der infizierten Zellen. Interessanterweise verwendet virale mRNA-Synthese zelluläre prä-mRNAs als Primer, und es wurde vorgeschlagen, dass dieser Prozess stattfindet Chromatin 1. Wechselwirkungen zwischen dem viralen Polymerase und der Wirts-RNA-Polymerase II, sowie zwischen NP und Host Nukleosomen wurden auch 1,2 ist.
Vor kurzem hat die Erzeugung von rekombinanten Influenza Viren, die für eine Ein-Strep-Tag genetisch mit dem C-Terminus des PB2-Untereinheit der viralen Polymerase (rWSN-PB2-Strep 3) fusioniert hat seinen beschrieben. Diese rekombinanten Viren können die Reinigung von PB2-enthaltende Komplexe, einschließlich vRNPs, aus infizierten Zellen. Zur Gewinnung gereinigter vRNPs werden Zellkulturen infiziert und vRNPs sind affinitätsgereinigt aus Lysaten aus diesen Zellen abgeleitet. Allerdings haben die Lyse bisher angewandten auf einer Schritt-Detergens-Lyse, die, trotz der Gegenwart eines allgemeinen Nuklease, oft zu extrahieren Chromatin-gebundene Material nur ineffizient gestützt.
Unsere Vorarbeiten schlug vor, dass ein großer Teil der nuklearen nicht vRNPs wurden während der traditionellen Zell-Lyse extrahiert, und konnte deshalb nicht Affinität gereinigt werden. Um diese Extraktion Effizienz zu steigern, und zur Trennung von nicht-nuklearen Chromatin-gebundenen vRNPs Chromatin-gebunden, passten wir einen stufenweisen Extraktion subzelluläre Protokoll, um Influenza-Virus-infizierten Zellen. Kurz gesagt, dieses Verfahren zuerst trennt die Kerne aus der Zelle und extrahiert dann lösliche nukleäre Proteine (hier als die "nukleoplasmatischen"-Fraktion).Das verbleibende unlösliche nukleare Material wird dann mit Benzonase, ein unspezifischer DNA / RNA-Nuclease, die von zwei Salzgewinnung Schritte befolgt verdaut: zuerst mit 150 mM NaCl (genannt "CH150"), dann 500 mM NaCl ("CH500") (Abb. 1 ). Diese Salzgewinnung Schritte wurden auf der Grundlage unserer Beobachtung gewählt, dass 500 mM NaCl ausreichend, um über 85% der nuklearen vRNPs solubilisieren dennoch erlauben die Bindung von getaggt vRNPs an die Affinitätsmatrix war.
Nach subzelluläre Fraktionierung der infizierten Zellen, ist es möglich, Affinitätsreinigung PB2-markierten vRNPs von jedem einzelnen Fraktion und Analyse der Protein und RNA-Komponenten unter Verwendung von Western-Blot und Primer-Extension, jeweils. Vor kurzem verwendeten wir diese Methode, um diese vRNP Export Komplexe Form während des späten Punkte nach der Infektion auf der Chromatin-Fraktion mit 500 mM NaCl (CH500) 3 extrahiert entdecken.
Protokoll
Ein schematisches Flussdiagramm des Protokolls ist in gezeigt. 1 und eine Tabelle von Reagenzien wird unten dargestellt.
1. Infection (16 - 24 h)
- Samen aus HeLa-Zellen in 5 150 mm Zellkulturschalen in Dulbeccos modifiziertem Eagle-Medium (DMEM) hohen Glucose-Medium, so dass sie erreichen etwa 80% Konfluenz wurden die folgenden Tag (etwa 5 × 10 8 Zellen). Es ist wichtig, dass die Zellen nicht erreichen 100% Konfluenz.
- Am nächsten Tag, verdünnen Strep-Tag Influenza Virus-Stock in Phosphat-gepufferter Salzlösung (PBS) mit 0,3% Rinderserumalbumin (BSA) mit einer Multiplizität der Infektion von 3.
- Entfernen des Wachstumsmediums von den Zellen einmal mit PBS gewaschen, und 10 ml PBS, enthaltend das Virus zu den Zellen. Inkubation für 1 h bei Raumtemperatur.
- Ersetzen Sie die Infektion mit DMEM Medium-High Glucose, und weiterhin Inkubation bei 37 ° C.
2. Subzelluläre Fraktionierung (3 h)
- Waschen der Zellen einmal mit kaltem PBS gewaschen und dann auszusetzen Zellen in kaltem PBS mit einem Gummischaber und in einen 50 ml konischen Röhrchen.
- Pellet-Zellen in einer Tischzentrifuge für 5 min bei 1.000 rpm und dann absaugen PBS.
- Resuspendieren Zellpellet wurde in 10 ml kaltem Saccharose-Puffer (10 mM HEPES pH 7,9, 10 mM KCl, 2 mM Mg-Acetat (MgOAc), 3 mM CaCl 2, 340 mM Saccharose, 1 mM Dithiothreitol (DTT), 1% Protease-Inhibitor-Mix) . Nach Aussetzung, passieren Zellen durch eine 200 pl Pipettenspitze an einem 10 ml Kunststoff-Pipette Serum zu Zellklumpen zu stören.
- Inkubieren Sie 10 min auf Eis. Sanft in regelmäßigen Abständen zu invertieren, um Zellen zu resuspendieren.
- In Nonidet P-40 (NP-40) bis zu einer Endkonzentration von 0,5% und Wirbel mit hoher Geschwindigkeit für 15 Sekunden. Sofort für 10 min zentrifugieren bei 3.500 × g bei 4 ° C in einer Table-Top-KreiselpumpenGE.
- Überstand entfernen und lagern bei 4 ° C. Dies ist die cytoplasmatische Fraktion. Das Pellet sollte kleiner und weißer als der ursprüngliche Zellpellet. Alle Fraktionen für mindestens 4 h bei 4 ° C gelagert werden
- Waschen Sie das Pellet in 5 ml kaltem Saccharose-Puffer, Re-Zentrifuge, wie in Schritt 2,5 und den Überstand verwerfen.
- Das Pellet (mit Kernen) in 1,5 ml kaltem nukleoplasmatischen Extraktionspuffer (50 mM HEPES pH 7,9, 150 mM KOAc, 1,5 mM MgCl 2, 0,1% NP-40, 1 mM DTT, 1% Protease-Inhibitor-Mix).
- Transfer zu einem gekühlten, Ganzglas-4 ml Dounce Homogenisator mit einem eng anliegenden Stößel und homogenisieren die Kerne mit 20 Schlägen.
Vermeiden Sie Schaumbildung während der Homogenisierung. Der Widerstand sollte nach etwa 10 Schläge erhöhen. Effiziente Homogenisierung kann unter einem Phasenkontrast-Mikroskop bestätigt werden. - Übertragen des homogenisierten Kerne in ein 1,5 ml Mikrozentrifugenröhrchen und Inkubieren 20 min auf einem rotierenden Rad bei 4 °C.
- Zentrifuge bei 16.000 × g in einer Mikrozentrifuge bei 4 ° C für 10 min.
- Überstand entfernen und lagern bei 4 ° C. Dies ist die nukleoplasmatischen Fraktion.
- Pellet in 1,5 ml Verdauung Puffer (50 mM HEPES pH 7,9, 10 mM NaCl, 1,5 mM MgCl 2, 1 mM DTT, 1% Protease-Inhibitor-Mix, 100 U / ml Benzonase (für RNA-Analyse, Ersatz mit 20 U / ml RNase -freier DNase I)) vorgewärmt auf 37 ° C, und die Platte 10 min bei 37 ° C
- In 42 ul 5 M NaCl (für Endkonzentration von 150 mM NaCl) und inkubieren 20 min weiter bei 4 ° C
- Zentrifuge bei 16.000 × g in einer Mikrozentrifuge bei 4 ° C für 10 min.
- Überstand entfernen und lagern bei 4 ° C. Dies ist der CH150-Fraktion.
- Pellet in 1,5 ml kaltem High-Salz-Puffer (50 mM HEPES pH 7,9, 500 mM NaCl, 1,5 mM MgCl 2, 0,1% NP-40, 1 mM DTT, 1% Protease-Inhibitor-Mix) und inkubieren 20 min auf Eis.
- Zentrifugieren bei 16.000 x g </ Em> in einer Mikrozentrifuge bei 4 ° C für 10 min.
- Überstand entfernen und lagern bei 4 ° C. Dies ist der CH500-Fraktion.
3. Strep-Tag Reinigung (2 h)
- Dann werden 300 ul 5 M NaCl zu der zytoplasmatischen Fraktion (für Endkonzentration von 150 mM NaCl).
- Aliquotieren 100 ul verpackt Strep-Tactin Sepharosekügelchen pro Fraktion in einem 15 ml konischen Röhrchen.
- 10 Volumina Strep Waschpuffer (20 mM HEPES pH 7,9, 150 mM NaCl (500 mM NaCl CH500 Proben), 1 mM EDTA), um die Kügelchen, das Röhrchen zu mischen, und 5 min zentrifugiert bei 1000 × g in einer Tischzentrifuge zentrifugieren.
- Überstand verwerfen, wiederholen Sie Schritt 3,3 und dann zweimal resuspendieren den Wulst Pellet in einem gleichen Volumen von Strep Waschpuffer.
- Fügen Sie 200 ul gewaschene Strep-Tactin Kügelchenaufschlämmung zu jeder Fraktion (in 1,5 ml Mikrozentrifugenröhrchen für nuceloplasmic, CH150, CH500 und Fraktionen und einem 15 ml konischen Röhrchen für cytoplasmatische Fraktion).
- DrehenRöhrchen 1 h bei 4 ° C auf einem rotierenden Rad.
- Zentrifugenröhrchen 1 min bei 1.000 × g bei 4 ° C Überstand verwerfen.
- 1 ml kaltem Strep Waschpuffer in jedes Röhrchen. Übertragen Sie Perlen aus 15 ml Tube (cytoplasmatische Fraktion) bis 1,5 ml Röhrchen. Alle Röhrchen 1 min bei 4 ° C, Zentrifugieren nach wie in Schritt 3.7. Wiederholen Sie diesen Waschschritt zweimal.
- Nach dem letzten Waschschritt, entfernen Sie alle Spuren von Waschpuffer mit einem flachen Pipettenspitze. Geben Sie 100 ul Elutionspuffer (Strep-Waschpuffer mit 2,5 mM Desthiobiotin) und vorsichtig mischen.
- Inkubieren für 15 Minuten auf Eis. Vorsichtig mischen Perlen gelegentlich durch Tippen auf den Boden der Rohre.
- Zentrifugenröhrchen 1 min bei 1000 x g bei 4 ° C in Mikrozentrifuge. Sammle Überstand mit eluierten Strep-getaggten vRNPs.
4. Repräsentative Ergebnisse
Die Effizienz der Fraktionierung besten durch Western-Blot-Analyse der aufgetrennten Lysaten unter Verwendung antib beurteiltOdies spezifischen Marker für subzelluläre (Abb. 2). Insbesondere sollte ein erfolgreiches subnuklearen Fraktionierung geringe oder keine zelluläre RNA-Polymerase II (Pol II) in der nukleoplasmatischen oder lösliche Kernfraktion 4 und am meisten sollte mit 150 mM NaCl 5 extrahiert werden. Ein Abbau von Pol II ist auch reproduzierbar in Influenza-Virus-infizierten Zellen im Vergleich zu nicht infizierten Zellen, in Übereinstimmung mit der Literatur 6 beobachtet.
Reinigung von vRNPs wird am besten durch Silber oder Coomassie-Färbung beurteilt, wie für eine zytoplasmatische Eluat in gezeigt. 3. Nach unserer Erfahrung wird das meiste Strep-PB2 in als Teil eines vollständig geformten vRNP, dh wenig löslich Polymerase wird gefangen genommen gereinigt. PA/PB1/PB2 Verhältnis von Silber oder Coomassie-Färbung beobachtet: Dies wird in der hohen NP wider. Ein geringeres Verhältnis schlägt Puffer Kontamination mit RNasen, da mehrere allgegenwärtige RNasen (wie RNase A) zu spalten viralen RNA in einer solchen Weise, dass Dissoziation bekanntaß die Polymerase von NP-Multimere 7. Interessanterweise führte die Behandlung mit Benzonase allein, während die ausreicht, um virale RNA zu verdauen, scheint keinen Einfluss auf die stöchiometrischen Verhältnisse der vRNP Proteine haben. Obwohl wir nicht erklären kann dieses Phänomen beobachteten wir Unterbrechung der vRNPs nach Verdau mit anderen RNasen (Daten nicht gezeigt), was darauf hindeutet, dass Benzonase verlassen können bestimmte strukturelle Bedeutung RNA-Regionen intakt. Nach Reinigung des vRNPs aus dem Zytoplasma und Zellkern (durchgeführt, ohne Nucleaseverdau) können 8 schwach, aber diskrete Banden bei hohen Molekulargewichten durch Silberfärbung, der den vorhergesagten Molekulargewichten der 8 Influenza Genomsegmente entsprechen (siehe ref. 3 beobachtet werden ).
Die relativen Mengen der vRNPs von jeder Fraktion bei 9 h nach Infektion gereinigt werden in gezeigt. 4. Die Verteilung der vRNPs ändert sich im Verlauf der Infektion, mit der stärksten Anreicherung in der CH150-Fraktion zu einem frühen Zeitpunkt, underhöhte Akkumulation im Zellkern und Zytoplasma zu spät Zeitpunkten.
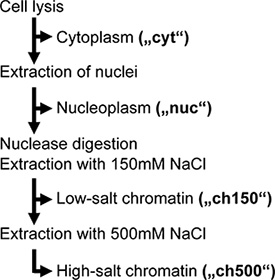
Abbildung 1. Flussdiagramm der subzellulären Fraktionierung. Abgewandelter Form aus Lit.. 3.
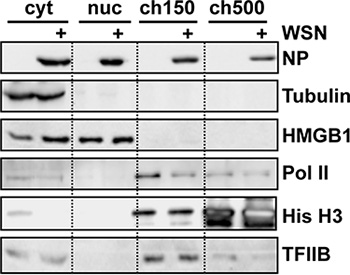
2. Western-Blot-Analyse der subzellulären Fraktionen aus Influenza-Virus-infizierten Zellen. 10 9 HeLa-Zellen wurden mit Virusstamm WSN 9 h vor subzelluläre Fraktionierung infiziert. Gleiche Mengen an Gesamtprotein wurden pro Spur geladen und unter Verwendung der Antikörper gezeigt. RNA Polymerase II (Pol II) wurde unter Verwendung von Klon 8WG16, die alle Formen des C-terminalen Domäne (die Band ist hypophosphorylierte prominentesten) erkennt. Abgewandelter Form aus Lit.. 3.

Abbildung 3.Silberfärbung Analyse der gereinigten vRNPs. HeLa-Zellen wurden mit rWSN (als Negativkontrolle) oder rWSN-PB2-Strep für 9 h, Strep Reinigung aus der zytoplasmatischen Fraktion gefolgt infiziert. Asterisk bezeichnet Bands, die nicht sichtbar sind nach RNase-Verdauung. Abgewandelter Form aus Lit.. 3.

Abbildung 4. Silberfärbung Analyse vRNPs aus verschiedenen zellulären Fraktionen gereinigt. 10 9 HeLa-Zellen wurden mit rWSN-PB2-Strep oder rWSN für 9 h infiziert, gefolgt von subzelluläre Fraktionierung und Reinigung Strep. Gleiche Mengen des Eluats aus jeder Fraktion wurden geladen. Abgewandelter Form aus Lit.. 3.
Diskussion
Während viele Studien, die kürzlich einzelne Proteine oder Mobilfunknetze in Influenzavirus-Infektion beteiligt sind 8 identifiziert haben, bleibt die funktionelle Bedeutung der meisten dieser Interaktionen unklar. Angesichts der absoluten Abhängigkeit von Chromatin-basierten Funktionen für Influenza-Virus RNA-Synthese und die komplexe biophysikalische und biochemische Natur des Kerns 9, werden neue Techniken erforderlich, um diese Funktionen aufzuklären. Die Fraktionierung subnuklearen ...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren bedanken sich bei Nada Naffakh und Marie-Anne Rameix-Welti (Institut Pasteur) für die rWSN-PB2-Strep Virus danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| DMEM-Hochglycose | Gibco | 11965-092 | |
| BSA | Sigma | A9418 | |
| Protease-Inhibitor Mix G | Serva | 39101 | |
| Benzonase Nuclease | Novagen | 71206 | 25 U / ul |
| DNase I, RNase-frei | ThermoScientific | EN0523 | 50 U / ul |
| Dounce Homogenisator | Wheaton | 432-1271 | Benutze Typ "B" Pistill |
| Strep-Tactin Sepharose | IBA GmbH | 2-1201-025 | 50% igen Suspension Spalte kann auch dazu verwendet werden |
| Desthiobiotin | IBAGmbH | 2-1000-002 |
Referenzen
- Engelhardt, O. G., Smith, M., Fodor, E. Association of the influenza A virus RNA-dependent RNA polymerase with cellular RNA polymerase II. J. Virol. 79, 5812-5812 (2005).
- Garcia-Robles, I., Akarsu, H., Müller, C. W., Ruigrok, R., Baudin, F. Interaction of influenza virus proteins with nucleosomes. Virology. 332, 329 (2005).
- Chase, G. P., Rameix-Welti, M. A., Zvirbilene, A., Zvirblis, G., Götz, V., Wolff, T., Naffakh, N., Schwemmle, M. Influenza virus ribonucleoprotein complexes gain preferential access to cellular export machinery through chromatin targeting. PLoS Pathogens. 7, e1002187 (2011).
- Kimura, H., Tao, Y., Roeder, R. G., Cook, P. R. Quantitation of RNA polymerase II and its transcription factors in an HeLa cell: little soluble holoenzyme but significant amounts of polymerases attached to the nuclear substructure. Mol. Cell. Biol. 19, 5383-5383 (1999).
- Henikoff, S., Henikoff, J. G., Sakai, A., Loeb, G. B., Ahmad, K. Genome-wide profiling of salt fractions maps physical properties of chromatin. Genome Res. 19, 460 (2009).
- Rodriguez, A., Perez-Gonzalez, A., Nieto, A. Influenza virus infection causes specific degradation of the largest subunit of cellular RNA polymerase II. J. Virol. 81, 5315-5315 (2007).
- Ye, Z., Liu, T., Offringa, D. P., McInnis, J., Levandowski, R. A. Association of influenza virus matrix protein with ribonucleoproteins. J. Virol. 73, 7467-7467 (1999).
- Watanabe, T., Watanabe, S., Kawaoka, T., Y, . Cellular networks involved in the influenza virus life cycle. Cell Host Microbe. 7, 427 (2010).
- Engelhardt, O. G., Fodor, E. Functional association between viral and cellular transcription during influenza virus infection. Rev. Med. Virol. 16, 329-345 (2006).
- Schwartz, L. B., Sklar, V. E., Jaehning, J. A., Weinmann, R., Roeder, R. G. Isolation and partial characterization of the multiple forms of deoxyribonucleic acid-dependent ribonucleic acid polymerase in the mouse myeloma, MOPC 315. J. Biol. Chem. 249, 5889 (1974).
- Lambert, J. P., Baetz, K., Figeys, D. Of proteins and DNA--proteomic role in the field of chromatin research. Mol. Biosyst. 6, 30 (2010).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J. Virol. 85, 6618 (2011).
- Liao, T. L., Wu, C. Y., Su, W. C., Jeng, K. S., Lai, M. M. Ubiquitination and deubiquitination of NP protein regulates influenza A virus RNA replication. EMBO J. 29, 3879 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten