É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Purificação por afinidade de Complexos de Influenza Virus ribonucleoproteína da cromatina de células infectadas
Neste Artigo
Resumo
Os vírus da gripe replicar seu genoma de RNA em associação com a célula hospedeira da cromatina. Aqui, apresentamos um método para purificar intactos os complexos de ribonucleoproteínas virais da cromatina das células infectadas. Purificou complexos virais podem ser analisados por Western blot e tanto de extensão do primer de proteína e do conteúdo de ARN, respectivamente.
Resumo
Como todos os vírus de RNA fita negativa-, o genoma dos vírus da gripe é embalado na forma de complexos de ribonucleoproteína virais (vRNP), em que o genoma de cadeia simples é encapsidado pelo nucleoproteína (NP), e associada com o complexo trimérico polimerase consistindo da PA, PB1, PB2 e subunidades. No entanto, em contraste com a maioria dos vírus de ARN, os vírus influenza realizar a síntese de RNA viral nos núcleos das células infectadas. Curiosamente, a síntese de mRNA virai utiliza celulares pré-mRNAs como iniciadores, e tem sido proposto que este processo tem lugar em cromatina 1. As interacções entre a polimerase virai e do hospedeiro a RNA polimerase II, bem como entre NP e nucleossomas hospedeiras foram também caracterizados 1,2.
Recentemente, a geração do vírus da influenza recombinantes que codificam um One-Strep-Tag geneticamente fundido com o terminal C da subunidade PB2 da polimerase virai (rWSN-PB2-Strep 3), seren descrito. Estes vírus recombinantes permitem a purificação de PB2 contendo complexos, incluindo vRNPs, a partir de células infectadas. Para obter purificado vRNPs, culturas de células são infectadas, e vRNPs são purificados por afinidade a partir de lisados derivados a partir destas células. Contudo, os procedimentos de lise utilizadas até à data têm sido baseados em um-passo de lise de detergente, o qual, apesar da presença de uma nuclease geral, muitas vezes extrair cromatina-bound único material de forma ineficiente.
O nosso trabalho preliminar sugeriu que uma grande porção de vRNPs nucleares não foram extraídos durante a lise das células tradicional, e, portanto, não poderia ser purificado por afinidade. Para aumentar este eficiência de extracção, e para separar cromatina-ligado a partir de não-ligados a cromatina vRNPs nucleares, adaptado um passo a passo do protocolo de extracção subcelular para influenza células infectadas por vírus. Resumidamente, este procedimento primeiro separa os núcleos a partir da célula e, em seguida, extrai solúveis proteínas nucleares (aqui denominado "a fracção nucleoplasmic").O material insolúvel remanescente nuclear é então digerido com Benzonase, um DNA inespecífica / RNA nuclease, seguido de dois passos de extracção de sal: em primeiro lugar utilizando 150 mM de NaCl (denominado "CH150"), em seguida, NaCl 500 mM ("ch500") (fig. 1 ). Estes passos de extracção de sal foram escolhidos com base na nossa observação de que 500 mM de NaCl foi suficiente para solubilizar mais de 85% de vRNPs nucleares e ainda assim permitir a ligação de vRNPs etiquetados para a matriz de afinidade.
Após fraccionamento subcelular das células infectadas, é possível purificar afinidade vRNPs pB2-marcados a partir de cada fracção individual e analisar a sua proteína e os componentes de RNA utilizando Western Blot e extensão do iniciador, respectivamente. Recentemente, utilizou este método para descobrir que vRNP exportação formar complexos durante pontos finais após a infecção sobre a fracção de cromatina extraiu-se com 500 mM de NaCl (ch500) 3.
Protocolo
Um fluxograma esquemático do protocolo é mostrado na fig. 1 e uma tabela de reagentes é apresentada abaixo.
1. Infecção (16 - 24 h)
- Semente para fora células HeLa em 5 de 150 mm pratos de plástico de células de cultura em meio Dulbecco modificado por Eagle (DMEM)-elevado médio de glicose, de modo que eles atingem cerca de 80% de confluência no dia seguinte (aproximadamente 5 x 10 células 8). É importante que as células não chegar a 100% de confluência.
- No dia seguinte, diluir a Strep-etiquetados com estoque de vírus influenza em salino tamponado com fosfato (PBS) contendo 0,3% de albumina sérica bovina (BSA) a uma multiplicidade de infecção de 3.
- Remover o meio de crescimento a partir das células, lavar uma vez com PBS, e adicionar 10 ml de PBS contendo o vírus às células. Incubar durante 1 h à temperatura ambiente.
- Substituir o meio infecção com DMEM de alto glicose, e continuar a incubação a 37 ° C.
2. Fraccionamento subcelular (3 h)
- Lave as células uma vez com PBS frio e, em seguida, suspender as células em PBS frio, com um raspador de borracha e transferência para um tubo cónico de 50 ml.
- As células da pelota em uma centrífuga de bancada durante 5 min a 1000 rpm e, em seguida, aspirar PBS.
- Ressuspender o sedimento de células em 10 ml de tampão de sacarose a frio (10 mM HEPES pH 7,9, 10 mM de KCl, 2 mg de acetato de mM (MgOAc), 3 mM de CaCl2, 340 mM de sacarose, 1 mM de ditiotreitol (DTT), mistura de 1% inibidor de protease) . Após a suspensão, passar as células através de uma ponta de pipeta 200 uL ligado a um 10 ml pipeta de plástico de soro de perturbar aglomerados de células.
- Incubar 10 min em gelo. Inverta suavemente periodicamente para ressuspender as células.
- Adicionar Nonidet P-40 (NP-40) até uma concentração final de 0,5% e vórtice a alta velocidade durante 15 seg. Imediatamente centrifugar durante 10 min a 3.500 xg, a 4 ° C em uma centrifuga de mesage.
- Remover o sobrenadante e armazenar a 4 ° C. Esta é a fracção citoplasmática. O sedimento deve ser menor e mais branco do que a original sedimento celular. Todas as fracções pode ser armazenado durante pelo menos 4 horas a 4 ° C.
- Lavar o sedimento em 5 ml de tampão frio de sacarose, re-centrífuga como no passo 2,5 e descartar o sobrenadante.
- Ressuspender o sedimento (contendo núcleos) em 1,5 ml de tampão de extracção a frio nucleoplasmic (HEPES 50 mM pH 7,9, 150 mM de KOAc, 1,5 mM de MgCl2, 0,1% de NP-40, 1 mM de DTT, mistura inibidor de protease de 1%).
- Transfira para uma gelada, todo em vidro 4 ml Dounce homogeneizador com um pilão apertada e homogeneizar os núcleos com 20 golpes.
Evitar a formação de espuma durante a homogeneização. A resistência deve aumentar depois de aproximadamente 10 pancadas. Homogeneização eficaz pode ser confirmado sob um microscópio de contraste de fase. - Transferir os núcleos homogeneizada para um tubo de microcentrífuga de 1,5 ml e incubar 20 min a uma roda giratória a 4 °C.
- Centrifugar a 16.000 x g numa microcentrífuga a 4 ° C durante 10 min.
- Remover o sobrenadante e armazenar a 4 ° C. Esta é a fracção nucleoplasmic.
- Ressuspender o sedimento em 1,5 ml de tampão de digestão (50 mM de HEPES pH 7,9, 10 mM de NaCl, 1,5 mM de MgCl2, 1 mM de DTT, 1% de mistura inibidor de protease, 100 U / ml Benzonase (para análise de RNA, substituto com 20 U / ml de RNase livre de DNase I)) pré-aquecida a 37 ° C, e incubar 10 min a 37 ° C.
- Adicionar 42 ul de NaCl 5 M (para uma concentração final de 150 mM de NaCl) e incubar 20 min mais a 4 ° C.
- Centrifugar a 16.000 x g numa microcentrífuga a 4 ° C durante 10 min.
- Remover o sobrenadante e armazenar a 4 ° C. Esta é a fracção CH150.
- Ressuspender o sedimento em 1,5 ml de tampão de sal de alta frio (50 mM HEPES pH 7,9, 500 mM de NaCl, 1,5 mM de MgCl2, 0,1% de NP-40, 1 mM de DTT, mistura inibidor de protease de 1%) e incubar 20 min em gelo.
- Centrifugar a 16.000 x g </ Em> numa microcentrífuga a 4 ° C durante 10 min.
- Remover o sobrenadante e armazenar a 4 ° C. Esta é a fracção ch500.
3. Strep-tag Purificação (2 h)
- Adicionar 300 ul de NaCl 5 M para a fracção citoplasmática (para uma concentração final de 150 mM de NaCl).
- Alíquota de 100 uL de concentrado contas Strep-Tactin Sepharose por fração em um tubo cônico de 15 ml.
- Adicionar 10 volumes de Strep tampão de lavagem (20 mM HEPES pH 7,9, 150 mM de NaCl (500 mM de NaCl durante ch500 amostras), EDTA 1 mM) a esferas, inverter o tubo de misturar, e centrifugar 5 min a 1000 x g em um tampo de mesa centrifugar.
- Descartar o sobrenadante, repetir o passo 3,3 duas vezes e, em seguida, ressuspender o sedimento grânulo em um volume igual de tampão de lavagem Strep.
- Adicionar 200 ul de suspensão lavada grânulo Strep-Tactin para cada fracção (em tubos de 1,5 ml de microcentrífuga de nuceloplasmic e CH150, e ch500 fracções e um tubo de 15 ml cónica para fracção citoplasmática).
- Rodartubos 1h a 4 ° C em uma roda rotativa.
- Centrifugar os tubos 1 min a 1000 x G a 4 ° C. Elimine o sobrenadante.
- Adicionar 1 ml de tampão de lavagem a frio Strep a cada tubo. Transferir grânulos a partir de 15 mL de tubo (fracção citoplasmática) a 1,5 mL tubo. Lavar todos os tubos 1 min a 4 ° C, centrifugando depois como na etapa 3,7. Repetir este passo de lavagem duas vezes.
- Após o passo de última lavagem, remover todos os vestígios de tampão de lavagem, utilizando uma pipeta de ponta plana. Adicionar 100 uL de tampão de eluição (tampão de lavagem Strep contendo 2,5 mM desthiobiotin) e misturar suavemente.
- Incubar 15 min em gelo. Misture suavemente contas ocasionalmente tocando no fundo dos tubos.
- Tubos de centrífuga 1 min a 1000 x G a 4 ° C em microcentrífuga. Recolher o sobrenadante contendo eluídas Strep-tag vRNPs.
4. Os resultados representativos
A eficiência do fraccionamento é melhor julgado por análise de Western blot dos lisados fraccionados usando antibodies específicos para marcadores subcelulares (Fig. 2). Especificamente, o fraccionamento subnuclear bem sucedida deveria mostrar pouca ou nenhuma celular RNA polimerase II (Pol II), na nucleoplasmic, ou fracção nuclear solúvel 4, e mais deve ser extraída com 150 mM de NaCl 5. Uma degradação de Pol II é também reprodutivelmente observada em células infectadas por vírus de influenza em comparação com células não infectadas, consistentes com a literatura 6.
A purificação do vRNPs é melhor julgado por prata ou coloração com Coomassie, como mostrado para um eluato citoplasmática na fig. 3. Na nossa experiência, a maioria Strep-PB2 é purificado como parte de um vRNP totalmente formado, isto é, polimerase pouco solúvel é capturado. Isso se reflete na alta NP: PA/PB1/PB2 proporção observada pela coloração de prata ou Coomassie. Uma relação mais baixa sugere a contaminação com tampão de RNases, como vários RNases ubíqua (tais como a RNase A) são conhecidos por RNA clivam virais, de tal forma a dissocicomi a polimerase de NP multímeros 7. Curiosamente, o tratamento com Benzonase sozinho, enquanto suficiente para digerir RNA viral, parece ter nenhum efeito sobre os rácios estequiométricas de proteínas vRNP. Embora não possamos explicar esse fenômeno, observou-se ruptura de vRNPs após digestão com RNases outros (dados não mostrados), sugerindo que Benzonase pode deixar certas regiões estruturalmente importantes RNA intacto. Após purificação do vRNPs a partir do citoplasma e nucleoplasma (realizada sem digestão com nuclease), 8 bandas fracas mas discreta de elevados pesos moleculares pode ser observada por coloração com prata, que correspondem aos pesos moleculares das preditos 8 segmentos do genoma de influenza (ver ref. 3 ).
As quantidades relativas de vRNPs purificadas a partir de cada fracção em 9 h após a infecção são mostrados na fig. 4. A distribuição de vRNPs varia ao longo do curso da infecção, com forte acumulação na fracção CH150 em pontos de tempo iniciais, eacumulação aumentada no nucleoplasma e citoplasma em pontos de tempo tardios.
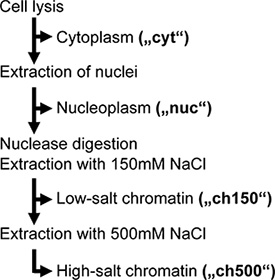
Figura Fluxograma 1. Do fracionamento subcelular. Adaptado da ref. 3.
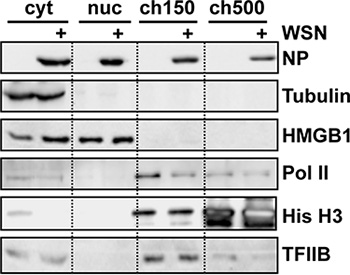
Figura 2. Ocidental análise de mancha de fracções subcelulares de influenza células infectadas por vírus. 10 9 células HeLa foram infectados com influenza WSN estirpe do vírus durante 9 h antes do fraccionamento subcelular. Quantidades iguais de proteína total foram carregados em cada pista e analisados utilizando os anticorpos indicados. RNA polimerase II (Pol II) foi detectada utilizando o clone 8WG16, que reconhece todas as formas do domínio C-terminal (a banda hypophosphorylated é o mais proeminente). Adaptado da ref. 3.

Figura 3.Análise de mancha de prata purificada vRNPs. As células HeLa foram infectadas com rWSN (como um controlo negativo) ou rWSN-PB2-Strep durante 9 horas, seguido de purificação Strep a partir da fracção citoplasmática. Asterisk denota bandas que não são visíveis após a digestão RNase. Adaptado da ref. 3.

Figura 4. Análise de manchas de Prata da vRNPs purificadas de diferentes frações celulares. 10 9 células HeLa foram infectadas com rWSN-PB2-Strep ou rWSN para 9 h, seguido por fraccionamento subcelular e purificação Strep. Quantidades iguais de eluato a partir de cada fracção foram carregados. Adaptado da ref. 3.
Discussão
Enquanto muitos estudos foram recentemente identificadas proteínas individuais ou redes celulares envolvidos na infecção por vírus influenza virus 8, o significado funcional da maioria destas interacções permanece obscura. Dada a dependência absoluta de cromatina funções baseadas para a síntese de RNA vírus influenza e da natureza complexa biofísicas e bioquímicas do núcleo 9, as novas técnicas serão necessários para elucidar estas funções. O fraccionamento subnuclear apresentamo...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores gostariam de agradecer Nada Naffakh e Marie-Anne Rameix-Welti (Institut Pasteur) para o vírus rWSN-PB2-Strep.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| DMEM de alto glicose | Gibco | 11965-092 | |
| BSA | Sigma | A9418 | |
| Inibidor da protease da mistura G | Serva | 39101 | |
| Benzonase Nuclease | Novagen | 71206 | 25 U / uL |
| DNase I, livre de RNase | ThermoScientific | EN0523 | 50 U / uL |
| Dounce homogeneizador | Wheaton | 432-1271 | Use tipo "B" pilão |
| Strep-Tactin Sepharose | IBA GmbH | 2-1201-025 | Formato de coluna de 50% de suspensão também pode ser usado |
| Desthiobiotin | IBAGmbH | 2-1000-002 |
Referências
- Engelhardt, O. G., Smith, M., Fodor, E. Association of the influenza A virus RNA-dependent RNA polymerase with cellular RNA polymerase II. J. Virol. 79, 5812-5812 (2005).
- Garcia-Robles, I., Akarsu, H., Müller, C. W., Ruigrok, R., Baudin, F. Interaction of influenza virus proteins with nucleosomes. Virology. 332, 329 (2005).
- Chase, G. P., Rameix-Welti, M. A., Zvirbilene, A., Zvirblis, G., Götz, V., Wolff, T., Naffakh, N., Schwemmle, M. Influenza virus ribonucleoprotein complexes gain preferential access to cellular export machinery through chromatin targeting. PLoS Pathogens. 7, e1002187 (2011).
- Kimura, H., Tao, Y., Roeder, R. G., Cook, P. R. Quantitation of RNA polymerase II and its transcription factors in an HeLa cell: little soluble holoenzyme but significant amounts of polymerases attached to the nuclear substructure. Mol. Cell. Biol. 19, 5383-5383 (1999).
- Henikoff, S., Henikoff, J. G., Sakai, A., Loeb, G. B., Ahmad, K. Genome-wide profiling of salt fractions maps physical properties of chromatin. Genome Res. 19, 460 (2009).
- Rodriguez, A., Perez-Gonzalez, A., Nieto, A. Influenza virus infection causes specific degradation of the largest subunit of cellular RNA polymerase II. J. Virol. 81, 5315-5315 (2007).
- Ye, Z., Liu, T., Offringa, D. P., McInnis, J., Levandowski, R. A. Association of influenza virus matrix protein with ribonucleoproteins. J. Virol. 73, 7467-7467 (1999).
- Watanabe, T., Watanabe, S., Kawaoka, T., Y, . Cellular networks involved in the influenza virus life cycle. Cell Host Microbe. 7, 427 (2010).
- Engelhardt, O. G., Fodor, E. Functional association between viral and cellular transcription during influenza virus infection. Rev. Med. Virol. 16, 329-345 (2006).
- Schwartz, L. B., Sklar, V. E., Jaehning, J. A., Weinmann, R., Roeder, R. G. Isolation and partial characterization of the multiple forms of deoxyribonucleic acid-dependent ribonucleic acid polymerase in the mouse myeloma, MOPC 315. J. Biol. Chem. 249, 5889 (1974).
- Lambert, J. P., Baetz, K., Figeys, D. Of proteins and DNA--proteomic role in the field of chromatin research. Mol. Biosyst. 6, 30 (2010).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J. Virol. 85, 6618 (2011).
- Liao, T. L., Wu, C. Y., Su, W. C., Jeng, K. S., Lai, M. M. Ubiquitination and deubiquitination of NP protein regulates influenza A virus RNA replication. EMBO J. 29, 3879 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados