Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Affinity очистки вируса гриппа рибонуклеопротеидных комплексов с хроматина инфицированных клеток
В этой статье
Резюме
Вирусы гриппа повторить их РНК-геном в связи с хост-клеточного хроматина. Здесь мы представляем метод, чтобы очистить нетронутыми вирусных комплексов рибонуклеопротеидных из хроматина инфицированных клеток. Очищенный вирусный комплексы могут быть проанализированы и Вестерн-блот и удлинения праймера белка и РНК, соответственно.
Аннотация
Like all negative-strand RNA viruses, the genome of influenza viruses is packaged in the form of viral ribonucleoprotein complexes (vRNP), in which the single-stranded genome is encapsidated by the nucleoprotein (NP), and associated with the trimeric polymerase complex consisting of the PA, PB1, and PB2 subunits. However, in contrast to most RNA viruses, influenza viruses perform viral RNA synthesis in the nuclei of infected cells. Interestingly, viral mRNA synthesis uses cellular pre-mRNAs as primers, and it has been proposed that this process takes place on chromatin1. Interactions between the viral polymerase and the host RNA polymerase II, as well as between NP and host nucleosomes have also been characterized1,2.
Recently, the generation of recombinant influenza viruses encoding a One-Strep-Tag genetically fused to the C-terminus of the PB2 subunit of the viral polymerase (rWSN-PB2-Strep3) has been described. These recombinant viruses allow the purification of PB2-containing complexes, including vRNPs, from infected cells. To obtain purified vRNPs, cell cultures are infected, and vRNPs are affinity purified from lysates derived from these cells. However, the lysis procedures used to date have been based on one-step detergent lysis, which, despite the presence of a general nuclease, often extract chromatin-bound material only inefficiently.
Our preliminary work suggested that a large portion of nuclear vRNPs were not extracted during traditional cell lysis, and therefore could not be affinity purified. To increase this extraction efficiency, and to separate chromatin-bound from non-chromatin-bound nuclear vRNPs, we adapted a step-wise subcellular extraction protocol to influenza virus-infected cells. Briefly, this procedure first separates the nuclei from the cell and then extracts soluble nuclear proteins (here termed the "nucleoplasmic" fraction). The remaining insoluble nuclear material is then digested with Benzonase, an unspecific DNA/RNA nuclease, followed by two salt extraction steps: first using 150 mM NaCl (termed "ch150"), then 500 mM NaCl ("ch500") (Fig. 1). These salt extraction steps were chosen based on our observation that 500 mM NaCl was sufficient to solubilize over 85% of nuclear vRNPs yet still allow binding of tagged vRNPs to the affinity matrix.
After subcellular fractionation of infected cells, it is possible to affinity purify PB2-tagged vRNPs from each individual fraction and analyze their protein and RNA components using Western Blot and primer extension, respectively. Recently, we utilized this method to discover that vRNP export complexes form during late points after infection on the chromatin fraction extracted with 500 mM NaCl (ch500)3.
протокол
Схема схема протокола показан на рис. 1 и таблицы реагентов приведены ниже.
1. Инфекция (16 - 24 ч)
- Семенной из клеток HeLa в 5 150 мм пластиковый культуре клеток блюд в среде Дульбекко изменения Орла (DMEM) высокого среднего глюкозы, например, что они достигнут примерно 80% слияния на следующий день (примерно 5 х 10 8 клеток). Очень важно, что клетки не достигают 100% слияния.
- На следующий день, развести Strep с метками акции вируса гриппа в фосфатно-солевом буфере (PBS), содержащего 0,3% бычьего сывороточного альбумина (БСА) до множества инфекций 3.
- Удалить рост средней из клеток, мыть один раз PBS, и добавить 10 мл PBS, содержащего вируса в клетки. Выдержите в течение 1 часа при комнатной температуре.
- Замените инфекции среды DMEM-высокий уровень глюкозы, и продолжают инкубацию при 37 ° C.
2. Субклеточных фракционирования (3 ч)
- Промойте клетки сразу с холодным ФСБ, а затем приостановить клетки в холодном PBS скребком резиновых и трансфер в 50 мл коническую трубку.
- Гранул клеток в настольной центрифуге в течение 5 мин при 1000 оборотов в минуту, а затем вдохнуть PBS.
- Ресуспендируют осадок клеток в 10 мл холодного буфера сахарозы (10 мМ HEPES рН 7,9, 10 мМ KCl, 2 мМ Mg ацетат (MgOAc), 3 мМ CaCl 2, 340 мМ сахарозы, 1 мМ ДТТ (DTT), 1% смеси ингибиторов протеазы) . После подвески, проходят через клетки в 200 мкл кончике пипетки прикреплен к 10 мл сыворотки пластиковых пипетки нарушить клетки сгустки.
- Выдержите 10 минут на льду. Аккуратно перевернуть периодически ресуспендирования клеток.
- Добавить Nonidet Р-40 (NP-40) до конечной концентрации 0,5% и вихря на высокой скорости в течение 15 сек. Сразу центрифуги в течение 10 мин при 3500 мкг на 4 ° C в настольный центрифугированияGE.
- Удалить супернатант и хранить при температуре 4 ° C. Это цитоплазматической фракции. Осадок должен быть меньше и светлее, чем исходный осадок клеток. Все фракции могут храниться в течение не менее 4 ч при температуре 4 ° C.
- Промойте осадок в 5 мл холодного буфера сахарозы, повторно центрифуги, как в пункте 2.5 и отказаться от супернатант.
- Ресуспендируют гранул (содержащих ядра) в 1,5 мл холодной нуклеоплазме буфера для экстракции (50 мМ HEPES рН 7,9, 150 мМ KOAc, 1,5 мМ MgCl 2, 0,1% NP-40, 1 мМ DTT, 1% смеси ингибиторов протеазы).
- Трансфер в охлажденном, все стекла 4 мл Dounce гомогенизатор с плотно закрывающейся пестик и гомогенизации ядер с 20 ударов.
Избегать образования пены во время гомогенизации. Сопротивление должно увеличиться после того, как около 10 ударов. Эффективность гомогенизации может быть подтверждено под микроскопом фазового контраста. - Передача усредненного ядер 1.5 трубки микроцентрифужных мл и инкубировать 20 минут на вращающееся колесо при 4 °C.
- Центрифуга при 16000 х г в микроцентрифуге при 4 ° С в течение 10 мин.
- Удалить супернатант и хранить при температуре 4 ° C. Это нуклеоплазме фракции.
- Ресуспендируют осадок в 1,5 мл пищеварения буфера (50 мМ HEPES рН 7,9, 10 мМ NaCl, 1,5 мМ MgCl 2, 1 мМ DTT, 1% смеси ингибиторов протеазы 100 ЕД / мл Benzonase (для анализа РНК, замена с 20 Ед / мл РНКазы без ДНКазы I)), предварительно нагревают до 37 ° C, и инкубировать 10 мин при 37 ° C.
- Добавить 42 мкл 5 М NaCl (для конечной концентрации 150 мМ NaCl) и инкубировать 20 мин дальнейшем при 4 ° C.
- Центрифуга при 16000 х г в микроцентрифуге при 4 ° С в течение 10 мин.
- Удалить супернатант и хранить при температуре 4 ° C. Это CH150 фракции.
- Ресуспендируют осадок в 1,5 мл холодной высокого соль буфера (50 мМ HEPES рН 7,9, 500 мМ NaCl, 1,5 мМ MgCl 2, 0,1% NP-40, 1 мМ DTT, 1% смеси ингибиторов протеазы) и инкубировать 20 минут на льду.
- Центрифуга при 16000 х г </ EM> в микроцентрифуге при 4 ° С в течение 10 мин.
- Удалить супернатант и хранить при температуре 4 ° C. Это ch500 фракции.
3. Strep-теги очистки (2 ч)
- Добавить 300 мкл 5 М NaCl в цитоплазматической фракции (для конечной концентрации 150 мМ NaCl).
- Алиготе 100 мкл упакованы Strep-Tactin бисером сефарозе в долю в 15 мл коническую трубку.
- Добавить 10 томов буфер Strep стирки (20 мМ HEPES рН 7,9, 150 мМ NaCl (500 мМ NaCl для ch500 образцов), 1 мМ ЭДТА) для бисера, переверните пробирку перемешать, и центрифуги 5 минут при 1000 х г в настольном центрифуги.
- Удалить супернатант, повторите шаг 3,3 раза, а затем ресуспендируют шарик шарик в равном объеме промывочного буфера Strep.
- Добавить 200 мкл промытый Strep-Tactin суспензии шарик для каждой фракции (в 1,5 мл микроцентрифужных труб для nuceloplasmic, CH150 и ch500 фракций и 15 мл коническую трубку для цитоплазматической фракции).
- ВращатьТрубы 1 час при температуре 4 ° C на вращающемся колесе.
- Пробирок 1 мин при 1000 х г при 4 ° C. Удалить супернатант.
- Добавить 1 мл холодной промывочного буфера Strep в каждую пробирку. Передача бусы из 15 мл трубку (цитоплазматической фракции) до 1,5 мл трубки. Вымойте все трубы 1 мин при 4 ° С, после центрифугирования, как в пункте 3.7. Повторите этот шаг мыть два раза.
- После того, как последний этап стирки, удаление всех следов моющего буфера с помощью плоской кончиком пипетки. Добавить 100 мкл буфера для элюции (Strep промывочного буфера, содержащего 2,5 мМ desthiobiotin) и аккуратно перемешать.
- Выдержите 15 минут на льду. Аккуратно перемешайте бисер иногда, нажав в нижней части трубы.
- Пробирки центрифужные 1 мин при 1000 х г при 4 ° С в микроцентрифужных. Соберите супернатант, содержащий элюированных Strep с метками vRNPs.
4. Представитель Результаты
Эффективность фракционирования лучше всего судить по Вестерн-блот анализе фракционированного лизатов использованием Антибodies специфичные для субклеточных маркеров (рис. 2). В частности, успешный субъядерных фракционирования должен показать мало или нет сотовой РНК-полимеразы II (Пол II) в нуклеоплазме или растворимый ядерной фракции 4, и большинство должны быть извлечены с 150 мМ NaCl, 5. Деградация Pol II также воспроизводимо наблюдается при гриппе инфицированных вирусом клеток по сравнению с неинфицированных клетках, в соответствии с литературными 6.
Очистка vRNPs лучше всего судить по серебра или окрашивание Кумасси, как показано на цитоплазматической элюата на рис. 3. По нашему опыту, наиболее Strep-PB2 очищается в качестве части полностью сформированный vRNP, т.е. мало растворим полимеразы в плен. Это нашло свое отражение в высоких Н.П.: PA/PB1/PB2 соотношение наблюдалось серебра или Кумасси окрашивания. Низкое соотношение предлагает буфер загрязнения РНКазы, а несколько вездесущий РНКазы (например, РНКазы), как известно, расщепляет вирусную РНК таким образом, чтобы диссоциацииели-полимеразы из НП мультимеры 7. Интересно, что лечение с Benzonase в одиночку, а достаточно, чтобы переварить вирусной РНК, по-видимому, не влияет на стехиометрических отношениях vRNP белков. Хотя мы не можем объяснить этот феномен, мы наблюдали нарушение vRNPs после переваривания с другими РНКазы (данные не представлены), предполагая, что Benzonase может оставить определенные структурно важных РНК регионах нетронутыми. После очистки vRNPs из цитоплазмы и нуклеоплазму (выполняется без пищеварение нуклеазы), 8 слабый, но дискретные группы с высоким молекулярным весом можно наблюдать, серебрения, которые соответствуют предсказанным молекулярным весом 8 сегментов генома гриппа (см. ссылку 3. ).
Относительные количества vRNPs очищенный от каждой фракции в 9 ч после инфекции, показаны на рис. 4. Распределение vRNPs меняется в течение инфекции, с сильным накоплением в CH150 долю на ранних моментов времени, иповышенное накопление в нуклеоплазме и цитоплазмы в конце моменты времени.
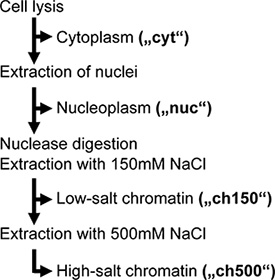
Рисунок 1. Блок-схема субклеточном фракционирования. Адаптировано из работы. 3.
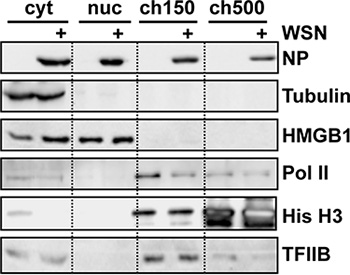
Рисунок 2. Вестерн-блот анализа субклеточных фракций из инфицированных вирусом гриппа клетки. 10 9 HeLa клетки были инфицированы штаммом вируса гриппа WSN за 9 ч до субклеточном фракционирования. Равные количества общего белка были загружены в каждом переулке и проанализированы с помощью антител показано на рисунке. РНК-полимеразы II (Пол II) было обнаружено использование клонов 8WG16, который признает все формы С-концевой домен (hypophosphorylated группы наиболее заметно). Адаптировано из работы. 3.

Рисунок 3.Серебряное пятно анализ очищенной vRNPs. HeLa клетки были инфицированы rWSN (в качестве отрицательного контроля) или rWSN-PB2-Strep за 9 ч, после чего Strep очистки от цитоплазматической фракции. Asterisk обозначает группы, которые не видны после переваривания РНКазы. Адаптировано из работы. 3.

Рисунок 4. Серебряный пятна анализ vRNPs очищается от различных клеточных фракциях. 10 9 HeLa клетки были инфицированы rWSN-PB2-стрептококками или rWSN за 9 ч следует субклеточном фракционирования и Strep очистки. Равное количество элюата от каждой фракции были загружены. Адаптировано из работы. 3.
Обсуждение
Хотя многие исследования недавно выявили отдельные белки или сотовой сети, участвующие в вирус гриппа инфекцию 8, функциональное значение большинства этих взаимодействий остается неясным. Учитывая абсолютную зависимость хроматина на основе функций для вируса гриппа синтез РНК...
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Авторы хотели бы поблагодарить Нада Naffakh и Мари-Анн-Rameix Вельти (Institut Pasteur) в rWSN-PB2-Strep вируса.
Материалы
| Name | Company | Catalog Number | Comments |
| Название реагента | Компания | Номер по каталогу | Комментарии |
| DMEM-высокий уровень глюкозы | Гибко | 11965-092 | |
| BSA | Сигма | A9418 | |
| Ингибитор протеазы Mix G | Serva | 39101 | |
| Benzonase нуклеазы | Novagen | 71206 | 25 U / мкл |
| ДНКазы I, без РНКазы | ThermoScientific | EN0523 | 50 ед / мкл |
| Dounce гомогенизатор | Уитон | 432-1271 | Используйте тип "B" пестик |
| Strep-Tactin сефарозы | IBA GmbH | 2-1201-025 | 50% Формат подвески колонка также может быть использована |
| Desthiobiotin | IBAGmbH | 2-1000-002 |
Ссылки
- Engelhardt, O. G., Smith, M., Fodor, E. Association of the influenza A virus RNA-dependent RNA polymerase with cellular RNA polymerase II. J. Virol. 79, 5812-5812 (2005).
- Garcia-Robles, I., Akarsu, H., Müller, C. W., Ruigrok, R., Baudin, F. Interaction of influenza virus proteins with nucleosomes. Virology. 332, 329 (2005).
- Chase, G. P., Rameix-Welti, M. A., Zvirbilene, A., Zvirblis, G., Götz, V., Wolff, T., Naffakh, N., Schwemmle, M. Influenza virus ribonucleoprotein complexes gain preferential access to cellular export machinery through chromatin targeting. PLoS Pathogens. 7, e1002187 (2011).
- Kimura, H., Tao, Y., Roeder, R. G., Cook, P. R. Quantitation of RNA polymerase II and its transcription factors in an HeLa cell: little soluble holoenzyme but significant amounts of polymerases attached to the nuclear substructure. Mol. Cell. Biol. 19, 5383-5383 (1999).
- Henikoff, S., Henikoff, J. G., Sakai, A., Loeb, G. B., Ahmad, K. Genome-wide profiling of salt fractions maps physical properties of chromatin. Genome Res. 19, 460 (2009).
- Rodriguez, A., Perez-Gonzalez, A., Nieto, A. Influenza virus infection causes specific degradation of the largest subunit of cellular RNA polymerase II. J. Virol. 81, 5315-5315 (2007).
- Ye, Z., Liu, T., Offringa, D. P., McInnis, J., Levandowski, R. A. Association of influenza virus matrix protein with ribonucleoproteins. J. Virol. 73, 7467-7467 (1999).
- Watanabe, T., Watanabe, S., Kawaoka, T., Y, . Cellular networks involved in the influenza virus life cycle. Cell Host Microbe. 7, 427 (2010).
- Engelhardt, O. G., Fodor, E. Functional association between viral and cellular transcription during influenza virus infection. Rev. Med. Virol. 16, 329-345 (2006).
- Schwartz, L. B., Sklar, V. E., Jaehning, J. A., Weinmann, R., Roeder, R. G. Isolation and partial characterization of the multiple forms of deoxyribonucleic acid-dependent ribonucleic acid polymerase in the mouse myeloma, MOPC 315. J. Biol. Chem. 249, 5889 (1974).
- Lambert, J. P., Baetz, K., Figeys, D. Of proteins and DNA--proteomic role in the field of chromatin research. Mol. Biosyst. 6, 30 (2010).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J. Virol. 85, 6618 (2011).
- Liao, T. L., Wu, C. Y., Su, W. C., Jeng, K. S., Lai, M. M. Ubiquitination and deubiquitination of NP protein regulates influenza A virus RNA replication. EMBO J. 29, 3879 (2010).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены