Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La detección de microARNs en la microglía por PCR en tiempo real en el SNC normal y durante la neuroinflamación
En este artículo
Resumen
La microglía se macrófagos residentes que proporcionan la primera línea de defensa y de vigilancia inmune del sistema nervioso central. Los microARN son moléculas reguladoras que juegan un papel importante en muchos procesos fisiológicos, incluida la activación y diferenciación de los macrófagos. En este artículo se describe el método para la medición de los microRNAs en la microglía.
Resumen
Microglia son las células del linaje mieloide que residen en el sistema nervioso central (SNC) 1. Estas células juegan un papel importante en patologías de muchas enfermedades asociadas con neuroinflamación tales como la esclerosis múltiple (MS) 2. Microglia en un SNC normales expresan los macrófagos CD11b marcador y exhiben un fenotipo de descanso por los bajos niveles de expresión de marcadores de activación como CD45. Durante los eventos patológicos en el SNC, la microglia se activan según lo determinado por la regulación positiva de CD45 y otros marcadores 3. Los factores que afectan el fenotipo microglia y funciones en el SNC no se ha estudiado bien. Los microARN (miRNA) son una familia creciente de moléculas conservadas (~ 22 nucleótidos de largo) que están involucradas en muchos procesos fisiológicos normales, como el crecimiento y la diferenciación celular 4 y patologías como la inflamación 5. MiRNAs downregulate la expresión de ciertos genes diana por unión secuencias complementarias de laARNm de ir a jugar un papel importante en la activación de las células inmunes, incluyendo los macrófagos y microglia 6 7. Con el fin de investigar las mediadas por miRNA-las vías que definen el fenotipo microglial función biológica, y para distinguir la microglía de otros tipos de macrófagos, es importante para evaluar cuantitativamente la expresión de microARN en particular en distintos subconjuntos de SNC-residente microglia. Los métodos comunes para medir la expresión de miRNAs en el SNC incluyen la PCR cuantitativa a partir de tejido neuronal conjunto y la hibridación in situ. Sin embargo, la PCR cuantitativa a partir de homogeneizado todo el tejido no permite la evaluación de la expresión de miRNA en la microglía, que representan sólo el 15.5% de las células del tejido neuronal. La hibridación in situ permite la evaluación de la expresión de microARN en tipos celulares específicos en las secciones de tejido, pero este método no es completamente cuantitativo. En este informe se describe un análisis cuantitativo y sensitive método para la detección de miRNA por PCR en tiempo real en la microglía aislada de SNC normales o durante la neuroinflamación con encefalomielitis autoinmune experimental (EAE), un modelo de ratón para la EM. El método descrito será útil para medir el nivel de expresión de microRNAs en la microglía en el SNC normal o durante la neuroinflamación asociada a diversas patologías como esclerosis múltiple, derrame cerebral, lesión traumática, la enfermedad de Alzheimer y tumores cerebrales.
Protocolo
1. El aislamiento de la microglia del SNC normales
Aislamiento de microglía se realiza como se ha descrito anteriormente con modificaciones 3.
- Preparar los instrumentos para la disección y la perfusión: tijeras, pinzas, jeringa de 10 ml con aguja 27G, 15 ml homogeneizador Dounce (teflón / vidrio), 40 micras de nylon filtro de células, el 40% y 70% de Percoll soluciones, 1X PBS.
- La eutanasia de los ratones por asfixia rápida (privación de oxígeno) en la cámara de CO 2 y de inmediato trasladarlos a mesa de laboratorio para la manipulación quirúrgica y la perfusión.
- Realizar manipulaciones quirúrgicas (unas tijeras y pinzas y el 70% de etanol como un antiséptico): cavidad peritoneal abierta, corte el diafragma, la cavidad abierta de tórax y cortar la aurícula derecha con unas tijeras.
- Realizar todo el cuerpo de perfusión intracardiaca mediante la inyección de 10 ml de la jeringa en el ventrículo izquierdo y la aplicación de presión. Pase 8-10 ml de 1X PBS a través de la circulación de la sangre en pacientes lenta y constantece.
- Repetir los pasos 1.2-1.4 para todos los ratones del grupo de ratones (típicamente 5-10). Después de la perfusión del ratón mantener en hielo hasta que todos los ratones son perfundidos.
- Después de la perfusión de todos los ratones, diseccionar el cerebro y la médula espinal. (Opcional: para estudiar la expresión de miRNAs en diferentes áreas del cerebro, la disección de estas áreas específicas, tales como el cerebelo, hipocampo, etc para el grupo de ratones de 5-10 y la piscina juntos para la homogeneización).
- Homogeneizar el cerebro y la médula espinal utilizando 15 ml Dounce homogeneizadora. Coloque un cerebro y la médula espinal en un homogeneizador y añadir 5 ml de PBS 1X. Realice 10-20 huelgas suaves; pasar homogeneizado a través de filtro de células en 50 ml-cónica del tubo y el certificado a 2000 rpm en una centrífuga de 5-7 minutos grandes.
- Desechar cerebro sobrenadante, resuspender / homogeneizado médula espinal de ratones en 2-3 ml de 5-7 de 70% de Percoll y superposición 7 ml de 40% de Percoll. Girar a 2000 rpm (sin freno!) Durante 30 minutos.
- Eliminar las células dañadas restos o la mielina en la cima de un 70% y recoger monolas células nucleares en / 40% al 70% de interfaz. Diluir las células recogidas por lo menos en una proporción 1:1 con PBS 1X y centrifugado a 2.000 rpm durante 5-7 minutos. Resuspender las células en 0,5 ml de PBS y transferirlas a 1,7 ml tubo Ependorf. Rendimiento típico es ~ 500 x 10 3 de las células mononucleares por ratón, lo que daría el número de células microgliales ~ 1x10 6 en 0,5 ml para la preparación de las células de cerebro y médula espinal de 2-3 ratones.
2. Control de la pureza de la microglía por citometría de flujo
La tinción con anticuerpos se realiza como se ha descrito anteriormente con modificaciones 7,8.
- Preparar tampón FACS (1X PBS con FBS al 2%), los anticuerpos que bloquean FcR, anti-CD11b - PE, anti-CD45-APC-Cy7 anticuerpos, 1% de paraformaldehído en PBS.
- Tomar 50 l de las células de paso 1,9 y transferirlos al nuevo tubo Eppendorf 1,7 ml y añadir 350 l de tampón FACS.
- Añadir l 5-10 de anti-Fc de anticuerpos e incubar 10-15minutos en hielo.
- Dividir celdas en dos tubos Eppendorf. Añadir a un tubo de 1 l de anti-CD11b - PE y 2 anticuerpos anti-CD45 l-APC-Cy7 e incubar 10-15 minutos en hielo. Segundo tubo se utiliza como un control negativo para la tinción de anticuerpos. Después de la incubación con los anticuerpos añadir 1 ml de tampón FACS a ambos tubos y el giro en la pequeña centrífuga a 5400 rpm durante 5 min. Después de girar fijar las células mediante resuspensión en 400 l de 1% de paraformaldehído en PBS y agitar.
- Realizar un análisis de citometría de flujo en el citómetro de flujo (LSR II, BD Biosciences) utilizando BD compubeads (BD Biosciences) para los controles de compensación como se ha descrito 8. Ejemplos de preparaciones buenas y malas se muestran en la figura. 1a, b.
3. El aislamiento de la microglía y los macrófagos periféricos del sistema nervioso central inflamadas en modelo de ratón de la neuroinflamación
Encefalitis autoinmune experimental (EAE) se induce como se describió anteriormenteen nuestro artículo 7; FACS clasificación se realiza de manera similar a protocolo publicado 8.
- Inducir EAE mediante la inmunización subcutánea con 150 mg MOG 35-55 péptido de 4 mg / ml de adyuvante completo de Freund (CFA) del grupo de 5-10 de 8 a 12 semanas de edad ratones C57BL / 6. La toxina pertussis se debe dar ip (150 ng / ratón) en los días cero y el segundo día, después de la inmunización. Los ratones se observaron signos de enfermedad a partir del día 10 después de la inmunización, y la gravedad de la enfermedad se califica en una escala numérica de 0 a 5 de la siguiente manera: 0) sin la enfermedad: 1) la cola a pie débil o inestable; 2) paresia extremidades posteriores; 3) parálisis de las extremidades traseras, 4) la parálisis posteriores y anteriores, y 5) la muerte o la eutanasia por razones humanitarias. Los ratones en el pico de la enfermedad (D21 después de la inmunización) se utilizan para experimentar con la puntuación de las enfermedades típicas que van desde 1,5 a 2,5.
- Aislar las células mononucleares de SNC de grupo de 5-10 ratones como en los pasos 1.1-1.4 y teñir las células con anticuerpos anti-CD11b y anti CD45-como en los pasos 2.1-2.4.En el paso 2.4 no solucionan las células, en lugar de volver a suspender en 400 l de PBS 1X en vez de 1% de paraformaldehído.
- La clasificación de células se realiza mediante FACSAria para las poblaciones de CD11b + CD45 baja microglia y las células CD45 + CD11b hi (~ 80% de ellos son los macrófagos periféricos y ~ 20% de ellos se activan microglia 3; en el futuro, nos referiremos a estas células como " macrófagos "). Utilizar 1,7 ml Ependorf tubos para la recogida de las muestras. 300 l de 1X PBS con FBS al 10%, debe añadirse a los tubos de recogida para evitar daños de las células clasificadas. Ejemplos de tres poblaciones de microglía, macrófagos y linfocitos se muestra en la figura. 2 bis, con la configuración correcta clasificación de compuerta se muestra en la figura. 2b. Purezas típicos de clasificación fueron en 98-100%, determinado por reanalizar poblaciones ordenados de microglia (Fig. 2c) y macrófagos (Fig. 2d). Rendimiento normal es 20 a 50 x 10 3 de la microglía y50-100 x 10 3 para los macrófagos aislados del grupo de cinco ratones.
4. Aislamiento de ARN
ARN se aisló utilizando el kit mirVana de acuerdo con las instrucciones del fabricante con modificaciones.
- Derivado de la microglia del SNC normales de paso 1.9 o subconjuntos ordenados de la microglía y los macrófagos de paso 3,3 en 1,7 ml tubos Eppendorf a 5400 rpm durante 5-7 minutos en centrífuga pequeña, cuidadosamente eliminar el sobrenadante y congelar los gránulos de las células en hielo seco. Proceder al paso 4,2 o transferir pastillas congeladas de células a -70 C, donde se puede almacenar durante 2-3 meses.
- Traslado gránulos congelados de células en hielo ordinario para la descongelación. Añadir 300 l de tampón de lisis del kit mirVana y mezcle suavemente por pipetear 5-7 veces.
- Añadir 30 l de aditivo homogeneizado de mirVana kit, vórtice de 30 segundos para mezclar, mantener en hielo durante 10 minutos
- Añadir 300 l de ácido Pehnol: cloroformo de mirVana kit, vórtice de 30 segundos, giro de 5 men a 10.000 rpm en centrífuga pequeña.
- Con cuidado, quite fase superior acuosa (300 l) y transferir a otro tubo Eppendorf 1,7 ml, mezclar con 375 l de etanol al 100%, traslado al cartucho del filtro de mirVana kit y vuelta durante 1 min a 10.000 rpm.
- Desechar flujo a través de tampón, añadir 700 l de solución de lavado I desde mirVana kit, y centrifugado durante 1 min a 10.000 rpm.
- Lavar dos veces más con 500 l de solución de lavado II a partir de mirVana kit y finalmente eluir ARN de cartucho con 60 l de pre-calentado agua libre de nucleasa.
- La cantidad y la calidad de ARN se evaluó mediante espectrofotómetro Nanodrop. La concentración de ARN debe ser de 5 a 20 ng / ml y OD λ260 / λ280 proporción dentro del intervalo 1.7-2.0. Si la concentración de ARN es inferior a 3 ng / ml y 260λ/280λ OD es menor que 1,6, las muestras se descartan. Muestras de ARN se puede almacenar a -70 ° C durante 6-12 meses.
- Para la estimación de más de la calidad del ARN, el ar muestrase analizaron mediante electroforesis en gel en TBE-gel de urea 15% (Life Technologies). Ejemplos típicos de calidad buena y mala de preparaciones de ARN se muestran en la figura. 3a y Fig.3b, respectivamente.
5. La detección de miRNA en macrófagos y microglia
Para el análisis de la expresión de miRNA, cuantitativa en tiempo real PCR transcriptasa inversa (QRT-PCR), los análisis se lleva a cabo utilizando los ensayos TaqMan con cebadores y sondas fluorescentes para microARN particular de interés y uno de los RNAs expresó doquier cortos (por ejemplo snoRNA-55, snoRNA -135 o U6) para la normalización. Los cebadores y sondas fluorescentes para conjuntos de miARN humanos o de ratón se pueden comprar de Life Technologies Inc. Aquí vamos a utilizar el miR-124 como un ejemplo de miRNA de interés y snoRNA 55-como un ejemplo para la normalización.
- Realice reacción de la transcriptasa inversa para hacer cDNA a partir de muestras de ARN utilizando el kit TaqMan transcripción reversa:
| Artículo | Volumen (l) por muestra |
| 5x Primer RT | 1,00 |
| ARN de la muestra | 1,67 |
| RT enzima Mix | |
| 25 mM de cada dNTPs (100 mm total) | 0,050 |
| MultiScribe la transcriptasa inversa) | 0,333 |
| 10X tampón de RT | 0,500 |
| AB Inhibidor de la RNasa | 0,063 |
| Agua libre de nucleasa | 1,388 |
| Total | 5,00 |
Diluir ARN muestras de concentración de 3 ng / l. Prepare la mezcla de la enzima RT y poner a los tubos de 3,33 l de PCR y añadir 1,67 l de muestra de RNA. Incubar en la PCR Instrumento (BioRad) a 16 ° Cdurante 30 min, 42 ° C durante 30 min, 85 ° C durante 5 min, y establece a 4 ° C en espera.
- Realizar en tiempo real de reacción de PCR utilizando TaqMan mezcla de PCR:
| Artículo | Volumen (l) por muestra |
| RT producto | 1.0 |
| 2X TaqMan Master Mix | 5.0 |
| Sonda de 5X | 2.0 |
| Agua libre de nucleasa | 2.0 |
| Total | 10,0 |
Use un plato óptico de reacción de 384 bien claro (Life Technologies) para cada reacción y la PCR en tiempo real instrumento de AB 7900 HT. Ciclismo condiciones: 95 ° C durante 10 min, [95 ° C durante 5 segundos, 60 ° C durante 60 seg] x 40 ciclos.
- Curvas típicas de la cantidad de fluorescently etiquetados específicos productos de la PCR (Y-ejes) para snoRNA-55 y miR-124-para las muestras de ARN (cada muestra es por duplicado) aisladas a partir de la microglía y los macrófagos derivados de médula ósea (BMDMs) frente al número de ciclos (eje X) se se muestra en la fig. 4a, b.
6. Normalización y Análisis de Datos
Los niveles relativos de expresión de miRNA de interés (MIR-124) se calculan utilizando el método ΔΔCT como se describió anteriormente con 7,9 snoRNA-55 para la normalización de la cantidad inicial de ARN.
- El ejemplo de cálculo del nivel relativo de miR-124 para la expresión de la microglía y BMDMs se muestra en la Tabla I.
- Como primer paso, los valores de Ct snoRNA-55 y miR-124 se determinan para la microglía y BMDMs de QRT-PCR curvas (Fig. 4, Tabla I, columnas: snoRNA-55 y miR-124).
- Como un segundo paso, los valores Ctse calculan para cada duplicado de cada muestra por sustracción de la TC para snoRNA-55 (normalización) de CT para miR-124 (experimento) (Tabla I, columnas: Ct (Norma), CT (Exp) y Ct).
- Como un tercer paso. Valores ΔΔCt se calcula por sustracción de Ct de la muestra de referencia (Ct (Ref.)) a partir de valores Ct de otras muestras (Tabla I, columna: ΔΔCt). Una muestra se define como una referencia; el nivel relativo de expresión miARN para esta muestra se define como 1,0. Todas las otras muestras se compara con esta muestra de referencia. En nuestro caso, se define el nivel de miR-124 en la primera muestra de la microglia como 1.0, por lo tanto, Ct (Ref) = 0,49 (Tabla I, la primera fila, marcado en rojo).
- Por último, el nivel relativo de expresión se calcula como el 2-ΔΔCt (Tabla I, columna: miR-124 eXpression).
- Todos QRT-PCR se realizaron por triplicado o en los duplicados, y el nivel relativo de expresión se presentan como media ± desviación estándar (SD) para triplicados (Fig. 5a, b) o como ambos lados duplicados por lado como se muestra en la fig. 5c.
7. Los resultados representativos
Curvas típicas para PCR en tiempo real y valores CT para MIR-124 (microARN de interés) y snoRNA 55-(normalización), así como los niveles relativos de expresión de miR-124 se muestran en la figura. 4 y la Tabla I. En nuestros experimentos hemos utilizado snoRNA-55 como un control para la normalización. Como hemos mencionado anteriormente, otros ARNs ubicuas también se puede utilizar para la normalización como snoRNA-135 y U6.
Los ejemplos de la expresión de miR-124 en adultos microglía vs médula ósea macrófagos derivados (BMDMs) se muestran en la figura. 5a (BMDMs se cultivaron en la presence de M-CSF como se describe 7). En la fig. 5b, se comparó la expresión de miR-124 en las poblaciones clasificadas de baja CD45 microglia vs CD45 macrófagos hi. En ambos de estos experimentos hemos demostrado que la microglía son diferentes de otros macrófagos expresando niveles más altos de MIR-124. Experimentos similares se pueden realizar en el futuro para la cinética de activación microglia en vivo o in vitro.
La expresión de miR-124 en la microglía aislada de SNC de ratones C57BL / 6 en las diferentes etapas de desarrollo se muestra en la figura. 5c. Estos datos demuestran que la microglia en las primeras etapas de desarrollo expresan niveles más bajos de miR-124, que se correlaciona con su fenotipo activado 7. Un análisis similar puede realizarse para estudiar las funciones de la microglía en el SNC normales tales como la comparación de la expresión de miRNAs en particular en la microglia aisladas de diferentes áreas del sistema nervioso central: el cerebelo, scable de Pinal, etc hipocampo
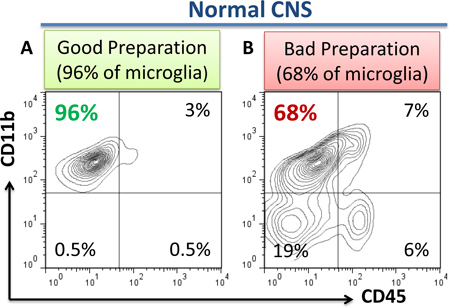
Figura 1. Los ejemplos de preparaciones de buenos y malos de la microglía según lo determinado por citometría de flujo. En el caso de "bueno" el aislamiento de la microglia del SNC normales, el 95-98% de las células CD45 + CD11b representan microgla bajo con 5.2% de CD11b + CD45 hi perivasculares macrófagos. Menos del 1% de CD11b - CD45 linfocitos hi o CD11b - CD45-células astrogliales o oligodendroglial o fragmentos de células deben estar presentes (uno). El caso de "mala" la preparación se muestra aquí (b) cuando el 18% de la contaminación de CD11b - CD45 - las células está presente (b, en el cuadrante inferior izquierdo) lo que sugiere una separación insuficiente gradiente de Percoll. En el caso de una buena preparación, 95-98% de las células debe representar CD11b + CD45bajo microglía (una, cuadrante superior izquierdo), 2-5% de todas las células deben representar CD11b + CD45 macrófagos perivasculares (un hi, cuadrante superior derecho), y menos del 1% de todas las células deben representar CD11b células negativas contaminantes (a, cuadrante inferior izquierdo). En el caso de "mala preparación", una contaminación significativa de CD11b - CD45 - células o fragmentos de células (astroglía, las partículas de la mielina, etc) están presentes (b, en el cuadrante inferior izquierdo), lo que hace que la preparación de las células no aptos para el aislamiento de ARN y más análisis. Además, CD11b - hi células CD45 podría ser también presente en el caso de "mala preparación" (b, en el cuadrante inferior derecho) que indica la contaminación por los linfocitos de la sangre debido a una perfusión insuficiente.
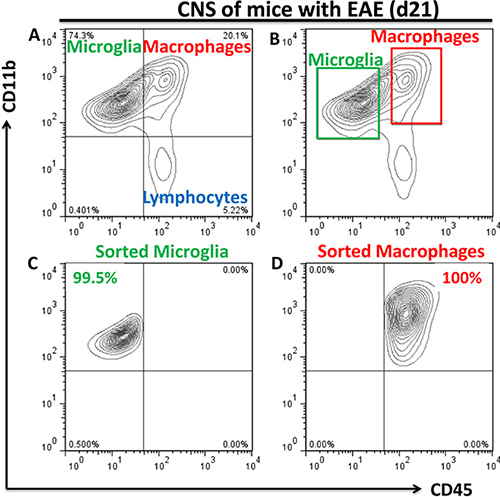
Figura 2. Citometría de flujo analysis y la clasificación de las poblaciones de la microglía y los macrófagos periféricos del sistema nervioso central enfermos (a) Durante la neuroinflamación tres poblaciones de células están presentes en el SNC: CD11b + CD45 baja (microglia), CD11b + CD45 hi (macrófagos), y CD11b - CD45. hi (linfocitos), como se muestra en la parte superior derecha izquierda, superior e inferior cuadrantes derecho, respectivamente. Gates para ordenar las poblaciones de CD11b + microglía CD45 baja y CD11b + CD45 macrófagos hi se muestra en (b) y las purezas de las poblaciones reanalizaron ordenados se muestran en (c, d). Gating preliminar sobre células viables sobre la base de FSC / SSC parámetros se llevó a cabo.
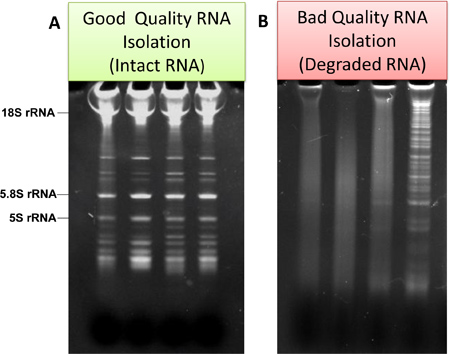
Figura 3. Ejemplos de qualit "bueno" y "malo"IES de ARN aislado de microglía y se analizaron por electroforesis en gel. En el caso de buena calidad, escalera de ARN y el ARN ribosomal es evidente (a). En el caso de mala calidad, muestra, y los productos de bajo peso molecular de degradación son evidentes (b).
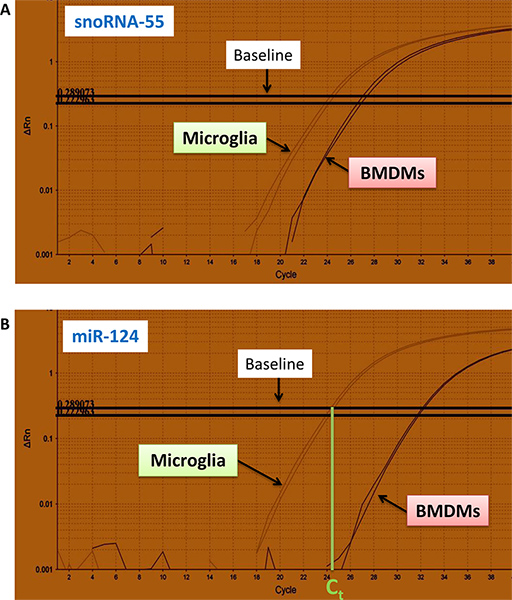
Figura 4. En tiempo real QRT-PCR marcado con fluorescencia para las curvas de miR-124 y snoRNA-55 productos de la PCR. Curvas de snoRNA-55 (a) y miR-124 (b) se muestran de la microglía y los macrófagos de la médula ósea derivados (BMDMs). El experimento se realizó por duplicado. Los valores de Ct se determina por la intersección de la línea de base con la parte inferior del rango lineal de QRT-PCR se curva como se muestra en (b). Haga clic aquí para ver más grande la figura .
| snoRNA-55 | MIR-124 | DCT = Ct (Exp.)-Ct (Norma) | DDCT = DCT-DCT (Ref) | Nivel relativo de expresión = 2-DDCT | |
| Ct (Norma) | Ct (Exp.) | DCT | DDCT | MIR-124 Expresión | |
| La microglía del SNC normales | 24.082 | 24.572 | 0,49 (Ref) | 0 | 1.0 |
| 23.766 | 24.291 | 0,525 | 0,035 | 1,02 | |
| De médula ósea (macrófagos derivados BMDMs) | 26.879 | 320.231 | 5,352 | 5,317 | 0,025 |
| 26.526 | 32.078 | 5,552 | 5,517 | 0,022 |
Tabla I. Cálculo de los niveles relativos de expresión de miR-124 en la microglía frente a los macrófagos derivados de médula ósea con base de los valores de C t para el miR-124 (micro ARN de interés) y snoRNA 55 (de control para la normalización).
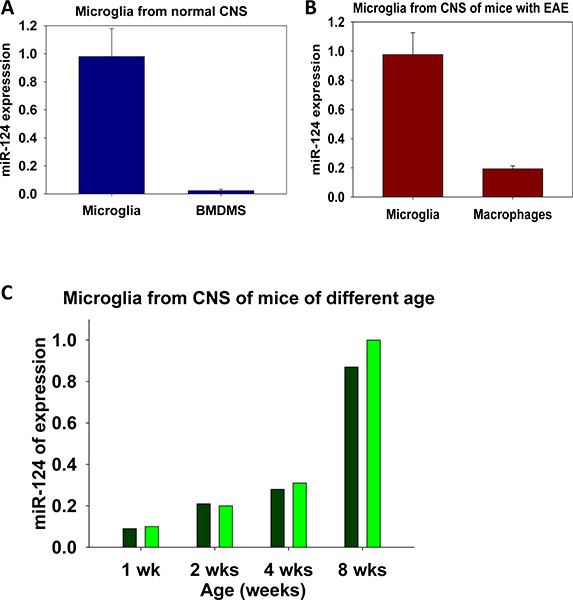
Figura 5. Análisis de miR-124 expresión en macrófagos y microglia. La comparación de la expresión de miR-124 en la microglía del SNC normales frente a los macrófagos derivados de médula ósea (BMDMs) se muestra en (a). Comparación de MIR-124 en la microglía vs macrófagos periféricos aisladas de SNC de en ratones con neuroinflamación se muestra en (b). Los cambios en elnivel de expresión de miR-124 en la microglía durante el desarrollo en ratones de 1, 2 y 4 semanas de edad, en comparación con ratones adultos (a las 8 semanas de edad) se muestra en (c). En (a, b) el experimento se realizó por triplicado con una media ± DE de tres reacciones PCR separadas se muestran. En (c) experimento se realizó en los duplicados como se indica.
Discusión
Recientemente un gran interés fue dado a la microglía y la participación de estas células en muchas patologías asociadas a la neuroinflamación y la neurodegeneración. Además, el papel de los microRNAs en la diferenciación de las células del sistema inmune y el cáncer ha crecido dramáticamente durante los últimos varios años 5,10. Hemos informado anteriormente el papel de miR-124 en el mantenimiento del fenotipo no activado o en reposo de la microglía en el SNC normal 7, pero el papel...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
El trabajo fue apoyado por el NIH R01-01A1 NS071039 beca de investigación.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| 1X PBS | GIBCO | 14190 | Puede ser preparado a partir de componentes secos en el laboratorio, pH: 7.2-7.4 |
| PBS 10 veces | Thermo Scientific | SH30258.02 | Puede ser preparado a partir de componentes secos en el laboratorio, pH: 7.2-7.4 |
| DMEM; FBS | GBCO | 11965, 10438 | |
| Mirvana kit | Invitrogen | AM1561 | |
| Percoll | Sigma | P4937-500 ml | 100 ml de 100% de Percoll se prepara mezclando 10 ml de 10X PBS y 90 ml de Percoll de Sigma. 50 ml de 70% de Percoll espreparado por mezcla de 35 ml de 100% de Percoll y 15 ml de DMEM 50 ml de 40% de Percoll se prepara mezclando 20 ml de 100% de Percoll y 30 ml de 1X PBS |
| MOG 35-55 péptido | AnaSpec Inc | 60130-5 | |
| Comité de Libertad Sindical | DIFCO | 263810 | |
| Toxina pertussis | Sigma | P7208 | |
| FcR anticuerpos bloqueantes | BD Biosciences | 553142 | Clon 2.4G2 |
| Anti-CD11b - PE MAB | BD Biosciences | 553311 | Puede ser utilizado con diferentes fluoróforos (por ejemplo, AF488) |
| Anti-CD45-APC-Cy7 mAbs | BioLegend | 103116 | Puede ser utilizado con diferentes fluoróforos (por ejemplo APC) |
| Paraformalformaldehído (16%) | EMS Inc. | 15710 | Diluir 1:15 en PBS 1X |
| 15% de TBE-gel de urea | Life Technologies Inc. | EC68855BOX | |
| Micro RNA TaqMan transcripción inversa Kit | Life Technologies Inc | 4366596 | |
| TaqMan Universal PCR Master Mix | Life Technologies Inc | 4304437 | |
| Los ensayos TaqMan mmu microARN miR-124a- | Life Technologies Inc | 4427975 ID 001182 | |
| Los ensayos TaqMan microARN snoRNA-55 | Life Technologies Inc | 4427975 ID 001228 | |
| Agua libre de nucleasa | Life Technologies Inc | AM9937 | Agua libre de nucleasa |
| 384 y placa de reacción clara óptica | Life Technologies Inc. | 4309849 | Puede ser sustituido con otras placas (por ejemplo, placa de 96 pocillos) en diferentes instrumentos en tiempo real de PCR |
| Homogeneizador Dounce (15 ml) | Wheaton Science Products. | 358044 | Teflón / vidrio |
| 40 μ de células colador de nylon | Halcón | 352340 | |
| Centrífuga grande | Sorval | RT 6000B | Diámetro del rotor 18,5 cm |
| Centrífuga pequeña | Eppendorf | 5415 R | Diámetro del rotor 6,5 cm |
| PCR en tiempo real del instrumento | Life Technologies Inc. | AB 7900 HT | Puede ser sustituido con otros instrumentos |
| Citómetro de flujo | BD Biosciencess | LSR II | Puede ser sustituido con otros instrumentos |
| FACS | BD Biosciences | FACSAria | Puede ser sustituido con otros instrumentos |
| Espectrofotómetro Nanodrop | Thermo Scientific | Nanodrop 1000 |
Referencias
- Tremblay, M. E. The Role of Microglia in the Healthy Brain. J. Neurosci. 31, 16064-16069 (2011).
- Graeber, M. B., Li, W., Rodriguez, M. L. Role of microglia in CNS inflammation. FEBS Lett. , (2011).
- Ponomarev, E. D., Shriver, L. P., Maresz, K., Dittel, B. N. Microglial cell activation and proliferation precedes the onset of CNS autoimmunity. J. Neurosci. Res. 81, 374-389 (2005).
- Perruisseau-Carrier, C., Jurga, M., Forraz, N., McGuckin, C. P. miRNAs stem cell reprogramming for neuronal induction and differentiation. Mol. Neurobiol. 43, 215-227 (2011).
- McCoy, C. E. The role of miRNAs in cytokine signaling. Front Biosci. 17, 2161-2171 (2010).
- He, M., Xu, Z., Ding, T., Kuang, D. M., Zheng, L. MicroRNA-155 regulates inflammatory cytokine production in tumor-associated macrophages via targeting C/EBPbeta. Cell Mol. Immunol. 6, 343-352 (2009).
- Ponomarev, E. D., Veremeyko, T., Barteneva, N., Krichevsky, A. M., Weiner, H. L. MicroRNA-124 promotes microglia quiescence and suppresses EAE by deactivating macrophages via the C/EBP-alpha-PU.1 pathway. Nat. Med. 17, 64-70 (2011).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). J. Vis. Exp. (41), e1546-3791 (2010).
- Gabriely, G. MicroRNA 21 promotes glioma invasion by targeting matrix metalloproteinase regulators. Mol. Cell Biol. 28, 5369-5380 (2008).
- Caffarelli, E., Filetici, P. Epigenetic regulation in cancer development. Front Biosci. 17, 2682-2694 (2011).
- Cardona, A. E., Huang, D., Sasse, M. E., Ransohoff, R. M. Isolation of murine microglial cells for RNA analysis or flow cytometry. Nat. Protoc. 1, 1947-1951 (2006).
- Gordon, R. A simple magnetic separation method for high-yield isolation of pure primary microglia. J. Neurosci. Methods. 194, 287-296 (2011).
- Moltzahn, F., Hunkapiller, N., Mir, A. A., Imbar, T., Blelloch, R. High Throughput MicroRNA Profiling: Optimized Multiplex qRT-PCR at Nanoliter Scale on the Fluidigm Dynamic ArrayTM IFCs. J. Vis. Exp. (54), e2552 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados