Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection des microARN dans la microglie par PCR en temps réel dans le SNC normal et pendant neuroinflammation
Dans cet article
Résumé
Les microglies sont les macrophages résidents qui fournissent la première ligne de défense et de la surveillance immunitaire du système nerveux central. Les microARN sont des molécules régulatrices qui jouent un rôle important dans de nombreux processus physiologiques, y compris l'activation et la différenciation des macrophages. Dans cet article, nous décrivons la méthode de mesure des microARN dans la microglie.
Résumé
Microglie sont des cellules de la lignée myéloïde qui résident dans le système nerveux central (SNC) 1. Ces cellules jouent un rôle important dans les pathologies de nombreuses maladies associées à la neuroinflammation tels que la sclérose en plaques (MS) 2. La microglie dans quelques SNC normal exprimer macrophages marqueur CD11b et présentent un phénotype de repos en exprimant de faibles niveaux de marqueurs d'activation tels que CD45. Lors d'événements pathologiques dans le système nerveux central, la microglie sont activés tel que déterminé par la régulation positive de CD45 et d'autres marqueurs 3. Les facteurs qui influent sur le phénotype des microglies et les fonctions dans le SNC ne sont pas bien étudiés. Les microARN (miARN) sont une famille de plus en plus de molécules conservées (~ 22 nucléotides de long) qui sont impliqués dans de nombreux processus physiologiques normaux tels que la croissance et la différenciation cellulaire 4 et pathologies telles que l'inflammation 5. MiARN régulent à la baisse l'expression de certains gènes cibles en se liant des séquences complémentaires de laARNm ir et de jouer un rôle important dans l'activation des cellules immunitaires innées dont les macrophages et la microglie 6 7. Afin d'étudier les voies médiées par miRNA qui définissent le phénotype des microglies, la fonction biologique, et de distinguer les microglies d'autres types de macrophages, il est important d'évaluer quantitativement l'expression de microARN spécifiques dans des sous-ensembles distincts de SNC-résident microglie. Les méthodes courantes pour mesurer l'expression des miARN dans le SNC comprennent la PCR quantitative à partir de tissu neuronal et de toute hybridation in situ. Cependant, la PCR quantitative à partir de broyat de tissu ensemble ne permet pas l'évaluation de l'expression de miARN dans la microglie, qui ne représentent que 5-15% des cellules de tissu neuronal. Hybridation in situ permet l'évaluation de l'expression de microARN dans des types cellulaires spécifiques dans les coupes de tissus, mais cette méthode n'est pas entièrement quantitative. Dans ce rapport, nous décrivons une amélioration quantitative et sensitive méthode pour la détection de miARN par PCR en temps réel dans la microglie isolés de SNC normal ou pendant l'utilisation neuroinflammation encéphalomyélite allergique expérimentale (EAE), un modèle de souris pour MS. La méthode décrite sera utile pour mesurer le niveau d'expression des microARN dans la microglie dans le SNC normal ou en neuroinflammation associée à diverses pathologies, y compris MS, accident vasculaire cérébral, un traumatisme, la maladie d'Alzheimer et les tumeurs cérébrales.
Protocole
1. Isolement des cellules microgliales du SNC normal
Isolement de la microglie est effectué comme cela a été décrit précédemment, avec les adaptations 3.
- Préparer les instruments de dissection et de la perfusion: ciseaux, pinces, une seringue de 10 ml avec aiguille 27g, 15 ml Dounce homogénéisateur (téflon / verre), 40 tamis cellulaire um nylon, 40% et 70% des solutions de Percoll, PBS 1X.
- Euthanasier les souris par asphyxie rapide (privation d'oxygène) dans le CO 2 la caméra et de les transférer immédiatement sur le banc de laboratoire pour les manipulations chirurgicales et de perfusion.
- Effectuer des manipulations chirurgicales (utiliser des ciseaux et des pinces et éthanol à 70% comme antiseptique): ouvert la cavité péritonéale, couper la membrane, de la cavité thoracique ouverte et coupez l'oreillette droite avec des ciseaux.
- Effectuer l'ensemble du corps perfusion intracardiaque en injectant 10 ml de la seringue dans le ventricule gauche et en appliquant une pression. Passez 8-10 ml de PBS 1X à travers la circulation sanguine au pa lente et régulièreCE.
- Répétez les étapes 1.2 à 1.4 pour toutes les souris du groupe (souris généralement 5-10). Après perfusion garder la souris sur la glace jusqu'à ce que toutes les souris sont perfusées.
- Après la perfusion de toutes les souris, disséquer le cerveau et la moelle épinière. (Facultatif: pour étudier l'expression des miARN dans les zones différentes du cerveau, disséquer ces domaines spécifiques tels que le cervelet, etc hippocampe pour le groupe de souris 5-10, et les réunir pour l'homogénéisation).
- Homogénéiser le cerveau et la moelle épinière à l'aide de 15 ml Dounce homogénéisateur. Placer un cerveau et la moelle épinière dans une homogénéisateur et ajouter 5 ml de PBS 1X. Effectuer 10-20 grèves douces; passer au tamis homogénat cellulaire dans 50 ml-conique du tube et d'un certificat à 2000 rpm sur quelques minutes de centrifugeuses grandes 5-7.
- Rejeter le surnageant, le cerveau resuspendre / homogénat la moelle épinière de souris 2-3 dans 5-7 ml de Percoll 70% et la superposition de 7 ml de Percoll 40%. Centrifuger à 2000 tours par minute (pas de frein!) Pendant 30 minutes.
- Jeter les cellules endommagées ou débris de myéline sur un sommet de 70% et de recueillir des monocellules nucléaires à 40% / 70% d'interface. Diluer les cellules recueillies au moins à un 1:1 avec du PBS 1X et de spin à 2000 rpm pendant 5-7 minutes. Remettre en suspension les cellules dans 0,5 ml de PBS et de les transférer dans 1,7 ml Ependorf tube. Rendement typique est de ~ 500 x 10 3 cellules mononucléées de la souris par, ce qui donnerait le nombre de cellules microgliales ~ 1x10 6 à 0,5 ml pour la préparation des cellules de cerveau et la moelle épinière de souris 2-3.
2. Contrôle de la pureté de la microglie par cytométrie en flux
La coloration avec des anticorps est effectué comme décrit précédemment, avec les adaptations 7,8.
- Préparer tampon FACS (PBS 1X avec FBS 2%), les anticorps FcR de blocage, anti-CD11b - PE, anti-CD45-APC-Cy7 anticorps, 1% de paraformaldéhyde dans du PBS.
- Prenez 50 ul des cellules de l'étape 1.9 et de les transférer à nouveau tube de 1,7 ml Eppendorf et ajouter 350 pi de tampon FACS.
- Ajouter ul de 5-10 anti-anticorps FcR et incuber 10-15minutes sur la glace.
- Fractionner les cellules dans des tubes Eppendorf deux. Ajouter à un tube 1 ul d'anticorps anti-CD11b - PE et 2 pi anti-CD45-APC-Cy7 anticorps et incuber 10-15 minutes sur la glace. Second tube sera utilisé comme un contrôle négatif pour immunofluorescence. Après une incubation avec des anticorps ajouter 1 ml de tampon FACS à deux tubes et de spin dans petite centrifugeuse à 5400 rpm pendant 5 min. Après avoir tourné fixer les cellules en les remettant en suspension dans 400 pi de 1% de paraformaldéhyde dans du PBS et vortex.
- Effectuer une analyse de cytométrie en flux sur cytomètre en flux (LSR II, BD Biosciences) en utilisant BD compubeads (BD Biosciences) pour les contrôles de compensation comme cela a été décrit 8. Des exemples de bonnes et de mauvaises préparations sont présentés dans la figure. 1a, b.
3. Isolement de la microglie et les macrophages périphériques du SNC enflammées dans le modèle souris de la neuroinflammation
Encéphalite auto-immune expérimentale (EAE) est induite comme cela a été décrit précédemmentdans notre papier 7; tri FACS est réalisé de manière similaire à publié protocole 8.
- Provoquer l'EAE par la vaccination sous-cutanée de 150 mg MOG 35-55 peptide dans 4 mg / ml de l'adjuvant complet de Freund (CFA) du groupe de 5-10 du 8-12-week-old souris C57BL / 6. Toxine de la coqueluche devrait être donnée ip (150 ng / souris), les jours zéro et le deuxième jour, après la vaccination. Les souris sont observées pour des signes de la maladie à partir du jour 10 post-vaccination, et sévérité de la maladie est notée sur une échelle numérique de 0 à 5 comme suit: 0) aucune maladie; 1) la queue faible ou à pied bancal; 2) parésie des membres postérieurs; 3) paralysie des membres postérieurs; 4) la paralysie des membres antérieurs et postérieurs, et 5) la mort ou l'euthanasie pour des raisons humanitaires. Souris à la pointe de la maladie (d21 post-immunisation) sont utilisés pour l'expérience avec un score de maladie typique allant de 1,5 à 2,5.
- Isoler les cellules mononucléaires du CNS d'un groupe de souris comme dans les étapes 5-10 1.1-1.4 et colorer les cellules avec des anticorps anti-CD11b et anti-CD45 comme dans les étapes 2.1-2.4.Dans l'étape 2.4 ne résout pas les cellules, au lieu de les remettre en suspension dans 400 ul de PBS 1X au lieu de 1% de paraformaldéhyde.
- Tri cellulaire est effectuée par FACSAria pour les populations de CD11b + CD45 faible microglie et les cellules CD45 + de CD11b Hi (~ 80% d'entre eux sont des macrophages périphériques et ~ 20% d'entre eux sont activés microglie 3; à l'avenir nous allons nous référer à ces cellules comme « macrophages "). Utilisez 1,7 ml Ependorf tubes pour la collecte des échantillons. 300 pi de PBS 1X avec 10% de FBS doit être ajouté à des tubes de prélèvement pour éviter tout dommage des cellules triées. Exemples de trois populations de la microglie, les macrophages et les lymphocytes sont présentés dans la figure. 2a avec de bonnes paramètres de grille de tri indiquées dans la Fig. 2b. Puretés de tri typiques étaient à 98-100% tel que déterminé par réanalyse populations triées de la microglie (Fig. 2c) et les macrophages (Fig. 2d). Rendement typique est de 20-50 x10 3 pour la microglie et les50-100 x 10 3 isolés pour les macrophages dans le groupe de cinq souris.
4. Isolement de l'ARN
ARN sera isolé en utilisant le kit Mirvana conformément aux instructions du fabricant, avec les adaptations.
- Spin microglie du SNC normal de l'étape 1.9 ou sous-ensembles de la microglie et triés macrophages de l'étape 3.3 dans des tubes Eppendorf de 1,7 ml à 5400 rpm pendant 5-7 minutes dans la centrifugeuse petite, soigneusement éliminer le surnageant et congeler les culots cellulaires sur la glace sèche. Passez à l'étape 4.2 ou transférer des culots de cellules congelés à -70 ° C, où il peut être stocké pendant 2-3 mois.
- Transfert culots cellulaires congelés à la glace régulière pour la décongélation. Ajouter 300 ul de tampon de lyse du kit Mirvana et mélanger délicatement par pipetage 5-7 fois.
- Ajouter 30 ul de l'additif homogénat de Mirvana kit, vortex 30 secondes pour mélanger, garder sur la glace pendant 10 minutes
- Ajouter 300 ul d'acide-Pehnol: chloroforme à partir Mirvana kit, vortex 30 secondes, de spin pour 5 mdans à 10.000 tours sur petite centrifugeuse.
- Prenez soigneusement phase supérieure aqueuse (300 pi) et de transférer à un autre tube de 1,7 ml Eppendorf, mélanger avec 375 ul d'éthanol à 100%, le transfert à la cartouche du filtre à partir Mirvana kit et de spin pendant 1 min à 10000 rpm.
- Jeter l'éluat tampon, ajouter 700 ul de solution de lavage I à partir de Mirvana kit, et de spin pendant 1 min à 10000 rpm.
- Laver deux fois plus avec 500 ul de Solution de lavage II à partir de Mirvana kit et enfin éluer l'ARN de la cartouche avec 60 ul de pré-chauffée d'eau sans nucléase.
- La quantité et la qualité de l'ARN est évaluée à l'aide spectrophotomètre Nanodrop. La concentration de l'ARN doit être comprise entre 5 et 20 ng / ml et de diamètre extérieur λ260 / λ280 rapport compris dans la plage de 1,7 à 2,0. Si la concentration de l'ARN est inférieure à 3 ng / ml et OD 260λ/280λ est inférieur à 1,6, les échantillons sont mis au rebut. Échantillons d'ARN peuvent être stockés à -70 ° C pendant 6-12 mois.
- Pour l'estimation supplémentaire de la qualité de l'ARN, l'ar échantillonse analysés par électrophorèse sur gel à 15% TBE-urée Gel (Life Technologies). Des exemples typiques de bonne qualité et mauvaise des préparations d'ARN sont présentés dans la figure. 3a et Fig.3b, respectivement.
5. Détection de miRNA dans la microglie et les macrophages
Pour l'analyse de l'expression du miARN, quantitative en temps réel RT-PCR (qRT-PCR) analyses est effectuée en utilisant des dosages TaqMan avec des amorces et des sondes fluorescentes pour microARN d'intérêt particulier et l'un des ARN exprimés de manière ubiquitaire de courte durée (par exemple snoRNA-55, snoRNA -135 ou U6) pour la normalisation. Les amorces et les sondes fluorescentes pour des ensembles particuliers de miARN humain ou de souris peuvent être achetés à partir de Life Technologies Inc Ici, nous allons utiliser miR-124 comme un exemple pour les miARN d'intérêt et snoRNA-55 comme un exemple pour la normalisation.
- Effectuer inverser la réaction de la transcriptase faire ADNc à partir d'échantillons d'ARN en utilisant le kit TaqMan transcription inverse:
| Point | Volume (pi) par échantillon |
| 5x amorce TI | 1,00 |
| Échantillon d'ARN | 1,67 |
| RT Enzyme Mix | |
| 25 mm de dNTP (100 mm au total) | 0,050 |
| MultiScribe la transcriptase inverse) | 0,333 |
| 10X tampon RT | 0,500 |
| AB inhibiteur de RNase | 0,063 |
| Eau sans nucléase | 1,388 |
| Total | 5,00 |
Diluer l'ARN des échantillons à une concentration de 3 ng / ul. Préparer le mélange enzymatique RT et de mettre 3,33 pi à tubes PCR et ajouter 1,67 ul de l'échantillon d'ARN. Incuber dans PCR Instrument (BioRad) à 16 ° Cpendant 30 min, 42 ° C pendant 30 min, 85 ° C pendant 5 min, et fixé à 4 ° C en attente.
- Effectuer en temps réel TaqMan PCR en utilisant les mélange PCR:
| Point | Volume (pi) par échantillon |
| RT produit | 1.0 |
| 2X Master Mix TaqMan | 5.0 |
| Sonde 5X | 2.0 |
| Eau sans nucléase | 2.0 |
| Total | 10,0 |
Utilisez une plaque de réaction de 384 puits clair optique (Life Technologies) pour chaque réaction et PCR en temps réel instrument AB 7900 HT. Conditions de cyclage: 95 ° C pendant 10 min, [95 ° C pendant 5 s et 60 ° C pendant 60 s] x 40 cycles.
- Courbes typiques pour la quantité de fluorescently étiquetés spécifiques des produits de PCR (Y-axes) pour snoRNA-55 et miR-124 pour les échantillons d'ARN (chaque échantillon est en double) isolés à partir de la microglie et les macrophages dérivés de moelle osseuse (BMDMs) par rapport au nombre de cycles (x-axes) sont Fig. 4a, b.
6. Analyse Normalisation et de données
Les niveaux d'expression relatifs de miARN d'intérêt (miR-124) sont calculés selon la méthode ΔΔCT comme décrit précédemment en utilisant 7,9 snoRNA-55 pour la normalisation de la quantité initiale de l'ARN.
- L'exemple de calcul du niveau relatif de miR-124 expression de la microglie et les BMDMs est indiqué dans le tableau I.
- Dans un premier temps, les valeurs de Ct pour snoRNA-55 et miR-124 sont déterminés pour la microglie et les BMDMs de qRT-PCR courbes (Fig.4; tableau I, colonnes: snoRNA-55 et miR-124).
- Dans un deuxième temps, les valeurs ΔCtsont calculés pour chaque exemplaire de chaque échantillon par soustraction de Ct pour snoRNA-55 (la normalisation) à partir de Ct pour miR-124 (expérience) (tableau I, colonnes: Ct (Norm), Ct (Exp), et ΔCt).
- Dans une troisième étape. Les valeurs ΔΔCt sont calculés par soustraction de ΔCt de l'échantillon de référence (ΔCt (Ref)) à partir des valeurs ΔCt des échantillons d'autres (tableau I, colonne: ΔΔCt). Un échantillon est définie comme une référence, le niveau d'expression par rapport miARN pour cet échantillon est défini comme 1,0. Tous les autres échantillons seront comparés à cet échantillon de référence. Dans notre cas, nous définissons le niveau de miR-124 dans le premier échantillon de la microglie que 1,0, donc ΔCt (Ref) = 0,49 (tableau I, première rangée, marqué en rouge).
- Enfin, le niveau relatif d'expression est calculé comme le 2-ΔΔCt (tableau I, colonne: miR-124 eXpression).
- Tous les QRT-PCR sont effectuées en triple ou en double, et le niveau relatif d'expression est présenté sous forme de moyenne ± écart-type (SD) pour trois exemplaires (Fig. 5a, b) ou comme les deux côtés des doublons à côte, comme le montre la figure. 5c.
7. Les résultats représentatifs
Courbes typiques pour PCR en temps réel et les valeurs CT pour miR-124 (microARN d'intérêt) et snoRNA-55 (la normalisation) ainsi que les niveaux relatifs d'expression de miR-124 sont présentés dans la figure. 4 et Tableau I. Dans nos expériences, nous avons utilisé snoRNA-55 en tant que témoin pour la normalisation. Comme nous l'avons mentionné ci-dessus, d'autres ARN omniprésents peut également être utilisé pour la normalisation tels que snoRNA-135 et U6.
Les exemples de l'expression de miR-124 chez les adultes microglies de la moelle osseuse par rapport aux macrophages dérivés (BMDMs) sont présentés dans la figure. 5a (BMDMs ont été cultivées dans le presence de M-CSF tel que décrit 7). Dans la figure. 5b, nous avons comparé l'expression de miR-124 dans les populations triées de CD45 faible microglies contre CD45 des macrophages hi. Dans ces deux expériences, nous avons démontré que les microglies sont différents de macrophages autres en exprimant des niveaux élevés de miR-124. Des expériences similaires peuvent être effectués à l'avenir pour la cinétique de l'activation des microglies in vivo ou in vitro.
L'expression de miR-124 dans la microglie isolés de SNC de souris C57BL / 6 à différents stades de développement est représenté sur la figure. 5c. Ces données démontrent que la microglie dans les premiers stades de développement expresse des niveaux plus faibles de miR-124, en corrélation avec leur phénotype activé 7. Une analyse similaire peut être effectuée pour étudier les fonctions de la microglie dans le SNC normales telles que la comparaison de l'expression des miARN particulier dans la microglie isolés à partir de différents domaines du système nerveux central: le cervelet, scordon Pinal, etc hippocampe
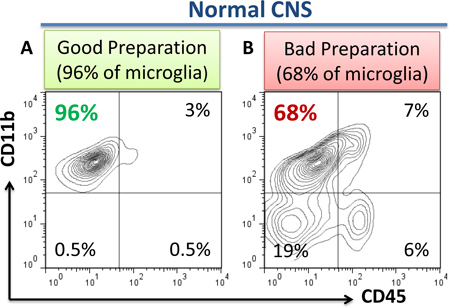
Figure 1. Les exemples de bonnes et de mauvaises préparations de la microglie tel que déterminé par cytométrie en flux. Dans le cas de «bonne» l'isolement de la microglie du SNC normal, 95-98% des cellules CD45 + représentent CD11b microgla faible avec 2-5% de CD11b + CD45 hi macrophages périvasculaires. Moins de 1% du CD11b - CD45 des lymphocytes hi ou CD11b - CD45 - cellules astrogliales ou oligodendrogliale ou des fragments cellulaires devraient être présents (a). Le cas de "mauvaise" la préparation est montré ici (b) où 18% de contamination CD11b - CD45 - cellules est présent (b, quadrant inférieur gauche), suggérant une séparation insuffisante gradient de Percoll. Dans le cas d'une bonne préparation, 95-98% des cellules devrait représenter CD11b + CD45faible microglie (a, dans le quadrant supérieur gauche), 2-5% de toutes les cellules doivent représenter CD11b + CD45 des macrophages périvasculaires hi (a, dans le quadrant supérieur droit), et moins de 1% de toutes les cellules doivent représenter CD11b négatifs cellules contaminantes (a, quadrant inférieur gauche). Dans le cas de "mauvaise préparation", une contamination importante de CD11b - CD45 - des cellules ou des fragments de cellules (astrocytes, des particules de myéline, etc) sont présents (b, quadrant inférieur gauche), ce qui rend la préparation des cellules pour l'isolement inadaptés ARN et plus analyse. En outre, CD11b - CD45 des cellules hi pourrait être également présent dans le cas de "mauvaise préparation" (b, quadrant inférieur droit) indiquant une contamination par des lymphocytes sanguins en raison de la perfusion insuffisante.
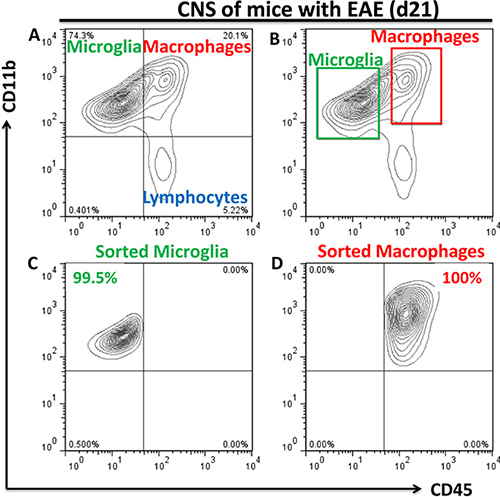
Figure 2. Cytométrie en flux analysis et de tri des populations de la microglie et les macrophages périphériques du SNC malades (a) Au cours de la neuroinflammation trois populations des cellules sont présents dans le SNC: CD11b + CD45 faible (microglie), CD11b + CD45 hi (macrophages), et CD11b - CD45. Salut (lymphocytes), comme indiqué dans le coin supérieur gauche, supérieur droit et inférieur droit quadrants, respectivement. Gates, pour le tri des populations de CD11b + microglie CD45 faible et CD11b + CD45 hi macrophages sont présentés dans (b) et les puretés des populations réanalysé triés sont présentés dans (c, d). Gating préliminaire sur les cellules viables sur la base de FSC / SSC paramètres a été mis en œuvre.
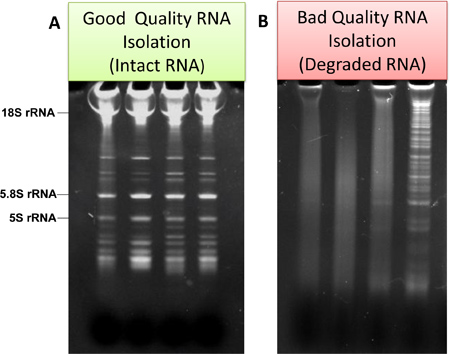
Figure 3. Des exemples de qualit "bon" et "mauvais"s des ARN isolés à partir de la microglie et analysés par électrophorèse sur gel. Dans le cas de bonne qualité, l'échelle de l'ARN et l'ARN ribosomal est évident (a). Dans le cas de mauvaise qualité, aux taches et à faible poids moléculaire des produits de dégradation de poids sont évidents (b).
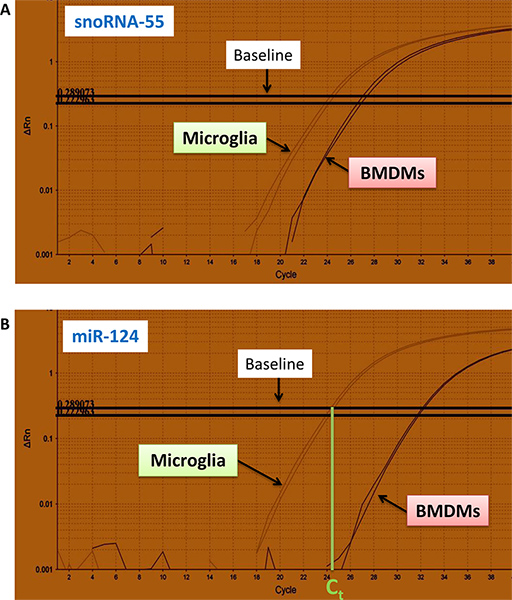
Figure 4. En temps réel QRT-PCR pour les courbes marquées par fluorescence miR-124 et snoRNA-55 produits de PCR. Courbes pour snoRNA-55 (a) et miR-124 (b) sont indiqués pour la microglie et les macrophages de la moelle osseuse dérivés (BMDMs). L'expérience a été réalisée en double. Les valeurs de Ct ont été déterminés par l'intersection de la ligne de base avec la partie inférieure de la gamme linéaire de qRT-PCR courbes comme indiqué dans (b). Cliquez ici pour agrandir la figure .
| snoRNA-55 | miR-124 | DCT = Ct (Exp)-Ct (Norm) | DDCT = DCT-DCT (Ref) | Niveau relatif d'expression = 2-DDCT | |
| Ct (Norm) | Ct (Exp) | DCT | DDCT | miR-124 Expression | |
| Les microglies du SNC normal | 24.082 | 24.572 | 0,49 (Ref) | 0 | 1.0 |
| 23.766 | 24.291 | 0,525 | 0,035 | 1,02 | |
| De la moelle osseuse macrophages dérivés (BMDMs) | 26.879 | 320.231 | 5,352 | 5,317 | 0,025 |
| 26.526 | 32.078 | 5,552 | 5,517 | 0,022 |
Tableau I. Calcul des niveaux relatifs de miR-124 l'expression dans les macrophages microglie la moelle osseuse par rapport aux dérivés fondés sur les valeurs t C pour miR-124 (microARN d'intérêt) et snoRNA-55 (contrôle de la normalisation).
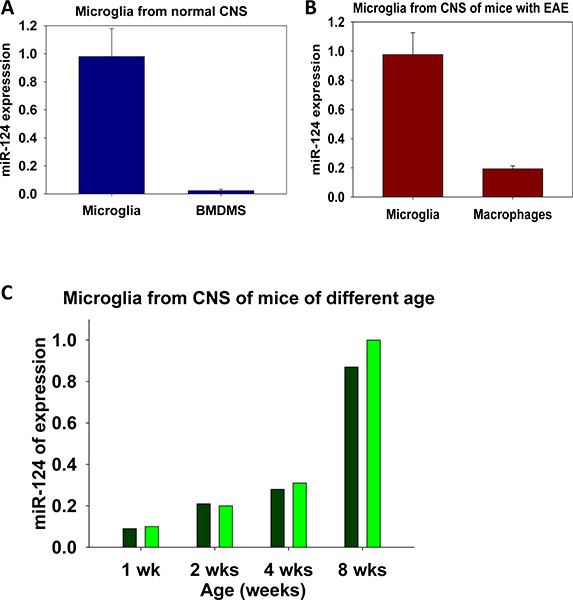
Figure 5. Analyse de miR-124 expression dans la microglie et les macrophages. Comparaison de miR-124 expression dans la microglie du SNC normal vs macrophages dérivés de moelle osseuse (BMDMs) est montré en (a). Comparaison de miR-124 dans la microglie vs macrophages périphériques isolées du SNC de la souris avec la neuroinflammation est montré dans (b). Les variations de laniveau d'expression de miR-124 dans la microglie au cours du développement chez la souris de 1, 2 et 4 semaines de vieux par rapport à des souris adultes (à 8 semaines) est montré dans (c). En (a, b) l'expérience a été réalisée en trois exemplaires, avec une moyenne ± écart-type de trois réactions PCR séparées montré. En (c) expérience a été réalisée en double comme indiqué.
Discussion
Récemment un intérêt significatif a été donné à la microglie et l'implication de ces cellules dans de nombreuses pathologies associées à la neuroinflammation et la neurodégénérescence. En outre, le rôle des microARN dans la différenciation des cellules immunitaires et le cancer a considérablement augmenté au cours des dernières années, plusieurs 5,10. Nous avons déjà signalé le rôle de miR-124 dans le maintien du phénotype non activé ou au repos de la microglie dans le SNC normales...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Le travail a été soutenu par le NIH R01 NS071039-01A1 subvention de recherche.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| PBS 1X | GIBCO | 14190 | Peuvent être préparés à partir de composants secs dans le laboratoire, pH: 7.2 à 7.4 |
| PBS 10X | Thermo Scientific | SH30258.02 | Peuvent être préparés à partir de composants secs dans le laboratoire, pH: 7.2 à 7.4 |
| DMEM; FBS | BCBG | 11965; 10438 | |
| Mirvana kit | Invitrogen | AM1561 | |
| Percoll | Sigma | P4937-500 ml | 100 ml de Percoll 100% est préparée en mélangeant 10 ml de PBS 10X et 90 ml de Percoll de Sigma. 50 ml de Percoll 70%préparé par le mélange de 35 ml de Percoll à 100% et 15 ml de DMEM 50 ml de Percoll 40% est préparée par mélange de 20 ml de Percoll 100% et 30 ml de 1X PBS |
| MOG 35-55 peptide | AnaSpec Inc | 60130-5 | |
| CFA | DIFCO | 263810 | |
| Toxine de la coqueluche | Sigma | P7208 | |
| FcR anticorps bloquants | BD Biosciences | 553142 | Clone 2.4G2 |
| Anti-CD11b - PE mAb | BD Biosciences | 553311 | Peut être utilisé avec différents fluorophores (par exemple AF488) |
| Anti-CD45-APC-Cy7 mABs | Biolegend | 103116 | Peut être utilisé avec différents fluorophores (par exemple APC) |
| Paraformalhyde (16%) | EMS Inc | 15710 | Diluer 1:15 dans du PBS 1X |
| 15% TBE-urée Gel | Life Technologies Inc | EC68855BOX | |
| MicroRNA TaqMan Kit de transcription inverse | Life Technologies Inc | 4366596 | |
| TaqMan Universal PCR Master Mix | Life Technologies Inc | 4304437 | |
| Les dosages TaqMan microARN miR-mmu-124a | Life Technologies Inc | 4427975 001182 ID | |
| Les dosages TaqMan microARN snoRNA-55 | Life Technologies Inc | 4427975 001228 ID | |
| Eau sans nucléase | Life Technologies Inc | AM9937 | Eau sans nucléase |
| Plaque 384 puits clair réaction optique | Life Technologies Inc | 4309849 | Peut être remplacé par d'autres plaques (par exemple une plaque de 96 puits) dans les différents instruments de PCR en temps réel |
| Dounce homogénéisateur (15 ml) | Wheaton Science Products. | 358044 | Teflon / verre |
| 40 tamis cellulaire μ en nylon | Faucon | 352340 | |
| Centrifugeuse Big | Sorval | RT 6000B | Diamètre du rotor 18,5 cm |
| Petite centrifugeuse | Eppendorf | 5415 R | Rotor diamètre de 6,5 cm |
| En temps réel instrument PCR | Life Technologies Inc | AB 7900 HT | Peut être remplacé par d'autres instruments |
| Cytomètre en flux | BD Biosciencess | LSR II | Peut être remplacé par d'autres instruments |
| FACS | BD Biosciences | FACSAria | Peut être remplacé par d'autres instruments |
| Spectrophotomètre Nanodrop | Thermo Scientific | Nanodrop 1000 |
Références
- Tremblay, M. E. The Role of Microglia in the Healthy Brain. J. Neurosci. 31, 16064-16069 (2011).
- Graeber, M. B., Li, W., Rodriguez, M. L. Role of microglia in CNS inflammation. FEBS Lett. , (2011).
- Ponomarev, E. D., Shriver, L. P., Maresz, K., Dittel, B. N. Microglial cell activation and proliferation precedes the onset of CNS autoimmunity. J. Neurosci. Res. 81, 374-389 (2005).
- Perruisseau-Carrier, C., Jurga, M., Forraz, N., McGuckin, C. P. miRNAs stem cell reprogramming for neuronal induction and differentiation. Mol. Neurobiol. 43, 215-227 (2011).
- McCoy, C. E. The role of miRNAs in cytokine signaling. Front Biosci. 17, 2161-2171 (2010).
- He, M., Xu, Z., Ding, T., Kuang, D. M., Zheng, L. MicroRNA-155 regulates inflammatory cytokine production in tumor-associated macrophages via targeting C/EBPbeta. Cell Mol. Immunol. 6, 343-352 (2009).
- Ponomarev, E. D., Veremeyko, T., Barteneva, N., Krichevsky, A. M., Weiner, H. L. MicroRNA-124 promotes microglia quiescence and suppresses EAE by deactivating macrophages via the C/EBP-alpha-PU.1 pathway. Nat. Med. 17, 64-70 (2011).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). J. Vis. Exp. (41), e1546-3791 (2010).
- Gabriely, G. MicroRNA 21 promotes glioma invasion by targeting matrix metalloproteinase regulators. Mol. Cell Biol. 28, 5369-5380 (2008).
- Caffarelli, E., Filetici, P. Epigenetic regulation in cancer development. Front Biosci. 17, 2682-2694 (2011).
- Cardona, A. E., Huang, D., Sasse, M. E., Ransohoff, R. M. Isolation of murine microglial cells for RNA analysis or flow cytometry. Nat. Protoc. 1, 1947-1951 (2006).
- Gordon, R. A simple magnetic separation method for high-yield isolation of pure primary microglia. J. Neurosci. Methods. 194, 287-296 (2011).
- Moltzahn, F., Hunkapiller, N., Mir, A. A., Imbar, T., Blelloch, R. High Throughput MicroRNA Profiling: Optimized Multiplex qRT-PCR at Nanoliter Scale on the Fluidigm Dynamic ArrayTM IFCs. J. Vis. Exp. (54), e2552 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon