Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Modelado específico del paciente del corazón: Estimación de las orientaciones de las fibras ventriculares
En este artículo
Resumen
Una metodología para estimar las orientaciones de fibras ventriculares de imágenes in vivo de las geometrías del corazón del paciente para el modelado personalizado se describe. Validación de la metodología a cabo utilizando normal y no corazones caninos demostrar que que no hay diferencias significativas entre las orientaciones de las fibras estimados y adquirido a un nivel clínicamente observable.
Resumen
Específicos del paciente simulaciones de corazón (dis) función dirigida a la personalización de la terapia cardiaca se ve obstaculizada por la ausencia de la tecnología de formación de imágenes in vivo para clínicamente adquirir orientaciones de fibra de miocardio. El objetivo de este proyecto fue desarrollar una metodología para estimar las orientaciones de fibras cardíacas de imágenes in vivo de las geometrías del corazón del paciente. Una representación precisa de la geometría ventricular y orientaciones de las fibras fue reconstruido, respectivamente, de alta resolución ex vivo resonancia estructural magnética (RM) y tensor de difusión (DT) imágenes de MR de un corazón humano normal, referido como el atlas. Geometría ventricular de un corazón paciente se extrajo, a través de la segmentación semiautomática, a partir de un estudio in vivo tomografía computarizada (TC) de imagen. Usando algoritmos de transformación de la imagen, la geometría ventricular atlas se deforma para coincidir con la del paciente. Por último, el campo de deformación se aplicó a la orientat atlas fibraiones para obtener una estimación de orientaciones de fibra de pacientes. La precisión de las estimaciones de fibra se evaluó utilizando seis normales y tres corazones insuficientes caninos. La diferencia absoluta media entre los ángulos de inclinación de las orientaciones de fibra adquiridos y estimado fue de 15,4 °. Simulaciones computacionales de mapas de activación ventricular y pseudo-ECG en ritmo sinusal y taquicardia ventricular se indica que no hay diferencias significativas entre las orientaciones de fibra estimados y adquiridos a clínicamente observables ideas nuevas level.The obtenidos del proyecto allanará el camino para el desarrollo de específicos del paciente modelos del corazón que puede ayudar a los médicos en el diagnóstico personalizado y decisiones sobre las intervenciones electrofisiológicos.
Introducción
El enfoque computacional se está convirtiendo central para el avance de la comprensión de la función del corazón en la salud y la enfermedad. State-of-the-art de todo corazón-modelos de electrofisiología y electromecánica se están utilizando para estudiar una amplia gama de fenómenos, tales como la propagación ventricular normal, la arritmia, la desfibrilación, el acoplamiento electromecánico, y la resincronización cardiaca 1. Sin embargo, para el enfoque computacional para ser directamente aplicables en el entorno clínico, es imperativo que los modelos de ser específica para el paciente, es decir, los modelos debe estar basado en la arquitectura específica y propiedades electrofisiológicas o electromecánicas de corazón enfermo del paciente. Simulación con estos modelos ayudarán a los médicos a llegar a decisiones altamente personalizadas para las intervenciones electrofisiológicos, así como la profilaxis, así dramáticamente mejorar la atención de la salud cardiaca 2-4.
contenido "> Creación de modelos realistas cardíacos requiere la adquisición de la geometría y la estructura de fibra de un paciente cardíaco. orientaciones de fibra determinar direcciones de propagación eléctrica y distribuciones de tensión en el corazón, y por lo tanto la adquisición de ellos es esencial para el modelado cardíaco 5, 6. Con los recientes avances en imagen médica, es ahora posible obtener la geometría de un corazón paciente, remodelación includingstructural tales como el infarto, in vivo con alta resolución usando imágenes por resonancia magnética (MRI) y tomografía computarizada tecnologías (CT). Sin embargo, no hay método práctico para la adquisición de estructura de la fibra de un corazón paciente in vivo. tensor de difusión (DT) RM 7, 8, la única técnica para adquirir orientaciones de las fibras del corazón intacto, no está ampliamente disponible en vivo debido a ciertas limitaciones 9. Una breve descripción de los esfuerzos anteriores para traducir DTMRI para el entorno clínico se pueden encontrar elsewhere 2. Aunque metodologías como regla basada en la asignación de las orientaciones de fibra de ofrecer alternativas a DTMRI, estos métodos tienen ciertas limitaciones graves 2, 10. Así, las dificultades en la adquisición de estructura de la fibra cardiaca in vivo en la actualidad impiden la aplicación de simulaciones cardíacos electrofisiológicos y electromecánicos en la clínica. El objetivo de esta investigación fue abordar directamente esta necesidad.La hipótesis de que las orientaciones de fibra ventricular de un corazón puede predecirse con precisión debido a la geometría del corazón y de un atlas, donde el atlas es un corazón cuya geometría y orientaciones de fibra están disponibles. En consecuencia, se utilizó el estado de la técnica para desarrollar una metodología para la estimación de las orientaciones de las fibras cardíacas en vivo, y probaron la hipótesis de normalidad y de no ventrículos caninos 2. La idea central de nuestra metodología de estimación de fibra es explotar similarities en orientaciones de las fibras, con respecto a la geometría, entre corazones diferentes con el fin de aproximarse a la estructura de la fibra de un corazón (objetivo) para que sólo la información de geometría se encuentra disponible. En el corazón de nuestra metodología de estimación es el registro de la geometría atlas con la geometría de destino mediante mapas difeomorfa gran deformación métrica (LDDMM) 11, y la transformación de las orientaciones de fibra atlas con la preservación de los componentes principales (PPD) 2, 12. El diffeomorphicproperty de garantías LDDMM que el atlas no "plegable" sí durante la deformación, preservando así las estructuras anatómicas integrityof. Figura 1 ilustra la canalización de procesamiento de nuestra metodología. La sección del texto protocolo § 1 describe los diversos componentes de la tubería mediante la demostración de cómo la estimación se puede realizar para un paciente ejemplo. Los números dentro de algunos de los bloques en la Figura 1 se refieren a la correspondientesubsecciones en la sección § 1 del texto del protocolo.
Se evaluó el desempeño de la metodología propuesta por la cuantificación del error de estimación, y medir el efecto de esta onsimulations de error de la electrofisiología cardiaca, por computacionalmente simulando locales mapas de activación eléctricas, así como pseudo-electrocardiogramas (ECGs pseudo-). Debido a la falta de disponibilidad de los corazones humanos, la evaluación del desempeño se realizó con corazones caninos disponibles a partir de estudios previos 13-15. El error de estimación se calcula por medio de ángulos de inclinación 16, la tradición followingthe de histología, donde las mediciones angulares son secciones performedon tejido que son cortadas en paralelo a la epicardialsurface. Desde el anglebetween la dirección de la fibra y el plano tangente epicárdica es generallysmall 17, 18, la pérdida de información en la descripción de un fiberdirection enteramente usando su ángulo de inclinación es insignificante. Para la computsimulaciones acionales, basados en imágenes modelos se construyeron como se informó anteriormente 19, 20, y el tejido cardíaco en los modelos fue representado establecidos sobre la base de técnicas matemáticas y los datos experimentales 21-25. El ritmo sinusal se simuló por la activación de replicación procedente de la red de Purkinje 26, y la taquicardia ventricular, por una estimulación S1-S2 protocolo 27. Los pseudo-ECG se calcularon 28 y compararon mediante la desviación media absoluta (MAD) métrica 29.
Protocolo
1. Orientaciones de fibra Estimación
- Adquirir imágenes de MRI estructurales y DTMRI de un corazón humano adulto normal en diástole, con una resolución de 1 mm 3. Usando ImageJ, extraer el miocardio ventricular de la imagen estructural atlas por ajuste, para cada rebanada de eje corto, cerrado splines a través de un conjunto de puntos de señal colocados a lo largo de los límites epicárdicos y endocárdicos en la rebanada (Figura 2A y la figura 2B). Realizar la colocación de puntos de señal manualmente para cada rebanada 10 ª en la imagen. Obtener los puntos de señal para las rebanadas restantes al interpolar linealmente los puntos manualmente identificados, usando MATLAB.Reconstruct las orientaciones de las fibras del corazón atlas mediante el cálculo de los vectores propios primarias de los DTS en la imagen DTMRI (Figura 2C).
- Adquirir una imagen de la geometría del corazón del paciente en diástole utilizando in vivo cardíaco CT o MRI. Reconstruct la geometría del corazón del paciente de la imagen de manera similar a la forma en que se construyó el atlas (Figura 3A y la Figura 3B). La imagen de paciente debe volver a tomar muestras antes de la reconstrucción de tal manera que la resolución en el plano es de 1 mm 2. De manera similar, el número de cortes para que puntos de referencia son recogidos manualmente, y el intervalo de interpolación fuera de plano debe ajustarse de modo que la imagen segmentada paciente corazón tiene un grosor de corte de 1 mm.
- Deformar la imagen ventricular atlas para que coincida con la imagen geometría paciente en dos pasos. En el primer paso, realizar una transformación afín sobre la base de un conjunto de puntos de puntos de referencia de trece: el ventrículo izquierdo (LV) ápice, el ventrículo dos a la derecha (RV) puntos de inserción en la base, los dos puntos de inserción de RV a mitad de camino entre la base y el ápice, y cuatro conjuntos de dos puntos que dividen uniformemente RV LV epicardio y los contornos en la base, ya mitad de camino entre la base y el ápice (Figura 4A y Figura 4B). En el segundo paso, deformar la transformada afín-atlas ventrículos aún más para que coincida con la geometría de la paciente, utilizando la correlación de gran difeomorfa deformación métrico (LDDMM) (Figura 4C).
- Morph la imagen DTMRI del atlas por el cambio de posición de voxels de imagen y reorientando los DTS de acuerdo con la matriz de transformación de la correspondencia afín y el campo de deformación de la transformación LDDMM. Realizar la re-orientación de los DTs con la preservación de las direcciones principales (PPD) método.
- Obtener la estimación de las orientaciones de las fibras de pacientes de la transformada imagen atlas DTMRI calculando el vector propio principal de la DT (Figura 5).
2. Medición del error de estimación
- Adquirir ex vivo estructural y MR imágenes DTMR de seis normales y tres corazones insuficientes caninos, a una resolución de 312,5 x 312,5 x 800 m 3. Aquí, el corazón faiseñuelo se debe generar en los caninos a través de la ablación por radiofrecuencia de la rama izquierda seguido de 3 semanas de tachypacing a 210 min -1.
- Segmento de los ventrículos del corazón canino de manera similar al corazón humano, atlas, tal como se describe en el § 1.1. Denotar ventrículos segmentados de corazones caninos normales como corazones 1 a 6, y los de no segmentado corazones caninos como corazones 7 a 9 (Figura 6).
- Obtener cinco diferentes estimaciones de orientaciones de las fibras ventriculares del corazón 1 mediante el uso de cada uno de los corazones de 2 a 6 como un atlas (Figura 7).
- Estimar orientaciones de las fibras para cada uno de los ventrículos del corazón fallan al usar 1 como el atlas (Figura 8).
- Foreach punto de datos de cada conjunto de orientaciones de fibra estimados, calcular el error de estimación como | θ c-θ a |, donde θ θ c y A son los ángulos de inclinación de un estimadod adquirido orientaciones de las fibras en ese punto, respectivamente.
- Para cada punto de datos en cada conjunto de orientaciones de fibra estimados, calcular el ángulo agudo betweenestimated y adquirió direcciones de las fibras en tres dimensiones (3D) por medio de thevector producto escalar.
3. La medición de los efectos del error de estimación en las simulaciones
- De corazón 1, la construcción de seis modelos, uno con las orientaciones de fibra DTMRI-adquiridas de corazón 1 (denominado modelo 1), y cinco con las cinco orientaciones de fibra estimados conjuntos de datos (modelos de 2 a 6). Para cada uno de el corazón tres defecto geometrías, construir dos modelos ventriculares, uno con los DTMRI adquiridas en orientaciones de fibra y el otro con las orientaciones de fibra estimados. Aquí, la resolución espacial de los modelos, calculado en términos de la longitud de borde media de las mallas, debe ser de aproximadamente 600 micras. Denotemos los modelos de insuficiencia cardiaca con fibras DTMRI adquiridas como modelos de 7 a 9, y los que tienen estifibras ed como modelos de 10 a 12.In los modelos, representación monodominio utilizar para describir el tejido cardíaco, con las ecuaciones que rigen:
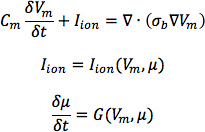
donde σ b es el tensor de conductividad a granel que se calcula a partir de los tensores de conductividad bidomain como se describe por Potse et al 30; V m es el potencial transmembrana; m C es la capacitancia de membrana específico, y de iones I es la densidad de la corriente transmembrana, que a su vez depende de V m y un conjunto de variables de estado μ describir la dinámica de los flujos iónicos a través de la m membrane.For C, utilizar un valor de 1 μ F / cm 2. Para i σ en los modelos normales del corazón canino, utilice longitudinalnales y transversales valores de conductividad de 0,34 S / m y 0,06 S / m, respectivamente. Representar lon l en los modelos Greenstein Winslow-iónicos del miocito ventricular canino. Disminuir las conductividades eléctricas en modelos caninos cardíacos ventriculares falla en un 30% (Figura 9).
- Usando el paquete de software CARP (CardioSolv, LLC), el ritmo sinusal simular con todos los modelos. Inducir taquicardia ventricular reentrante (VT) en los seis modelos fallan al usar un protocolo de estimulación S1-S2. Elige la sincronización entre S1 y S2 para obtener actividad TV sostenida durante 2 segundos después de la entrega S2. Si VT no se induce para cualquier sincronización S1-S2, disminuir las conductividades de hasta un 70% hasta VT fue inducida (Figura 10).
- Para cada simulación, calcular pseudo-ECGs tomando la diferencia de los potenciales extracelulares entre dos puntos en un baño de isotrópica que rodea el corazón. Coloque los dos puntos cerca de la base del corazón separadospor 18 cm, de tal manera que la línea que conecta ellos es perpendicular al plano de base-vértice del septo como se ilustra en la Figura 10. Para cada simulación con orientaciones de fibra estimados, calcular la métrica como MAD
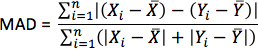
donde X es la forma de onda de ECG de la simulación con orientaciones de fibra estimados, Y es la forma de onda de ECG de thecorresponding simulación con orientaciones de fibra adquiridos, X es el valor medio de X, Y es el valor medio de Y, y n es la longitud de X e Y.
Resultados
La figura 11, muestra AC visualizaciones estilizadas de estimados, así como orientaciones de fibra DTMRI derivados en corazones normales y fallando. Examen cualitativo muestra que las orientaciones de fibra estimados alinean bien con DTMRI derivados de los. Panel D ilustra, superpuesta a la geometría del corazón 1, la distribución del error en ángulos de inclinación de los corazones normales ', promediado en los cinco estimaciones. Panel E muestra la distribución media de error de no haber á...
Discusión
Esta investigación demuestra cuantitativamente que, en ausencia de DTMRI, las orientaciones de fibra de miocardio ventrículos normales y no puede ser estimado a partir de imágenes in vivo de sus geometrías para uso en las simulaciones de la electrofisiología cardiaca. La metodología propuesta se demuestra con datos de la TC en vivo, pero es igualmente aplicable a las imágenes de RM in vivo de la geometría ventricular, frente a la falta de capacidad para adquirir directamente del pacien...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Agradecemos a los Dres. Raimond Winslow, Elliot McVeigh, y Helm Patrick de la Johns Hopkins University de proporcionar conjuntos de datos ex vivo la investigación online.This fue apoyada por los Institutos Nacionales de Salud de subvención R01-HL082729 y National Science Foundation de subvención CBET-0.933.029.
Materiales
| Name | Company | Catalog Number | Comments |
| LDDMM | Johns Hopkins University | http://cis.jhu.edu/software/lddmm-volume/index.php | |
| MATLAB | Mathworks, Inc. | R2011b | http://www.mathworks.com/products/matlab/ |
| ImageJ | Los Institutos Nacionales de la Salud | http://rsbweb.nih.gov/ij/ | |
| Tarántula | CAE Software Solutions | http://www.meshing.at/Spiderhome/Tarantula.html | |
| CARP | CardioSolv | http://cardiosolv.com/ | |
| Imágenes caninas | Johns Hopkins University | http://www.ccbm.jhu.edu/research/DTMRIDS.php |
Referencias
- Trayanova, N. Whole Heart Modeling: Applications to Cardiac Electrophysiology and Electromechanics. Circulation Research. 108, 113-128 .
- Vadakkumpadan, F., Arevalo, H., Ceritoglu, C., Miller, M., Trayanova, N. Image-Based Estimation of Ventricular Fiber Orientations for Personalized Modeling of Cardiac Electrophysiology. IEEE Transactions on Medical Imaging. 31 (5), 1051-1060 .
- Vadakkumpadan, F., Gurev, V., Constantino, J., Arevalo, H., Trayanova, N., Kerckhoffs, R. Modeling of Whole-Heart Electrophysiology and Mechanics: Towards Patient-Specific Simulations. Patient-Specific Modeling of the Cardiovascular System: Technology-Driven Personalized Medicine. , 145-165 (2010).
- Buxton, A. E., Lee, K. L., DiCarlo, L., Gold, M. R., Greer, G. S., Prystowsky, E. N., O'Toole, M. F., Tang, A., Fisher, J. D., Coromilas, J., Talajic, M., Hafley, G. Electrophysiologic testing to identify patients with coronary artery disease who are at risk for sudden death. Multicenter Unsustained Tachycardia Trial Investigators. The New England Journal of Medicine. 342 (26), 1937-1945 (2000).
- Wei, D., Okazaki, O., Harumi, K., Harasawa, E., Hosaka, H. Comparative simulation of excitation and body surface electrocardiogram with isotropic and anisotropic computer heart models. IEEE Transactions on Biomedical Engineering. 42 (4), 343-357 (1995).
- Leon, L. J., Horacek, B. M. Computer model of excitation and recovery in the anisotropic myocardium. II. Excitation in the simplified left ventricle. Journal of Electrocardiology. 24 (1), 17-31 (1991).
- Rohmer, D., Sitek, A., Gullberg, G. T. Reconstruction and Visualization of Fiber and Laminar Structure in the Normal Human Heart from Ex Vivo Diffusion Tensor Magnetic Resonance Imaging (DTMRI) Data. Investigative Radiology. 42 (11), 777-789 (2007).
- Daubert, J. P., Zareba, W., Hall, W. J., Schuger, C., Corsello, A., Leon, A. R., Andrews, M. L., McNitt, S., Huang, D. T., Moss, A. J., Investigators, M. I. S. Predictive value of ventricular arrhythmia inducibility for subsequent ventricular tachycardia or ventricular fibrillation in Multicenter Automatic Defibrillator Implantation Trial (MADIT) II patients. Journal of Americal College of Cardiology. 47 (1), 98-107 (2006).
- Sosnovik, D. E., Wang, R., Dai, G., Reese, T. G., Wedeen, V. J. Diffusion MR tractography of the heart. Journal of Cardiovascular Magnetic Resonance. 11 (1), 47-61 (2009).
- Sundar, H., Shen, D., Biros, G., Litt, H., Davatzikos, C. Estimating myocardial fiber orientations by template warping. Proc. IEEE International Symposium on Biomedical Imaging. , 73-76 (2006).
- Beg, M. F., Helm, P. A., McVeigh, E., Miller, M. I., Winslow, R. L. Computational Cardiac Anatomy Using MRI. Magnetic Resonance in Medicine. 52 (5), 1167-1174 (2004).
- Alexander, D. C., Pierpaoli, C., Basser, P. J., Gee, J. C. Spatial Transformations of Diffusion Tensor Magnetic Resonance Images. IEEE Transactions on Medical Imaging. 20, 1131-1139 (2001).
- Helm, P. A., Younes, L., Beg, M. F., Ennis, D. B., Leclercq, C., Faris, O. P., McVeigh, E., Kass, D., Miller, M. I., Winslow, R. L. Evidence of Structural Remodeling in the Dyssynchronous Failing Heart. Circulation Research. 98, 125-132 (2006).
- Helm, P., Beg, M. F., Miller, M., Winslow, R. Measuring and mapping cardiac fiber and laminar architecture using diffusion tensor MR imaging. Annals of the New York Academy of Sciences. 1047, 296-307 (2005).
- Helm, P. A., Tseng, H. -. J., Younes, L., McVeigh, E. R., Winslow, R. L. Ex vivo 3D diffusion tensor imaging and quantification of cardiac laminar structure. Magnetic Resonance in Imaging. 54, 850-859 (2005).
- Scollan, D. F., Holmes, A., Winslow, R., Forder, J. Histological validation of myocardial microstructure obtained from diffusion tensor magnetic resonance imaging. American Journal of Physiology - Heart and Circulatory Physiology. 275 (6), H2308-H2318 (1998).
- Lombaert, H., Peyrat, J., Croisille, P., Rapacchi, S., Fanton, L., Cheriet, F., Clarysse, P., Magnin, I., Delingette, H., Ayache, N. Human Atlas of the Cardiac Fiber Architecture: Study on a Healthy Population. IEEE Transactions on Medical Imaging. 31 (7), 1436-1447 (2012).
- Streeter, D. D. . Gross morphology and fiber geometry of the heart. , (1979).
- Vadakkumpadan, F., Rantner, L. J., Tice, B., Boyle, P., Prassl, A. J., Vigmond, E., Plank, G., Trayanova, N. Image-Based Models of Cardiac Structure with Applications in Arrhythmia and Defibrillation Studies. Journal of Electrocardiology. 42, 151.e1-151.e10 (2009).
- Plank, G., Zhou, L., Greenstein, J. L., Plank, G., Zhou, L., Greenstein, J. L., Cortassa, S., Winslow, R. L., O'Rourke, B., Trayanova, N. A. From mitochondrial ion channels to arrhythmias in the heart: computational techniques to bridge the spatio-temporal scales. Philosophical Transactions Series A, Mathematical, Physical, and Engineering Sciences. 366 (1879), 3381-3409 (2008).
- Roberts, D. E., Scher, A. M. Effect of tissue anisotropy on extracellular potential fields in canine myocardium in situ. Circulation Research. 50, 342-351 (1982).
- Greenstein, J., Wu, R., Po, S., Tomaselli, G. F., Winslow, R. L. Role of the Calcium-Independent Transient Outward Current I(to1) in Shaping Action Potential Morphology and Duration. Circulation Research. 87, 1026-1033 (2000).
- Winslow, R., Rice, J., Jafri, S., Marbán, E., O'Rourke, B. Mechanisms of altered excitation-contraction coupling in canine tachycardia-induced heart failure, II: model studies. Circulation Research. 84 (5), 571-586 (1999).
- Akar, F., Nass, R., Hahn, S., Cingolani, E., Shah, M., Hesketh, G., DiSilvestre, D., Tunin, R., Kass, D., Tomaselli, G. Dynamic Changes in Conduction Velocity and Gap Junction Properties During Development of Pacing-Induced Heart Failure. American Journal of Physiology - Heart and Circulatory Physiology. 293 (2), H1223-H1230 (2007).
- Gurev, V., Constantino, J., Rice, J. J., Trayanova, N. Distribution of Electromechanical Delay in the Ventricles:Insights from a 3D Electromechanical Model of the Heart. Biophysical Journal. 99 (3), 745-754 .
- Ten Tusscher, K. H. W. J., Hren, R., Panfilov, A. V. Organization of Ventricular Fibrillation in the Human Heart. Circulation Research. 100 (12), e87-e101 (2007).
- Gima, K., Rudy, Y. Ionic Current Basis of Electrocardiographic Waveforms. Circulation Research. 90, 889-896 (2002).
- Gerstenfeld, E., Dixit, S., Callans, D., Rajawat, Y., Rho, R., Marchlinski, F. Quantitative comparison of spontaneous and paced 12-lead electrocardiogram during right ventricular outflow tract ventricular tachycardia. Journal of Americal College of Cardiology. 41 (11), 2046-2053 (2003).
- Potse, M., Dube, B., Richer, J., Vinet, A., Gulrajani, R. M. A comparison of monodomain and bidomain reaction-diffusion models for action potential propagation in the human heart. IEEE Transactions on Biomedical Engineering. 53 (12), 2425-2435 (2006).
- Peyrat, J. -. M., Sermesant, M., Pennec, X., Delingette, H., Chenyang, X., McVeigh, E. R., Ayache, N. A Computational Framework for the Statistical Analysis of Cardiac Diffusion Tensors: Application to a Small Database of Canine Hearts. IEEE Transactions on Medical Imaging. 26, 1500-1514 (2007).
- Chen, J., Song, S. -. K., Liu, W., McLean, M., Allen, S. J., Tan, J., Wickline, S. A., Yu, X. Remodeling of cardiac fiber structure after infarction in rats quantified with diffusion tensor MRI. American Journal of Physiology - Heart and Circulatory Physiology. 285 (3), H946-H954 (2003).
- Stecker, E. C., Chugh, S. S. Prediction of sudden cardiac death: next steps in pursuit of effective methodology. Journal of Interventional Cardiac Electrophysiolog. 31 (2), 101-107 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados