È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Paziente-specifica modellazione del Cuore: Stima di orientamento delle fibre ventricolari
In questo articolo
Riepilogo
Una metodologia per la stima orientamento delle fibre ventricolari da immagini in vivo di geometrie del cuore del paziente per la modellazione personalizzata è descritto. Validazione della metodologia effettuata con una normale e in mancanza di cuori canini dimostrare che non ci sono differenze significative tra orientamento delle fibre stimati e ha acquisito a livello clinicamente osservabile.
Abstract
Paziente-specifiche simulazioni di cuore (dis) funzione volta a personalizzare la terapia cardiaca sono ostacolati dalla mancanza di tecnologia di imaging in vivo per l'acquisizione clinicamente orientamento delle fibre miocardiche. L'obiettivo di questo progetto era quello di sviluppare una metodologia per la stima orientamento delle fibre cardiache dal vivo in immagini di geometrie del cuore del paziente. Una rappresentazione accurata della geometria ventricolare e orientamento delle fibre è stato ricostruito, rispettivamente, di alta risoluzione risonanza ex vivo strutturale magnetica (RM) e del tensore di diffusione (DT) immagini RM di un cuore umano normale, indicato come l'atlante. Geometria ventricolare di un cuore paziente è stato estratto, tramite segmentazione semiautomatico, da una in vivo la tomografia computerizzata (TC) delle immagini. Utilizzando algoritmi di trasformazione di immagini, la geometria ventricolare atlante è stato deformato in base a quella del paziente. Infine, il campo di deformazione è stato applicato alla fibra atlante Orientationioni per ottenere una stima di orientamento delle fibre del paziente. La precisione delle stime di fibre è stata valutata utilizzando sei cuori in mancanza di normali e tre cani. La differenza media assoluta tra angoli di inclinazione di orientamento delle fibre acquisite e stimata è stata di 15,4 °. Simulazioni computazionali di mappe di attivazione ventricolare e pseudo-ECG in ritmo sinusale e tachicardia ventricolare indicato che non vi sono differenze significative tra orientamento delle fibre stimati e acquisito in generale clinicamente osservabili intuizioni level.The nuove ottenuti dal progetto aprirà la strada per lo sviluppo di paziente-specifici modelli del cuore che può aiutare i medici nella diagnosi personalizzata e le decisioni in materia di interventi di elettrofisiologia.
Introduzione
L'approccio computazionale sta diventando centrale per l'avanzamento della comprensione della funzione del cuore in salute e malattia. State-of-the-art con tutto il cuore i modelli di elettrofisiologia ed elettromeccanica sono attualmente utilizzate per studiare una vasta gamma di fenomeni, come ad esempio la propagazione ventricolare normale, aritmia, defibrillazione, accoppiamento elettromeccanico, e di resincronizzazione cardiaca 1. Tuttavia, per l'approccio computazionale per essere direttamente applicabile in ambiente clinico, è imperativo che i modelli di essere paziente-specifico, cioè i modelli deve essere basata sull'architettura specifico e proprietà elettrofisiologiche o elettromeccanici del cuore malato del paziente. Simulazione con tali modelli sarà di aiuto ai medici di arrivare a decisioni altamente personalizzati per gli interventi elettrofisiologiche così come profilassi, in tal modo notevolmente migliorare l'assistenza sanitaria cardiaco 2-4.
contenuto "> Creazione di modelli realistici cardiaci richiede l'acquisizione della struttura di geometria e fibre di un cuore paziente. orientamento delle fibre determinare direzioni di propagazione elettrica e distribuzioni di deformazione nel cuore, e quindi il loro acquisto è essenziale per la modellazione cardiaca 5, 6. Con recenti progressi nella diagnostica per immagini, è ora possibile ottenere la geometria di un cuore paziente, rimodellamento includingstructural come l'infarto, in vivo con alta risoluzione mediante risonanza magnetica (MRI) e la tomografia computerizzata (CT) tecnologie. Tuttavia, non vi è alcuna metodo pratico per acquisire struttura fibrosa di un cuore paziente in vivo. Diffusion tensor (DT) MRI 7, 8, l'unica tecnica per acquisire orientamento delle fibre del cuore intatto, non è ampiamente disponibile in vivo a causa di alcune limitazioni 9. Breve descrizione degli sforzi precedenti per tradurre DTMRI alla clinica può essere trovato elsewhere 2. Anche se metodologie come basato su regole assegnazione di orientamento delle fibre di offrire alternative a DTMRI, queste metodologie hanno alcune limitazioni gravi 2, 10. Così difficoltà nell'acquisire struttura cardiaca in vivo attualmente in fibra di ostacolare l'applicazione delle simulazioni elettrofisiologici cardiaci ed elettromeccanica in ambiente clinico. L'obiettivo di questa ricerca è stato quello di rivolgersi direttamente a questa esigenza.Abbiamo ipotizzato che orientamenti ventricolari fibra di un cuore può essere previsto con precisione, data la geometria del cuore e un atlante, dove l'atlante è un cuore cui geometria e fibra orientamenti sono disponibili. Di conseguenza, abbiamo usato lo stato dell'arte delle tecniche di sviluppare una metodologia per la stima di orientamento delle fibre cardiache in vivo, e testato l'ipotesi in condizioni normali e in mancanza di ventricoli canino 2. L'idea centrale della nostra metodologia di stima delle fibre è quello di sfruttare similarities in orientamento delle fibre, relativi alla geometria, tra le diverse anime, al fine di avvicinare la struttura fibrosa di un (target) di cuore per cui solo le informazioni sulla geometria è disponibile. Al centro della nostra metodologia di stima è la registrazione della geometria atlante con la geometria di destinazione utilizzando grandi mappatura diffeomorphic metrica deformazione (LDDMM) 11, e l'orientamento delle fibre di morphing atlante utilizzando conservazione delle componenti principali (PPD) 2, 12. La diffeomorphicproperty di garanzie LDDMM che l'atlante non "deployante" stesso durante la deformazione, in modo da preservare le strutture anatomiche integrityof. Figura 1 illustra la pipeline di elaborazione della nostra metodologia. La sezione di testo protocollo § 1 descrive i vari componenti della pipeline dimostrando come la stima può essere eseguita per esempio un paziente. I numeri all'interno alcuni dei blocchi in figura 1 si riferiscono alla corrispondentesottosezioni di cui alla sezione § 1 del testo del protocollo.
Abbiamo valutato le prestazioni della metodologia proposta, che quantifica l'errore di stima, e misurare l'effetto di questo onsimulations errore di elettrofisiologia cardiaca, per la simulazione computazionale locali mappe di attivazione elettrici così come pseudo-elettrocardiogrammi (pseudo-ECG). A causa della indisponibilità dei cuori umani, la valutazione delle prestazioni è stata condotta utilizzando i cuori canine disponibili da studi precedenti 13-15. L'errore di stima è stata calcolata mediante angoli di inclinazione 16, tradizione followingthe di istologia, dove le misure angolari sono sezioni di tessuto performedon che sono tagliati parallelamente al epicardialsurface. Poiché il anglebetween direzione delle fibre ed epicardici piano tangente è generallysmall 17, 18, la perdita di informazioni nel descrivere un fiberdirection interamente con il suo angolo di inclinazione è insignificante. Per il computnazionali di simulazioni, image-based modelli sono stati costruiti come riportato in precedenza 19, 20, e il tessuto cardiaco nei modelli è stata rappresentata sulla base stabiliti tecniche matematiche e dati sperimentali 21-25. Ritmo sinusale è stato simulato attivazione replica proveniente dalla rete Purkinje 26, e la tachicardia ventricolare, da un S1-S2 stimolazione protocollo 27. Pseudo-ECG sono stati calcolati 28 e confrontati utilizzando la deviazione media assoluta (MAD) metrica 29.
Protocollo
1. Fibra Orientamenti Stima
- Acquisire strutturali immagini MRI e DTMRI di un cuore umano adulto normale in diastole, con una risoluzione di 1 mm 3. Uso ImageJ, estrarre il miocardio ventricolare dall'immagine atlante strutturale raccordo, per ciascun asse corto slice, spline chiusa attraverso una serie di punti limite disposte lungo i confini epicardici ed endocardici nella fetta (Figura 2A e 2B Figura). Eseguire il posizionamento dei punti limite manualmente per ogni fetta 10 th nell'immagine. Ottenere i punti limite per le rimanenti fette interpolando linearmente i punti individuati manualmente, utilizzando i MATLAB.Reconstruct orientamento delle fibre del cuore atlante calcolando gli autovettori primari dei DTs nell'immagine DTMRI (Figura 2C).
- Acquisire un'immagine della geometria del cuore del paziente in diastole usando in vivo cardiaca CT o MRI. Reconstruire la geometria paziente cuore dall'immagine simile al modo in cui è stato costruito l'atlante (Figura 3A e 3B Figura). L'immagine paziente deve essere nuovamente campionati prima ricostruzione tale che la risoluzione in piano è di 1 mm 2. Analogamente, il numero di fette per cui punti di riferimento sono raccolte manualmente, e l'intervallo di fuori del piano di interpolazione deve essere regolato in modo che l'immagine segmentata cuore paziente ha uno spessore di strato di 1 mm.
- Deforma l'immagine ventricolare atlante in modo che corrisponda all'immagine paziente geometria in due fasi. Nella prima fase, eseguire una trasformazione affine basato su un insieme di tredici punti monumenti: il ventricolo sinistro (LV) apice, le due ventricolare destro (RV) punti di inserimento alla base, l'inserimento due metà RV punti tra base e apice, e quattro serie di due punti che dividono in modo uniforme per camper e contorni LV epicardici alla base, ed a metà strada tra la base e il vertice (Figura 4A & Figura 4B). Nella seconda fase, la deformazione affine-atlante trasformato ventricoli ulteriore geometria adatta al paziente, utilizzando grandi mappatura diffeomorphic metrica deformazione (LDDMM) (Figura 4C).
- Morph l'immagine DTMRI dell'atlante riposizionando di voxel immagine e riorientamento DTS secondo la matrice di trasformazione affine della corrispondenza e il campo di deformazione della trasformazione LDDMM. Eseguire la ri-orientamento dei DTs con la conservazione di direzioni principali (PPD) metodo.
- Ottenere la stima degli orientamenti fibra del paziente dal morphed immagine atlante DTMRI calcolando l'autovettore principale del DTs (Figura 5).
2. Misurazione di errore di stima
- Acquisire ex vivo strutturale MR e immagini DTMR di sei cuori fallimento normali e tre canini, con una risoluzione di 312,5 × 312,5 × 800 micron 3. Qui, cuore fairichiamo deve essere generata in canini mediante ablazione con radiofrequenza della branca sinistro seguito da 3 settimane di tachypacing a 210 min -1.
- Segmento i ventricoli dal cuore canino simile al cuore umano atlante, come descritto nel § 1.1. Indichiamo ventricoli segmentati dalle normali cuore canino come cuori da 1 a 6, e quelli segmentato dalla mancata cuore canino come cuori da 7 a 9 (Figura 6).
- Ottenere cinque diverse stime orientamenti ventricolari fibra di cuore 1 utilizzando ciascuno dei cuori 2 a 6 come un atlante (Figura 7).
- Stimare orientamento delle fibre per ciascuno dei ventricoli fallimento usando cuore 1 come l'atlante (Figura 8).
- Foreach punto dati di ogni serie di orientamento delle fibre stimati, calcolare l'errore di stima come | θ c-θ un |, dove θ c e θ sono un gli angoli di inclinazione di una stimad acquisito orientamento delle fibre in quel punto, rispettivamente.
- Per ciascun punto di dati in ogni serie di orientamento delle fibre stimati, calcolare l'angolo acuto betweenestimated e acquisito indicazioni fibra in tre dimensioni (3D) mediante prodotto thevector dot.
3. Misurazione degli effetti di errore di stima sulle Simulazioni
- Dal cuore 1, costruire sei modelli, uno con gli DTMRI-acquisiti orientamento delle fibre del cuore 1 (indicato come modello 1), e cinque con cinque serie di dati stimati orientamento delle fibre (modelli 2 a 6). Per ciascuno dei tre omettendo cuore geometrie, costruire due modelli ventricolari, uno con i DTMRI acquisite orientamento delle fibre e l'altra con gli orientamenti in fibra stimati. Qui la risoluzione spaziale dei modelli, calcolata in termini di lunghezza del bordo media delle maglie, dovrebbe essere di circa 600 micron. Indichiamo i modelli di scompenso cardiaco con fibre DTMRI-acquisiti e modelli 7 a 9, e quelli con estimatEd fibre come modelli da 10 a 12.In i modelli, utilizzare la rappresentazione monodominio per descrivere il tessuto cardiaco, con le equazioni che disciplinano:
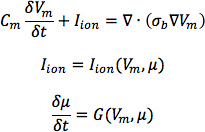
dove σ b è il tensore di conduttività che è calcolato dai tensori conducibilità bidomain come descritto da Potse et al 30; V m è il potenziale transmembrana; C m è la capacità di membrana specifico e io Ion è la densità della corrente transmembrana, che a sua volta dipende V m e un insieme di variabili di stato μ descrivere le dinamiche di flussi ionici attraverso il C membrane.For m, utilizzare un valore di 1 μ F / cm 2. Per i normali modelli σ in cuore canini, utilizzare longitudinalivalori di conducibilità nali e trasversale di 0,34 S / m e 0,06 S / m, rispettivamente. Rappresentare l lon dai Greenstein-Winslow modelli ionici del miociti ventricolari canino. Diminuire le conducibilità elettrica nei modelli canini insufficienza cardiaca ventricolare del 30% (Figura 9).
- Utilizzo del pacchetto software CARP (CardioSolv, LLC), ritmo sinusale con tutti i modelli di simulazione. Indurre tachicardia rientrante ventricolare (VT) nei sei modelli fallimento utilizzando un S1-S2 protocollo di stimolazione. Scegliere il tempo tra S1 e S2 per ottenere l'attività VT sostenuta per 2 secondi dopo la consegna S2. Se VT non viene indotta per qualsiasi S1-S2 temporizzazione, diminuire la conducibilità fino al 70% fino VT è stato indotto (Figura 10).
- Per ogni simulazione, il calcolo pseudo-ECG prendendo la differenza di potenziale tra due punti extracellulari in bagno isotropo che circonda il cuore. Posizionare i due punti vicino alla base del cuore separatoda 18 cm, in modo tale che la linea che li collega è perpendicolare al piano di base-apice del setto come illustrato in Figura 10. Per ogni simulazione con orientamento delle fibre stimati, calcolare la metrica come MAD
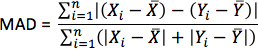
dove X è la forma d'onda ECG della simulazione con orientamento delle fibre stimati, Y è la forma d'onda ECG thecorresponding di simulazione con orientamento delle fibre acquisite, X è il valore medio di X, Y è il valore medio di Y, ed n è la lunghezza di X e Y.
Risultati
Figura 11, AC visualizza visualizzazioni semplificati di stima e DTMRI derivati orientamento delle fibre nei cuori normali e in mancanza. Esame qualitativa mostra che gli orientamenti in fibra stimati allineano bene con DTMRI derivati da quelli. Pannello D illustra, sovrapposto sulla geometria del cuore 1, la distribuzione di errore in angoli di inclinazione cuori normali ", mediato su tutte e cinque le stime. Pannello E mostra la distribuzione media di errore omettendo angoli di inclin...
Discussione
Questa ricerca dimostra quantitativamente che, in assenza di DTMRI, orientamento delle fibre miocardiche di ventricoli normali e mancanza può essere stimata in vivo immagini delle loro geometrie per uso in simulazioni di elettrofisiologia cardiaca. La metodologia proposta è stata dimostrata in vivo dati CT, ma è ugualmente applicabile a immagini RM in vivo della geometria ventricolare, affrontando la mancanza di capacità di acquisire direttamente orientamento delle fibre del paziente. E &#...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Drs. Raimond Winslow, Elliot McVeigh, Helm e Patrick presso la Johns Hopkins University per fornire il set di dati ex vivo online.This ricerca è stata sostenuta dal National Institutes of Health di sovvenzione R01-HL082729, e la National Science Foundation concessione CBET-0933029.
Materiali
| Name | Company | Catalog Number | Comments |
| LDDMM | Johns Hopkins University | http://cis.jhu.edu/software/lddmm-volume/index.php | |
| MATLAB | MathWorks, Inc. | R2011b | http://www.mathworks.com/products/matlab/ |
| ImageJ | National Institutes of Health | http://rsbweb.nih.gov/ij/ | |
| Tarantola | CAE Software Solutions | http://www.meshing.at/Spiderhome/Tarantula.html | |
| CARP | CardioSolv | http://cardiosolv.com/ | |
| Immagini Canine | Johns Hopkins University | http://www.ccbm.jhu.edu/research/DTMRIDS.php |
Riferimenti
- Trayanova, N. Whole Heart Modeling: Applications to Cardiac Electrophysiology and Electromechanics. Circulation Research. 108, 113-128 .
- Vadakkumpadan, F., Arevalo, H., Ceritoglu, C., Miller, M., Trayanova, N. Image-Based Estimation of Ventricular Fiber Orientations for Personalized Modeling of Cardiac Electrophysiology. IEEE Transactions on Medical Imaging. 31 (5), 1051-1060 .
- Vadakkumpadan, F., Gurev, V., Constantino, J., Arevalo, H., Trayanova, N., Kerckhoffs, R. Modeling of Whole-Heart Electrophysiology and Mechanics: Towards Patient-Specific Simulations. Patient-Specific Modeling of the Cardiovascular System: Technology-Driven Personalized Medicine. , 145-165 (2010).
- Buxton, A. E., Lee, K. L., DiCarlo, L., Gold, M. R., Greer, G. S., Prystowsky, E. N., O'Toole, M. F., Tang, A., Fisher, J. D., Coromilas, J., Talajic, M., Hafley, G. Electrophysiologic testing to identify patients with coronary artery disease who are at risk for sudden death. Multicenter Unsustained Tachycardia Trial Investigators. The New England Journal of Medicine. 342 (26), 1937-1945 (2000).
- Wei, D., Okazaki, O., Harumi, K., Harasawa, E., Hosaka, H. Comparative simulation of excitation and body surface electrocardiogram with isotropic and anisotropic computer heart models. IEEE Transactions on Biomedical Engineering. 42 (4), 343-357 (1995).
- Leon, L. J., Horacek, B. M. Computer model of excitation and recovery in the anisotropic myocardium. II. Excitation in the simplified left ventricle. Journal of Electrocardiology. 24 (1), 17-31 (1991).
- Rohmer, D., Sitek, A., Gullberg, G. T. Reconstruction and Visualization of Fiber and Laminar Structure in the Normal Human Heart from Ex Vivo Diffusion Tensor Magnetic Resonance Imaging (DTMRI) Data. Investigative Radiology. 42 (11), 777-789 (2007).
- Daubert, J. P., Zareba, W., Hall, W. J., Schuger, C., Corsello, A., Leon, A. R., Andrews, M. L., McNitt, S., Huang, D. T., Moss, A. J., Investigators, M. I. S. Predictive value of ventricular arrhythmia inducibility for subsequent ventricular tachycardia or ventricular fibrillation in Multicenter Automatic Defibrillator Implantation Trial (MADIT) II patients. Journal of Americal College of Cardiology. 47 (1), 98-107 (2006).
- Sosnovik, D. E., Wang, R., Dai, G., Reese, T. G., Wedeen, V. J. Diffusion MR tractography of the heart. Journal of Cardiovascular Magnetic Resonance. 11 (1), 47-61 (2009).
- Sundar, H., Shen, D., Biros, G., Litt, H., Davatzikos, C. Estimating myocardial fiber orientations by template warping. Proc. IEEE International Symposium on Biomedical Imaging. , 73-76 (2006).
- Beg, M. F., Helm, P. A., McVeigh, E., Miller, M. I., Winslow, R. L. Computational Cardiac Anatomy Using MRI. Magnetic Resonance in Medicine. 52 (5), 1167-1174 (2004).
- Alexander, D. C., Pierpaoli, C., Basser, P. J., Gee, J. C. Spatial Transformations of Diffusion Tensor Magnetic Resonance Images. IEEE Transactions on Medical Imaging. 20, 1131-1139 (2001).
- Helm, P. A., Younes, L., Beg, M. F., Ennis, D. B., Leclercq, C., Faris, O. P., McVeigh, E., Kass, D., Miller, M. I., Winslow, R. L. Evidence of Structural Remodeling in the Dyssynchronous Failing Heart. Circulation Research. 98, 125-132 (2006).
- Helm, P., Beg, M. F., Miller, M., Winslow, R. Measuring and mapping cardiac fiber and laminar architecture using diffusion tensor MR imaging. Annals of the New York Academy of Sciences. 1047, 296-307 (2005).
- Helm, P. A., Tseng, H. -. J., Younes, L., McVeigh, E. R., Winslow, R. L. Ex vivo 3D diffusion tensor imaging and quantification of cardiac laminar structure. Magnetic Resonance in Imaging. 54, 850-859 (2005).
- Scollan, D. F., Holmes, A., Winslow, R., Forder, J. Histological validation of myocardial microstructure obtained from diffusion tensor magnetic resonance imaging. American Journal of Physiology - Heart and Circulatory Physiology. 275 (6), H2308-H2318 (1998).
- Lombaert, H., Peyrat, J., Croisille, P., Rapacchi, S., Fanton, L., Cheriet, F., Clarysse, P., Magnin, I., Delingette, H., Ayache, N. Human Atlas of the Cardiac Fiber Architecture: Study on a Healthy Population. IEEE Transactions on Medical Imaging. 31 (7), 1436-1447 (2012).
- Streeter, D. D. . Gross morphology and fiber geometry of the heart. , (1979).
- Vadakkumpadan, F., Rantner, L. J., Tice, B., Boyle, P., Prassl, A. J., Vigmond, E., Plank, G., Trayanova, N. Image-Based Models of Cardiac Structure with Applications in Arrhythmia and Defibrillation Studies. Journal of Electrocardiology. 42, 151.e1-151.e10 (2009).
- Plank, G., Zhou, L., Greenstein, J. L., Plank, G., Zhou, L., Greenstein, J. L., Cortassa, S., Winslow, R. L., O'Rourke, B., Trayanova, N. A. From mitochondrial ion channels to arrhythmias in the heart: computational techniques to bridge the spatio-temporal scales. Philosophical Transactions Series A, Mathematical, Physical, and Engineering Sciences. 366 (1879), 3381-3409 (2008).
- Roberts, D. E., Scher, A. M. Effect of tissue anisotropy on extracellular potential fields in canine myocardium in situ. Circulation Research. 50, 342-351 (1982).
- Greenstein, J., Wu, R., Po, S., Tomaselli, G. F., Winslow, R. L. Role of the Calcium-Independent Transient Outward Current I(to1) in Shaping Action Potential Morphology and Duration. Circulation Research. 87, 1026-1033 (2000).
- Winslow, R., Rice, J., Jafri, S., Marbán, E., O'Rourke, B. Mechanisms of altered excitation-contraction coupling in canine tachycardia-induced heart failure, II: model studies. Circulation Research. 84 (5), 571-586 (1999).
- Akar, F., Nass, R., Hahn, S., Cingolani, E., Shah, M., Hesketh, G., DiSilvestre, D., Tunin, R., Kass, D., Tomaselli, G. Dynamic Changes in Conduction Velocity and Gap Junction Properties During Development of Pacing-Induced Heart Failure. American Journal of Physiology - Heart and Circulatory Physiology. 293 (2), H1223-H1230 (2007).
- Gurev, V., Constantino, J., Rice, J. J., Trayanova, N. Distribution of Electromechanical Delay in the Ventricles:Insights from a 3D Electromechanical Model of the Heart. Biophysical Journal. 99 (3), 745-754 .
- Ten Tusscher, K. H. W. J., Hren, R., Panfilov, A. V. Organization of Ventricular Fibrillation in the Human Heart. Circulation Research. 100 (12), e87-e101 (2007).
- Gima, K., Rudy, Y. Ionic Current Basis of Electrocardiographic Waveforms. Circulation Research. 90, 889-896 (2002).
- Gerstenfeld, E., Dixit, S., Callans, D., Rajawat, Y., Rho, R., Marchlinski, F. Quantitative comparison of spontaneous and paced 12-lead electrocardiogram during right ventricular outflow tract ventricular tachycardia. Journal of Americal College of Cardiology. 41 (11), 2046-2053 (2003).
- Potse, M., Dube, B., Richer, J., Vinet, A., Gulrajani, R. M. A comparison of monodomain and bidomain reaction-diffusion models for action potential propagation in the human heart. IEEE Transactions on Biomedical Engineering. 53 (12), 2425-2435 (2006).
- Peyrat, J. -. M., Sermesant, M., Pennec, X., Delingette, H., Chenyang, X., McVeigh, E. R., Ayache, N. A Computational Framework for the Statistical Analysis of Cardiac Diffusion Tensors: Application to a Small Database of Canine Hearts. IEEE Transactions on Medical Imaging. 26, 1500-1514 (2007).
- Chen, J., Song, S. -. K., Liu, W., McLean, M., Allen, S. J., Tan, J., Wickline, S. A., Yu, X. Remodeling of cardiac fiber structure after infarction in rats quantified with diffusion tensor MRI. American Journal of Physiology - Heart and Circulatory Physiology. 285 (3), H946-H954 (2003).
- Stecker, E. C., Chugh, S. S. Prediction of sudden cardiac death: next steps in pursuit of effective methodology. Journal of Interventional Cardiac Electrophysiolog. 31 (2), 101-107 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon