Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Screening siRNA para identificar ubiquitina y reguladores del sistema ubiquitina-como de las vías biológicas en células de mamífero en cultivo
En este artículo
Resumen
Aquí se describe una metodología para llevar a cabo un siRNA dirigidos pantalla "ubiquitome" para identificar nuevos ubiquitina y reguladores-ubiquitina de la respuesta celular mediada HIF1A a la hipoxia. Esto puede ser adaptado a cualquier vía biológica, donde una lectura robusta de la actividad del indicador está disponible.
Resumen
Modificación posterior a la traducción de las proteínas con ubiquitina y ubiquitina-como las moléculas (Ubls) se está convirtiendo en una red de señalización celular dinámica que regula diversos procesos biológicos, incluyendo la respuesta a la hipoxia, proteostasis, la respuesta al daño del ADN y la transcripción. Para entender mejor cómo UBLs regulan las vías pertinentes a la enfermedad humana, hemos recopilado un siRNA humana "ubiquitome" biblioteca que consta de 1.186 piscinas siRNA dúplex dirigidos todos conocidos y predijo componentes de las vías del sistema de UBL. Esta biblioteca se puede criba frente a una gama de líneas celulares que expresan los reporteros de diversas vías biológicas para determinar qué componentes UBL actúan como reguladores positivos o negativos de la vía en cuestión. Aquí se describe un protocolo que utiliza esta biblioteca para identificar ubiquitome-reguladores de la respuesta celular mediada HIF1A a la hipoxia mediante un indicador de luciferasa basado en la transcripción. Una etapa inicial de desarrollo de ensayosse lleva a cabo para establecer los parámetros de detección adecuados de la línea celular antes de realizar la pantalla en tres etapas: detección / deconvolución primaria, secundaria y terciaria. El uso de objeto a través de siRNA bibliotecas de todo el genoma se está convirtiendo cada vez más popular, ya que ofrece la ventaja de la cobertura del sólo miembros de la vía con la que los investigadores están más interesados. A pesar de las limitaciones inherentes de la detección siRNA, en particular, los falsos positivos causados por siRNA fuera de objetivo efectos, la identificación de auténticos nuevos reguladores de las vías en cuestión superan estas deficiencias, que pueden ser superadas mediante la realización de una serie de experimentos de control llevadas a cabo cuidadosamente.
Introducción
La modificación de proteínas con ubiquitina y ubiquitina-como las moléculas (Ubls) representa un sistema bioquímico expansiva que regula diversos procesos biológicos y las respuestas al estrés. La unión covalente de UBLs a sus proteínas diana puede tener diversos resultados que regulan la estabilidad, la localización, función o interactoma del sustrato 1. Los pasos enzimáticos subyacentes UBL modificación se establecieron por primera vez para la ubiquitina, y ahora sirven como paradigma para la modificación con la mayoría de UBLs, incluyendo SUMO, NEDD8, ISG15 y FAT10. Para que se produzca la modificación, el grupo carboxilato del motivo diglicina UBL se activa por primera vez por una enzima de activación E1 para formar un tiol de alta energía que se transfiere a la cisteína del sitio activo de una enzima de conjugación E2. El E2 entonces interactúa con un E3 ligase sustrato determinado para mediar la transferencia de la LBM en (generalmente) un residuo de lisina objetivo la creación de una cadena ramificada (isopeptídico) vinculación 2. Las rondas sucesivas de modificación pueden ocurrir para construir cadenas isopeptídicos sobre el sustrato, que para la ubiquitina puede ocurrir a través de cualquiera de sus siete lisinas, o a través de su metionina N-terminal para crear cadenas de ubiquitina lineales. Estas modificaciones forman topologías discretas con diversos fines, tales como la creación de nuevos motivos de interacción y dirigir proteínas para la degradación antes de la remoción UBL por proteasas especializados. En el caso de la ubiquitina hay dos enzimas E1, E2 30-40 enzimas de conjugación, al menos 600 ligasas E3 y aproximadamente 100 enzimas deubiquitylating (DUBS). Mientras que las vías son menos expansiva para las otras 10 o así UBLs, la complejidad general ubiquitome ofrece gran diversidad en el resultado biológico de una modificación particular UBL. Sin embargo, mientras que los principales avances de la biología UBL se han hecho, las funciones celulares precisas de la mayoría de estos componentes ubiquitome siguen siendo desconocidos.
El uso de short interfering ácido ribonucleico (siRNAs) ha emergido como una herramienta poderosa en la genética inversa, debido a la capacidad de los siRNAs para dirigirse específicamente a los ARNm celulares para la destrucción, permitiendo que el papel de los genes individuales que se examinó en diferentes contextos biológicos 3. Pantallas de todo el genoma se han utilizado para identificar y validar nuevos reguladores de muchos procesos celulares, y han creado una gran cantidad de datos útiles al alcance de la comunidad científica en general. Sin embargo, mientras que las pantallas de todo el genoma han demostrado ser extremadamente útil, pantallas específicas son cada vez más populares, ya que son más baratos, más rápidos implican menos la gestión de datos y un informe sólo sobre los miembros del genoma en el que el investigador está más interesado. Por lo tanto, para entender mejor los componentes de la familia los procesos celulares UBL están involucrados en, hemos recopilado una biblioteca humana siRNA dirigido todos los conocidos y predijo componentes del ubiquitome. Esto incluye los UBLs, la activación de enzimas E1, E2 conjugaciónenzimas, ligasas E3, dominio de unión a ubiquitina (UBD) que contienen proteínas y DUBs. Esta biblioteca se puede utilizar para la pantalla contra una amplia gama de líneas celulares indicadoras de problemas biológicos distintos, permitiendo así la identificación imparcial de componentes UBL novedosos que rigen estas vías.
El siguiente protocolo describe cómo realizar una rigurosa siRNA dirigidos ubiquitome pantalla para identificar nuevos reguladores de la respuesta HIF1A dependiente a la hipoxia. Bajo la tensión de oxígeno normal, HIF1A está sujeta a hidroxilación prolilo que hace que sea reconocida y destinada a la degradación por el Von Hippel Lindau (VHL) E3 ligase complejo 4. La hipoxia inhibe la hidroxilación prolil que conduce a la estabilización de HIF1A y su posterior unión a elementos de respuesta a la hipoxia (HRE) para conducir la expresión de genes. Aquí, se describe una pantalla utilizando células de osteosarcoma U20S de forma estable que expresa luciferasa de luciérnaga bajo el control de tres copias en tándem de la Hypoxia Elemento de Respuesta (células U20S-HRE) 5. Este protocolo se puede adaptar para cualquier vía biológica si un robusto lectura de salida de la actividad del indicador es alcanzable y se puede acoplar con controles positivos y negativos apropiados.
Protocolo
1. Fase de Desarrollo de Ensayos
Nota: antes de iniciar la pantalla de siRNA, una etapa de desarrollo de ensayos es fundamental establecer parámetros importantes para el cribado con el reportero línea celular. Es esencial invertir esfuerzo importante en esta etapa, ya que será la base del futuro éxito de la pantalla.
- Para caracterizar la hipoxia-capacidad de respuesta de U20S-HRE reportero línea celular, creciendo 2 x 75 cm 2 frascos de células U20S-HRE a 80-90% de confluencia en medio de Eagle modificado de Dulbecco (DMEM) suplementado con 10% de FBS.
- Aspirar los medios de comunicación de un matraz de células, se lava dos veces con 10 ml de PBS y separar las células mediante la adición de 2 ml de solución de tripsina-EDTA 0,05% e incubando a 37 ° C durante 5 min. Resuspender las células en 8 ml de DMEM 10% de SFB y la pipeta hacia arriba y hacia abajo para crear una suspensión celular homogénea.
- Pipetear 20 l de la suspensión a una cámara de recuento de células, por duplicado, e inserte la cámara en un cou células automatizadonter. Haga clic en "Mostrar Imagen" en el software del contador de células y asegurar que las células están en foco. Haga clic en "Cuenta" y tome nota de la densidad celular. Repita esto para el otro duplicado y calcular la densidad celular promedio. Diluir las células con DMEM 10% de FBS a una concentración de 60.000 células / ml.
- Con una pipeta multicanal, añadir 100 ml (6.000 células) de las células diluidas a los mismos tres columnas (por ejemplo A5-H5, H6-A6 y A7-H7) de 5 placas de 96 pocillos de ensayo de paredes blancas estériles, y la transferencia a un humidificado 37 ° C incubadora a 5% de CO 2 durante la noche. Nota: estas placas se utilizarán para determinar la salida de la luciferasa hipoxia que dependen de una serie de exposiciones de hipoxia: 0 h, 2 h, 6 h, 10 hy 24 h.
- Al final del día siguiente, anote la hora y agregue la placa de la hipoxia de 24 horas a la estación de trabajo conjunto hipoxia al 1% de oxígeno. A la mañana siguiente, calcule el tiempo de 10 horas, 6 horas y 2 horas antes de que la placa de 24 horas se debe a ser eliminado frOM la estación de trabajo hipoxia. En estos momentos, añadir el 10 horas, 6 horas y 2 placas hr a la estación de trabajo de la hipoxia.
- Preparar 30 ml de 2x tampón de lisis de luciferasa / ensayo combinado (50 mM de Tris fosfato de pH 7,8; 16 mM de MgCl 2; DTT 2 mM; 2% de Triton-X-100; 30% de glicerol; mM de ATP 1; 1% de BSA; 0,25 mM luciferina y 8 mM Na 4 P 2 O 7). Nota: agregar los componentes en el orden de la lista y permitir que la BSA al menos 30 minutos para disolver antes de la adición de luciferina y Na 4 P 2 O 7.
- Eliminar todas las placas de la estación de trabajo de la hipoxia en el momento apropiado. Añadir 100 l de 2x tampón de lisis de luciferasa / ensayo a los pocillos apropiados de la placa de ensayo y cubrir con film transparente. Agitar las placas en un agitador de placas durante 10 min a 500 rpm para lisar completamente las células.
- Transfiera la pila de placas de 96 pocillos a una placa de bastidor automatizado luminómetro. Bajo el menú "Protocolos", seleccione "abs 595" y poner de relieve todo wells para ser leídos en el mapa de placa en la pantalla del menú "bien la selección". Haga clic en "Ejecutar" para medir la luminiscencia de cada pocillo y luego calcular el promedio de lectura de cada placa. Calcular la hipoxia que dependen plegable aumento de la actividad del indicador dividiendo el promedio de lectura de cada placa hipoxia por la lectura promedio de la normoxia (0 hr hipoxia) Placa de control. Nota: es esencial observar una robusta respuesta de luciferasa dependiente de hipoxia de ser adecuado para el cribado. A veces más metro setenta y cinco, en la actividad del indicador se considera excelente.
- Crear una placa de control maestro que contiene cuatro (HIF1A siRNA) réplicas (pozos A1-D1), cuatro bajo control (FIH1 siRNA) replica (pozos A12-D12) de alto control, cuatro almacenamientos intermedios sólo controlar repeticiones (pozos E1-H1) y cuatro réplicas de control de siRNA no objetivo (E12-H12), donde todos los grupos de siRNA se encuentran a una concentración de 200 nM. Nota: Los controles de alta y baja se establecen sobre la base de los reguladores de la vía conocidos que aumentan y decrfacilitar la respuesta a la hipoxia, respectivamente. Alternativamente, el desarrollo del ensayo usando un subconjunto de la biblioteca se puede utilizar para identificar los controles adecuados.
- Con una pipeta multicanal, transferir 10 l de cada siRNA control a un blanco placa de ensayo amurallada estéril. Prepare la mezcla de transfección de reactivos de transfección de 0,1 l en 10 l medio reducido de suero (1:100) por pocillo, y la transferencia de 10 l de mezcla de transfección para cada pocillo de control. Pipetear hacia arriba y hacia abajo brevemente para mezclar, y dejar la placa en reposo durante 20 a 60 minutos para permitir la formación de complejos de transfección.
- Preparar una suspensión de células de 75.000 células / ml con el segundo recipiente con células U20S-HRE. Usando una pipeta multicanal, añadir 80 l (6.000 células) de la suspensión celular a cada mezcla de transfección. Colocar la placa en un humidificado incubadora a 37 º C con CO2 al 5% durante 24 horas, luego se transfieren a una estación de trabajo hipoxia durante 24 horas más y realizar ensayos de luciferasa como se describe en los pasos 1.6-1.8.
- Calcular el factor Z para los controles alto y bajo utilizando la fórmula Z = 1 - [3 x (desviación estándar de alta Control + desviación estándar de bajo el control) / (media de control de alta - media de baja de control)]. Nota: un valor de entre 0,5 a 1 indica un excelente ensayo y debe ser esforzó para.
2. Pantalla Principal
Nota: una vez que estas condiciones básicas de la etapa de desarrollo del ensayo están en su lugar, la pantalla principal se puede realizar por triplicado en formato de 96 pocillos de la placa usando el siguiente protocolo.
- Grow 7 x 75 cm 2 frascos de células U20S-HRE a 80-90% de confluencia en DMEM suplementado con 10% v / v de suero bovino fetal (FCS).
Nota: Los siguientes pasos 2.2 a 2.13 deben llevarse a cabo en el Día 1. - Iniciar réplica 1 preparando una placa de control maestro que contiene 200 l de cada control como se describe en el paso 1.9 y descongelación a cabo una serie de diluciones del ubiquitome l siRNAibrary por un mínimo de 30 min. Nota: Cada pocillo contiene una piscina de 4 siRNAs reunidos en 17 placas de 96 pocillos que con las columnas 1 y 12 se deja vacío para los controles.
- En una campana de flujo laminar, la etiqueta (o de código de barras) 17 de paredes blancas placas de ensayo estériles con tapas de los números 1-17, correspondiente a cada placa de la serie de siARN.
- Centrifugar la placa de control maestro y el ARNsi ubiquitome brevemente biblioteca (1 min a 2.000 xg) para asegurar que todos los siRNAs se acumulan en la parte inferior del pozo.
- Utilice un dispensador de líquido automatizado para "estampar" robóticamente 10 l de cada control de la placa de control principal en las 17 placas de ensayo, usando la misma pila de punta de 96 pocillos para cada plato.
- El uso de una pila fresca de 96 pocillos punta para cada una de las 17 placas de biblioteca, la transferencia de 10 l de cada ubiquitome ARNsi a su placa de ensayo correspondiente usando un dispensador de líquido automatizado.
- Preparar 40 ml de reactivo de transfección para las 17 placas de ensayo utilizando los ES relacióntablished en 1.10. Nota: esto incluye un adicional de 20 ml para tener en cuenta el "volumen muerto" en el dispensador de la célula. El "volumen muerto" se refiere al volumen de líquido continuamente presente dentro de la red de tubos en el dispensador de células.
- Utilice un dispensador de células automatizado para transferir 10 l de reactivo de transfección para cada pocillo en las placas de ensayo 17. Brevemente agitar las placas de ensayo en un agitador (1 min a 500 rpm) para permitir la mezcla completa de siRNA y reactivo de transfección, a continuación, dejar todavía a temperatura ambiente durante 20-60 minutos para permitir la ARNsi: la formación de complejos reactivo de transfección.
- Lavar dos matraces de 75 cm2 de células U20S-HRE dos veces con 10 ml de PBS y separar con 2 ml de tripsina-EDTA a 37 ° C durante 5 min. Añadir 8 ml de DMEM 10% de FBS a cada matraz y la pipeta arriba y abajo varias veces para crear una suspensión celular homogénea. Combinar las células de ambos matraces y transferir a un tubo de plástico de 50 ml estéril.
- Calcular la concentración de células pipeteando20 células mu l por duplicado a una cámara de recuento de células y calcular la concentración celular media de dos lecturas de un contador automático de células como se describe en el paso 1.3.
- Preparar 155 ml de suspensión de células por dilución con DMEM 10% de FBS a una concentración de 75.000 células por ml en un recipiente de plástico estéril. Nota: este volumen es suficiente para 17 placas y se incluye un adicional de 25 ml para el volumen muerto en el dispensador de células.
- Añadir un agitador magnético estéril a la suspensión celular y el lugar en un agitador instalado en el interior de la campana de flujo laminar para limitar la formación de grumos de células. Equilibrar el dispensador automático de células con la suspensión celular y configurarlo para dispensar 80 l de células por pocillo (6.000 células).
- Coloque cada una de las 17 placas, a su vez, en el dispensador automático de células y dispensar células. Registre el tiempo y las placas de la pila en grupos de 5 a continuación, coloque las pilas en un húmedo 37 ° C incubadora a 5% de CO 2 durante 24 horas. Nota: la concentración final depiscina siRNA será 20 nM en 100 l de volumen total.
Nota: Los siguientes pasos 02.14 a 02.15 deben llevarse a cabo en el Día 2. - Inicie la segunda réplica en el día 2, siguiendo el procedimiento indicado en el Día 1 para la replicación 1 (02.02 a 02.13).
- Después de 24 horas de la transfección, las placas de transferencia de réplica 1 en un ambiente estéril a una estación de trabajo hipoxia fijaron en 1% de oxígeno y dejan durante 24 horas para inducir el reportero EDH.
Nota: Los siguientes pasos 02.16 a 02.20 deben llevarse a cabo en el Día 3. - Inicie la tercera réplica, siguiendo el procedimiento indicado en el Día 1 para la replicación 1 (2.2 a 2.13).
- Después de 24 horas de la transfección, las placas de transferencia de réplica 2 en un ambiente estéril a una estación de trabajo hipoxia fijaron en 1% de oxígeno y dejan durante 24 horas para inducir el reportero EDH.
- Dos horas antes de replicar 1 placas son para ser retirado de la estación de trabajo de la hipoxia, se preparan 200 ml de tampón de lisis 2x luciferasa / ensayo como se describe en 1.6. Nota: este volumen encluye un adicional de 20 ml para asegurar el depósito de regulación se mantiene bien cubierto.
- Retire replicar 1 placas de ensayo de la estación de trabajo de la hipoxia después de la exposición hipoxia 24 horas y el uso de un dispensador de líquido automatizado, añadir 100 l de tampón de lisis 2x luciferasa / ensayo a cada placa de ensayo y cubrir con film transparente.
- Agitar las placas en un agitador de células durante 10 min a 500 rpm para lisar completamente las células. Transfiera cada placa a su vez a un lector de placas luminómetro y luminiscencia registro tal como se describe en el paso 1.8.
Nota: Los siguientes pasos 02.21 a 02.22 deben llevarse a cabo en el Día 4. - Después de 24 horas de la transfección, las placas de transferencia de réplica 3 en un ambiente estéril a una estación de trabajo hipoxia fijaron en 1% de oxígeno y dejan durante 24 horas para inducir el reportero EDH.
- Lea la réplica 2 placas siguiendo los pasos utilizados para la replicación 02.18 a 02.20 1.
Nota: el siguiente paso 2.23 debe llevarse a cabo en el Día 5. - Lea las réplicas de 3 platos siguiendo02.18-02.20 paso utilizado para la réplica 1.
- Compilar los 3 repeticiones de la pantalla principal, y calcular el factor Z de cada placa utilizando la fórmula dada en 1.12. Nota: si cualquier placa tiene un factor Z de menos de 0,5, considerar la repetición de esta placa para mejorar la calidad de los datos.
- Calcular el coeficiente de varianza (CV) para el alta y los controles bajos mediante el uso de la siguiente fórmula: 100 x desviación estándar del control / promedio de control. Nota: CV debe ser lo más bajo posible, es razonable esperar que la variación del 10-15%. Si la variación es significativamente mayor, considere la repetición de la placa.
- Calcular ciento activación de cada siRNA por placa utilizando la siguiente fórmula: [(media de alto control - score siRNA) / (media de alto control - media de baja control)] x 100 Además, el cálculo de las especies no objetivo (NT. ) veces de cada siRNA utilizando la fórmula [Sample data / media de NT]. Por tanto la activación por ciento y NT veces, calcular la media de los tres replicaates y compilar los valores.
- Utilizando los datos en bruto, el cálculo de Posición Coeficiente de correlación de Spearman (SRCC) para determinar cómo de cerca las tres placas replicadas se correlacionan con la fórmula:
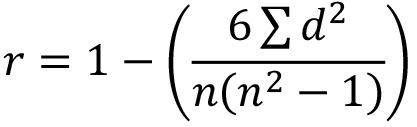
donde r = SRCC; d = diferencia entre los dos números en cada par de filas y n = número de pares de datos. Nota: una buena correlación entre las réplicas de la placa dará valores cercanos a 1 Si una réplica muestra bajo SRCC contra los otros 2 platos, investigar más a fondo y considerar la repetición de ese plato.. - Decida qué siRNAs son de suficiente interés para la investigación secundaria (hasta el 80). Emplear puntos de corte definidos por el usuario (por ejemplo, la selección de todos los siRNAs que muestran menos del 5% o más del 95% de activación por ciento, o menos de 0,5 NT veces o más de 1,5 veces el NT) o aplicar la bioinformática o razonamiento basado en el conocimiento para buscar grupos de golpea comprendidos en la misma vía de UBL. Nota:normalmente, una combinación de estos enfoques determina que se toman siRNA en el cribado secundaria.
3. Pantalla secundaria
Nota: una pantalla secundaria de confirmación se lleva a cabo sobre la base de un máximo de 80 siRNAs de interés desde la pantalla principal. Este número se puede llevar a cabo convenientemente con cada réplica chapada en una sola placa de 96 pocillos con controles completos como por la pantalla principal (ver 2.2). Es muy útil para confirmar que los reguladores identificados en la pantalla principal de manera reproducible provocan el mismo fenotipo, y esto le ayudará a perfeccionar las decisiones de triage en el que golpea debe realizarse a través de la pantalla final terciaria / deconvolución.
- Grow 1 x 75 cm 2 frasco de células U20S-HRE a 80-90% de confluencia en DMEM con 10% de SFB.
- Anote el número de la placa y la ubicación de los éxitos de la pantalla principal, y planificar su nueva ubicación en la cereza pantalla secundaria recogió la placa principal, dejando las columnas 1, unad 12 libre para los controles. Preparar la placa de control maestro como en 1.9 pero con un volumen total de cada control de 50 l.
- Descongele una segunda serie de dilución de la biblioteca ubiquitome (reservado para piscinas siRNA individuales cherry-picking) por un mínimo de 30 min. Centrifugar brevemente placas (1 min a 2.000 xg) a continuación, añadir 50 l de los siRNAs pantalla secundaria a sus nuevas ubicaciones de la placa en la cereza recogido placa maestra.
- Llevar a cabo la pantalla secundaria utilizando el mismo protocolo básico descrito para la pantalla primaria con las siguientes modificaciones: (a) ejecutar todas las tres repeticiones en el mismo día en una placa de ensayo por repetición y (b) ajustar los volúmenes de reactivos en consecuencia.
4. Terciario Deconvolution Screen /
Nota: el cribado terciario o deconvolución se realiza en un máximo de 20 siRNAs de la pantalla secundaria. Este paso es examinar el efecto de la caída en el uso de cada siRNA individuo del poo originalesl de cuatro. Normalmente, al menos, dos dúplex siRNA individuales de cada grupo deben ilícito el mismo fenotipo que tener un grado razonable de confianza de que el fenotipo observado no se debe a siRNA fuera de objetivo efectos. Para aumentar aún más la confianza en esta etapa, dúplex siRNA dirigidos individuales adicionales del gen en cuestión pueden ser diseñados y probados por su capacidad de provocar el fenotipo dado. Los resultados de estos experimentos pueden usarse entonces para calcular la puntuación H, donde H = 0,6 o más (es decir, donde al menos 3 de 5 siRNAs individuales obtener la fenotipo) se considera aceptable 6.
- Crecer un 75 cm 2 frasco de células U20S-HRE a 80-90% de confluencia en DMEM con 10% de SFB.
- Crear un mapa de la placa de la pantalla superior mediante la planificación de la ubicación de los cuatro dúplex siRNA individuales de cada golpe en la placa principal de pantalla superior, dejando las columnas 1 y 12 libre para los controles. Preparar la placa principal de control como en la 1.9 pero con un totalvolumen de cada control de 50 l.
- Descongelar las placas deconvolución / siRNA individual y añadir 50 l de los siRNAs individuales (200 nM) en cada pocillo de la placa maestra pantalla terciario según el mapa de la placa (4.2) para permitir tres x 10 repeticiones ly un cómodo 20 l exceso .
- Realizar la pantalla superior utilizando el mismo protocolo básico esbozado para la pantalla principal con las siguientes modificaciones: (a) ejecute las tres repeticiones en el mismo día en una placa de ensayo por repetición y (b) ajustar los volúmenes de reactivos en consecuencia
Nota: Lo ideal sería que el umbral para ARNsi dúplex individuales debe fijarse en la misma rigurosidad de corte como para la piscina. Sin embargo, puede ser aceptable para relajar el umbral en un 10-20% de siRNAs individuales, especialmente donde al menos otro dúplex cae dentro del umbral establecido. Es cojinete vale la pena en cuenta que el efecto individual de siRNAs puede ser menor que la dela piscina, y por el contrario, en algunos casos siRNAs individuales pueden mostrar un mayor efecto sobre el fenotipo celular en aislamiento que cuando existe en la piscina (incluso cuando la concentración se explica).
Resultados
Antes de la detección, se establece la hipoxia-respuesta de las células U20S-HRE. Células U20S-HRE expresan un constructo indicador que consiste en la luciferasa de luciérnaga fundida aguas abajo de tres copias en tándem del elemento de respuesta a la hipoxia, que está obligado por el heterodímero HIF1A/HIF1B tras la exposición a la hipoxia (Figura 1A). Las células se colocan en una estación de trabajo hipoxia para una serie de veces para establecer que la exposición hipoxia produce la respue...
Discusión
El uso de pantallas de siRNA en todo el genoma de células de mamíferos se ha demostrado ser de gran valor en la identificación de nuevos reguladores de las vías biológicas distintas. Aquí, hemos descrito el uso de una pantalla de ARNsi ubiquitome dirigido a identificar reguladores de la respuesta celular mediada por HIF1A a la hipoxia. Pantallas dirigidas están volviendo cada vez más atractivo, ya que son generalmente más baratos, más rápidos, más fáciles de manejar y un informe sólo sobre los componentes ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Wellcome Trust, GlaxoSmithKline (GSK) y el Instituto Escocés de Señalización Celular (ahora parte de la fosforilación de la proteína MRC y la unidad Ubiquitylation).
Materiales
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
Referencias
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados