このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
培養哺乳動物細胞内の生物学的経路のユビキチンおよびユビキチン様システムレギュレータを識別するために、siRNAのスクリーニング
要約
ここでは、低酸素にHIF1A媒介細胞応答の新規ユビキチンおよびユビキチン様レギュレータを識別するための画面」ubiquitome」を標的としたsiRNAを実行するための方法論を説明します。これはレポーター活性のうち、堅牢な読み取りが可能な任意の生物学的経路に適合させることができる。
要約
ユビキチンおよびユビキチン様分子(のUbl)を有するタンパク質の翻訳後修飾は、低酸素応答、タンパク質恒常性、DNA損傷応答及び転写を含む多様な生物学的経路を調節ダイナミック細胞シグナル伝達ネットワークとして浮上している。より良いのUblは、ヒトの疾患に関連する経路を調節する方法を理解するために、我々は、UBLシステム経路のすべての既知の予測成分を標的1186 siRNAの二重のプールからなるヒトのsiRNA」ubiquitome」ライブラリをコンパイルしている。このライブラリは、UBL成分は、当該経路の正又は負の調節因子として作用するかを決定するために、多様な生物学的経路のレポーターを発現する細胞株の範囲に対してスクリーニングすることができる。ここでは、転写ベースのルシフェラーゼレポーターを使用して低酸素に対するHIF1A媒介細胞応答のubiquitomeレギュレーターを識別するために、このライブラリを利用したプロトコルを記述します。最初のアッセイ開発段階第一、第二および第三級/デコンボリューションスクリーニング:三段階でのスクリーニングを実施する前に、細胞株の適切なスクリーニングパラメータを確立するために行われる。それが唯一の研究者が最も興味を持っているとの経路のメンバーに報告するという利点を提供するように全ゲノムsiRNAライブラリーを介してターゲットの使用は、ますます人気が高まっています。 siRNAのスクリーニングの固有の制限にもかかわらず、siRNAのオフターゲット効果に起因する特定の偽陽性で、問題となっている経路の本物の小説制御因子の同定は慎重に行っ制御一連の実験を実施することによって克服することができるこれらの欠点を上回る。
概要
ユビキチンおよびユビキチン様分子(のUbl)とのタンパク質の修飾は、多様な生物学的経路とストレス反応を調節広大な生化学システムを表します。彼らの標的タンパク質に対するのUblの共有結合は、安定性、局在性、機能または基板1のインタラクトームを規制する様々な成果を持つことができます。 UBLの変更の基礎となる酵素工程は、まず、ユビキチンのために設立され、現在ではSUMO、NEDD8、ISG15およびFAT10を含むほとんどのUbl、との修正のためのパラダイムとして機能した。修飾が起こるためには、UBLのジグリシンモチーフのカルボキシレート基は、第E2結合酵素の活性部位システインへ転送される高エネルギーチオールを形成するためにE1活性化酵素により活性化される。 E2は、その後、(通常は)分枝鎖(イソ)を作成し、ターゲットのリジン残基が結合2にUBLの転写を媒介する基質結合E3リガーゼと相互作用する。修正の連続ラウンドは、ユビキチンリニアユビキチン鎖を作成し、その7リジンのいずれかを介して、またはそのN末端メチオニンを介して起こり得る基板上にイソペプチドチェーンを構築するために発生する可能性があります。これらの変更は、このような従来の専門プロテアーゼによるUBLの除去に新たな相互作用モチーフを作成し、分解のためにタンパク質を標的として、多様な目的に離散トポロジーを形成している。ユビキチンの場合、2つのE1酵素、30〜40のE2結合酵素、少なくとも600のE3リガーゼと約100脱ユビキチン化酵素(DUBの)があります。経路は、他の10かそこらのUblはあまり広大ですが、全体的なubiquitomeの複雑さは、特定のUBL改変の生物学的結果に大きな多様性を与える。 UBL生物学における大きな進歩がなされてきたがしかし、これらubiquitome部品の大多数の正確な細胞の役割は不明のまま。
short int型の使用erferingリボ核酸(siRNA)が原因、特に個々の遺伝子の役割は、さまざまな生物学的状況3で調べることができるように、破壊のための細胞mRNAを標的とするsiRNAの能力に逆遺伝学の強力なツールとして浮上している。全ゲノムスクリーンは、多くの細胞プロセスの新たな規制当局を特定し、検証するために使用されており、より広い科学界にアクセス可能な有用なデータが豊富に作成しました。全ゲノムスクリーンは非常に有用であることが証明されているがしかし、対象となる画面は、彼らが安くなっていますように、速く、唯一の研究者が最も興味のあるゲノムのメンバーにはあまりデータ管理とレポートを伴い、ますます人気が高まっている。そのため、より良い細胞プロセスUBLファミリーの構成要素が関与しているかを理解するために、我々はすべての知られてubiquitomeのコンポーネントを予測を対象とした、人間のsiRNAライブラリーをコンパイルしている。これはのUbl、E1活性化酵素、E2共役が含まれてい酵素、E3リガーゼ、ユビキチン結合ドメイン(UBD)含有タンパク質とのDUB。このライブラリは、このように、これらの経路を支配する新規なUBL成分の公平な同定を可能にする、異なる生物学的問題のレポーター細胞株の広い範囲に対してスクリーニングすることができる。
以下のプロトコルは、低酸素に対するHIF1A依存性応答の新規制御因子を特定するために、厳格な標的としたsiRNA ubiquitome画面を実行する方法について説明します。通常の酸素分圧の下では、HIF1Aは、フォン·ヒッペル·リンドウ(VHL)E3リガーゼ複合体4によって認識され、分解の標的させるプロリルヒドロキシル化の対象となります。低酸素症は、HIF1Aの安定化につながるプロリルヒドロキシル化を阻害し、その後の低酸素応答エレメント(HRE)に結合する遺伝子の発現を駆動する。ここでは、安定的にハイの三タンデムコピーの制御下でホタルルシフェラーゼを発現U20S骨肉腫細胞を使用して画面を記述poxia応答エレメント(U20S-HRE細胞)5。レポーター活性の強固な読み出しが達成可能であり、適切な陽性および陰性対照とカップリングすることができる場合に、このプロトコルは、任意の生物学的な経路に適合させることができる。
プロトコル
1。アッセイ開発ステージ
注意:事前のsiRNAスクリーニングを開始する、アッセイ開発段階はレポーター細胞株を用いたスクリーニングのための重要なパラメータを設定することが重要です。これは、画面の将来の成功を支えるので、この段階では多大な労力を投資することが不可欠である。
- U20S-HREリポーター細胞系の低酸素応答性を特徴付けるために、10%FBSを補充したイーグル培地(DMEM)改変ダルベッコ中80〜90%コンフルエンスに2×75センチメートルをU20S-HRE細胞の2フラスコを成長させる。
- 吸引物セルの一つのフラスコから培地を10mlのPBSで2回洗浄し、2mlの0.05%トリプシン-EDTA溶液を添加し、5分間37℃でインキュベートすることにより細胞を剥離。均質な細胞懸濁液を作成するために8ミリリットル、上下のDMEM、10%FBSとピペット内の細胞を再懸濁。
- ピペット20連で細胞計数室へのサスペンションのμL、および自動細胞カップにチャンバーを挿入NTER。セルカウンタソフトウェアの「表示画像を "クリックし、セルにフォーカスがあることを確認。 「カウント」をクリックして、細胞密度をメモします。他の重複のためにこれを繰り返し、平均セル密度を計算。 60,000細胞/ mlの濃度になるようにDMEM、10%FBSで細胞を希釈する。
- マルチチャンネルピ ペットを使用して、同じ3列( 例:A5-H5、A6-H6およびA7-H7)5無菌白壁96ウェルアッセイプレートの、転送にまで希釈し100μlの細胞(6000細胞)を追加一晩、5%CO 2で加湿した37℃のインキュベーター。注:0時間、2時間、6時間、10時間および24時間これらのプレートは、低酸素曝露の範囲の低酸素依存性ルシフェラーゼ出力を決定するために使用される。
- 次の日の終わりに、時間に注意し、1%の酸素に設定され、低酸素ワークステーションに24時間の低酸素プレートを追加します。 24時間プレートはFRを削除される予定である前に、次の朝には、時間が10時間、6時間、2時間を計算する低酸素ワークステーションOM。これらの時間に、低酸素ワークステーションに10時間、6時間、2時間プレートを追加します。
- 2×30mlの組み合わせルシフェラーゼ溶解/アッセイ緩衝液(50mMトリスリン酸pH7.8で調製し、16のMgCl 2、2mMのDTT、2%トリトン-X-100、30%グリセロール、1mMのATP、1%BSA、0.25ミリモルルシフェリンおよび8μMのNa 4 P 2 O 7)。注:リストされている順序でコンポーネントを追加し、BSA、少なくとも30分は、ルシフェリンおよびNa 4 P 2 O 7を添加する前に溶解させる。
- 適切な時期に低酸素ワークステーションからすべてのプレートを取り外します。アッセイプレートの適切なウェルに2倍のルシフェラーゼ溶解/アッセイバッファー100μlを加え、透明フィルムでカバーしています。完全に細胞を溶解し、500rpmで10分間、プレートシェーカー上でプレートを振る。
- 自動化された96ウェルプレートルミノメーターラックにプレートスタックを転送します。 「プロトコル」メニューの下に、「ABS 595」を選択し、すべてのWELを強調LSは「よく選択」メニューの下に、画面上のプレートマップに読み込まれる。各プレートの平均測定値を計算し、各ウェルの発光を測定するために、「実行」をクリックしてください。酸素正常状態(0時間の低酸素)制御板の平均読み取りによって、各低酸素プレートの平均測定値を除算することにより、レポーター活性の低酸素依存増加倍率を計算します。注:スクリーニングに適するように堅牢な低酸素依存性ルシフェラーゼ反応を観察することが必須である。レポーター活性の五から十倍の増加が優れたと認めている。
- 4高いコントロール(HIF1A siRNA)を反復し(ウェルA1-D1)、(FIH1 siRNAは)(ウェルA12〜D12)に複製4低コントロールを含むマスター制御板を作成し、4バッファのみの複製物(井戸E1-H1)を制御し、4すべてのsiRNAプールが200nmの濃度であり、非標的siRNA対照の複製物(E12〜-H12)、。注記:ハイとローのコントロールが向上し、DECR既知の経路制御因子に基づいて確立されてそれぞれ低酸素応答を緩和する。あるいは、ライブラリーのサブセットを使用してアッセイの開発は、適切なコントロールを識別するために使用することができる。
- マルチチャンネルピペットを用いて、滅菌した白い壁のアッセイプレートに、各コントロールsiRNAを10μlを移す。ウェルあたり10μlの減少血清培地(1:100)で0.1μLトランスフェクション試薬のトランスフェクションミックスを調製し、各ウェルコントロールにトランスフェクション混合物10μlを移す。アップピペットミックスダウンする簡単に言うと、トランスフェクション複合体形成を可能にするために20〜60分間休ませるプレートを残す。
- HRE-U20S細胞の第二のフラスコに75,000細胞/ mlの細胞懸濁液を調製する。マルチチャンネルピペットを使用して、各トランスフェクション混合物を細胞懸濁液を80μl(6,000細胞)を加える。 24時間、5%CO 2の加湿した37℃のインキュベーターにプレートを移し、その後さらに24時間低酸素ワークステーションに転送し、段階的に説明したようにルシフェラーゼアッセイを実施する1.6 -1.8。
- 式Z = 1を使用してハイとローのコントロールのZ係数を計算する - [3×(高コントロール+低コントロールの標準偏差の標準偏差)/(高対照の平均 - 低コントロールの平均を)]。注:0.5〜1の間の値は、優れた分析を示し、ために努力しなければならない。
2。一次スクリーニング
注:アッセイ開発段階からこれらの基本的な条件が整っている一旦、一次スクリーニングは、以下のプロトコルを用いて96ウェルプレートフォーマットで三重に実施することができる。
- DMEM中の80〜90%に7×75センチメートルにHRE-U20S細胞の2フラスコを成長させる合流10%v / vのウシ胎児血清(FCS)を補充した。
注:以下は、2.2から2.13が1日目に行われるべきで繰り返します。 - ステップ1.9で説明およびsiRNA ubiquitome Lの希釈系列を解凍として各コントロールの200μLを含むマスター制御板を用意することで1を複製開始最低30分間ibrary。注:各ウェルのコントロールの空のまま列1と12と17×96ウェルプレートで組み立て4 siRNAのプールが含まれています。
- 層流フードでは、ラベル(またはバーコード)の数字1月17日からの蓋つきの17無菌白壁アッセイプレートを、siRNAライブラリーシリーズの各プレートに対応する。
- マスターコントロールプレートを遠心分離し、siRNAは、すべてのsiRNAは井戸の底に蓄積確実にするためのライブラリを簡単(2,000×gで1分間)ubiquitome。
- 各プレートについて、同じ96ウェルチップスタックを使用して、17アッセイプレート上に自動液体分注にロボットにより「スタンプ」のマスターコントロールプレートから各コントロールの10μlを使用する。
- 17ライブラリープレートの各々のための新しい96ウェルチップスタックを用いて、転写、各10μlを、自動液体ディスペンサーを用いて、その対応するアッセイプレートに対するsiRNAをubiquitome。
- 比ESを使用して、17アッセイプレートを40 mLのトランスフェクション試薬を調製1.10にtablished。注:これは、細胞ディスペンサの「デッドボリューム」を考慮するための追加の20ミリリットルを含む。 「デッドボリューム」は、細胞ディスペンサ内チューブネットワーク内で継続的に存在する液体の体積を意味する。
- 17アッセイプレート内の各ウェルにトランスフェクション試薬を10μlを転送する自動細胞ディスペンサーを使用してください。トランスフェクション試薬の複合体形成:簡単に言うと、その後のsiRNAができるように20〜60分間室温で、まだ残して、siRNAおよびトランスフェクション試薬の完全な混合を可能にするためにシェーカー(500 rpmで1分)でアッセイプレートを振る。
- 10ミリリットルPBSで2回U20S-HRE細胞の2 75cm 2のフラスコを洗い、5分間、37℃で2ミリリットルのトリプシン-EDTAで切り離し。均質な細胞懸濁液を作成し、各フラスコとピペットまでに、数回上下8ミリリットルのDMEM、10%FBSを追加します。両フラスコから細胞を結合し、滅菌50ミリリットルのプラスチックチューブに移す。
- ピペッティングして細胞濃度を計算20μlの細胞計数室への重複のセルと、ステップ1.3で説明したように、自動化細胞カウンターの2値から平均細胞濃度を計算する。
- 滅菌プラスチック容器1ml当たり75,000細胞の濃度にDMEM、10%FBSで希釈して、細胞懸濁液の155ミリリットルを調製する。注意:このボリュームは、17枚のために十分であり、細胞ディスペンサーのデッドボリュームのための追加25ミリリットルが含まれています。
- 細胞凝集を制限するために、層流フードの内側に設定する攪拌機で、細胞懸濁液と場所に、無菌磁気攪拌機を追加します。細胞懸濁液を自動細胞ディスペンサーを平衡化し、ウェルあたりの細胞を80μl(6,000細胞)を分配するように設定します。
- 自動細胞ディスペンサー上に、今度は、17枚のそれぞれを置き、細胞を分注する。 5つのグループで、時間とスタックプレートを記録し、その後24時間、5%CO 2で湿度の高い37℃のインキュベーター内でスタックを配置します。注:終濃度siRNAプールを100μl総容量で20 nmとなります。
注意:以下は、2.14から2.15の2日目に行われるべきで繰り返します。 - 反復1(2.2から2.13)のために、1日目に概説された手順に従い、2日目の2番目の反復を開始します。
- トランスフェクションの24時間後、低酸素ワークステーションに無菌環境下での反復1からの転送プレートを1%酸素に設定し、HREレポーターを誘導するために24時間のままにしておきます。
注意:以下は、2.16から2.20が3日目に行われるべきで繰り返します。 - 反復1(2.2から2.13)のために、1日目に概説された手順に従い、第三の複製を開始します。
- トランスフェクションの24時間後、低酸素ワークステーションに無菌環境内のレプリケート2からの転送プレートを1%酸素に設定し、HREレポーターを誘導するために24時間のままにしておきます。
- 二時間前1プレートを複製1.6に記載したように2×ルシフェラーゼ溶解/アッセイ緩衝液200mlを調製し、低酸素ワークステーションから除去される。注:このボリュームバッファリザーバがよく覆われたまま確実にするために、追加の20ミリリットルをcludes。
- 24時間の低酸素暴露後に低酸素ワークステーションから1アッセイプレートの複製を削除し、自動液体ディスペンサーを用いて、各アッセイプレートに2倍のルシフェラーゼ溶解/アッセイバッファー100μlを加え、透明フィルムでカバーしています。
- 完全に細胞を溶解し、500rpmで10分間、細胞シェーカー上でプレートを振る。ステップ1.8で説明したように、照度計プレートリーダーとレコード発光に順番に各プレートを転送します。
注意:以下は、2.21から2.22の4日目に行われるべきで繰り返します。 - トランスフェクションの24時間後、低酸素ワークステーションに無菌環境内のレプリケート3からの転送プレートを1%酸素に設定し、HREレポーターを誘導するために24時間のままにしておきます。
- 複製1に使用し、次のステップ2.18から2.20での複製を2枚をお読みください。
注:次のステップ2.23が5日目に行われるべきである。 - 以下、レプリケート3版を読むレプリケート1に使用される2.18から2.20のステップ。
- 一次スクリーニングのすべての3回の反復をコンパイルし、1.12で示した式を用いて、各プレートについて、Z係数を計算。注意:プレートは、0.5未満のZ因子を有する場合、データの品質を向上させるために、このプレートを繰り返す考える。
- 制御の制御/平均の100×標準偏差:次の式を使用してハイとローのコントロールの変動係数(CV)を計算します。注:CVはできるだけ低くあるべきで、それは10〜15%の変動を期待することは合理的である。変動が有意に高い場合には、プレートを反復することを検討してください。
- 。×100また、非標的を算出する(NT [ - / - (ハイコントロールの平均低コントロールの平均値)(siRNAのスコア高対照の平均)]:以下の式を用いてプレートあたり各siRNAのパーセント活性を計算式[NTのサンプルデータ/平均]を用いて、各siRNAの)倍。パーセントの活性化とNT倍の両方で、3 replicの平均を計算するのATEと値をコンパイルします。
- 生データを用いて、3つの複製プレートを、式を用いてどのように相関するかを決定するために密接にスピアマンの順位相関係数(SRCC)を計算する。
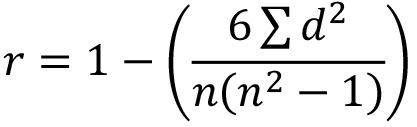
ここで、r = SRCC; D =ランクの各ペアの2つの数値の差であり、n =データのペアの数。注:プレートの複製物との間に良好な相関関係が1反復は、他の2枚に対して低いSRCCが表示されている場合は、1に近い値を与え、さらに調査し、そのプレートを繰り返して検討していきます。 - (80まで)二次スクリーニングのために十分な関心のあるsiRNAを決定します。 ( 例えば 5%未満以上95%パーセントの活性化、または0.5未満のNT倍または1.5以上のNT倍を示したすべてのsiRNAを選択)、ユーザー定義のカットオフを採用するかのクラスターを検索するために、バイオインフォマティクスや知識ベース推論を適用同じUBL経路内に入るヒット。注意:通常、これらのアプローチの組み合わせは、siRNAは二次スクリーニングに取り込まれるかを決定する。
3。二次スクリーニング
注:確認の二次スクリーニングは、一次スクリーニングから関心のある80のsiRNAの最大に基づいて行われる。この数は、都合よく、一次スクリーニングに従ってフルコントロールを有する単一の96穴プレートに、各レプリケート(2.2参照)を用いて行うことができる。それは、一次スクリーニングで同定された規制当局が再現可能に同じ表現型を誘発し、これが最終的な三次/デコンボリューション画面に至るまで実施されるべきで当たる上トリアージの決定を精緻に支援することを確認するために非常に便利です。
- U20S-HRE細胞の1×75cm 2のフラスコを、10%FBSを含むDMEM中で80〜90%のコンフルエンスまで成長。
- プレート番号と一次スクリーニングのヒットの位置に注意して、列の1を残して、二次スクリーニングチェリーがマスタープレートを選んだ上で、その新しい場所を計画するDコントロールのための無料の12。 1.9と同様であるが、50μLの各コントロールの総量をコントロールマスタープレートを準備します。
- 最低30分間(チェリーピッキング、個々のsiRNAプール用に確保して)ubiquitomeライブラリの第二の希釈系列を解凍する。遠心分離機のプレートの短時間(2,000×gで1分)、その後、桜に彼らの新しいプレートの位置に二次スクリーニングのsiRNAの50μlを加えるには、マスタープレートを選んだ。
- 以下の変更を加えて一次スクリーニングのために概説したものと同じ基本的なプロトコルを使用して二次スクリーニングを実施する:(a)は、反復ごとに1つのアッセイプレートを同じ日に3つのすべての複製を実行し、(b)は、それに応じて試薬の量を調整します。
4。第三紀/デコンボリューション画面
注:三次スクリーニングまたはデコンボリューションは、二画面から20のsiRNAの最大値に対して行われる。この手順は、元のうんちから、個々のsiRNAを用いたノックダウンの効果を調べることです4リットル。通常は、各プールから少なくとも2個々のsiRNA二本鎖は、観察された表現型がsiRNAオフターゲット効果によるものではないことを確信の合理的な学位を持っているため、同じ表現型を誘発(illicit)する必要があります。さらに、この段階での信頼性を高めるために、問題の遺伝子を標的とする付加的な個々のsiRNA二重鎖を設計し、所定の表現型を誘発する能力について試験することができる。これらの実験からの結果は、H = 0.6以上( すなわち、5つの個々のsiRNAのうち、少なくとも3つの表現型を誘発する場合)が許容されると考えられる6 Hスコアを計算するために使用されてもよい。
- U20S-HRE細胞の1 75cm 2のフラスコを、10%FBSを含むDMEM中で80〜90%のコンフルエンスまで成長。
- コントロールのための無料のカラム1と12を残して、三次スクリーンマスタープレート上の各ヒットの4個々のsiRNA二本鎖の位置を計画することによって第三のスクリーンのためのプレートマップを作成します。 1.9と同様であるが、トータルでコントロールマスタープレートを準備します50μLの各コントロールのボリューム。
- デコンボリューション/個々のsiRNAのプレートを解凍し、3×10μLの複製、快適な20μL過剰を可能にするためにプレートマップ(4.2)によると、第三級スクリーンマスタープレートの各ウェルに個々のsiRNA(200 nM)を50μlを加える。
- (A)を複製につき1アッセイプレートを同じ日に3つのすべての複製を実行し、(b)は、それに応じて試薬の量を調整します。以下の変更を加えて一次スクリーニングのために概説したものと同じ基本的なプロトコルを使用して三次の画面を実施
注:理想的には、個々のsiRNA二本鎖のためのしきい値は、カットオフのプールと同じストリンジェンシーで設定する必要があります。しかしながら、少なくとも一つの他の二重鎖が設定された閾値内にある場合、特に、個々のsiRNAについて10〜20%の閾値を緩和する許容可能であり得る。これは、siRNAの個々の効果はのそれより小さくてもよいことに注意して価値のある軸受で逆にプールし、、いくつかのケースでは、個々のsiRNAは、それが(濃度が考慮されていても)プール内に存在する場合に比べて孤立した細胞の表現型に対する強い効果を示すことがある。
結果
スクリーニングの前に、HRE-U20S細胞の低酸素応答性が確立される。 U20S-HRE細胞が低酸素にさらされるとHIF1A/HIF1Bヘテロ二量体( 図1A)に拘束された低酸素応答エレメントの3タンデムコピーの下流に融合ホタルルシフェラーゼからなるレポーター構築物を発現する。細胞を低酸素暴露は、スクリーニングのための最も効果的な応答を生成する確立する時間の範囲の低酸素ワークステ...
ディスカッション
哺乳動物細胞におけるゲノムワイドなsiRNAスクリーンの使用は、異なる生物学的経路の新規な調節因子を同定するのに非常に有益であることが証明された。ここでは、低酸素に対するHIF1A媒介性細胞応答の調節因子を同定するための標的ubiquitome siRNAスクリーニングの使用が記載されている。対象となる画面は、彼らは一般的に安価であるように、より速く、ますます魅力的になってきて簡単?...
開示事項
著者らは、開示することは何もありません。
謝辞
この作品は、ウェルカムトラスト、グラクソ·スミスクライン(GSK)とセルシグナリングのためのスコットランド研究所(MRCのタンパク質リン酸化とユビキチン化ユニットの一部)によってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
参考文献
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved