Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
siRNA contrôle pour la ubiquitine et les régulateurs du système ubiquitine-comme des voies biologiques dans des cellules de mammifères en culture
Dans cet article
Résumé
Ici, nous décrivons une méthode pour effectuer un siRNA ciblés écran "ubiquitome" pour identifier de nouveaux régulateurs de l'ubiquitine et ubiquitine de la réponse cellulaire médiée HIF1A à l'hypoxie. Ceci peut être adapté à n'importe quelle voie biologique où une lecture robuste de l'activité du rapporteur est disponible.
Résumé
Modification post-traductionnelle des protéines avec l'ubiquitine et ubiquitine (UBL) molécules est en train de devenir un réseau de signalisation cellulaire dynamique qui régule diverses voies biologiques, y compris la réponse à l'hypoxie, proteostasis, la réponse aux dommages de l'ADN et la transcription. Pour mieux comprendre comment UBL réglementer les filières pertinentes à la maladie humaine, nous avons compilé un siRNA humain "ubiquitome" bibliothèque composée de 1186 siARN piscines duplex ciblant tous connus et prévus des composants de voies du système UBL. Cette bibliothèque peut être projeté contre un éventail de lignées cellulaires exprimant journalistes de diverses voies biologiques pour déterminer quels composants UBL agissent comme régulateurs positifs ou négatifs de la voie en question. Ici, nous décrivons un protocole utilisant cette bibliothèque pour identifier ubiquitome-régulateurs de la réponse cellulaire médiée par HIF1A à l'hypoxie en utilisant un rapporteur de luciférase à base de transcription. Un stade de développement de l'essai initialest effectuée pour établir les paramètres de dépistage appropriés de la lignée cellulaire avant d'effectuer l'écran en trois étapes: dépistage / déconvolution primaire, secondaire et tertiaire. L'utilisation de bibliothèques ciblée sur génome entier de siRNA est de plus en plus populaire car il offre l'avantage de rendre compte uniquement sur les membres de la voie avec laquelle les enquêteurs sont plus intéressés. Malgré les limites inhérentes de dépistage siRNA, en particulier les faux positifs causés par siARN effets hors-cible, l'identification des véritables nouveaux régulateurs des voies en question l'emportent sur ces lacunes, qui peuvent être surmontées par l'exécution d'une série d'expériences de contrôle attentivement entreprises.
Introduction
Modification des protéines avec l'ubiquitine et ubiquitine (UBL) molécules représente un système biochimique expansive qui régule diverses voies biologiques et des réactions de stress. La fixation covalente d'UBL à leurs protéines cibles peut avoir divers effets de régulation de la stabilité, la localisation, la fonction ou interactome du substrat 1. Les étapes enzymatiques sous-jacents UBL modification ont d'abord été établies pour l'ubiquitine, et maintenant servir de paradigme pour la modification avec la plupart des UBL, y compris SUMO, NEDD8, ISG15 et FAT10. Pour la modification de se produire, le groupe carboxylate de la diglycine UBL motif est d'abord activée par une enzyme activant E1 pour former un thiol de haute énergie qui est transférée à la cystéine du site actif d'une enzyme de conjugaison E2. Le E2 interagit ensuite avec une ligase E3 lié au substrat de servir de médiateur de transfert de la LBM sur (habituellement) un résidu de lysine cible création d'une chaîne ramifiée (isopeptidique) 2 tringlerie. Les cycles successifs de modification peuvent se produire pour construire des chaînes isopeptidiques sur le substrat, ce qui peut se produire pour l'ubiquitine à travers l'une de ses sept lysines, ou par l'intermédiaire de sa méthionine N-terminale pour créer des chaînes linéaires d'ubiquitine. Ces modifications font topologies discrètes avec des fins diverses telles que la création de nouveaux motifs d'interaction et le ciblage des protéines pour la dégradation avant l'enlèvement UBL par des protéases spécialisées. Dans le cas de l'ubiquitine E1, il existe deux enzymes, enzymes de conjugaison E2 30 à 40, au moins 600 ligases E3 et environ 100 enzymes deubiquitylating (DUB). Alors que les voies sont moins expansive pour les 10 ou si d'autres UBL, la complexité globale de ubiquitome offre énorme diversité biologique dans le résultat d'une modification UBL particulier. Cependant, alors que des avancées majeures dans UBL biologie ont été faits, les rôles cellulaires précises de la majorité de ces composants ubiquitome restent inconnus.
L'utilisation de short interfering acide ribonucléique (siRNA) a émergé comme un outil puissant pour la génétique inverse en raison de la capacité de siRNA pour cibler spécifiquement ARNm cellulaires pour destruction, permettant le rôle des gènes individuels à examiner dans différents contextes biologiques 3. Criblage du génome entier ont été utilisés pour identifier et valider de nouveaux régulateurs de nombreux processus cellulaires, et ont créé une foule de données utiles accessibles à la communauté scientifique au sens large. Cependant, alors que les écrans du génome entier ont révélé extrêmement utile, écrans ciblés sont de plus en plus populaires car ils sont moins chers, plus rapides, moins impliquer la gestion de données et de rapport uniquement sur les membres du génome où le chercheur est le plus intéressé. Par conséquent, afin de mieux comprendre les processus cellulaires UBL composants de la famille sont impliqués, nous avons constitué une bibliothèque siRNA humain ciblant tous connus et prévus des composants de la ubiquitome. Cela inclut les UBL, E1, E2 activation des enzymes conjugaisondes enzymes, des ligases E3, domaine de liaison à l'ubiquitine (DLC) contenant des protéines et des DUBs. Cette bibliothèque peut être utilisée pour l'écran contre un large éventail de lignées cellulaires rapporteurs de problèmes biologiques distinctes, permettant ainsi l'identification objective des nouveaux composants de l'UBL régissant ces voies.
Le protocole suivant décrit comment effectuer une rigoureuse siRNA ciblés ubiquitome écran pour identifier de nouveaux régulateurs de la réponse de HIF1A dépendant à l'hypoxie. Sous tension normale d'oxygène, HIF1A est soumise à prolyl hydroxylation qui l'amène à être reconnue et ciblée pour la dégradation par la maladie de Von Hippel Lindau (VHL) E3 complexe ligase 4. L'hypoxie inhibe l'hydroxylation prolyl conduisant à la stabilisation des HIF1A et sa liaison ultérieure à des éléments de réponse à l'hypoxie (HRE) de conduire l'expression du gène. Ici, nous décrivons un écran à l'aide des cellules d'ostéosarcome U20S exprimant de façon stable la luciférase de luciole sous le contrôle de trois copies en tandem de l'HyResponse Element poxia (cellules U20S-EDH) 5. Ce protocole peut être adapté pour n'importe quelle voie biologique si un solide lecture de l'activité du rapporteur est réalisable et peut être couplé avec des témoins positifs et négatifs appropriés.
Protocole
1. Phase de démarrage de dosage
Remarque: avant de lancer l'écran de siRNA, un stade de développement de dosage est essentiel de définir des paramètres importants pour le dépistage de la lignée cellulaire rapporteur. Il est essentiel d'investir des efforts importants à ce stade que cette sous-tendra la réussite future de l'écran.
- Pour caractériser l'hypoxie-réactivité de U20S-EDH lignée cellulaire rapporteur, grandir 2 x 75 cm2 de cellules U20S-EDH à 80-90% de confluence dans Dulbecco Modified Eagle (DMEM) supplémenté avec 10% de FBS.
- Aspirer le support d'un flacon de cellules, laver deux fois avec 10 ml de PBS et détacher les cellules par addition de 2 ml de solution à 0,05% de trypsine-EDTA et incubation à 37 ° C pendant 5 min. Resuspendre les cellules dans 8 ml de DMEM 10% de FBS et de la pipette vers le haut et vers le bas pour créer une suspension cellulaire homogène.
- Pipette 20 ul de la suspension à une chambre de comptage de cellules, en double exemplaire, et insérez la chambre dans un cou de cellules automatisénter. Cliquez sur "Afficher l'image" sur le logiciel de compteur de cellules et de s'assurer que les cellules sont en discussion. Cliquez sur "comte" et faire une note de la densité cellulaire. Répétez cette opération pour l'autre exemplaire et calculer la densité moyenne des cellules. Diluer les cellules avec du DMEM 10% FBS à une concentration de 60 000 cellules / ml.
- En utilisant une pipette multicanaux, ajouter 100 pi (6000 cellules) des cellules diluées aux trois mêmes colonnes (par exemple A5-H5, A6-H6 et A7-H7) de 5 plaques à 96 puits essai murs blancs stériles, et le transfert à une humidifié à 37 ° C incubateur à 5% de CO2 pendant une nuit. Remarque: ces plaques seront utilisés pour déterminer la sortie de la luciférase hypoxie dépend pour une gamme d'expositions de l'hypoxie: 0 h, 2 h, 6 h, 10 h et 24 h.
- A la fin de la journée suivante, noter l'heure et ajouter la plaque de l'hypoxie de 24 heures à l'ensemble des postes de travail à l'hypoxie 1% d'oxygène. Le lendemain matin, calculer le temps h 10, 6 h et 2 h avant que la plaque de 24 heures devrait être retiré from le poste de travail de l'hypoxie. Pendant ces périodes, ajouter le h 10, 6 h et 2 plaques h à la station de travail de l'hypoxie.
- Préparer 30 ml de tampon 2x combiné luciférase lyse / dosage (50 mM Tris phosphate pH 7,8, 16 mM MgCl2, 2 mM DTT, 2% de Triton-X-100, 30% glycérol, 1 mM ATP, 1% de BSA; 0,25 mM luciférine et 8 uM Na 4 P 2 O 7). Remarque: ajouter les composants dans l'ordre indiqué et permettre à la BSA au moins 30 min pour dissoudre avant l'addition de luciférine et Na 4 P 2 O 7.
- Retirez toutes les plaques du poste de travail de l'hypoxie au moment opportun. Ajouter 100 ul de tampon de lyse luciférase / test 2x pour les puits appropriés de la plaque d'essai et couvrir avec un film transparent. Secouer les plaques sur un agitateur de plaque pendant 10 minutes à 500 tours par minute pour lyser les cellules à fond.
- Transférer la pile de plaques à une 96 crémaillère plaque de luminomètre et automatisé. Dans le menu "Protocoles", sélectionnez "abs 595" et mettre en évidence tout wells à lire dans la carte à l'écran plaque sous le menu "bien la sélection". Cliquez sur "Exécuter" pour mesurer la luminescence de chaque puits, puis calculer la valeur moyenne de chaque plaque. Calculer la fois l'augmentation de l'hypoxie-dépendante de l'activité du rapporteur en divisant la valeur moyenne de chaque plaque d'hypoxie par la valeur moyenne de la normoxie (0 h hypoxie) de la plaque de commande. Remarque: il est essentiel d'observer une réponse de la luciférase de l'hypoxie-dépendante robuste pour convenir pour le dépistage. Une augmentation de multipliés par cinq fois dix activité de journaliste est considéré comme excellent.
- Créer une plaque de commande maître qui contient quatre haut contrôle (HIF1A siARN) répétitions (puits A1-D1), quatre bas contrôle (FIH1 siRNA) réplique (puits A12-D12), quatre tampon ne contrôle répétitions (puits E1-H1) et quatre réplicats de commande de siRNA non-cible (E12-H12), où tous les pools siARN sont à une concentration de 200 nM. Remarque: les contrôles haut et bas sont établis sur la base de la réglementation de la voie connus qui augmentent et decrfaciliter respectivement la réponse à l'hypoxie. En variante, le développement du dosage en utilisant un sous-ensemble de la bibliothèque peut être utilisée pour identifier des contrôles appropriés.
- En utilisant une pipette multicanaux, transférer 10 pi de chaque siRNA contrôle à blanc plaque d'essai clos stérile. Préparer mélange de transfection de 0,1 pi réactif de transfection dans 10 pi de milieu-sérum réduite (1:100) par puits, et le transfert de 10 pi de mélange de transfection à chaque contrôle bien. Introduire à la pipette de haut en bas rapidement pour mélanger, et laisser la plaque au repos pendant 20-60 minutes pour permettre la formation du complexe transfection.
- Préparer une suspension cellulaire de 75 000 cellules / ml avec le second flacon de cellules U20S-HRE. En utilisant une pipette multicanaux, ajouter 80 ul (6000 cellules) de la suspension cellulaire à chaque mélange de transfection. Transférer la plaque à un incubateur humidifié à 37 ° C avec 5% de CO2 pendant 24 heures, puis les transférer à un poste de travail de l'hypoxie pendant encore 24 h et d'effectuer des dosages de la luciférase de la manière décrite dans les étapes 1.6-1.8.
- Calculer le facteur Z pour les contrôles haut et bas à l'aide de la formule Z = 1 - [3 x (écart-type de contrôle élevé + écart-type de contrôle faible) / (moyenne de haute lutte - moyen de faible contrôle)]. Remarque: une valeur comprise entre 0,5-1 indique un excellent test et devrait être essayé d'obtenir.
2. Écran primaire
Remarque: une fois que ces conditions de base de la phase de développement du test sont en place, l'écran principal peut être effectué en trois exemplaires au format 96 puits plaque en utilisant le protocole suivant.
- Cultiver 7 x 75 cm2 de cellules U20S-HRE à 80-90% de confluence dans du DMEM supplémenté avec 10% v / v de sérum de veau foetal (SVF).
Note: ce qui suit les étapes 2.2 à 2.13 doivent être effectués le jour 1. - Initier la réplication par une préparation d'une plaque de commande maître contenant 200 ul de chaque commande de la manière décrite dans l'étape 1.9 et de décongélation à une série de dilution de la ubiquitome l siRNAIBLIOTHÈQUE pour un minimum de 30 minutes. Remarque: chaque puits contient une piscine de 4 siRNA assemblés en 17 plaques 96 puits avec des colonnes 1 et 12 laissé vide pour les contrôles.
- Dans une hotte à flux laminaire, l'étiquette (ou code à barres) 17 blancs plaques de dosage à paroi stériles avec des couvercles à partir de numéros de 1 à 17, correspondant à chaque plaque de la série de la bibliothèque siRNA.
- Centrifuger la plaque de commande maître et le siRNA ubiquitome brièvement bibliothèque (1 min à 2000 xg) pour s'assurer que tous les siRNA s'accumulent au fond du puits.
- Utilisez un distributeur de liquide automatisé pour "stamp" robot 10 pi de chaque contrôle de la plaque de commande de maître sur les plaques d'essai 17, en utilisant la même pointe pile de 96 puits pour chaque plaque.
- L'utilisation d'un 96 puits d'empilement pointe douce pour chacune des plaques de la bibliothèque 17, le transfert de 10 ul de chaque siRNA ubiquitome à sa plaque d'essai correspondant en utilisant un distributeur de liquide automatisé.
- Préparer 40 ml de réactif de transfection pour les plaques d'essai en utilisant le rapport 17 established en 1.10. Remarque: cela inclut un supplément de 20 ml pour tenir compte de la "volume mort" dans le distributeur de cellule. Le «volume mort» désigne le volume de liquide présent en permanence dans le réseau de tubes dans le distributeur de la cellule.
- Utiliser un distributeur de cellules automatisé pour transférer 10 pi de réactif de transfection à chaque puits dans les plaques de dosage 17. Agiter brièvement les plaques de dosage sur un agitateur (1 min à 500 rpm) pour permettre un mélange intime de siRNA et un réactif de transfection, puis laisser encore à la température ambiante pendant 20 à 60 min pour permettre siRNA: réactif de transfection formation du complexe.
- Laver deux flacons de 75 cm2 de cellules U20S-HRE deux fois avec 10 ml de PBS et détacher avec 2 ml de trypsine-EDTA à 37 ° C pendant 5 min. Ajouter 8 ml de DMEM 10% FBS à chaque flacon et la pipette de haut en bas plusieurs fois pour créer une suspension de cellules homogène. Combiner des cellules des deux flacons et les transférer dans un tube de plastique de 50 ml stérile.
- Calculer la concentration des cellules par pipetage20 pi de cellules en double exemplaire, à une chambre de comptage de cellules et de calculer la concentration moyenne des cellules à partir de deux mesures d'un compteur de cellules automatisé tel que décrit à l'étape 1.3.
- Préparer 155 ml de suspension cellulaire par dilution avec du DMEM 10% FBS à une concentration de 75 000 cellules par ml dans un récipient en plastique stérile. Remarque: ce volume est suffisant pour 17 plaques et comprend une 25 ml supplémentaires pour le volume mort dans le distributeur de la cellule.
- Ajouter un agitateur magnétique stérile à la suspension cellulaire et le placer sur un agitateur mis en place à l'intérieur de la hotte à flux laminaire pour limiter l'agglutination des cellules. Equilibrer le distributeur de cellules automatisé avec la suspension cellulaire et le mettre à distribuer 80 ul de cellules par puits (6000 cellules).
- Placer chacune des plaques 17, à son tour, sur le distributeur de cellules automatique et distribuer des cellules. Noter l'heure et plaques de cheminée en groupes de 5, puis placez les piles dans un humide 37 ° C incubateur à 5% de CO 2 pendant 24 heures. Remarque: la concentration finale depiscine de siRNA sera de 20 nM dans 100 pi volume total.
Note: ce qui suit les étapes 2.14 à 2.15 doivent être effectués le jour 2. - Initier la seconde répétition, le jour 2, suivant la procédure décrite dans Jour 1 pour la réplication 1 (02.02 à 02.13).
- Après 24 heures de transfection, plaques de transfert de répétition 1 dans un environnement stérile à un poste de travail de l'hypoxie fixé à 1% d'oxygène et de laisser pendant 24 heures pour induire le journaliste EDH.
Note: ce qui suit les étapes 2.16 à 2.20 doivent être effectués le jour 3. - Initier la troisième répétition, suivant la procédure décrite dans Jour 1 pour la réplication 1 (02.02 à 02.13).
- Après 24 heures de transfection, plaques de transfert de répétition 2 dans un environnement stérile à un poste de travail de l'hypoxie fixé à 1% d'oxygène et de laisser pendant 24 heures pour induire le journaliste EDH.
- Deux heures avant de reproduire une des plaques doivent être retirés de la station de travail de l'hypoxie, de préparer 200 ml de tampon de lyse luciférase 2x / dosage comme décrit au point 1.6. Remarque: ce volumeinclut une période supplémentaire de 20 ml d'assurer le réservoir tampon reste bien couverte.
- Retirer reproduire une des plaques d'essai à partir du poste de l'hypoxie après une exposition de 24 heures à l'hypoxie et à l'aide d'un distributeur de liquide automatisé, ajouter 100 ul de tampon 2x luciférase lyse / test pour chaque plaque d'essai et couvrir avec un film transparent.
- Secouer les plaques sur un agitateur de cellules pendant 10 minutes à 500 tours par minute pour lyser les cellules à fond. Transférer chaque plaque à son tour à un lecteur de plaque de luminomètre et enregistrer luminescence comme décrit dans l'étape 1.8.
Note: ce qui suit les étapes 2.21 à 2.22 doivent être effectués le jour 4. - Après 24 heures de transfection, plaques de transfert de répétition 3 dans un environnement stérile à un poste de travail de l'hypoxie fixé à 1% d'oxygène et de laisser pendant 24 heures pour induire le journaliste EDH.
- Lire la répétition 2 plaques en suivant les étapes 2.18 à 2.20 utilisées pour la réplication 1.
Remarque: l'étape suivante 2.23 doit être effectuée le jour 5. - Lire les répliques 3 plaques en suivantl'étape 2.18 à 2.20 utilisé pour la réplication 1.
- Compiler tous les 3 répétitions de l'écran principal, et de calculer le facteur Z pour chaque plaque à l'aide de la formule donnée au point 1.12. Remarque: si aucune plaque a un facteur de Z de moins de 0,5, envisager de répéter cette plaque pour améliorer la qualité des données.
- Calculer le coefficient de variation (CV) pour le haut et les bas des contrôles à l'aide de la formule suivante: 100 x écart-type de contrôle / moyen de contrôle. Remarque: CV doit être aussi faible que possible, il est raisonnable de s'attendre à ce 10 à 15% de variation. Si la variation est nettement plus élevé, envisager de renouveler la plaque.
- Calculer pour cent activation de chaque siRNA par plaque à l'aide de la formule suivante: [(moyenne de haute lutte - note siRNA) / (moyenne de haute lutte - moyen de faible contrôle)] x 100 En outre, le calcul de la non-cible (NT. ) fois de chaque siRNA en utilisant la formule [Exemple de données / moyen de NT]. Pour les deux activation de pour cent et NT fois, le calcul de la moyenne des trois replicAtes et compiler les valeurs.
- En utilisant les données brutes, calculer corrélation de rang le coefficient de Spearman (SRCC) pour déterminer dans quelle mesure les trois plaques répétés corrélation en utilisant la formule:
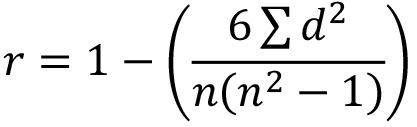
où r = SRCC; d = la différence entre les deux numéros de chaque paire de rangs et n = nombre de paires de données. Remarque: une bonne corrélation entre les répétitions de plaques donnera des valeurs proches de 1 Si une répétition présente une faible SRCC contre les deux autres plaques, d'approfondir et d'envisager de renouveler cette plaque.. - Décider qui siRNA sont d'un intérêt suffisant pour le dépistage secondaire (jusqu'à 80). Employer offs de coupe définis par l'utilisateur (par exemple, la sélection de tous les siRNA montrant moins de 5% ou plus de 95% d'activation de pour cent, soit moins de 0,5 NT fois ou plus de 1,5 NT fois) ou appliquer la bioinformatique ou le raisonnement de la connaissance à la recherche de groupes de frappe relevant de la même voie UBL. Remarque:normalement, une combinaison de ces approches qui détermine siRNA sont pris en criblage secondaire.
3. L'écran secondaire
Remarque: un écran secondaire de confirmation est réalisée sur la base d'un maximum de 80 siRNA d'intérêt de l'écran principal. Ce numéro peut être facilement réalisée avec chaque réplique plaquée sur une seule plaque de 96 puits de contrôles complets que par l'écran principal (voir 2.2). Il est très utile pour confirmer que les régulateurs identifiés dans l'écran principal suscitent reproductible le même phénotype, et ce sera d'aider à préciser les décisions de triage sur lequel doit être effectuée par le biais de l'écran final tertiaire / de déconvolution Hits.
- Cultiver 1 x 75 cm 2 flacon de cellules U20S-HRE à 80-90% de confluence dans du DMEM avec 10% de FBS.
- Notez le numéro de plaque et l'emplacement des résultats d'écran primaires, et planifier leur nouvel emplacement sur la cerise écran secondaire ramassé plaque de maître, laissant colonnes 1 and 12 ans gratuit pour les contrôles. Préparer la plaque de maître de contrôle comme dans 1.9 mais avec un volume total de chaque commande de 50 pi.
- Décongeler une deuxième série de la bibliothèque ubiquitome de dilution (mis de côté pour les piscines de siARN individuels cherry-picking) pour un minimum de 30 minutes. Centrifuger les plaques brièvement (1 min à 2000 xg) puis ajouter 50 ul de siRNA de l'écran secondaire vers leurs nouveaux emplacements de la plaque sur la cerise cueillies plaque de maître.
- Effectuer l'écran secondaire en utilisant le même protocole de base décrit à l'écran principal avec les modifications suivantes: (a) exécuter tous les trois répétitions, le même jour dans une plaque d'essai par répétition et (b) ajuster les volumes de réactifs en conséquence.
4. L'écran supérieur / de déconvolution
Remarque: le dépistage tertiaire ou déconvolution est effectuée sur un maximum de 20 siRNA de l'écran secondaire. Cette étape est d'examiner l'effet de l'utilisation de chaque siRNA knockdown individu de la poo originalel de quatre. Normalement, au moins deux duplex de siRNA individuels de chaque groupe devraient illicite le même phénotype d'avoir un degré de confiance raisonnable que le phénotype observé n'est pas dû à siARN effets hors-cible. Pour augmenter encore la confiance à ce stade, des duplex de siRNA individuels supplémentaires ciblant le gène en question peuvent être conçus et testés pour leur capacité à induire le phénotype donné. Les résultats de ces expériences peuvent alors être utilisés pour calculer le score de H, où H = 0,6 ou plus (c. où au moins 3 des 5 siRNA individuels susciter le phénotype) est considéré comme acceptable 6.
- Cultiver un flacon de 75 cm2 de cellules U20S-HRE à 80-90% de confluence dans du DMEM avec 10% de FBS.
- Créer une carte de plaque pour l'écran supérieur de la planification de l'emplacement des quatre duplex de siRNA spécifiques de chaque coup sur la plaque de maître d'écran supérieur, laissant les colonnes 1 et 12 ans gratuit pour les contrôles. Préparer la plaque de maître de contrôle comme dans 1.9 mais avec un totalvolume de chaque commande de 50 pi.
- Décongeler les plaques déconvolution / siRNA individu et ajouter 50 ul de siRNA individuels (200 nM) dans chaque puits de la plaque de maître d'écran tertiaire selon le plan de la plaque (4.2) pour permettre à trois x 10 répétitions de pi et un confortable 20 pl excès .
- Effectuer l'écran supérieur en utilisant le même protocole de base décrit à l'écran principal avec les modifications suivantes: (a) exécuter tous les trois répétitions, le même jour dans une plaque d'essai par répétition et (b) ajuster les volumes de réactifs en conséquence
Note: Idéalement, le seuil de duplex de siRNA individuels devrait être fixé à la même rigueur de coupure que pour la piscine. Cependant, il peut être acceptable de se détendre le seuil de 10-20% pour les siRNA individuels, en particulier là où au moins un autre duplex en deçà du seuil fixé. Il est intéressant de palier à l'esprit que l'effet individuel de siRNA peut être inférieure à celle dela piscine, et à l'inverse, dans certains cas individuels siRNA peut montrer un effet plus fort sur le phénotype cellulaire dans l'isolement que quand il existe dans la piscine (même lorsque la concentration est comptabilisé).
Résultats
Avant le dépistage, l'hypoxie-réponse des cellules U20S-EDH est établi. Cellules U20S-EDH expriment une construction rapporteur constitué de la luciférase de luciole fusionné en aval de trois copies en tandem de l'élément de réponse à l'hypoxie, qui est lié par l'hétérodimère HIF1A/HIF1B lors de l'exposition à l'hypoxie (figure 1A). Les cellules sont placées dans un poste de travail de l'hypoxie pour une plage de temps pour établir laquelle l'exposition ?...
Discussion
L'utilisation d'écrans de siRNA du génome dans les cellules de mammifères s'est avérée extrêmement utile dans l'identification de nouveaux régulateurs de voies biologiques distinctes. Ici, nous avons décrit l'utilisation d'un écran de siRNA ciblé ubiquitome pour identifier des régulateurs de la réponse cellulaire médiée par HIF1A à l'hypoxie. Écrans ciblés sont de plus en plus attrayant car ils sont généralement moins cher, plus rapide, plus facile à gérer et rapport uniq...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par le Wellcome Trust, GlaxoSmithKline (GSK) et l'Institut écossais pour Signalisation cellulaire (qui fait maintenant partie de la phosphorylation de la protéine MRC et de l'unité de ubiquitylation).
matériels
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
Références
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon