É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Screening siRNA identificar Ubiquitin e ubiquitina-como reguladores do sistema de caminhos biológicos em células de mamíferos cultivadas
Neste Artigo
Resumo
Aqui, descrevemos uma metodologia para realizar uma siRNA alvo "ubiquitome" tela para identificar romance ubiquitina e reguladores de ubiquitina-like da resposta celular mediada por HIF1A à hipóxia. Isto pode ser adaptado para qualquer via biológica, onde uma leitura robusta de actividade repórter está disponível.
Resumo
Modificação pós-tradução de proteínas com ubiquitina e ubiquitina como moléculas (UBLs) está a emergir como uma rede de sinalização celular dinâmica que regula diversos processos biológicos incluindo a resposta a hipoxia, Proteostase, a resposta a danos no ADN e a transcrição. Para entender melhor como UBLs regular caminhos relevantes para doenças humanas, nós compilamos uma siRNA humano "ubiquitome" biblioteca composta de 1.186 siRNA piscinas duplex segmentação todos conhecidos e componentes das vias do sistema UBL previsto. Esta biblioteca pode ser pesquisada contra uma gama de linhas de células que expressam os repórteres de diversas vias biológicas para determinar quais os componentes UBL actuam como reguladores positivos ou negativos da via em questão. Aqui, nós descrevemos um protocolo utilizando esta biblioteca para identificar ubiquitome-reguladores da resposta celular mediada pelo HIF1A a hipóxia através de um repórter de luciferase baseados em transcrição. Uma fase inicial de desenvolvimento do ensaioé realizado para estabelecer os parâmetros de rastreio adequados para a linha celular antes de realizar a tela em três fases: triagem / desconvolução primário, secundário e terciário. O uso de alvo sobre bibliotecas genoma siRNA inteiros está se tornando cada vez mais popular, pois oferece a vantagem de relatar apenas sobre os membros da via com a qual os pesquisadores estão mais interessados. Apesar das limitações inerentes de triagem siRNA, em particular falso-positivos causados por siRNA fora do alvo efeitos, a identificação dos verdadeiros inovadores reguladores das vias em questão superam essas deficiências, que podem ser superados através da realização de uma série de experimentos de controle cuidadosamente desenvolvidas.
Introdução
Modificação de proteínas com ubiquitina e ubiquitina-like moléculas (UBLs) representa um sistema bioquímico expansivo que regula diversos caminhos biológicos e respostas ao estresse. A ligação covalente de UBLs para as suas proteínas alvo pode ter vários resultados que regulam a estabilidade, localização ou função interactoma do substrato 1. Foram estabelecidas as etapas enzimáticas subjacentes modificação UBL primeiro para ubiquitina, e agora servem como paradigma para a modificação com a maioria dos UBLs, incluindo SUMO, NEDD8, ISG15 e FAT10. Para a modificação ocorrer, o grupo carboxilato da diglicina motivo UBL é activado pela primeira vez por uma enzima E1 activação para formar um tiol de alta energia que é transferida para a cisteína do sítio activo de uma enzima de conjugação de E2. O E2 depois interage com uma ligase E3-bound substrato para mediar a transferência de LBM para (geralmente) um resíduo de lisina alvo criando uma cadeia ramificada (isopéptido) ligação 2. Rodadas sucessivas de modificação pode ocorrer para construir cadeias isopeptídicas sobre o substrato, o que para ubiquitina pode ocorrer através de qualquer uma de suas sete Usinas, ou através de sua metionina N-terminal para criar cadeias de ubiquitina lineares. Essas modificações formar topologias discretos com diversos fins, tais como a criação de novos motivos de interação e direcionamento de proteínas para a degradação antes da remoção UBL por proteases especializadas. No caso da ubiquitina existem duas enzimas E1, E2 enzimas de conjugação de 30-40, pelo menos, 600 E3 ligases e aproximadamente 100 enzimas deubiquitylating (DUBs). Enquanto os caminhos são menos expansivo para os outros 10 ou mais UBLs, a complexidade geral ubiquitome oferece enorme diversidade biológica no resultado de uma modificação especial Usama Bin Laden. No entanto, enquanto as principais avanços na biologia UBL têm sido feitas, as funções celulares precisas da maioria destes componentes ubiquitome permanecem desconhecidos.
O uso de short interfering ácido ribonucleico (siRNAs) emergiu como uma ferramenta poderosa na genética inversa, devido à capacidade de siRNAs para alvejar especificamente mRNAs celulares para a destruição, permitindo que o papel dos genes individuais para ser analisada em diversos contextos biológicos 3. Telas do genoma inteiro têm sido utilizados para identificar e validar novos reguladores de muitos processos celulares, e criou uma riqueza de dados úteis acessível à comunidade científica em geral. No entanto, enquanto as telas de genomas inteiros já demonstrou ser extremamente útil, telas direcionados estão se tornando cada vez mais popular como eles são mais baratos, mais rápidos, envolvem menos de gerenciamento de dados e relatório apenas sobre os membros do genoma na qual o investigador é o mais interessado. Portanto, para entender melhor quais componentes da família processos celulares UBL estão envolvidos, nós compilamos uma biblioteca siRNA humano visando todos os conhecidos e os componentes do ubiquitome previsto. Isso inclui os UBLs, E1, E2 enzimas de activação conjugandoenzimas, ligases E3, domínio de ligação da ubiquitina (UBD) contendo proteínas e DUBs. Esta biblioteca pode ser utilizada para a tela de encontro a uma vasta gama de linhas de células repórter de problemas biológicas distintas, permitindo assim a identificação de componentes imparcial UBL novos regulam estas vias.
O protocolo a seguir descreve como executar uma rigorosa siRNA direcionado ubiquitome tela para identificar novos reguladores da resposta dependente da HIF1A à hipóxia. Sob tensão normal de oxigênio, HIF1A está sujeita a hidroxilação prolyl que faz com que ela seja reconhecida e direcionada para a degradação pelo Von Hippel Lindau (VHL) E3 ligase complexo 4. Hipóxia inibe hidroxilação prolil levando à estabilização de HIF1A e sua subsequente ligação a elementos de resposta a hipoxia (Hres) para conduzir a expressão do gene. Aqui, nós descrevemos um ecrã usando células de osteosarcoma U20S estavelmente expressando a luciferase do pirilampo sob o controlo de três cópias em tandem da HyElemento de Resposta poxia (células U20S-HRE) 5. Este protocolo pode ser adaptado para qualquer via biológica se uma leitura robusta de actividade repórter é viável e pode ser acoplado com os controlos positivos e negativos adequados.
Protocolo
1. Ensaio Etapa de Desenvolvimento
Nota: antes de iniciar a tela de siRNA, uma fase de desenvolvimento do ensaio é fundamental para definir os parâmetros importantes para a triagem com a linha de células repórter. É essencial investir esforço significativo nesta fase, pois isso contribuirá para o sucesso futuro da tela.
- Para caracterizar a capacidade de resposta de hipoxia o HRE-U20S linha de células repórter, crescer 2 x 75 cm dois frascos de células U20S-HRE a 80-90% de confluência em Dulbeccos Modified Eagle Médium (DMEM) suplementado com 10% de FBS.
- Aspirar os meios de comunicação a partir de um frasco de células, lava-se duas vezes com 10 ml de PBS e separar as células por adição de 2 ml de solução de tripsina-EDTA a 0,05% e incubando a 37 ° C durante 5 min. Ressuspender as células em 8 ml de DMEM 10% FBS e pipeta-se para baixo e para criar uma suspensão de células homogéneas.
- Pipetar 20 mL da suspensão a uma câmara de contagem de células, em duplicado, e inserir a câmara em um cou células automatizadonter. Clique em "Imagem Display" no software contador de células e assegurar que as células estão em foco. Clique em "Contagem" e tome nota da densidade celular. Repita este procedimento para o outro duplicado e calcular a densidade celular média. Dilui-se as células com DMEM FBS a 10% a uma concentração de 60.000 células / ml.
- Usando uma pipeta de canais múltiplos, adicionar 100 ul (6000 células) das células de diluído para as mesmas três colunas (por exemplo, A5-H5, A6 e A7-H6-H7) de 5 placas estéreis de paredes brancas de 96 cavidades de ensaio, e a transferência humidificada a 37 ° C na incubadora de 5% de CO 2 durante a noite. Nota: estas placas vão ser usadas para determinar a produção de luciferase dependente da hipoxia para uma variedade de posições de hipoxia: 0 horas, 2 horas, 6 horas, 10 horas e 24 horas.
- No final do dia seguinte, observar o tempo e adicionar a chapa de hipoxia de 24 horas para o conjunto da estação de trabalho para a hipoxia 1% de oxigénio. Na manhã seguinte, calcular o tempo de 10 horas, 6 horas e 2 horas antes do prato hr 24 deverá ser removido from a estação de trabalho de hipóxia. Nessas ocasiões, adicione a 10 horas, 6 horas e 2 placas de RH para a estação de trabalho de hipóxia.
- Preparar 30 ml de 2 x tampão de lise de luciferase / ensaio combinado (50 mM Tris pH 7,8 fosfato; mM de MgCl 2 16; DTT 2 mM, 2% de Triton-X-100, 30% de glicerol; ATP 1 mM, 1% BSA, 0,25 mM luciferina e 8 uM de Na 4 P 2 O 7). Nota: adicionar os componentes na ordem indicada e permitir que a BSA, pelo menos, 30 minutos para se dissolver antes da adição de luciferina e Na 4 P 2 O 7.
- Remover todas as placas a partir da estação de trabalho de hipoxia no momento apropriado. Adicionar 100 ul de tampão de lise de luciferase / 2x ensaio aos poços apropriados da placa de ensaio e cobrir com película transparente. Agitar as placas sobre um agitador de placas durante 10 minutos a 500 rpm para lisar completamente as células.
- Transferir a pilha de placas para um sistema automatizado de 96 cavidades cremalheira placa luminómetro. Sob o menu "Protocolos", selecione "abs 595" e destacar todos wells para ser lido na placa de mapa na tela no menu "bem a seleção." Clique em "Executar" para medir a luminescência de cada poço, em seguida, calcular a média de leitura de cada placa. Calcule o fold-aumento dependente da hipóxia da atividade repórter dividindo a leitura média de cada placa hipóxia pela leitura média da normóxia (0 hr hipoxia) placa de controle. Nota: é essencial para observar uma resposta de luciferase dependente de hipoxia robusta para ser adequado para o rastreio. Um aumento de cinco a dez vezes na atividade de repórter é considerado excelente.
- Criar uma placa de controle mestre que contém quatro alto controle (HIF1A siRNA) repetições (poços A1-D1), quatro baixo controle (FIH1 siRNA) replica (poços A12-D12), quatro tampão só controlar repetições (poços E1-H1) e quatro repetições de controlo siRNA não-alvo (E12-H12), onde todos os pools de siRNA estão a uma concentração de 200 nM. Nota: controles de altos e baixos são estabelecidos com base na via reguladores conhecidos que aumentam e DECRaliviar a resposta hipoxia, respectivamente. Em alternativa, o desenvolvimento de ensaio utilizando um subconjunto da biblioteca podem ser utilizados para identificar os controlos adequados.
- Usando uma pipeta de canais múltiplos, transferir 10 ml de cada um siRNA de controlo para uma placa de ensaio de paredes brancas estéril. Preparar a mistura de transfecção de reagente de transfecção de 0,1 mL em 10 ul de meio de soro reduzido (1:100) por poço, e a transferência de 10 uL de mistura de transfecção a cada poço de controlo. Pipeta cima e para baixo rapidamente para misturar, e deixar a placa em repouso por 20-60 min para permitir a formação de complexos de transfecção.
- Prepara-se uma suspensão de células de 75.000 células / ml com o segundo balão de células U20S-HRE. Usando uma pipeta de canais múltiplos, adicionar 80 ul (6000 células) de suspensão de células a cada mistura de transfecção. Transferir a placa para um incubador humidificado a 37 ° C com 5% de CO 2 durante 24 horas, em seguida a transferência para uma estação de trabalho para uma hipoxia ainda mais 24 horas e realizar ensaios de luciferase, tal como descrito nas etapas 1.6-1.8.
- Calcular o fator Z para os controles de alta e baixa, usando a fórmula Z = 1 - [3 x (desvio padrão de alto controle + desvio padrão de baixo controle) / (média de controle de alta - média de baixo controle)]. Nota: um valor de entre 0,5-1 indica uma excelente ensaio e deve ser esforçado para.
2. Tela Principal
Nota: uma vez que estas condições alcalinas a partir da fase de desenvolvimento do ensaio estão no lugar, o ecrã primário pode ser realizado em triplicado, em formato de 96 poços da placa usando o seguinte protocolo.
- Grow 7 x 75 cm 2 frascos de células U20S-HRE a 80-90% de confluência em meio DMEM suplementado com 10% v / v de soro fetal bovino (FCS).
Nota: os passos a seguir 2,2-2,13 deve ser levada a cabo no Dia 1. - Iniciar uma repetição através da preparação de uma placa de controlo principal contendo 200 ul de cada controlo, tal como descrito no passo 1.9 e de descongelamento para uma série de diluições do siRNA ubiquitome library por um período mínimo de 30 min. Nota: cada poço contém uma piscina de 4 siRNAs reunidos em 17 placas x 96 poços com colunas 1 e 12 deixados em branco para controles.
- Em uma câmara de fluxo laminar, a etiqueta (ou código de barras) 17 placas de ensaio de paredes brancas estéreis com tampa de números 1-17, que corresponde a cada uma das placas da série biblioteca de siRNA.
- Centrifugar a placa de controlo principal e o siRNA ubiquitome brevemente biblioteca (1 min a 2000 xg) para assegurar que todos os siRNAs se acumulam no fundo do poço.
- Use um líquido dispensador automático para roboticamente "selo" 10 ml de cada controle da placa de controle principal para as placas de ensaio 17, utilizando-se o mesmo de 96 poços pilha dica para cada placa.
- Usando um de 96 poços frescas pilha ponta de cada uma das placas da biblioteca 17, a transferência de 10 uL de cada ubiquitome siRNA para a sua placa de ensaio correspondente, utilizando um dispensador de líquido automatizado.
- Preparar 40 ml de reagente de transfecção para as 17 placas de ensaio utilizando os es ráciolecido em 1.10. Nota: isto inclui um adicional de 20 ml para explicar o "volume morto" no distribuidor celular. O "volume morto" refere-se ao volume de líquido continuamente presente no interior da rede de tubagem para o dispensador de células.
- Usar um dispensador de células automatizado para transferir 10 ul de reagente de transfecção a cada poço nas placas de ensaio 17. Agitar rapidamente as placas de ensaio num agitador (1 min a 500 rpm) para permitir que a mistura completa do siRNA e reagentes de transfecção, em seguida, deixar ainda à temperatura ambiente durante 20-60 minutos para permitir que o siRNA: reagente de transfecção a formação do complexo.
- Lavar duas 75 centímetros dois frascos de células U20S-HRE duas vezes com 10 ml de PBS e destacam com 2 ml de tripsina-EDTA, a 37 ° C durante 5 min. Adicionar 8 mL de DMEM 10% FBS a cada balão e pipeta cima e para baixo várias vezes para criar uma suspensão de células homogéneas. Combinar as células de ambos os frascos e transferir para um tubo de 50 ml de plástico estéril.
- Calcular a concentração de células por pipetagem20 ul de células em duplicado a uma câmara de contagem de células e calcular a concentração de célula média de duas leituras do contador de células automatizado como descrito no passo 1.3.
- Preparar 155 ml de suspensão de células por diluição com meio DMEM FBS a 10% a uma concentração de 75.000 células por ml, em um recipiente de plástico estéril. Nota: este volume é suficiente para 17 placas e inclui um adicional de 25 ml de volume morto no distribuidor célula.
- Adicionar um agitador magnético estéril à suspensão de células e colocar em um agitador configurado no interior da câmara de fluxo laminar, para limitar a acumulação de células. Equilibrar o dispensador de células automatizado com a suspensão celular e configurá-lo para dispensar 80 mL de células por poço (6.000 células).
- Colocar cada uma das 17 placas, por sua vez, para o distribuidor de células automático e dispensar as células. Gravar a hora e as placas da pilha, em grupos de 5, em seguida, colocar as pilhas num húmido 37 ° C numa estufa a 5% de CO 2 durante 24 horas. Nota: a concentração final desiRNA piscina será de 20 nM em 100 ul de volume total.
Nota: os passos a seguir 2,14-2,15 deve ser levada a cabo no Dia 2. - Inicie a segunda réplica no dia 2, seguindo o procedimento descrito no Dia 1 para replicar 1 (2,2-2,13).
- Após 24 horas de transfecção, placas de transferência de replicar 1 em um ambiente estéril para uma estação de trabalho hipóxia fixado em 1% de oxigênio e deixe por 24 horas para induzir o repórter HRE.
Nota: os passos a seguir 2,16-2,20 deve ser realizada no dia 3. - Inicia a terceira réplica, seguindo o procedimento descrito no Dia 1 para replicar 1 (2,2-2,13).
- Após 24 horas de transfecção, placas de transferência de replicar 2 em um ambiente estéril para uma estação de trabalho hipóxia fixado em 1% de oxigênio e deixe por 24 horas para induzir o repórter HRE.
- Duas horas antes de replicar uma placas estão a ser removidos da estação de trabalho de hipoxia, preparar 200 ml de tampão de lise 2x luciferase / ensaio como descrito em 1.6. Nota: este volumemente, incluem um adicional de 20 ml para assegurar o reservatório tampão permanece bem coberto.
- Retirar replicar uma placas de ensaio a partir da estação de trabalho depois de exposição a hipoxia hipoxia e 24 horas utilizando um dispensador de líquido automatizado, adicionar 100 mL de tampão de lise 2x luciferase / ensaio a cada placa de ensaio e cobrir com película transparente.
- Agitar as placas sobre um agitador de células durante 10 minutos a 500 rpm para lisar completamente as células. Transferir cada placa em vez de um leitor de placa de luminómetro e ficha luminescência, tal como descrito na etapa 1.8.
Nota: os passos a seguir 2,21-2,22 deve ser realizada no dia 4. - Após 24 horas de transfecção, placas de transferência de replicar 3 em um ambiente estéril para uma estação de trabalho hipóxia fixado em 1% de oxigênio e deixe por 24 horas para induzir o repórter HRE.
- Leia a réplica 2 placas seguindo os passos 2,18-2,20 utilizados para replicar 1.
Nota: o seguinte passo 2.23 deve ser levada a cabo no Dia 5. - Leia as réplicas 3 placas, seguindopasso 2,18-2,20 usado para replicar 1.
- Compilar todos os 3 repetições da tela principal, e calcular o fator Z para cada placa usando a fórmula dada em 1.12. Nota: se qualquer placa tem um factor de Z de menos que 0,5, considerar repetindo esta placa para melhorar a qualidade dos dados.
- O cálculo do coeficiente de variação (CV) para o alto e os controlos baixos, utilizando a seguinte fórmula: 100 x desvio padrão de controlo / média de controlo. Nota: CV deve ser tão baixo quanto possível, é razoável esperar variação de 10-15%. Se a variação é significativamente mais elevada, considere repetindo o prato.
- Calcule por cento ativação de cada siRNA por placa utilizando a seguinte fórmula: [(média de alto controle - pontuação siRNA) / (média de controle de alta - média de baixo controle)] x 100 Além disso, o cálculo do não-alvo (NT. ) vezes de cada siRNA usando a fórmula [Amostra de dados / média de NT]. Para ambos os por cento de activação e NT-dobra, calcular a média dos três replicates e compilar os valores.
- Utilizando os dados brutos, calcular Ranking coeficiente de correlação de Spearman (SRCC) para determinar o quão perto as três placas idênticas, correlacionar com a fórmula:
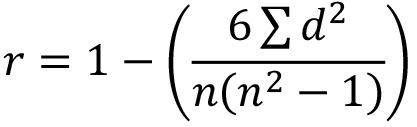
onde r = SRCC; d = diferença entre os dois números em cada par de fileiras e n = número de pares de dados. Nota: uma boa correlação entre réplicas de placas vai dar valores próximos a 1 Se uma réplica apresenta baixa SRCC contra as outras duas placas, investigar e considerar repetir aquele prato.. - Decida qual siRNAs são de interesse suficiente para a triagem secundária (até 80). Empregar offs corte definidos pelo usuário (por exemplo, a seleção de todos os siRNAs mostrando menos de 5% ou mais de 95% de ativação por cento, ou menos de 0,5 NT vezes ou mais de 1,5 NT-fold) ou aplicar bioinformática ou raciocínio baseado em conhecimento para procurar aglomerados de atinge abrangidos pelo mesmo caminho Usama Bin Laden. Nota:normalmente, uma combinação destas abordagens determina que siRNA são tomados em rastreio secundário.
3. Tela secundária
Nota: uma tela secundária de confirmação é realizada com base em um máximo de 80 siRNAs de interesse a partir da tela principal. Este número pode ser convenientemente levada a cabo com cada repetição banhado numa única placa de 96 poços com os controlos completos como por o ecrã primário (ver 2.2). É muito útil para confirmar que os reguladores identificados na tela principal reproducibly obter o mesmo fenótipo, e isso vai ajudar a refinar as decisões de triagem em que bate deve ser levada até a tela final terciário / deconvolução.
- Grow 1 x 75 cm 2 balão de células U20S-HRE a 80-90% de confluência em DMEM com 10% de FBS.
- Anote o número da placa e localização dos acessos de tela primários e planear a sua nova localização no ecrã secundário cereja pegou placa mestre, deixando colunas 1, umad 12 livre para controles. Preparar a placa mestre de controlo como na secção 1.9, mas com um volume total de cada controlo de 50 ul.
- Descongele uma segunda série de diluição da biblioteca ubiquitome (reservado para piscinas siRNA individuais cherry-picking) para um mínimo de 30 min. Centrifugar brevemente a placas (1 min a 2.000 xg), em seguida, adicionar 50 ul dos siRNAs tela secundária para seus novos locais de placa na cereja pegou placa mestre.
- Realizar o ecrã secundário utilizando o mesmo protocolo de base descrito para o ecrã primário com as seguintes modificações: (a) executar todas as três repetições, no mesmo dia em uma placa de ensaio de cada repetição e (b) ajustar os volumes de reagentes em conformidade.
4. Terciário Tela / Deconvolution
Nota: triagem terciário ou deconvolução é realizada em um máximo de 20 siRNAs a partir do ecrã secundário. Este passo é examinar o efeito do uso de cada siRNA knockdown indivíduo do poo originaisl de quatro. Normalmente, pelo menos, duas cadeias duplas de siRNA individuais de cada grupo deverá ilícito o mesmo fenótipo, que apresentam um grau de confiança razoável que o fenótipo observado não é devido ao siRNA efeitos fora do alvo. Para aumentar ainda mais a confiança nesta fase, ARNic em cadeia dupla individuais adicionais que visam o gene em questão pode ser projectado e testado quanto à sua capacidade para induzir o fenótipo determinado. Os resultados destas experiências pode então ser usado para calcular a pontuação de H, em que H = 0,6 ou mais (isto é, pelo menos 3 dos 5 siRNAs individuais induzir o fenótipo) é considerada aceitável 6.
- Crescer uma 75 centímetros 2 balão de células U20S-HRE a 80-90% de confluência em DMEM com 10% de FBS.
- Criar um mapa da placa para a tela superior, planejando a localização dos quatro sobrados siRNA individuais de cada hit na placa mestre tela superior, deixando colunas 1 e 12 livre para controles. Prepare a placa mestre de controle como na 1.9, mas com um totalvolume de cada controlo de 50 ul.
- Descongelar as placas desconvolução / siRNA individuais e adicionar 50 ul de os siRNAs individuais (200 nm) em cada poço da placa mestre tela terciário de acordo com o mapa da placa (4.2) para permitir a três repetições x 10 ul e um excesso de 20 ul confortável .
- Efectuar a tela superior, usando o mesmo protocolo básico traçado para a tela principal com as seguintes modificações: (a) executar todas as três repetições, no mesmo dia em uma placa de ensaio por repetição e (b) ajustar os volumes de reagentes de acordo
Nota: O ideal seria o limite para siRNA duplex individuais devem ser definidos ao mesmo rigor de corte como para a piscina. No entanto, pode ser aceitável para relaxar o limite de 10-20% por siRNA individuais, especialmente em que pelo menos um outro duplex cai dentro do limite definido. É vantajoso rolamento em mente que o efeito individual de siRNAs pode ser menor do que aa piscina, e, inversamente, em alguns casos, siRNAs individuais podem mostrar um efeito mais forte sobre o fenótipo celular isoladamente do que quando ela existe na piscina (mesmo quando a concentração é contabilizado).
Resultados
Antes do rastreio, a hipóxia-responsividade das células U20S-HRE é estabelecida. Células U20S-HRE expressar uma construção repórter consistindo de luciferase de vaga-lume fundido a jusante da três cópias em tandem do elemento de resposta a hipóxia, que é obrigado pelo heterodímero HIF1A/HIF1B após a exposição à hipóxia (Figura 1A). As células são colocadas numa estação de trabalho de hipoxia para uma série de vezes para estabelecer que a exposição a hipoxia produz a resposta mais...
Discussão
O uso de telas siRNA ampla do genoma nas células de mamíferos tem provado ser extremamente útil na identificação de novos reguladores de vias biológicas distintas. Aqui, nós descrevemos o uso de um ecrã ubiquitome siRNA alvo para identificar reguladores da resposta celular mediada pelo HIF1A a hipóxia. Telas alvejados estão se tornando cada vez mais atraente como eles são geralmente mais barato, mais rápido, mais fácil de gerenciar e relatório apenas sobre os componentes da via em que os investigadores est...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi financiado pelo Wellcome Trust, Glaxosmithkline (GSK) eo Instituto Escocês de Sinalização Celular (agora parte da Fosforilação MRC e unidade ubiquitinação).
Materiais
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
Referências
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados