Method Article
Espectrometría de masas enfoque para estudiar la estructura de la proteína y las interacciones en liofilizadas Polvos
* Estos autores han contribuido por igual
En este artículo
Resumen
Here, we present detailed protocols for solid-state amide hydrogen/deuterium exchange mass spectrometry (ssHDX-MS) and solid-state photolytic labeling mass spectrometry (ssPL-MS) for proteins in solid powders. The methods provide high-resolution information on protein conformation and interactions in the amorphous solid-state, which may be useful in formulation design.
Resumen
Amida de hidrógeno / intercambio de deuterio (ssHDX-MS) y fotolítica etiquetado de la cadena lateral (SSPL-MS), seguido por análisis de espectrometría de masas pueden ser valiosos para la caracterización de formulaciones liofilizadas de proteínas terapéuticas. Etiquetado seguido por la digestión proteolítica adecuada permite que la estructura de proteínas y las interacciones que se asignan con la resolución a nivel de péptido. Dado que la proteína elementos estructurales se estabilizan mediante una red de enlaces químicos de las principales cadenas y cadenas laterales de aminoácidos, un etiquetado específico de los átomos en los residuos de aminoácidos da una idea de la estructura y la conformación de la proteína. En contraste con los métodos de rutina utilizados para estudiar las proteínas en sólidos liofilizados (por ejemplo, FTIR), ssHDX-MS-MS y SSPL proporcionan información cuantitativa y específica de sitio. El grado de incorporación de deuterio y los parámetros cinéticos se puede relacionar con el intercambio rápidamente y lentamente piscinas de amida (N rápido, N lento) y refleja directamente los degree de plegamiento de proteínas y estructura en formulaciones liofilizadas. Etiquetado fotolítico Estable no sufre de respaldo de cambio, una ventaja sobre ssHDX-MS. Aquí, ofrecemos protocolos detallados para ambos ssHDX-MS y SSPL-MS, utilizando la mioglobina (Mb) como una proteína modelo en formulaciones liofilizadas que contienen trehalosa o sorbitol.
Introducción
Medicamentos de proteínas son el sector de más rápido crecimiento de la industria biofarmacéutica y ofrecen nuevos tratamientos prometedores para enfermedades hasta ahora intratables, como los trastornos hormonales, cáncer y enfermedades autoinmunes 1. En 2012, el mercado de productos bioterapéuticos global alcanzó $ 138 mil millones y se espera que llegue a $ 179 mil millones para el año 2018 2. Las proteínas son más grandes y más frágiles que los fármacos de molécula pequeña convencionales y por lo tanto son más susceptibles a muchos tipos de degradación 3. Para asegurar la vida útil y la estabilidad adecuada, fármacos de proteínas se formulan a menudo como liofilizado (es decir, liofilizado) polvos sólidos. Sin embargo, una proteína puede todavía sufrir degradación en el estado sólido, en particular si su estructura nativa no se conserva durante el proceso de liofilización 4,5. Asegurar que la estructura se ha conservado es factible sólo si hay métodos analíticos que pueden sondear conformación de la proteína en el estado sólido con suficient resolución.
Espectroscopía de RMN 6 y cristalografía de rayos X 7 son los métodos comúnmente utilizados de alta resolución para evaluar la estructura de proteínas en solución y los sólidos cristalinos 8. Debido a la naturaleza de los excipientes y los métodos de procesamiento utilizados, las formulaciones de proteínas liofilizadas son generalmente amorfa en vez de cristalina 9. La falta de homogeneidad y el orden microscópico hace que las técnicas mencionadas anteriormente poco práctico para las proteínas en sólidos amorfos. Espectroscopía infrarroja por transformada de Fourier (FTIR) 10, espectroscopia de Raman 11 y la espectroscopia de infrarrojo cercano (NIR) 12 han sido utilizados regularmente por la industria biofarmacéutica para comparar la estructura secundaria de proteínas en polvos liofilizados a la de la estructura de estado de solución nativa. Sin embargo, estos métodos son de baja resolución y sólo pueden proporcionar información sobre los cambios globales en la estructura secundaria. Caracterización estructural de estado sólido utilizando FTIRha mostrado ya sea débil o pobre 13,14 15 correlación con la estabilidad de almacenamiento a largo plazo. Estas limitaciones ponen de relieve la necesidad de métodos de alta resolución conveniente para identificar perturbaciones estructurales de proteínas en el estado sólido.
Etiquetado químico junto con la proteólisis y el análisis de espectrometría de masas ha surgido como un enfoque poderoso para el seguimiento de la estructura de proteínas y las interacciones moleculares en solución acuosa. En el desarrollo farmacéutico, HDX-MS se ha utilizado para el mapeo de epítopo en las interacciones antígeno-anticuerpo 16,17, para mapear las interacciones receptor-drogas 18, para vigilar los efectos de las modificaciones post-traduccionales en la conformación de los fármacos proteicos 19, y comparar variación de lote a lote en el desarrollo de los biosimilares 20. Del mismo modo, los ligandos fotoactivables se han utilizado para identificar dianas de fármacos y para determinar la afinidad de unión y la especificidad de las interacciones fármaco-receptor 21,22. Para enviarxtend la aplicación de estos métodos a formulaciones liofilizadas, nuestro grupo ha desarrollado la espectrometría de masas de intercambio de deuterio hidrógeno en estado sólido (ssHDX-MS) y espectrometría de masas etiquetado fotolítico de estado sólido (SSPL-MS) para el estudio de las conformaciones de proteínas y las interacciones de excipiente en muestras liofilizadas con alta resolución.
En tanto ssHDX-MS-MS y SspI, la proteína está marcada en condiciones de reacción ideales en sólidos liofilizados, y las muestras se reconstituye a continuación, y se analizó mediante espectrometría de masas con o sin digestión proteolítica. ssHDX-MS proporciona información sobre la exposición principal cadena de vapor de deuterio, mientras SSPL-MS proporciona información sobre el medio ambiente de las cadenas laterales (Figura 1). Los dos métodos de este modo pueden proporcionar información complementaria sobre la conformación de proteínas en el estado sólido. Aquí, ofrecemos un protocolo general para el estudio de las proteínas en los sólidos liofilizados utilizando ssHDX-MS y SSPL-MS (Figura 2), utilizando como Mbuna proteína modelo. Mostramos la capacidad de los dos métodos para distinguir las diferencias en las formulaciones con dos excipientes diferentes.
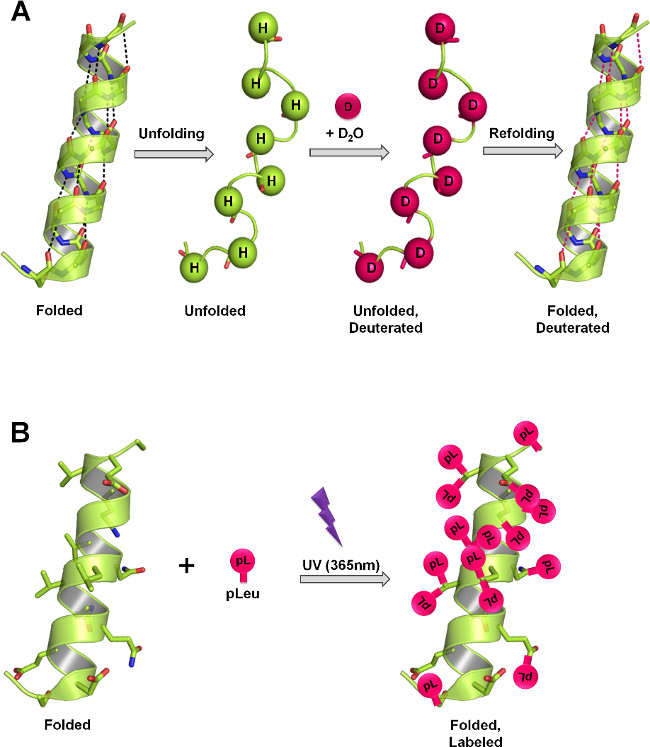
Figura 1:. SsHDX y SSPL estructura de la proteína medida en sólidos liofilizados a través de diferentes mecanismos de etiquetado (A) En HDX, la columna vertebral amida hidrógenos intercambio con deuterio como una función de la estructura de proteínas y D 2 O accesibilidad. En el estado sólido, la velocidad y el grado de intercambio de deuterio dependen del nivel de D 2 O sorción, la movilidad de proteínas (despliegue y replegamiento de eventos) y la naturaleza de los excipientes presentes en la matriz sólida. (B) En PL, la irradiación UV a 365 nm inicia la formación de un carbeno intermedio reactivo del grupo funcional diazirina de pleu del Sáhel y se inserta de forma no específica a cualquier enlace XH (X = cualquier átomo), o added a través de un enlace C = C en sus inmediaciones. En el estado sólido, la velocidad y extensión de etiquetado dependen de la concentración local del agente de marcaje, el tiempo de irradiación, la estructura de proteínas y la naturaleza de los excipientes presentes en la matriz sólida. Los paneles A y B muestran el etiquetado teórico máximo que puede ocurrir en la columna vertebral y las cadenas laterales, respectivamente, en la proteína.
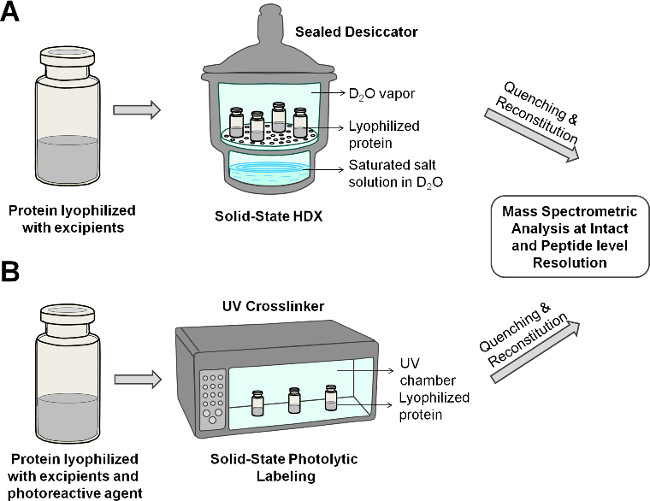
Figura 2: muestra de estado sólido esquemática HDX-MS (A) y PL-MS (B) para la proteína en la formulación liofilizada.
Protocolo
1. Preparación de muestras y liofilización
- Se dializa el volumen requerido de solución madre Mb contra un tampón y filtro adecuado a través de un filtro estéril de 0,22 micras.
- Preparar el volumen necesario de excipientes y foto-leucina (ácido L-2-amino-4,4-azi-pentanoico; pleu del Sáhel) soluciones madre en tampón adecuado. Filtrar las soluciones madre a través de un filtro estéril de 0,22 micras.
- Preparar formulaciones como se muestra en la Tabla 1 usando las soluciones madre de proteínas, excipientes, pleu del Sáhel, y tampón.
- Filtrar las muestras a través de un filtro estéril de 0,22 micras para eliminar cualquier partículas formadas en el paso 1.3. Llenar las muestras por separado como 0,2 ml en viales de vidrio de 2 ml. Utilice viales de vidrio que son transparentes a los rayos UV (365 nm) de luz para activar pleu del Sáhel en los estudios SSPL-MS.
- Viales de carga en un liofilizador y la liofilización inician mediante el diseño de un ciclo de liofilización apropiado.
- Aquí, conservar las muestras a -40 ° C, seguidopor secado primario a vacío (70 Pa) a -35 ° C durante 12 horas y secado secundario a 25 ° C durante 12 horas. Otros ciclos de liofilización y métodos de secado (por ejemplo, secado por pulverización) puede también ser utilizado.
- Rellene los viales que contienen muestras liofilizadas con nitrógeno antes de la limitación.
| Formulaciones | Composición (mg / ml) antes de la liofilización | ||||
| Mb | La trehalosa | Sorbitol | pleu del Sáhel c | De potasio, fosfato, pH 7,4 | |
| MBT un | 1.7 | 3.4 | - | - | 0.4 |
| MBS una | 1.7 | - | 3.4 | - | 0.4 |
| MBT + pleu del Sáhel b | 1.7 | 3.4 | - | 14,3 x 10 -3-1,43 | 0.4 |
| MBS + pleu del Sáhel b | 1.7 | - | 3.4 | 14,3 x 10 -3-1,43 | 0.4 |
Tabla 1:... Composición de formulaciones liofilizadas Mb a formulaciones utilizadas para el estudio ssHDX-MS Formulaciones B utilizados para el estudio SSPL-MS c L-2-amino-4,4-azipentanoic ácido o foto-leucina (pleu del Sáhel). pleu del Sáhel a cinco concentraciones diferentes (14,3 x 10 -3 a 1,43 mg / ml) correspondientes a 1x, 10x, 20x, 50x y 100x exceso molar con respecto a Mb fueron co-liofilizado con formulaciones MBT y MBS.
2. ssHDX-MS para la proteína intacta
- Añadir una cantidad saturante (~ 440 g) de K 2 CO 3 a 200 ml de D 2 O previously coloca en el compartimiento inferior de un desecador. Sellar el hermético desecador y deje que se equilibre a 5 ° C hasta una humedad relativa estable (RH) de ~ 43% se alcanza. Otros valores de HR de interés se pueden obtener mediante la selección de diferentes soluciones salinas saturadas 23,24.
- Iniciar reacciones ssHDX mediante la colocación de los viales destapados que contienen la proteína liofilizada en el compartimiento superior del desecador. Sellar el desecador hermético e incubar a 5 ° C para permitir que se produzca la HDX (Figura 2A).
- Recoger muestras ssHDX en varias ocasiones, por triplicado. Para formulaciones Mb, recoger muestras en nueve puntos 1, 2, 4, 8, 16, 32, 56, 92 y 144 h de tiempo.
- Tape los frascos inmediatamente después de retirarse del desecador y calmar las reacciones por el flash de congelación de los viales en nitrógeno líquido. Guarde los viales a -80 ° C hasta su análisis por espectrometría de masas.
- Analizar las muestras usando una cromatografía de masas de alta resolución líquido Spectrom adecuadométodo etría (LC-MS). Diseño o comprar un sistema LC refrigerada adecuada para reducir al mínimo la espalda de intercambio durante el análisis de la muestra. Utilice la configuración de la unidad de refrigeración de la columna y el método LC-MS informó anteriormente 25.
NOTA: Dado que la tasa de amida de intercambio de protones depende del pH y la temperatura, deuterones incorporados en la proteína pueden intercambiar con hidrógeno presente en la fase móvil ("intercambio de vuelta"), causando una pérdida de información. Aunque un pH ácido (pH 2,5) de tampón de enfriamiento y disolventes de HPLC puede minimizar la espalda de intercambio en gran medida, la reducción de la temperatura (≤0 ° C) por medio de un sistema de refrigeración columna adecuada puede proteger aún más la proteína de la parte posterior de intercambio . - Conectar el bucle de muestra y la trampa de la proteína a la válvula que controla automáticamente el proceso de desalación y la elución. Calibrar el espectrómetro de masas mediante la inyección de un TOF de baja concentración de la mezcla de sintonía en el espectrómetro de masas en el rango de m / z de 200-3,200. El inmovilizadola columna de pepsina y columna analítica no son necesarios para el análisis de proteínas intactas.
- Ajuste la temperatura en el sistema de refrigeración a ≤0 ° C y espere a que el sistema alcance una temperatura de funcionamiento estable de ~ 0 ° C.
- Transferir rápidamente las muestras de -80 ° C en nitrógeno líquido para el análisis de espectrometría de masas. El uso de pinzas, retirar cuidadosamente cada vial del nitrógeno líquido y reconstituir la muestra mediante la adición de un volumen específico de hielo frío tampón de enfriamiento rápido que contiene ácido fórmico al 0,2% (FA) (pH 2,5) y 5% de metanol en agua al vial.
- Programe un HPLC adecuado y el método de espectrometría de masas utilizando el software de control. Para las formulaciones Mb, muestra desalt que contiene 20 pmol Mb en la trampa de proteína para 1,7 min con 5% de acetonitrilo, 95% de agua y 0,1% de ácido fórmico (FA), y eluyen usando un gradiente aumentó a 80% de acetonitrilo, 20% de agua y 0,1 % FA en 3,3 min. Recoge los espectros de masas en todo el rango m / z 200-3,200.
- Para determinar la masa de intactaproteínas, adquirir datos de una muestra de proteína no deuterado (es decir, la proteína no sometido a ssHDX) en solución acuosa utilizando el método de la etapa 2.9.
- Obtener las masas de las muestras no deuterado y deuterados por deconvoluting los espectros brutos, utilizando el software de análisis de datos. En este caso, ajuste el rango de masas de 15,000-18,000 Da, la resolución de masa de 1,0 Da, y la altura del pico de 90% para el cálculo de la masa de Mb.
- Calcular el número de deuterones incorporados en la proteína intacta (aquí, Mb) restando la masa de la proteína no deuterado de la masa de proteína deuterado en cada punto de tiempo de intercambio.
- Calcular la absorción por ciento de deuterio con relación a la máxima teórica utilizando la siguiente ecuación (ecuación 1)
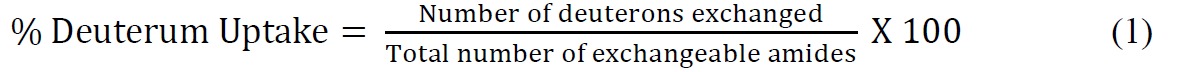
donde el número total de amidas intercambiables = número total de aminoácidos - número de residuos de prolina - 2 ("2" representa la N-grupo amino terminal y el hidrógeno amida que se someten a un rápido intercambio de vuelta). - Montar los datos cinéticos ssHDX utilizando una ecuación exponencial adecuado. Una ecuación biexponencial (Ecuación 2) es generalmente la más simple que proporciona un ajuste razonable a los datos ssHDX. En este estudio, para el MBT y MBS, ajustar los datos a un modelo biexponencial que asigna deuterones a "rápido" y "lento" el intercambio de piscinas.
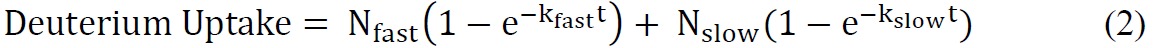
donde N rápido y lento N son el número de amidas intercambiables en el "rápido" y "lento" el intercambio de piscinas, respectivamente, yk rápido y lento k son las constantes de velocidad de primer orden asociadas a las dos piscinas.
3. ssHDX-MS para la Proteína en Péptido Nivel
- Realizar ssHDX siguiendo los pasos 2.1 a 2.8, con las siguientes modificaciones en el paso 2.6. Conecte el col pepsina inmovilizadaUMN y la columna analítica a la válvula como se informó anteriormente 25 y reemplazar la trampa proteína conectado a la válvula con una trampa de péptido. Calibrar el espectrómetro de masas mediante el establecimiento de la relación masa a carga que van desde 100 a 1.700.
NOTA: Durante la etapa de reconstitución (paso 2.8), un agente desnaturalizante y agente reductor se puede incluir en la memoria intermedia de enfriamiento para facilitar la digestión con pepsina de proteínas con enlaces disulfuro (por ejemplo, anticuerpos monoclonales). - La programación de una HPLC adecuado y el método de espectrometría de masas utilizando el software de control. Para formulaciones Mb, digerir las muestras que contienen 20 pmol Mb en línea con el 0,1% FA, atrapar y desalar péptidos de 1.7 min con acetonitrilo al 10%, el 90% de agua y un 0,1% FA en una trampa de péptidos. La elución de los fragmentos en la columna analítica con un aumento de gradiente a 60% de acetonitrilo, 40% de agua y 0,1% FA en 4,0 min. Adquirir los espectros de masas en todo el rango m / z 100-1,700.
- Identificar los fragmentos pépticas por análisis MS / MS de unmuestra de proteína no deuterado. Utilice el software de la espectrometría de masas para comparar masas experimentales de iones de fragmentos de péptidos a las masas predichos de fragmentos de péptidos en una base de datos personalizada. Establezca un punto de masa (por ejemplo, 10 ppm) de corte para identificar masas con baja error. Para los péptidos emparejados, preparar una lista compuesta de (i) la secuencia del péptido, (ii) el estado de carga, y (iii) tiempo de retención.
- Utilice la lista generada en el paso 3.3 para mapear y determinar el número promedio de deuterones incorporados para cada fragmento de pepsina digerir. Esto se puede lograr mediante el empleo adecuado HDX-MS software de análisis de datos 24.
- Para calcular la absorción de deuterio por ciento y para ajustar los datos cinéticos ssHDX para cada uno de los fragmentos pépticas, siga los pasos 2.13 y 2.14. En este estudio, HDX datos cinéticos para cada uno de seis pepsina no redundante digieren los fragmentos de formulaciones se ajustaron MBT y MBS a un modelo de asociación biexponencial (Ecuación 2).
4. SSPL-MS para Intacto Protein
- Para comenzar la reacción de marcaje fotolítico, encienda por primera vez el agente de reticulación UV y permitir que las lámparas se calienten durante 5 min. Asegúrese de que la fuente de UV está equipado con lámparas de longitud de onda de 365 nm para activar el grupo diazirina de pleu del Sáhel.
PRECAUCIÓN: No abra la puerta del agente de reticulación UV cuando las luces están encendidas. Proteger los ojos y la piel de la exposición a la luz UV si la fuente no está encerrado por una puerta de cristal de protección UV. - Apague el agente de reticulación UV antes de abrir la puerta. Una vez que las lámparas se apagan, destape los viales que contienen la formulación liofilizada y colocarlos dentro de la cámara de agente de reticulación UV, como se muestra en la Figura 2B. Irradiar las muestras con luz UV durante 40 minutos.
- Realizar experimentos de control siguiendo el paso 4.1 a 4.3 para (i) de muestras liofilizadas sin pleu del Sáhel y (ii) de muestras liofilizadas con pleu del Sáhel reconstituyeron en agua.
- Cap y almacenar los viales a -20 ° C hasta su análisis por espectrometría de masas.
- ReconstitUTE La muestras sólidas mediante la adición de un volumen adecuado de agua destilada de grado espectrometría de masa para llevar la concentración de 2 mM.
- Para comenzar el análisis de muestras, siga los pasos 2.6 y 2.9.
NOTA: Desde atrás de intercambio no es un problema con el etiquetado covalente, SSPL-MS no requiere un sistema especial LC refrigerado. - Para determinar la masa nativa de la proteína, para adquirir datos de una muestra de proteína que no ha sido sometido a SSPL siguiendo el paso 2.9. Obtener las masas de las muestras no marcados y etiquetados por deconvoluting los espectros prima como se explica en el paso 2.11.
- Calcular el número de pleu del Sáhel Incorporated utilizando la siguiente fórmula:
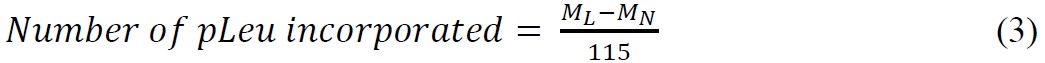
donde M L es la masa de proteína marcada, M N es la masa de la proteína nativa y 115 es la masa media (Da) añadió a la proteína nativa siguiente single incorporación pleu del Sáhel. Tenga en cuenta que la reacción de marcaje se produce con ésimoe pérdida de N 2 (28 Da). La masa monoisotópico de pleu del Sáhel es 143.07. - Calcular los porcentajes de poblaciones de proteínas con diferentes números de etiquetas usando alturas de los picos de los cromatogramas de iones extraídos.

donde "i" indica el número de etiquetas, PH i denota la altura del pico de proteína marcada L i y PH u denota la altura del pico de la proteína no marcada como se observa por espectrometría de masas.
5. SSPL-MS para la proteína en el nivel de péptidos
- Realizar SSPL siguiendo los pasos 4.1 a 4.4.
- Para el análisis de nivel de péptido, reconstituir las muestras de sólidos en tampón de bicarbonato de amonio (100 mM, pH 8,0).
- Después de la reconstitución, mezclar la solución de proteína marcada con tripsina a 10: 1 relación molar de la proteína a la tripsina y se incuba a 60 ° C durante 16 hr.
- Interrumpir la reacción mediante la adición de 0,1% FAen agua a la muestra para dar una concentración final de 2 mM proteína.
- Conectar el bucle de muestra, trampa de péptido y la columna analítica a la válvula vinculada al sistema de HPLC.
- Programe un HPLC adecuado y el método de espectrometría de masas utilizando el software de control. Para las formulaciones Mb, inyectar 20 pmol de la proteína digerida en el bucle de muestra, y desalar los péptidos en una trampa de péptido durante 1,5 min con 5% de acetonitrilo, 95% de agua y 0,1% FA, seguido de una elución en la columna analítica con gradiente aumentar a 55% de acetonitrilo, 45% de agua y 0,1% FA en 22 min. Recoge los espectros de masas en todo el rango m / z 100-1,700.
- Prepare una lista masa teórica de péptido-pleu del Sáhel aductos utilizando una herramienta en línea como ExPASy 26 con número de pleu del Sáhel previamente calculadas a partir del análisis de proteínas intactas. Incluir al menos 4 divisiones perdidas. Tenga en cuenta que la reacción de marcaje se produce con la pérdida de N 2. Por lo tanto, la masa de péptido-pleu del Sáhel aducto = masa de prueba no marcadopéptido PTIC + n (masa de pleu del Sáhel) - n (masa de N 2), donde "n" es el número de pleu del Sáhel incorporado.
NOTA: Si el análisis masivo de proteínas intactas mostró hasta tres poblaciones marcadas de proteína, considere hasta tres posibles etiquetas pleu del Sáhel por péptido. Para un péptido con masa de 1000 Da, la masa teórica de péptido-pleu del Sáhel aducto con 1 pleu del Sáhel incorporación sería 1000 + 1 (143) - 1 (28) = 1115 Da. Del mismo modo, las masas teóricas de péptido-pleu del Sáhel aductos con 2 pleu del Sáhel, 3 pleu del Sáhel, etc. sería 1.230 Da, 1.345 Da, etc., respectivamente. - Utilice el software de análisis de masas para que coincida con la lista masa teórica generada en el paso 5.7 con las masas observadas experimentalmente. Establezca un punto de masa (por ejemplo, 50 ppm) de corte para identificar masas con baja error.
- Para los péptidos emparejados, determinar el número real de pleu del Sáhel incorpora usando la fórmula en el paso 4.8 (Ecuación 3), donde M L y M N son las masas de marcadoy el péptido nativo, respectivamente.
Resultados
Aquí, ssHDX-MS-MS y SSPL se han utilizado para estudiar el efecto de los excipientes sobre la conformación y de estado sólido interacciones de liofilizados formaciones MB. Las concentraciones de proteína y los excipientes utilizados en este estudio se dan en la Tabla 1. Los resultados representativos del análisis ssHDX-MS-MS y SSPL de Mb liofilizados obtenidos siguiendo los protocolos anteriores se presentan.
Captación de deuterio a nivel proteína intacta
ssHDX-MS es capaz de distinguir entre las formulaciones Mb a nivel intacto. Los espectros de masas de deconvoluted Mb intactas siguientes 144 horas de ssHDX desde MBS formulación mostró una mayor captación de deuterio que la formulación MBT (Figura 3A). En promedio, MBS mostraron 46% mayor captación deuterio que el MBT (Tabla 2).
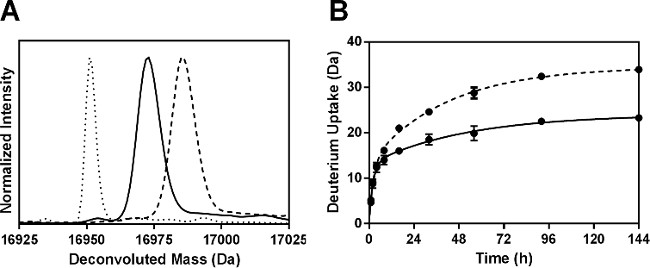
Figura 3: ssHDX-MS para Mb intacta: (A) deconvoluted espectros de masas de Mb intactas deuterados de formulaciones MBT (línea continua) y MBS (línea discontinua) después de 144 horas de ssHDX. El espectro de masas de deconvoluted Mb intactos no deuterado también se muestra (línea de puntos). (B) la cinética ssHDX para Mb intacta en formulaciones MBT (línea continua) y MBS (línea discontinua). El curso temporal de ssHDX se ajustó a una ecuación para dos fase de asociación exponencial utilizando la versión de software Graph Pad Prism 5 (n = 3, ± SD).
La cinética de deuteración para MBS intactas y MBT son similares en puntos de tiempo tempranos (1-4 h), pero MBS mostraron una mayor intercambio de deuterio con el aumento en el tiempo (8-144 h) (Figura 3B). Esto sugiere la importancia de seleccionar los puntos de tiempo más largos para ssHDX en condiciones inferiores de humedad relativa y temperatura. Además, el proceso de sorción y difusión 2 O D puede afectar la tasa de ssHDX en la po tiempo tempranoints. Nuestros estudios previos han demostrado que la absorción de humedad en ssHDX es completa en un período de horas, y tiene contribución mínima para intercambiar cinética más allá de este tiempo. La tasa observada y medida de cambio, por lo tanto no son simplemente medidas de D 2 O adsorción 27,28. Las pequeñas barras de error en la Figura 3B, con indicación de las desviaciones estándar de tres muestras ssHDX-MS independientes, muestran que el experimento es altamente reproducible.
| El deuterio Captación (%) b | N rápido c | k c rápido | N lenta c | k c lenta | |
| MBT un | 15,9 ± 0,5 | 13,1 (0,8) | 0,43 (0,03) | 11,0 (0,9) | 0.019 (0.001) |
| MBS una | 23,2 ± 0,5 | 15,4 (0,7) | 0,49 (0,04) | 19,2 (0,6) | 0,024 (0,002) |
| % Cambio d | 46% | 18% | 14% | 75% | 26% |
Tabla 2:. Las medidas cuantitativas de la captación de deuterio en estudios ssHDX de formulaciones Mb una Ver Tabla 1 para la composición de deuterio b Porcentaje captación relativa a la máxima teórica por Mb intactas después de 144 horas de HDX a 5 ° C, 43% HR (n = 3. , Parámetros de media ± DE). c determinados por regresión lineal de los datos cinéticos ssHDX-MS. Evolución temporal de intercambio de deuterio para Mb intacta fue ajustado a un modelo de asociación biexponencial (. Ecuación 2). Los valores entre paréntesis son los errores estándar de los parámetros de regresión. D El porcentaje de cambio en las mismas estuvieron calculATED como 100 x [(valor de los MBS - valor del MBT) / (valor de MBT)].
Los parámetros de regresión (N rápido, N lento, k rápido y lento k) para la cinética de absorción de deuterio para MBT y MBS se dan en la Tabla 2. Aunque el N valores rápidos y N lentas son más grandes de MBS que MBT, las diferencias en la N lenta valores fueron mayores que las diferencias en los valores Fast N. En concreto, el valor rápido N es sólo un 18% mayor en MBS que en MBT, mientras que el valor lento N es 75% mayor en MBS que en MBT. Esto sugiere que los N valores más pequeños en lentas MBT puede ser debido a la mayor retención de la estructura o la protección de grupos amida Mb por excipientes que están expuestos a D 2 O en MBS. Sin embargo, los mecanismos detallados no se entienden claramente. Las constantes de velocidad (k rápido y lento k) para ambas formulaciones son muy similares.
la captación de deuterio en el nivel de péptido
Después de la digestión con pepsina, se identificaron un total de 52 péptidos. Seis fragmentos no redundantes correspondientes a 100% de la secuencia Mb se utilizaron para el análisis reportados aquí. Información adicional se puede obtener mediante el uso de fragmentos superpuestos, según lo informado por nuestro grupo con anterioridad 24. Se calculó la captación por ciento de deuterio para cada péptido y los resultados de 144 muestras hr traza (Figura 4A). Cinética de HDX para los seis fragmentos pépticas mostraron un comportamiento biexponencial (Figura 4B), consistente con subpoblaciones de hidrógenos amida sometidos a "rápido" y "lento" de cambio.
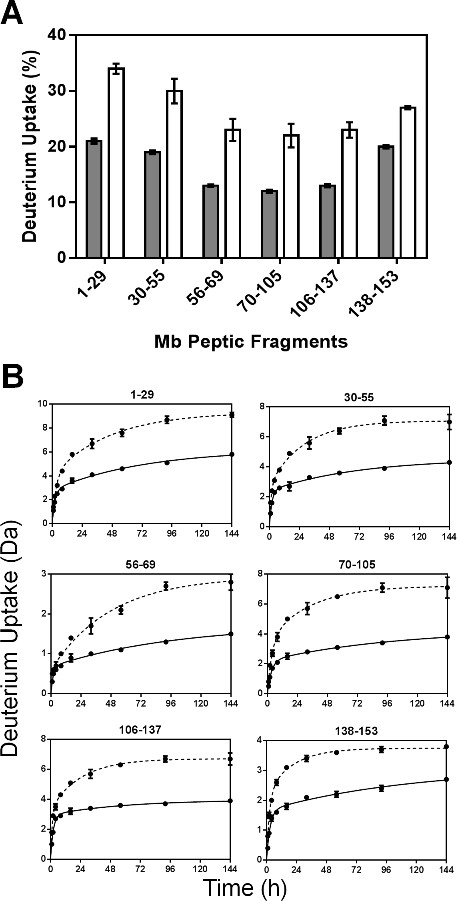
Figura 4: ssHDX-MS para Mb en el nivel de péptido: (A) la captación de deuterio Porcentaje de 6 no redufragmentos pépticas ndant de Mb en formulaciones MBT (gris) y MBS (blanco) después de 144 horas de HDX. cinética (B) ssHDX para seis fragmentos pépticas no redundantes de Mb en formulaciones MBT (línea continua) y MBS (línea discontinua). El curso temporal de ssHDX se ajustó a una ecuación para dos fase de asociación exponencial utilizando la versión de software Graph Pad Prism 5 (n = 3, ± SD).
Los parámetros de regresión para los péptidos no redundantes se presentan en la Figura 5. Como las constantes de velocidad para armarios fragmentos peptídicos no son la constante de velocidad promedio para las amidas individuales, las constantes de velocidad observadas para los fragmentos pépticas no pueden ser linealmente relacionadas a las de la proteína intacta. Los N valores rápido para la mayoría de los fragmentos pépticas (excepto fragmento 56-69) en las formulaciones de MBS fueron ligeramente mayores que los de MBT (Figura 5A). Del mismo modo, los valores de k rápidas generalmente mostraron poca diferencia entre formulations y en diferentes regiones de la molécula Mb (Figura 5B). Sin embargo, los N valores lento lento y K para el MBS son significativamente mayores en todos los fragmentos que para MBT (Figura 5C y 5D). El considerable aumento de N lento y k lento para MBS puede reflejar una mayor movilidad de los grupos amida en los "lentos" piscinas intercambio.
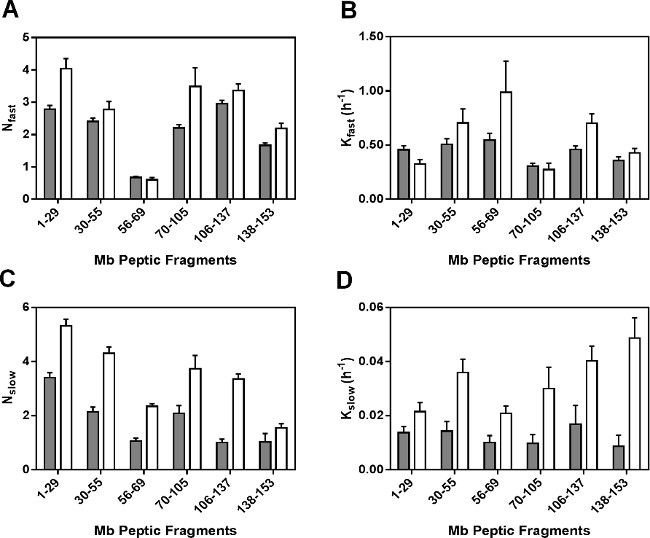
Figura 5: parámetros cinéticos ssHDX para Mb péptidos pépticas: N rápido (A), k rápido (B), N lento (C) y k valores lentos (D) obtenidos de la regresión lineal de los datos cinéticos ssHDX-MS durante seis péptidos pépticas no redundantes de Mb en formulaciones MBT (gris) y MBS ( blanco) (n = 3, ± SE).
Etiquetado Fotolítica a nivel de proteína intacta
Mb irradia en presencia de exceso de 20x pleu del Sáhel formado Mb-pleu del Sáhel múltiples aductos, como se detecta por LC-MS (Figura 6A). Los espectros deconvoluted para MBT irradió durante 40 minutos con 20x pleu del Sáhel mostró hasta 3 etiquetas con la adición de 115, 230 y 345 Da a la masa de Mb sin etiquetar. MBS irradiados de manera similar con 20x pleu del Sáhel mostraron menos absorción pleu del Sáhel a nivel intacta, con un máximo de 2 poblaciones marcados detectados por LC-MS.
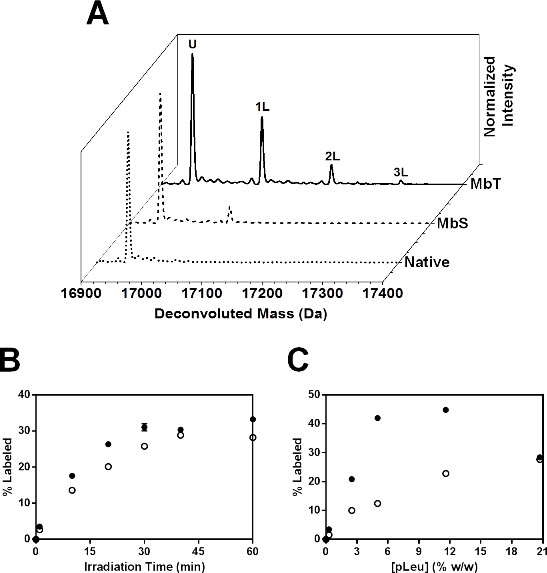
Figura 6: SSPL-MS para Mb intacta: (A) deconvoluted espectros de masas de MBT (línea continua) y MBS (línea discontinua) etiquetados con 20x exceso (5% w / w) pleu del Sáhel. Deconvoluted espectro de masas del Mb nativo (Mb liofilizó y se irradió en ausencia de pleu del Sáhel) se muestra como la línea de puntos. U denota una población de proteína que queda sin marcar después de la irradiación. Las poblaciones de proteína transportadora de 1, 2 y 3 etiquetas pleu del Sáhel se representan como 1L, 2L y 3L respectivamente. cinética (B) SSPL-MS para Mb intactos en formulaciones MBT (círculos cerrados) y MBS (círculos abiertos) como una función de la concentración pleu del Sáhel. Todas las muestras se irradiaron durante 40 min. Las barras de error están dentro de los símbolos. (C) SSPL-MS cinética para Mb intactos en formulaciones MBT (círculos cerrados) y MBS (círculos abiertos) liofilizó y irradiados en la presencia de exceso de 100x pleu del Sáhel (20,7% w / w) como una función del tiempo de irradiación. Las barras de error están dentro de los símbolos.
En estudios cinéticos, el porcentaje de proteína marcada aumentó exponencialmente tanto para MBT y MBS con el aumento de tiempo de irradiación (Figura 6B). MBS mostraron menos captación pleu del Sáhel de MBT en cada tiempo de irradiación. Ambas formulaciones parecían llegar a una meseta a 40 min. Así, un estudio cinético puede ser nosotros eful para determinar la duración de la irradiación necesario para obtener la activación completa pleu del Sáhel. La cinética de etiquetado también se estudiaron como una función de la concentración pleu del Sáhel (Figura 6C). El porcentaje de proteína marcada aumentó con la concentración pleu del Sáhel tanto MBT y MBS. Sin embargo, en 20,7% w / w pleu del Sáhel, MBT mostró una disminución en la captación de pleu del Sáhel. Esto puede ser debido a la exclusión de pleu del Sáhel de la superficie de la proteína a alta concentración pleu del Sáhel. Por lo tanto, un estudio con diferentes concentración de pleu del Sáhel se debe realizar para seleccionar la concentración pleu del Sáhel apropiado que permite el etiquetado suficiente a través de la superficie de la proteína sin exclusión superficie. En este estudio, 20x exceso pleu del Sáhel fue seleccionado para realizar nuevos estudios a nivel de péptido.
El general disminuyó etiquetado observado para MBS sugiere mala accesibilidad de la cadena lateral de la matriz que contiene pleu del Sáhel. Esto es consistente con un cambio conformacional en la presencia de sorbitol que resulta en el etiquetado reducida.
contenido "> etiquetado Fotolítica a nivel péptidoCon base en los estudios de etiquetado proteínas intactas, 20x exceso pleu del Sáhel fue seleccionado para comparar MBT y MBS en el nivel de péptidos. Se digirieron muestras marcadas con tripsina y se analizaron por LC-MS. Se detectaron un total de 40 péptidos correspondientes a 100% de la secuencia Mb para muestras MBT y MBS. En algunos casos, la digestión tríptica puede proporcionar cobertura limitada secuencia de la proteína si residuos de Lys y / o Arg están fuertemente etiquetados. Para mejorar la cobertura de secuencia, una mezcla de tripsina y quimotripsina puede ser utilizado para digerir la proteína marcada.
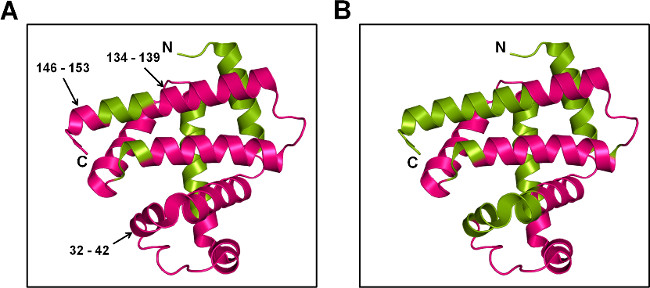
Figura 7: SSPL-MS para Mb a nivel de péptido: la representación de dibujos animados de Mb etiquetado con 20x exceso de pleu del Sáhel (5% w / w) en presencia de trehalosa (A) y sorbitol (B). La proteína marcada se digirió con trypsin y péptidos marcados fueron asignadas a la estructura cristalina de Mb (PDB ID 1WLA). Las regiones etiqueta y etiqueta son de color magenta y verde, respectivamente.
SSPL-MS con digestión con tripsina ofrece información cualitativa sobre los péptidos están etiquetados. Dadas las diferentes poblaciones marcadas a nivel intacta, el mecanismo de etiquetado promiscua pleu del Sáhel y diferencias en la eficiencia de ionización de péptidos marcados y no marcados, es difícil obtener mediciones cuantitativas para SSPL-MS después de la digestión. Sin embargo, la información cualitativa todavía puede dar una idea de los cambios conformacionales de proteínas a nivel de péptido. En este estudio, las formulaciones tanto MBT y MBS mostraron pleu del Sáhel absorción a través de la mayor parte de la superficie de la proteína. Cuando se compara con MBS, fragmentos de péptidos 32-42, 134-139 y 146-153 de MBT mostraron etiquetado pleu del Sáhel (Figura 7). Esto sugiere que las cadenas laterales de estos aminoácidos están expuestos a pleu del Sáhel, como las hélices en estos regions están intactos en la matriz de MBT. En contraste, la protección de etiquetado pleu del Sáhel en la matriz de MBS es consistente con perturbaciones estructurales en estas regiones.
En general, los resultados de ssHDX-MS-MS y SSPL sugieren que los métodos pueden proporcionar información de alta resolución a nivel de péptido complementario sobre la columna vertebral (ssHDX-MS) y la cadena lateral (SSPL-MS) de exposición y efectos de excipiente en las formulaciones de proteínas liofilizadas .
Discusión
Varios estudios han sugerido que el entorno local en muestras liofilizadas afecta a la degradación de proteínas 5,29,30. Sin embargo, establecer una relación directa entre la estructura de la proteína y la estabilidad en el estado sólido no ha sido posible debido a la falta de métodos analíticos de alta resolución. La aplicación de métodos de alta resolución existentes, tales como HDX y PL a polvos liofilizados requiere la modificación de los protocolos de la solución y la interpretación de los datos cuidado. HDX-MS y PL-MS han sido adoptados sucesivamente para monitorear conformaciones de las proteínas en el estado sólido. Los resultados presentados aquí y en otros lugares 27,28,31-33 han demostrado la capacidad de estos métodos para controlar la conformación de proteínas con alta resolución en el entorno sólido. Aunque los pasos críticos en el análisis de los datos no varían de etiquetado en solución 34-36, se requieren consideraciones importantes durante la instalación experimental y la interpretación de datos para químico de estado sólidoetiquetado cal.
La selección del reactivo de marcaje debe basarse en el tamaño y el mecanismo de etiquetado. El pequeño tamaño de deuterio permite que el esqueleto peptídico a palpar fácilmente, mientras que el tamaño relativamente grande de pleu del Sáhel limita etiquetado a las cadenas laterales. Tanto ssHDX y SSPL no muestran ninguna preferencia por cualquier aminoácido, por lo que el etiquetado depende sólo de la columna vertebral y de la cadena lateral de la exposición a la matriz. Para sondear con eficacia conformaciones de proteínas en estado sólido, los factores externos que afectan el proceso de etiquetado deben ser cuidadosamente controlados. La cantidad total y la distribución espacial de agente de marcaje en los sólidos liofilizados es diferente de soluciones acuosas.
En ssHDX, la cantidad de D 2 O en la matriz sólida puede afectar la tasa de desplegamiento de la proteína (o despliegue parcial), el replegamiento, y el intercambio de deuterio. Este no es el caso con solución HDX, en el que la muestra de proteína normalmente se diluye con un volumen amplio de D 2 O.Detección cuidadosa de los efectos de la hidratación en la tasa de ssHDX puede informar a la selección de las condiciones ideales de SR. Para controlar la velocidad de absorción de humedad y evitar el colapso del polvo en formulaciones que contienen excipientes higroscópicos (por ejemplo, sacarosa y trehalosa), ssHDX puede llevarse a cabo bajo condiciones de refrigeración (2-8 ° C). Nuestro estudio anterior sobre los efectos de hidratación mostró velocidad y el grado de intercambio aumentó con el aumento en el contenido de humedad, como se esperaba. En gran parte de nuestro trabajo, una HR intermedio del 43% a los 5 ° C ha demostrado ser ideal para distinguir formulaciones en un tiempo razonable 24. La reacción se lleva a cabo normalmente hasta que se alcanza una meseta. Esto asegura que la sorción de humedad y la difusión en el sólido no controlan la tasa de HDX. El uso de tamaños de muestra pequeños sólidos con un volumen de pre-liofilización de ≤2 ml también ayuda a asegurar que D 2 O sorción de vapor es esencialmente completa al comienzo del período de cambio. Aunque ssHDX-MS ofreceinformación cuantitativa sobre la conformación de la proteína en estado sólido, hay ciertas condiciones en que la interpretación de los datos no puede basarse exclusivamente en el estudio ssHDX solo. Es posible que la absorción de deuterio disminución observada en una muestra (en comparación con el control) puede ser debido a la mayor retención de la estructura de la proteína o la cantidad significativa de agregados de proteínas presentes en la muestra. En tal caso, la interpretación de los datos ssHDX requiere los resultados de otros métodos complementarios. Se observó ensanchamiento de los picos en los espectros de masas deuterado para varias formulaciones Mb 27,28. Esto podría ser debido a varios factores como la presencia de la población de proteína parcialmente desplegada, la heterogeneidad espacial en la muestra, o los gradientes espaciales de la concentración de D 2 O. Sin embargo, estos factores no se distinguían en ssHDX-MS y necesita más investigación.
Como SSPL-MS es relativamente nueva en comparación con otros métodos, el aprendizaje continuo absus aplicaciones y limitaciones que se requiere. En SSPL, el foto-reticulante se liofiliza con la proteína. La falta de humedad limita la movilidad de los componentes dentro de la matriz sólida, y la relajación estructural parcial que puede ocurrir con sorción de humedad en ssHDX no es un fenómeno en SSPL. Esto limita el etiquetado en SSPL a la vecindad inmediata de la foto-reticulante. Sin embargo, a diferencia de HDX-MS, MS / MS análisis de la proteína covalentemente etiquetados puede proporcionar información estructural a nivel de residuos. Desde etiquetado SSPL es covalente e irreversible, de vuelta de cambio no se produce y se puede preparar muestras y se analizaron sin la preocupación por la pérdida de la etiqueta. Para facilitar la difusión de agente de marcaje y mejorar la eficiencia de etiquetado en matriz sólida, SSPL se puede realizar con el aumento de% de humedad relativa. pleu del Sáhel absorción también se puede mejorar mediante el aumento de la concentración de agente fotorreactivo. La relación molar de proteína para pleu del Sáhel puede variarse según se desee. En general, un exceso molar de 100x pleu del Sáhel a protein asegurará un etiquetado adecuado. Sin embargo, la alta concentración pleu del Sáhel puede resultar en la pérdida de la estructura terciaria de proteínas en la matriz sólida. Por lo tanto, además de la cinética de etiquetado y composición de la formulación, la selección de la concentración de pleu del Sáhel debe también estar basada en el mantenimiento de la integridad estructural de la proteína. Como pleu del Sáhel etiquetas no selectiva XH (donde X = C, N, O), es posible que los excipientes con sitios de etiquetado similares pueden afectar en gran medida el nivel de etiquetado de proteínas. La interferencia de excipientes en la disponibilidad de pleu del Sáhel para el etiquetado de proteínas aún no se ha caracterizado. Se sabe que el carbeno generada a partir de la activación no se diazirina residuo específico, sin embargo un estudio informa sesgo hacia Asp y Glu 36. Mientras que es bueno para aprender acerca de las interacciones de residuos específica, la información de péptido nivel también es útil y se puede utilizar para diseñar excipientes para bloquear regiones con alta exposición de la matriz en el estado sólido. SSPL-MS proporciona información cualitativa detallada, sin embargodatos cuantitativos debe ser obtenida y métricas robustas necesita ser desarrollado para analizar las diferencias de formulación a través de una variedad de sistemas liofilizados.
El uso de una etiqueta de residuo específico combinado con el análisis MS / MS se puede mejorar aún más la resolución al nivel de aminoácidos. Reactivos de etiquetado tales como 2,3-butanodiona para etiquetar Arg, N hidroxisuccinimida derivados de Lys y N derivados -alkylmaleimide para Cys se puede utilizar para mapear con precisión las interacciones moleculares en polvo liofilizado. Sin embargo, estos reactivos son dependiente del pH y las reacciones pueden no ser tan bien controlados como el etiquetado fotolítica en estado sólido. Un enfoque alternativo es incorporar el foto-reticulante en la secuencia de proteína con el uso de líneas de células auxotróficas, mutagénesis dirigida al sitio o derivatización de la cadena lateral.
Nuestros estudios previos ssHDX-MS-MS y SSPL han demostrado que el etiquetado de proteínas depende de la naturaleza y cantidad de excipientesutilizado 24,27,28,31-33,37,38. ssHDX-MS de Mb coliofilizado con clorhidrato de guanidina (Gdn.HCl) mostraron una mayor captación de deuterio que Mb coliofilizado con bajo peso molecular que no contienen azúcar 32. En un estudio SSPL-MS separada, Mb coliofilizado con Gdn.HCl mostró una mayor protección de etiquetado fotolítico de Mb con sacarosa 33. Además, las mediciones cuantitativas de ssHDX-MS han sido altamente correlacionado con la estabilidad de la proteína durante el almacenamiento a largo plazo 28. Estos estudios sugieren que ssHDX o SSPL de la proteína refleja la medida de la retención estructural de la proteína en polvo liofilizado. Creemos que la retención de la estructura secundaria en polvos liofilizados proporciona un entorno favorable para el etiquetado de cadena lateral con pleu del Sáhel y protección de hidrógeno amida de intercambio de deuterio. Sin embargo, la comparación detallada del contenido informativo de estos métodos se debe realizar en el futuro. Aunque el establecimiento de la utilidad de ssHDX-MS-MS y SSPLcomo herramienta de detección formulación finalmente exigir que se aplica a muchas proteínas, los resultados de nuestros estudios recientes apoya su adopción más amplia. Con un mayor desarrollo, se espera que estos métodos son ampliamente útil para caracterizar las formulaciones de proteínas de estado sólido en la industria biofarmacéutica.
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
The authors gratefully acknowledge financial support from NIH R01 GM085293 (PI: E. M. Topp) and from the College of Pharmacy at Purdue University.
Materiales
| Name | Company | Catalog Number | Comments |
| Equine myoglobin | Sigma-Aldrich | M0630-5G | |
| D-(+)-Trehalose dihydrate | Sigma Aldrich | #T9531 | |
| D-Sorbitol | Sigma Aldrich | #240850 | |
| L-Photo-leucine | Thermo Scientific | #22610 | |
| Potassium phosphate monobasic | Sigma-Aldrich | #P0662 | |
| Potassium phosphate dibasic | Sigma-Aldrich | #P3786 | |
| Deuterium Oxide | Cambridge Isotope Laboratories | #DLM-4-PK | Alternate (Cat. No.: 151882, Sigma-Aldrich) |
| Immobilized pepsin | Applied Biosystems | #2-3132-00 | |
| Trypsin | Promega | #V511A | Chymotrypsin (Cat. No.: #V1062, Promega) can be additionally used |
| Water, Optima LC/MS grade | Fisher Chemical | #7732-18-5 | |
| Acetonitrile | Sigma-Aldrich | #34998 | |
| Formic acid | Thermo Scientific | #28905 | |
| ESI-TOF Calibrant | Agilent Technologies | #G1969-85000 | Highly flammable liquid |
| Protein microtrap | Michrom Bioresources | TR1/25108/03 | |
| Peptide microtrap | Michrom Bioresources | TR1/25109/02 | |
| Analytical column | Agilent Technologies | Zorbax 300SB-C18 | |
| Freeze dryer | VirTis AdVantage Plus | ||
| Stratalinker equipped with five 365 nm lamps | Stratagene Corp. | Stratalinker 2400 | |
| HPLC | Agilent Technologies | 1200 series LC | Refrigerated LC system for HDX-MS |
| ESI-qTOF MS | Agilent Technologies | 6520 qTOF | |
| HDExaminer (HDX-MS data analysis software) | Sierra Analytics | http://www.massspec.com/HDExaminer.html |
Referencias
- Lawrence, S. Billion dollar babies--biotech drugs as blockbusters. Nat. Biotechnol. 25 (4), 380-382 (2007).
- . . Global Markets and Manufacturing Technologies for Protein Drugs. , BIO021D (2013).
- Lai, M. C., Topp, E. M. Solid-state chemical stability of proteins and peptides. J. Pharm. Sci. 88 (5), 489-500 (1999).
- Carpenter, J. F., Pikal, M. J., Chang, B. S., Randolph, T. W. Rational design of stable lyophilized protein formulations: some practical advice. Pharm. Res. 14 (8), 969-975 (1997).
- Carpenter, J. F., Chang, B. S., Garzon-Rodriguez, W., Randolph, T. W. Rational design of stable lyophilized protein formulations: theory and practice. Pharm. Biotechnol. 13, 109-133 (2002).
- Wüthrich, K. Protein structure determination in solution by NMR spectroscopy. J. Biol. Chem. 265 (36), 22059-22062 (1990).
- Ilari, A., Savino, C. Protein structure determination by x-ray crystallography. Methods. Mol. Biol. 452, 63-87 (2008).
- Brunger, A. T. X-ray crystallography and NMR reveal complementary views of structure and dynamics. Nat. Struct. Biol. 4, 862-865 (1997).
- Yu, L. Amorphous pharmaceutical solids: preparation, characterization and stabilization. Adv Drug. Deliv. Rev. 48 (1), 27-42 (2001).
- Manning, M. C. Use of infrared spectroscopy to monitor protein structure and stability. Expert. Rev. Proteomics. 2 (5), 731-743 (2005).
- Grohganz, H., Gildemyn, D., Skibsted, E., Flink, J. M., Rantanen, J. Rapid solid-state analysis of freeze-dried protein formulations using NIR and Raman spectroscopies. J. Pharm. Sci. 100 (7), 2871-2875 (2011).
- Bai, S., Nayar, R., Carpenter, J. F., Manning, M. C. Noninvasive determination of protein conformation in the solid state using near infrared (NIR) spectroscopy. J. Pharm. Sci. 94 (9), 2030-2038 (2005).
- Pikal, M. J., et al. Solid state chemistry of proteins: II. The correlation of storage stability of freeze-dried human growth hormone (hGH) with structure and dynamics in the glassy solid. J. Pharm. Sci. 97 (12), 5106-5121 (2008).
- Wang, B., Tchessalov, S., Cicerone, M. T., Warne, N. W., Pikal, M. J. Impact of sucrose level on storage stability of proteins in freeze-dried solids: II. Correlation of aggregation rate with protein structure and molecular mobility. J. Pharm. Sci. 98 (9), 3145-3166 (2009).
- Schule, S., Friess, W., Bechtold-Peters, K., Garidel, P. Conformational analysis of protein secondary structure during spray-drying of antibody/mannitol formulations. Eur. J. Pharm. Biopharm. 65 (1), 1-9 (2007).
- Baerga-Ortiz, A., Hughes, C. A., Mandell, J. G., Komives, E. A. Epitope mapping of a monoclonal antibody against human thrombin by H/D-exchange mass spectrometry reveals selection of a diverse sequence in a highly conserved protein. Protein. Sci. 11 (6), 1300-1308 (2002).
- Coales, S. J., Tuske, S. J., Tomasso, J. C., Hamuro, Y. Epitope mapping by amide hydrogen/deuterium exchange coupled with immobilization of antibody, on-line proteolysis, liquid chromatography and mass spectrometry. Rapid. Commun. Mass. Spectrom. 23 (5), 639-647 (2009).
- Pacholarz, K. J., Garlish, R. A., Taylor, R. J., Barran, P. E. Mass spectrometry based tools to investigate protein-ligand interactions for drug discovery. Chem. Soc. Rev. 41 (11), 4335-4355 (2012).
- Houde, D., Peng, Y., Berkowitz, S. A., Engen, J. R. Post-translational modifications differentially affect IgG1 conformation and receptor binding. Mol. Cell. Proteomics. 9 (8), 1716-1728 (2010).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. J. Pharm. Sci. 100 (6), 2071-2086 (2011).
- Dorman, G., Prestwich, G. D. Using photolabile ligands in drug discovery and development. Trends. Biotechnol. 18 (2), 64-77 (2000).
- Robinette, D., Neamati, N., Tomer, K. B., Borchers, C. H. Photoaffinity labeling combined with mass spectrometric approaches as a tool for structural proteomics. Expert. Rev. Proteomics. 3 (4), 399-408 (2006).
- Greenspan, L. Humidity fixed points of binary saturated aqueous solutions. Journal of Research of the National Bureau of Standards. 81A (1), 8 (1977).
- Sophocleous, A. M., Zhang, J., Topp, E. M. Localized hydration in lyophilized myoglobin by hydrogen-deuterium exchange mass spectrometry. 1. Exchange mapping. Mol. Pharm. 9 (4), 718-726 (2012).
- Keppel, T. R., Jacques, M. E., Young, R. W., Ratzlaff, K. L., Weis, D. D. An efficient and inexpensive refrigerated LC system for H/D exchange mass spectrometry. J. Am. Soc. Mass. Spectrom. 22 (8), 1472-1476 (2011).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic. Acids. Res. 31 (13), 3784-3788 (2003).
- Sophocleous, A. M., Topp, E. M. Localized hydration in lyophilized myoglobin by hydrogen-deuterium exchange mass spectrometry. 2. Exchange kinetics. Mol. Pharm. 9 (4), 727-733 (2012).
- Moorthy, B. S., Schultz, S. G., Kim, S. G., Topp, E. M. Predicting Protein Aggregation during Storage in Lyophilized Solids Using Solid State Amide Hydrogen/Deuterium Exchange with Mass Spectrometric Analysis (ssHDX-MS). Mol. Pharm. 11 (6), 1869-1879 (2014).
- Wang, W. Lyophilization and development of solid protein pharmaceuticals. Int. J. Pharm. 203 (1-2), 1-60 (2000).
- Sarciaux, J. M., Mansour, S., Hageman, M. J., Nail, S. L. Effects of buffer composition and processing conditions on aggregation of bovine IgG during freeze-drying. J. Pharm. Sci. 88 (12), 1354-1361 (1999).
- Li, Y., Williams, T. D., Schowen, R. L., Topp, E. M. Characterizing protein structure in amorphous solids using hydrogen/deuterium exchange with mass spectrometry. Anal. Biochem. 366 (1), 18-28 (2007).
- Sinha, S., Li, Y., Williams, T. D., Topp, E. M. Protein conformation in amorphous solids by FTIR and by hydrogen/deuterium exchange with mass spectrometry. Biophys. J. 95 (12), 5951-5961 (2008).
- Iyer, L. K., Moorthy, B. S., Topp, E. M. Photolytic labeling to probe molecular interactions in lyophilized powders. Mol. Pharm. 10 (12), 4629-4639 (2013).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. J. Vis. Exp. (81), e50839 (2013).
- Kaltashov, I. A., Bobst, C. E., Abzalimov, R. R. H/D exchange and mass spectrometry in the studies of protein conformation and dynamics: is there a need for a top-down approach. Anal. Chem. 81 (19), 7892-7899 (2009).
- Jumper, C. C., Schriemer, D. C. Mass spectrometry of laser-initiated carbene reactions for protein topographic analysis. Anal. Chem. 83 (8), 2913-2920 (2011).
- Li, Y., Williams, T. D., Topp, E. M. Effects of excipients on protein conformation in lyophilized solids by hydrogen/deuterium exchange mass spectrometry. Pharm. Res. 25 (2), 259-267 (2008).
- Li, Y., Williams, T. D., Schowen, R. L., Topp, E. M. Trehalose and calcium exert site-specific effects on calmodulin conformation in amorphous solids. Biotechnol. Bioeng. 97 (6), 1650-1653 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados