Method Article
Mass spettrometrica Approcci alla struttura studio e interazioni proteina in liofilizzati Polveri
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Here, we present detailed protocols for solid-state amide hydrogen/deuterium exchange mass spectrometry (ssHDX-MS) and solid-state photolytic labeling mass spectrometry (ssPL-MS) for proteins in solid powders. The methods provide high-resolution information on protein conformation and interactions in the amorphous solid-state, which may be useful in formulation design.
Abstract
Amide idrogeno / scambio di deuterio (ssHDX-MS) e della catena laterale fotolitico etichettatura (SSPL-MS), seguita da spettrometria di massa possono essere utili per caratterizzare formulazioni liofilizzate di proteine terapeutiche. Etichettatura seguita da adeguata digestione proteolitica permette la struttura delle proteine e le interazioni da mappare con una risoluzione a livello di peptide. Poiché la proteina elementi strutturali sono stabilizzate da una rete di legami chimici dalle principali catene e catene laterali di aminoacidi, etichettatura specifica degli atomi nei residui amminoacidici permette di comprendere meglio la struttura e la conformazione della proteina. In contrasto con i metodi di routine utilizzati per studiare le proteine in solidi liofilizzati (ad esempio, FTIR), ssHDX-MS e SSPL-MS fornisce informazioni quantitative e specifiche del sito. Il grado di incorporazione di deuterio e parametri cinetici può essere correlato a scambiare rapidamente e lentamente piscine ammide (N veloce, N lento) e riflette direttamente le degree di protein folding e struttura in formulazioni liofilizzati. Etichettatura fotolitico Stabile non subisce back-cambio, un vantaggio rispetto ssHDX-MS. Qui, forniamo protocolli dettagliati sia per ssHDX-MS e SSPL-MS, utilizzando mioglobina (Mb) come modello di proteine in formulazioni liofilizzati contenenti sia trealosio o sorbitolo.
Introduzione
Farmaci proteici sono il settore in più rapida crescita del settore biofarmaceutico e offrono promettenti nuovi trattamenti per le malattie precedentemente incurabili, tra cui disturbi ormonali, tumori e malattie autoimmuni 1. Nel 2012, il mercato globale ha raggiunto biotherapeutics 138.000 milioni dollari e si prevede di raggiungere i 179 miliardi dollari entro il 2018 2. Le proteine sono più grandi e più fragile rispetto ai tradizionali farmaci a piccole molecole e così sono più sensibili a molti tipi di degrado 3. Per assicurare un'adeguata shelf-life e stabilità, farmaci proteici sono spesso formulati come liofilizzato (cioè, liofilizzato) polveri solide. Tuttavia, una proteina può ancora subire degradazione allo stato solido, in particolare se la sua struttura nativa non viene mantenuto durante il processo di liofilizzazione 4,5. Assicurare che la struttura è stata mantenuta è possibile solo se ci sono metodi analitici che possono sondare conformazione proteica nello stato solido con sufficienRisoluzione t.
Spettroscopia NMR 6 e cristallografia a raggi X 7 sono i metodi ad alta risoluzione comunemente usati per valutare la struttura delle proteine in soluzione e cristalline solidi 8. A causa della natura degli eccipienti e metodi di trasformazione utilizzati, formulazioni proteiche liofilizzati sono solitamente amorfi anziché cristallina 9. La mancanza di omogeneità e di ordine microscopico rende le tecniche sopra menzionate impraticabile per le proteine nei solidi amorfi. Spettroscopia in trasformata di Fourier (FTIR) 10, spettroscopia Raman 11 e spettroscopia vicino infrarosso (NIR) 12 hanno regolarmente utilizzati dall'industria biofarmaceutica di confrontare proteina struttura secondaria in polveri liofilizzate a quello della struttura in soluzione-stato nativo. Tuttavia, questi metodi sono a bassa risoluzione e in grado di fornire solo informazioni sui cambiamenti globali nella struttura secondaria. Solid-state caratterizzazione strutturale mediante FTIRha dimostrato sia debole 13,14 o poveri 15 correlazione con la stabilità di archiviazione a lungo termine. Queste limitazioni mettono in evidenza la necessità di metodi ad alta risoluzione adatte per identificare proteine perturbazioni strutturali nello stato solido.
Etichettatura chimica accoppiato con proteolisi e della spettrometria di massa è emerso come un approccio efficace per il controllo struttura delle proteine e le interazioni molecolari in soluzione acquosa. In sviluppo farmaceutico, HDX-MS è stato utilizzato per la mappatura degli epitopi nelle interazioni antigene-anticorpo 16,17, per mappare le interazioni recettore-droga 18, per monitorare gli effetti delle modificazioni post-traslazionali sulla conformazione dei farmaci proteici 19, e per confrontare lotto a lotto variazione sviluppare biosimilari 20. Analogamente, ligandi fotoattivabile sono stati utilizzati per identificare bersagli farmacologici e determinare l'affinità di legame e la specificità delle interazioni farmaco-recettore 21,22. Per inviarextend l'applicazione di questi metodi a formulazioni liofilizzati, il nostro gruppo ha sviluppato a stato solido spettrometria di massa di scambio idrogeno deuterio (ssHDX-MS) e allo stato solido spettrometria di massa etichettatura fotolitico (SSPL-MS) per studiare conformazioni di proteine e le interazioni eccipiente in campioni liofilizzati con alta risoluzione.
In entrambi ssHDX-MS e SSPL-MS, la proteina è etichettato in condizioni di reazione ideali in solidi liofilizzati, ei campioni sono poi ricostituito e analizzato mediante spettrometria di massa con o senza digestione proteolitica. ssHDX-MS fornisce informazioni sulla catena principale di esposizione al vapore di deuterio, mentre SSPL-MS fornisce informazioni sull'ambiente delle catene laterali (Figura 1). I due metodi possono perciò fornire informazioni complementari su conformazione proteica in stato solido. Qui, forniamo un protocollo generale per lo studio delle proteine nei solidi liofilizzati utilizzando ssHDX-MS e SSPL-MS (figura 2), utilizzando come Mbun modello della proteina. Noi mostriamo la capacità dei due metodi per distinguere differenze di formulazioni con due diversi eccipienti.
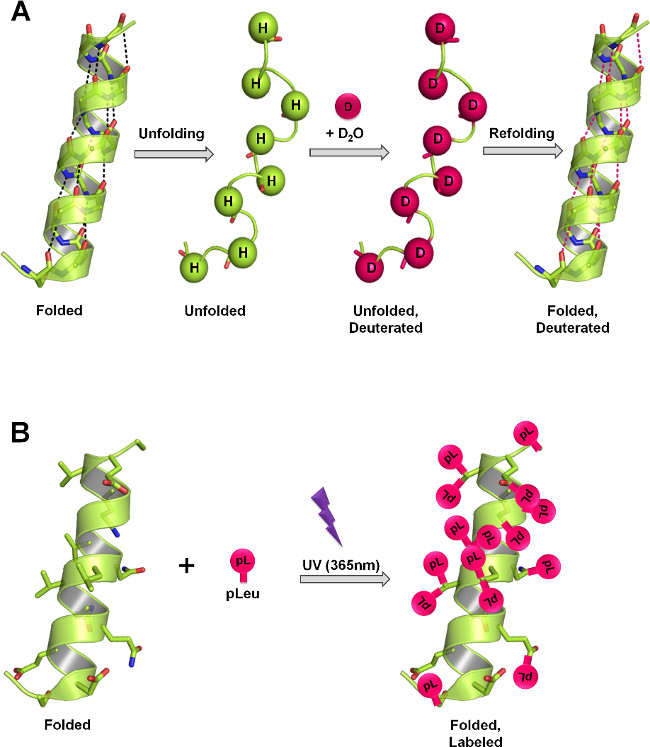
Figura 1:. SsHDX e SSPL struttura proteica misura in solidi liofilizzati attraverso diversi meccanismi di etichettatura (A) In HDX, l'ammide backbone idrogeni scambio con deuterio in funzione della struttura delle proteine e D 2 O accessibilità. Nello stato solido, la velocità ed il grado di conversione di deuterio dipendono dal livello di D 2 O assorbimento, la mobilità della proteina (e sulla ripiegatura eventi) e la natura degli eccipienti presenti nella matrice solida. (B) In PL, irraggiamento UV a 365 nm inizia la formazione di un intermedio reattivo carbene dal gruppo funzionale diazirine di pLeu ed è inserito in modo non specifico in nessuna legame XH (X = qualsiasi atomo), o added attraverso un legame C = C nelle sue immediate vicinanze. Nello stato solido, la velocità e l'entità etichettatura dipendono dalla concentrazione locale dell'agente etichettatura, tempo di irradiazione, struttura proteica e la natura degli eccipienti presenti nella matrice solida. Pannelli A e B mostrano l'etichettatura teorica massima che può verificarsi su backbone e catene laterali rispettivamente di proteine.
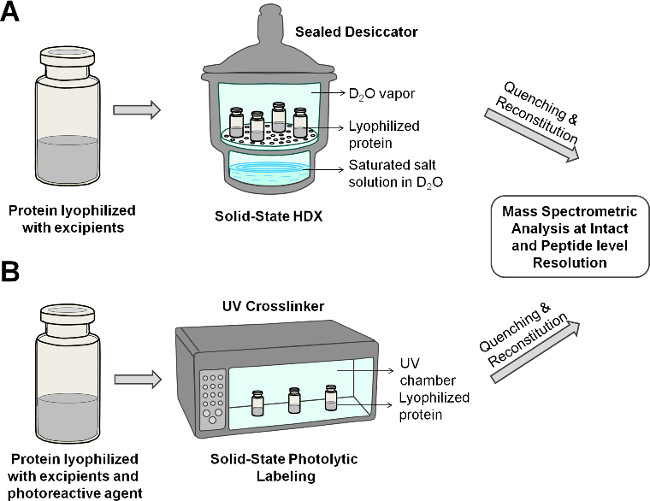
Figura 2: Schema a stato solido che mostra HDX-MS (A) e PL-MS (B) per la proteina nella formulazione liofilizzata.
Protocollo
1. Preparazione del campione e liofilizzazione
- Dializzare il volume necessario di soluzione madre Mb contro un tampone adatto e filtro attraverso un filtro sterile 0,22 micron.
- Preparare il volume richiesto di eccipienti e foto-leucina (acido L-2-amino-4,4-azi-pentanoic; pLeu) soluzioni madre a tampone adatto. Filtrare le soluzioni madre attraverso un filtro sterile 0,22 micron.
- Preparare formulazioni come mostrato nella Tabella 1 usando le soluzioni di stock di proteine, eccipienti, pLeu, e buffer.
- Filtrare i campioni attraverso un filtro sterile 0,22 micron per rimuovere eventuali particelle al punto 1.3. Riempire i campioni separatamente come 0,2 ml in fiale di vetro 2 ml. Utilizzare fiale di vetro che sono trasparenti alla luce UV (365 nm) per attivare pLeu negli studi SSPL-MS.
- Fiale di carico in un liofilizzatore e avviare liofilizzazione per la progettazione di un ciclo di liofilizzazione appropriata.
- Qui, congelarli a -40 ° C, seguitamediante essiccazione sotto vuoto primaria (70 mTorr) a -35 ° C per 12 ore ed essiccamento secondario a 25 ° C per 12 ore. Altro cicli di liofilizzazione e metodi di essiccazione (pe, spray drying) può anche essere usato.
- Backfill i flaconi contenenti campioni liofilizzati con azoto prima del livellamento.
| Formulazioni | Composizione (mg / ml) prima liofilizzazione | ||||
| Mb | Trealosio | Sorbitolo | pLeu c | Potassio, fosfato, pH 7.4 | |
| MbT un | 1.7 | 3.4 | - | - | 0.4 |
| Mbs un | 1.7 | - | 3.4 | - | 0.4 |
| MbT + pLeu b | 1.7 | 3.4 | - | 14,3 x 10 -3 a 1,43 | 0.4 |
| MBS + pLeu b | 1.7 | - | 3.4 | 14,3 x 10 -3 a 1,43 | 0.4 |
Tabella 1:... Composizione di formulazioni Mb liofilizzati a formulazioni utilizzate per lo studio ssHDX-MS Formulazioni B utilizzati per lo studio SSPL-MS c L-2-ammino-4,4-azipentanoic acido o foto-leucina (pLeu). pLeu a cinque concentrazioni diverse (14,3 x 10 -3 a 1,43 mg / ml) corrispondenti a 1x, 10x, 20x, 50x e 100x eccesso molare rispetto al Mb sono stati co-liofilizzato con MBT e Mbs formulazioni.
2. ssHDX-MS per Protein Intact
- Aggiungere una quantità saturante (~ 440 g) di K 2 CO 3 a 200 ml di D 2 O precedente ay collocato nel vano inferiore di un essiccatore. Sigillare la tenuta d'aria essiccatore e lasciarlo equilibrare a 5 ° C fino a una umidità relativa stabile (RH) del ~ 43% è raggiunto. Altri valori RH di interesse possono essere ottenuti selezionando differenti soluzioni saline sature 23,24.
- Avviare reazioni ssHDX posizionando fiale contenenti la proteina non ridotti liofilizzato nel vano superiore del essiccatore. Sigillare l'essiccatore a tenuta d'aria e incubare a 5 ° C per permettere HDX avvenga (Figura 2A).
- Raccogliere campioni ssHDX in vari momenti in triplice copia. Per formulazioni Mb, raccogliere campioni a nove punti di tempo 1, 2, 4, 8, 16, 32, 56, 92 e 144 hr.
- Tappare le fiale immediatamente dopo il ritiro dal essiccatore e placare le reazioni da Flash congelamento le fiale in azoto liquido. Conservare le fiale a -80 ° C fino a spettrometria di massa.
- Analizzare i campioni con un adeguato cromatografia-massa Spectrom alta risoluzione liquidoMetodo metria (LC-MS). Progettazione o acquistare un sistema idoneo LC refrigerato per minimizzare back-scambio durante l'analisi del campione. Utilizzare l'impostazione del gruppo frigorifero colonna e il metodo LC-MS precedentemente segnalato 25.
NOTA: Poiché il tasso di ammide scambio protonico dipende dal pH e dalla temperatura, deuteroni incorporati nella proteina possono scambiare con idrogeno presente nella fase mobile ("scambio back"), causando una perdita di informazioni. Sebbene un pH acido (pH 2,5) di tampone quench e solventi HPLC può minimizzare back-scambio in larga misura, riducendo la temperatura (≤0 ° C) per mezzo di un sistema di refrigerazione colonna adatto può proteggere ulteriormente la proteina da back-scambio . - Collegare il loop del campione e intrappolare proteine per la valvola che controlla automaticamente il processo di dissalazione e eluizione. Calibrare lo spettrometro di massa iniettando una TOF bassa concentrazione mix di sintonia nello spettrometro di massa nel range m / z di 200-3,200. Il immobilizzatoColonna pepsina e la colonna analitica non sono necessari per l'analisi delle proteine intatte.
- Impostare la temperatura nel sistema refrigerato per ≤0 ° C e attendere che il sistema raggiunga una temperatura di funzionamento stabile di ~ 0 ° C.
- Trasferire rapidamente i campioni da -80 ° C in azoto liquido per la spettrometria di massa. Utilizzando pinze, estrarre con attenzione ogni flaconcino da azoto liquido e ricostituire il campione aggiungendo un volume specifico di ghiaccio freddo quench tampone contenente acido formico al 0,2% (FA) (pH 2,5) e 5% di metanolo in acqua per flaconcino.
- Programmare una HPLC e metodo adatto spettrometria di massa utilizzando il software di controllo. Per formulazioni Mb, desalt campione contenente 20 pmol Mb nella trappola proteine per 1,7 min con 5% di acetonitrile, 95% di acqua e 0,1% acido formico (FA), ed eluire utilizzando un gradiente aumentato al 80% acetonitrile, 20% di acqua e 0,1 % FA in 3.3 min. Raccogliere spettri di massa nel range m / z 200-3,200.
- Per determinare la massa di intattoproteine, acquisire dati per un campione undeuterated proteine (cioè, proteina non sottoposto a ssHDX) in soluzione acquosa con il metodo del punto 2.9.
- Ottenere le masse di campioni undeuterated e deuterati dai deconvoluting gli spettri prime utilizzando il software di analisi dei dati. Qui, impostare l'intervallo di massa al 15,000-18,000 Da, la risoluzione di massa di 1,0 Da, e l'altezza del picco a 90% per il calcolo della massa di Mb.
- Calcolare il numero di deuteroni incorporati nella proteina intatta (qui, Mb) sottraendo la massa di proteine undeuterated dalla massa di proteine deuterato ad ogni tempo di cambio.
- Calcolare la percentuale di deuterio assorbimento rispetto al massimo teorico utilizzando la seguente equazione (Equazione 1)
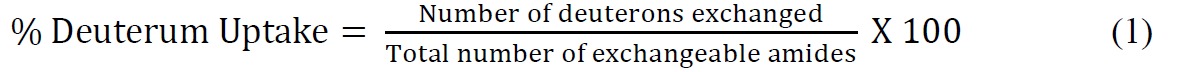
dove totale numero di amidi intercambiabili = numero totale di amminoacidi - numero di residui di prolina - 2 ("2" conti per l'N-gruppo terminale amino e ammide idrogeno che subiscono un rapido back-scambio). - Montare i dati cinetici ssHDX utilizzando un'equazione esponenziale adeguato. Un'equazione biesponenziale (Equazione 2) è di solito la più semplice che fornisce una misura ragionevole i dati ssHDX. In questo studio, per MbT e Mbs, adattare i dati a un modello biesponenziale che assegna deutoni a "veloce" e "lento" scambio di piscine.
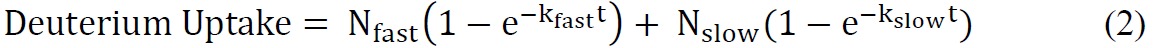
dove N veloce e lento N è il numero di ammidi intercambiabili nel "veloce" e "lento" scambio piscine, rispettivamente, e k veloce e lento k sono le costanti di velocità di primo ordine associati alle due piscine.
3. ssHDX-MS per proteine a Peptide Level
- Eseguire ssHDX seguendo i passaggi da 2.1 a 2.8, con le seguenti modifiche al punto 2.6. Collegare il colle di pepsina immobilizzatoumn colonna analitica e alla valvola come riferito in precedenza 25 e sostituiscono la trappola proteina collegata alla valvola con una trappola peptide. Calibrare lo spettrometro di massa impostando la massa per caricare rapporto compreso tra 100 e 1.700.
NOTA: Durante la fase di ricostituzione (passo 2.8), un agente riducente e denaturazione agente può essere inclusa nel tampone di raffreddamento per facilitare la pepsina digestione delle proteine con legami disolfuro (ad esempio, anticorpi monoclonali). - Un programma HPLC appropriata e il metodo di spettrometria di massa con il software di controllo. Per formulazioni Mb, digerire campioni contenenti 20 pmol Mb online con 0,1% FA, trappola e desalt peptidi per 1,7 minuti con il 10% acetonitrile, 90% di acqua e 0,1% FA in una trappola peptide. Eluire i frammenti sulla colonna analitica con un gradiente di aumento al 60% acetonitrile, 40% di acqua e 0,1% FA in 4,0 min. Acquisire spettri di massa nel range m / z 100-1,700.
- Identificare i frammenti peptiche mediante analisi MS / MS di uncampione di proteine undeuterated. Utilizzare software spettrometria di massa per confrontare masse sperimentali di ioni frammento peptide alle masse previsti di frammenti peptidici in un database personalizzato. Impostare un punto materiale cut-off (ad esempio 10 ppm) per identificare masse con basso errore. Per peptidi abbinati, preparare una lista che comprende (i) sequenza peptidica, (ii) stato di carica, e (iii) tempo di ritenzione.
- Utilizzare l'elenco generato al punto 3.3 per mappare e determinare il numero medio di deutoni incorporati per ogni pepsina digerire frammento. Ciò può essere ottenuto impiegando adatto software di analisi dati HDX-MS 24.
- Per calcolare la percentuale di deuterio assorbimento e per adattare i dati cinetici ssHDX per ognuno dei frammenti peptica, seguire i passi 2.13 e 2.14. In questo studio, HDX dati cinetici per ciascuno dei sei pepsina non ridondante digerire frammenti da MBT e Mbs formulazioni sono stati montati su un modello di associazione biesponenziale (Equazione 2).
4. SSPL-MS per Pro IntactTEIN
- Per iniziare la reazione di marcatura fotolitico, prima accendere il reticolante UV e permettere le lampade per scaldare per 5 min. Verificare che la sorgente UV è dotato di lampade di lunghezza d'onda 365 nm per attivare il gruppo di diazirine pLeu.
ATTENZIONE: Non aprire mai la porta del reticolante UV quando le lampade sono accese. Proteggere gli occhi e la pelle dall'esposizione ai raggi UV se la fonte non è racchiuso da una porta di vetro UV-protezione. - Spegnere il reticolante UV prima di aprire la porta. Una volta che le lampade sono spente, uncap fiale contenenti la formulazione liofilizzata e metterli all'interno della camera reticolante UV come mostrato nella Figura 2B. Irradiare l'campioni con luce UV per 40 min.
- Effettuare esperimenti di controllo seguendo passo da 4.1 a 4.3 per (i) campioni liofilizzati senza pLeu e (ii) i campioni liofilizzati con pLeu ricostituito in acqua.
- Cap e conservare le fiale a -20 ° C fino a spettrometria di massa.
- Reconstitute campioni solidi aggiungendo un volume adeguato di acqua distillata grado spettrometria di massa per portare la concentrazione di 2 mM.
- Per iniziare l'analisi del campione, seguire i passi 2.6 e 2.9.
NOTA: Dal back-scambio non è un problema con l'etichettatura covalente, SSPL-MS non richiede uno speciale sistema LC refrigerato. - Per determinare la massa della proteina nativa, acquisire dati per un campione proteina che non è stato sottoposto a SSPL seguendo passo 2.9. Ottenere le masse di campioni non etichettati e marcati dalla deconvoluting spettri grezzo come spiegato al punto 2.11.
- Calcolare il numero di pLeu inserite utilizzando la seguente formula:
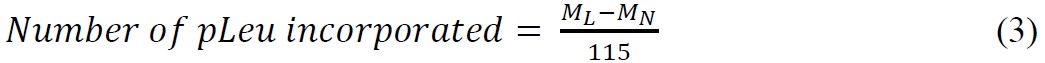
dove M L è la massa di proteina marcata, M N è la massa di proteina nativa e 115 è la massa media (Da) aggiunto alla proteina nativa seguente singola incorporazione pLeu. Si noti che la reazione di marcatura avviene con the perdita di N 2 (28 Da). La massa monoisotopico di pLeu è 143,07. - Calcolare le percentuali di popolazioni della proteina con un diverso numero di etichette utilizzando altezze del picco dei cromatogrammi ionici estratti.

dove "i" indica il numero di etichette, PH i denota l'altezza di picco per etichettato proteina L i e PH u denota l'altezza di picco della proteina non marcato come osservato mediante spettrometria di massa.
5. SSPL-MS per proteica a livello Peptide
- Eseguire SSPL seguendo i passaggi da 4.1 a 4.4.
- Per l'analisi livello di peptide, ricostituire campioni solidi in tampone di bicarbonato di ammonio (100 mM, pH 8,0).
- Dopo ricostituzione, miscelare la soluzione di proteina marcata con tripsina a 10: 1 rapporto molare di proteine di tripsina e incubare a 60 ° C per 16 ore.
- Placare la reazione aggiungendo 0,1% FAin acqua al campione di cedere concentrazione finale di 2 mM proteine.
- Collegare il loop del campione, trap peptide e colonna analitica alla valvola collegata al sistema HPLC.
- Programmare una HPLC e metodo adatto spettrometria di massa utilizzando il software di controllo. Per formulazioni Mb, iniettare 20 pmol della proteina digerita nel circuito di campionamento e desalificare i peptidi in una trappola peptide per 1,5 min con 5% di acetonitrile, 95% di acqua e 0,1% FA, seguita da una eluizione in colonna analitica con gradiente aumentare fino a 55% di acetonitrile, 45% di acqua e 0,1% FA in 22 min. Raccogliere spettri di massa nel range m / z 100-1,700.
- Preparare una lista teorica di massa per peptide-pLeu addotti utilizzando uno strumento online come ExPASy 26 con i numeri di pLeu precedentemente calcolati dall'analisi delle proteine intatte. Includi almeno 4 perse divisioni. Si noti che la reazione di marcatura si verifica con la perdita di N 2. Pertanto, la massa del peptide-pLeu addotto = massa di prova non marcatopeptide PTIC + n (massa pLeu) - n (massa di N 2), dove "n" è il numero di pLeu incorporato.
NOTA: Se l'analisi di massa di proteine intatte mostrato fino a tre popolazioni etichettati di proteine, in considerazione fino a tre possibili etichette pLeu per peptide. Per un peptide con massa di 1000 Da, la massa teorica di peptide-pLeu addotto con 1 pLeu incorporazione sarebbe 1.000 + 1 (143) - 1 (28) = 1.115 Da. Analogamente, le masse teoriche di peptide-pLeu addotti con 2 pLeu, 3 pLeu, ecc sarebbe 1.230 Da, 1.345 Da, ecc rispettivamente. - Utilizzare il software di analisi di massa in modo che corrisponda alla lista massa teorica generata al passo 5.7 con le masse osservati sperimentalmente. Impostare un punto materiale cut-off (ad esempio, 50 ppm) per identificare masse con basso errore.
- Per peptidi abbinati, determinare il numero effettivo di pLeu incorporato usando la formula di cui passo 4.8 (Equazione 3), dove M e L M N sono le masse di etichettae peptide nativo, rispettivamente.
Risultati
Qui, ssHDX-MS e SSPL-MS sono stati utilizzati per studiare l'effetto degli eccipienti sulle interazioni di conformazione e dello stato solido di formazioni Mb liofilizzati. Le concentrazioni di proteine e eccipienti utilizzati in questo studio sono riportati in Tabella 1. I risultati rappresentativi dall'analisi ssHDX-MS e SSPL-MS di Mb liofilizzati ottenuti seguendo i protocolli di cui sopra sono presentate.
Deuterio assorbimento a livello proteico intatto
ssHDX-MS è in grado di distinguere tra le formulazioni Mb a livello intatto. Gli spettri di massa deconvoluto di Mb intatte seguito 144 ore di ssHDX da MBS formulazione ha mostrato maggiore assorbimento di deuterio di formulazione MbT (Figura 3A). In media, MBS ha mostrato il 46% maggiore assorbimento di deuterio di MbT (Tabella 2).
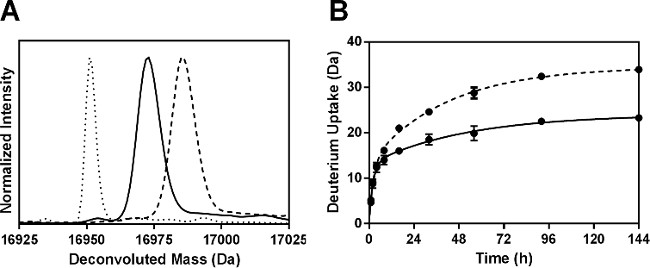
Figura 3: ssHDX-MS per Mb intatte: (A) deconvoluzione massa spettri Mb intatte deuterati da formulazioni MbT (linea continua) e MBS (linea tratteggiata) dopo 144 ore di ssHDX. Lo spettro di massa deconvoluto di Mb intatte undeuterated è anche mostrato (linea tratteggiata). (B) cinetica ssHDX per Mb intatti a formulazioni MbT (linea continua) e MBS (linea tratteggiata). La durata di ssHDX è stato montato ad un'equazione a due fasi associazione esponenziale utilizzando software Graph Pad Prism versione 5 (n = 3, ± SD).
La cinetica deuterazione per Mbs intatti e MBT sono simili in momenti iniziali (1-4 hr), ma MBS hanno mostrato un aumento di scambio di deuterio con aumento del tempo (8-144 ore) (Figura 3B). Ciò suggerisce l'importanza di selezionare i punti di tempo più lungo per ssHDX a basse RH e temperatura condizioni. Inoltre, il processo di assorbimento e diffusione D 2 O può influenzare il tasso di ssHDX all'epoca po precoceint. Nostri studi precedenti hanno dimostrato che assorbimento di umidità in ssHDX è completa in un periodo di ore, e ha minimo contributo di scambiare cinetiche oltre questo tempo. Il tasso osservato e l'estensione dello scambio, pertanto, non sono semplicemente misure di D 2 O adsorbimento 27,28. Le piccole barre di errore in Figura 3B, indicano le deviazioni standard di tre campioni ssHDX-MS indipendenti, mostrano che l'esperimento è altamente riproducibile.
| Deuterio assorbimento (%) b | N veloce c | k veloce c | N lento c | k lento c | |
| MbT un | 15.9 ± 0.5 | 13.1 (0.8) | 0,43 (0,03) | 11.0 (0.9) | 0.019 (0.001) |
| Mbs un | 23.2 ± 0.5 | 15,4 (0,7) | 0.49 (0.04) | 19.2 (0.6) | 0.024 (0.002) |
| % Variazione d | 46% | 18% | 14% | 75% | 26% |
Tabella 2:. Le misure quantitative di deuterio assorbimento negli studi ssHDX di formulazioni Mb per la composizione di una Vedi tabella 1 b percentuale di deuterio assorbimento rispetto al massimo teorico per Mb intatti dopo 144 ore di HDX a 5 ° C, 43% di umidità relativa (n = 3. , Parametri media ± SD). c determinati dalla regressione lineare dei dati cinetici ssHDX-MS. Naturalmente il tempo di cambio di deuterio per intatto Mb è stato montato su un modello di associazione biesponenziale (Eqn. 2). I valori tra parentesi sono errori standard dei parametri di regressione. Ð la variazione percentuale misurazioni erano calculATED come 100 x [(valore da MBS - valore da MBT) / (valore da MBT)].
I parametri di regressione (N veloce, lento N, k veloce e lento k) per la cinetica di assorbimento di deuterio MBT e Mbs sono riportati nella tabella 2. Anche se il N valori veloci e N lenti sono più grandi di MBS di MBT, le differenze di N lento valori erano superiori alle differenze nei valori veloci N. In particolare, il valore veloce N è solo il 18% più grande in MB che in MbT, mentre il valore lento N è il 75% più grande in MB che in MBT. Questo suggerisce che i più piccoli N valori lenti a MbT può essere dovuto alla maggiore ritenzione di struttura di Mb o di protezione di gruppi ammidici da eccipienti che sono esposti a D 2 O in MB. Tuttavia, i meccanismi dettagliati non sono ancora chiaramente definiti. Le costanti di velocità (K veloce e K lento) per entrambe le formulazioni sono molto simili.
deuterio assorbimento a livello peptide
Dopo digestione con pepsina, un totale di 52 peptidi sono stati identificati. Sei frammenti non ridondanti corrispondenti al 100% della sequenza Mb sono stati utilizzati per l'analisi qui riportato. Ulteriori informazioni possono essere ottenute utilizzando frammenti sovrapposti, come riportato dal nostro gruppo in precedenza 24. L'assorbimento percentuale di deuterio per ciascun peptide è stato calcolato e i risultati di 144 campioni hr tracciata (Figura 4A). Cinetica HDX per i sei frammenti peptiche hanno mostrato comportamenti biesponenziale (Figura 4B), in linea con sottopopolazioni di idrogeno amidici sottoposti "veloce" e "lento" scambio.
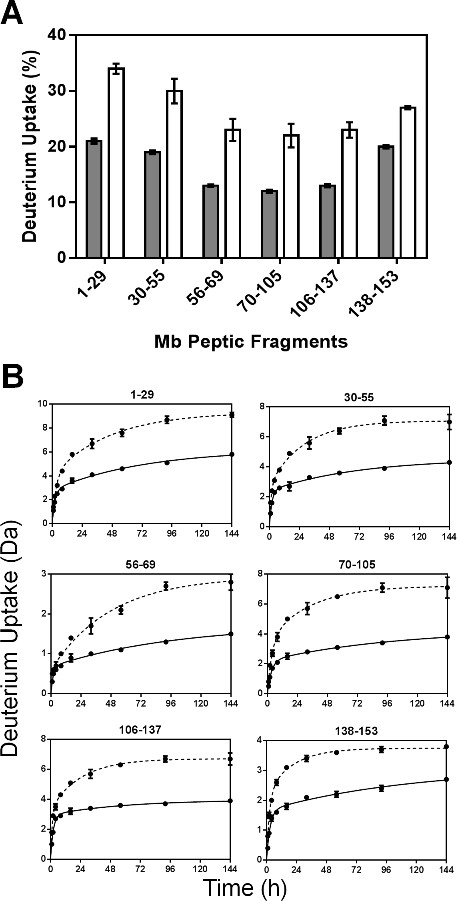
Figura 4: ssHDX-MS per Mb a livello peptide: (A) uptake Percent deuterio per 6 non reduframmenti peptica ndant di Mb in formulazioni MbT (grigio) e Mbs (bianco) dopo 144 ore di HDX. cinetica (B) ssHDX per sei frammenti peptiche non ridondanti di Mb in formulazioni MbT (linea continua) e MBS (linea tratteggiata). La durata di ssHDX è stato montato ad un'equazione a due fasi associazione esponenziale utilizzando software Graph Pad Prism versione 5 (n = 3, ± SD).
Parametri di regressione per i peptidi non ridondanti sono presentati nella Figura 5. Come le costanti di velocità a muro per frammenti peptidici non sono la costante tasso medio per i singoli amidi, le costanti di velocità osservati per i frammenti peptica non possono essere linearmente correlati a quelli per la proteina intatto. I N valori veloci per la maggior parte dei frammenti peptica (tranne frammento 56-69) in formulazioni MBS sono leggermente superiori a quelli ottenuti MbT (Figura 5A). Allo stesso modo, i valori veloci k generalmente mostrato poca differenza tra formulations e in diverse regioni della molecola Mb (Figura 5B). Tuttavia, i valori N lenta lenta e K per MBS sono significativamente maggiore in tutti i frammenti che per MbT (Figura 5C e 5D). Il notevole incremento in N k lento e lento per Mbs può riflettere una maggiore mobilità di gruppi ammidici nelle "lenti" piscine scambio.
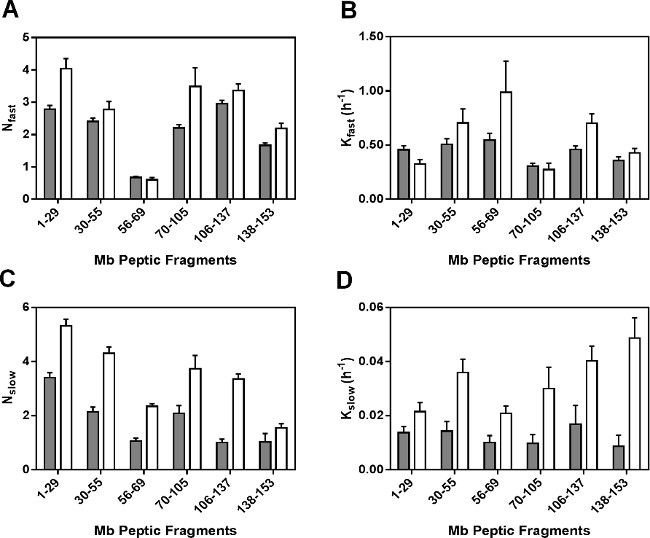
Figura 5: parametri cinetici ssHDX per Mb peptidi peptica: N veloce (A), k veloce (B), N lento (C) e k valori lento (D) ottenuti dalla regressione lineare dei dati cinetici ssHDX-MS per sei peptidi peptica non ridondanti di Mb in formulazioni MbT (grigio) e MBS ( bianco) (n = 3, ± SE).
Etichettatura fotolitica a livello proteico intatto
Mb irradiato in presenza di eccesso 20x pLeu formata multipli Mb-pLeu addotti, come rilevato da LC-MS (Figura 6A). Gli spettri deconvoluzione per MbT irradiato per 40 min con 20x pLeu ha mostrato fino a 3 etichette con l'aggiunta di 115, 230 e 345 Da alla massa di Mb senza etichetta. Mbs irradiati simile con 20x pLeu mostrato meno pLeu assorbimento a livello intatto, con un massimo di 2 popolazioni etichettati rilevati da LC-MS.
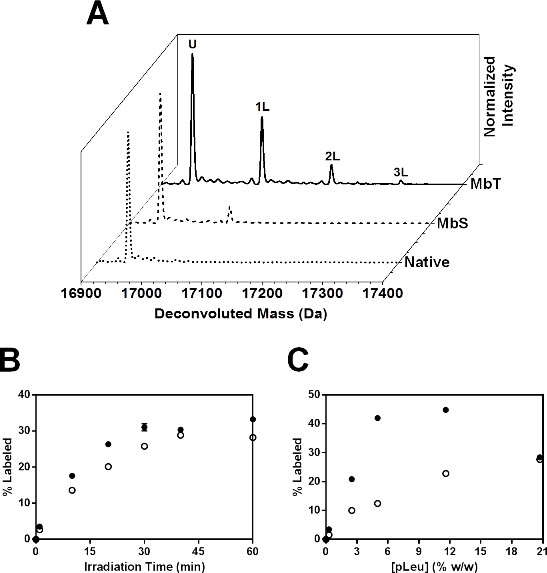
Figura 6: SSPL-MS per Mb intatte: (A) deconvoluzione spettri di massa per MbT (linea continua) e MBS (linea tratteggiata) etichettati con 20x eccesso (5% w / w) pLeu. Spettro di massa deconvoluto nativo Mb (Mb liofilizzata e irradiata in assenza di pLeu) è indicata come la linea tratteggiata. U indica una popolazione di proteine che rimane senza etichetta dopo l'irradiazione. Le popolazioni di proteine che trasportano 1, 2 e 3 etichette pLeu sono rappresentati come 1L, 2L e 3L rispettivamente. cinetica (B) SSPL-MS per Mb intatti a formulazioni MbT (circoli chiusi) e MBS (cerchi aperti) in funzione della concentrazione pLeu. Tutti i campioni sono stati irradiati per 40 min. Le barre di errore sono all'interno dei simboli. (C) cinetica SSPL-MS per Mb intatti a formulazioni MbT (circoli chiusi) e MBS (cerchi aperti) liofilizzati e irradiate in presenza di 100x eccesso pLeu (20,7% w / w) in funzione del tempo di irradiazione. Le barre di errore sono all'interno dei simboli.
In studi cinetici, la percentuale di proteina marcata aumentato esponenzialmente sia MbT e MBS all'aumentare del tempo di irraggiamento (Figura 6B). Mbs mostrato meno assorbimento pLeu di MbT in ogni tempo di irradiazione. Entrambe le formulazioni sembravano raggiungere un plateau a 40 min. Così, uno studio cinetico può essere noi eful per determinare la durata di irradiazione necessaria per ottenere la completa attivazione pLeu. Cinetiche etichettatura sono stati studiati come funzione della concentrazione pLeu (figura 6C). La percentuale di proteina marcata è aumentato con la concentrazione pLeu sia MbT e Mbs. Tuttavia, al 20,7% w / w pLeu, MbT mostrato una diminuzione pLeu assorbimento. Questo può essere dovuto ad esclusione pLeu dalla superficie della proteina ad alta concentrazione pLeu. Quindi, uno studio con vari concentrazione di pLeu deve essere eseguita per selezionare la concentrazione pLeu appropriato che consenta la un'etichettatura adeguata sulla superficie proteica senza esclusione di superficie. In questo studio, 20x eccesso pLeu stato selezionato per ulteriori studi di livello peptide.
La complessiva diminuzione etichettatura osservato per MBS suggerisce poveri della catena laterale accessibilità alla matrice contenente pLeu. Questo è coerente con un cambiamento conformazionale in presenza di sorbitolo che provoca etichettatura ridotta.
content "> etichettatura fotolitica a livello peptideSulla base degli studi in materia di etichettatura di proteine intatte, 20x eccesso pLeu è stato scelto per confrontare MbT e Mbs a livello peptide. Campioni marcati sono stati digeriti con tripsina e analizzati da LC-MS. Un totale di 40 peptidi corrispondenti al 100% della sequenza Mb stati rilevati per MBT e Mbs campioni. In alcuni casi, la digestione con tripsina può fornire una copertura limitata sequenza proteica se Lys e / o Arg residui sono fortemente etichettati. Per migliorare la copertura sequenza, una miscela di tripsina e chimotripsina può essere utilizzato per digerire la proteina marcata.
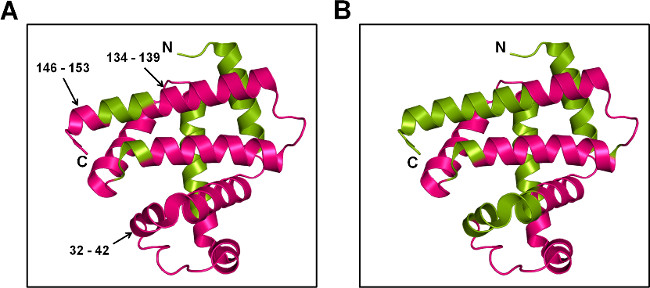
Figura 7: SSPL-MS per Mb a livello peptide: Rappresentazione fumetto di Mb etichettato con 20x eccesso pLeu (5% w / w) in presenza di trealosio (A) e sorbitolo (B). La proteina marcata è stato digerito con trypsin e peptidi marcati sono stati mappati al struttura cristallina di Mb (PDB ID 1WLA). Le regioni etichettati e senza etichetta sono di colore magenta e verde, rispettivamente.
SSPL-MS con digestione tripsina fornisce informazioni qualitative sui peptidi di essere etichettati. Date le diverse popolazioni etichettati a livello intatto, il meccanismo promiscua di etichettatura pLeu e le differenze di efficienza di ionizzazione di peptidi etichettati e senza etichetta, è difficile ottenere metriche quantitative per SSPL-MS dopo la digestione. Tuttavia, le informazioni qualitative può ancora fornire una conoscenza proteiche cambiamenti conformazionali a livello peptide. In questo studio, sia MBT e Mbs formulazioni mostrato pLeu assorbimento attraverso la maggior parte della superficie proteica. Rispetto a Mbs, frammenti peptidici 32-42, 134-139 e 146-153 da MBT mostrato etichettatura pLeu (Figura 7). Questo suggerisce che le catene laterali di questi aminoacidi sono esposti a pLeu, come le eliche in questi regions sono intatte nella matrice MBT. Al contrario, la protezione da etichettatura pLeu nella matrice MBS è coerente con perturbazioni strutturali in queste regioni.
Nel complesso, i risultati di ssHDX-MS e SSPL-MS suggeriscono che i metodi in grado di fornire informazioni complementari ad alta risoluzione a livello di peptide su backbone (ssHDX-MS) e della catena laterale (SSPL-MS) di esposizione e di eccipiente effetti in formulazioni proteiche liofilizzati .
Discussione
Diversi studi hanno suggerito che l'ambiente locale in campioni liofilizzati colpisce proteine degrado 5,29,30. Tuttavia, stabilendo una relazione diretta tra struttura proteica e stabilità allo stato solido non è possibile a causa della mancanza di metodi analitici ad alta risoluzione. L'applicazione di metodi di risoluzione elevate esistenti come HDX e PL di polveri liofilizzate richiede la modifica dei protocolli di soluzioni e un'attenta interpretazione dei dati. HDX-MS e PL-MS sono stati successivamente adottati per monitorare conformazioni di proteine nello stato solido. I risultati qui presentati e altrove 27,28,31-33 hanno dimostrato la capacità di questi metodi per monitorare conformazione proteica con alta risoluzione in ambiente solido. Anche se i passaggi critici in analisi dei dati non si discostano da etichettatura in soluzione 34-36, importanti considerazioni durante l'installazione sperimentale e l'interpretazione dei dati sono necessari per chemi a stato solidoetichettatura cal.
Selezione del reagente etichettatura deve essere basata sulla dimensione e il meccanismo di etichettatura. Le piccole dimensioni del deuterio permette al backbone peptidico per tastare facilmente, considerando le dimensioni relativamente maggiore di pLeu limita etichettatura alle catene laterali. Sia ssHDX e SSPL mostrano alcuna preferenza per qualsiasi amminoacido, in modo che l'etichettatura dipende solo backbone e catena laterale esposizione alla matrice. Per sondare efficacemente conformazioni di proteine a stato solido, i fattori esterni che influenzano il processo di etichettatura devono essere controllati attentamente. L'importo totale e la distribuzione spaziale di agente etichettatura in solidi liofilizzati è diverso da soluzioni acquose.
In ssHDX, la quantità di D 2 O nella matrice solida può influenzare la velocità di svolgimento proteine (o parziale dispiegamento), ripiegando e cambio deuterio. Questo non è il caso con la soluzione HDX, in cui il campione di proteine è normalmente diluito con un ampio volume di D 2 O.Proiezione accurata degli effetti di idratazione sul tasso ssHDX può informare la selezione di condizioni ideali RH. Per controllare la velocità di assorbimento di umidità ed evitare il collasso della polvere in formulazioni contenenti eccipienti igroscopici (ad esempio saccarosio e trealosio), ssHDX può essere effettuata in condizioni refrigerate (2-8 ° C). Il nostro studio precedente sugli effetti di idratazione ha mostrato un aumento dei tassi e l'entità di scambio con aumento del contenuto di umidità, come previsto. In gran parte del nostro lavoro, un RH intermedio del 43% a 5 ° C si è dimostrata ideale distinguere formulazioni in un tempo ragionevole 24. La reazione viene solitamente effettuata fino a raggiungere un plateau. Questo assicura che assorbimento di umidità e la diffusione nel solido non controllano il tasso HDX. L'uso di piccole dimensioni dei campioni solidi con volume di pre-liofilizzazione di ≤2 ml aiuta anche a garantire che D 2 O vapore assorbimento è sostanzialmente completo all'inizio del periodo di scambio. Anche se ssHDX-MS fornisceinformazioni quantitative sulla conformazione della proteina in stato solido, ci sono alcune condizioni in cui l'interpretazione dei dati non può essere interamente basato sullo studio ssHDX solo. È possibile che l'assorbimento di deuterio diminuzione osservata in un campione (in confronto al controllo) può essere dovuta alla maggiore ritenzione della struttura proteica o la notevole quantità di aggregati proteici presenti nel campione. In tal caso, l'interpretazione dei dati ssHDX richiede risultati ottenuti con altri metodi complementari. Ampliamento Peak in spettri di massa deuterato è stato osservato per diverse formulazioni Mb 27,28. Questo potrebbe essere dovuto a vari fattori come la presenza della popolazione proteine parzialmente unfolded, eterogeneità spaziale nel campione oppure i gradienti spaziali nella concentrazione D 2 O. Tuttavia, questi fattori non sono stati distinti in ssHDX-MS e necessita di ulteriori indagini.
Come SSPL-MS è relativamente nuovo rispetto ad altri metodi, ab apprendimento continuole sue applicazioni e le limitazioni è necessario. In SSPL, foto-cross-linker viene liofilizzata con la proteina. La mancanza di umidità limita la mobilità dei componenti all'interno della matrice solida, e il rilassamento strutturale parziale che può verificarsi con assorbimento di umidità in ssHDX non è un fenomeno in SSPL. Questo limita etichettatura SSPL alle immediate vicinanze della foto-cross-linker. Tuttavia, a differenza HDX-MS, analisi MS / MS della proteina covalentemente etichettato può fornire livello residuo informazioni strutturali. Dal momento che l'etichettatura SSPL è covalente e irreversibile, di nuovo cambio non si verifica e campioni può essere preparato e analizzato senza preoccupazione per la perdita di etichetta. Per facilitare la diffusione dell'agente etichettatura e migliorare l'efficienza di etichettatura in solido-matrice, SSPL può essere eseguita con l'aumentare% RH. pLeu assorbimento può essere migliorata aumentando la concentrazione dell'agente fotoreattivo. Il rapporto molare di proteine per pLeu può essere variato come desiderato. In generale, un eccesso 100x molare di pLeu a proTEIN garantirà un'adeguata etichettatura. Tuttavia, alta concentrazione pLeu può causare la perdita di proteine struttura terziaria nella matrice solida. Quindi, Oltre a cinetiche di etichettatura e composizione formulazione, selezione di concentrazione pLeu deve basarsi sul mantenimento proteine integrità strutturale. Come pLeu etichette non selettivamente XH (dove X = C, N, O) gruppo, è possibile che gli eccipienti con siti di etichettatura simili possono influenzare notevolmente il livello di etichettatura proteine. L'interferenza degli eccipienti nella disponibilità di pLeu per l'etichettatura proteina è ancora da caratterizzare. È noto che la carbene generato dall'attivazione diazirine non è specifica residui tuttavia uno studio riporta propensione Asp e Glu 36. Mentre è bene conoscere interazioni specifiche residui, informazioni peptide-livello è anche utile e può essere utilizzato per progettare eccipienti di bloccare regioni con elevata esposizione matrice allo stato solido. SSPL-MS fornisce informazioni qualitative dettagliate, tuttaviaha bisogno di dati quantitativi che si vuole ottenere e metriche robuste devono essere sviluppate per analizzare le differenze di formulazione attraverso una varietà di sistemi di liofilizzati.
L'uso di un'etichetta specifico residuo combinato con analisi MS / MS può migliorare ulteriormente risoluzione al livello amminoacido. Reagenti Etichettatura quali 2,3-butanedione di etichettare Arg, N derivati -hydroxysuccinimide per Lys e N derivati -alkylmaleimide per Cys può essere utilizzato per mappare con precisione le interazioni molecolari in polvere liofilizzata. Tuttavia questi reagenti sono pH-dipendenti e le reazioni non possono essere così controllati, come l'etichettatura fotolitico in stato solido. Un approccio alternativo è di incorporare la foto-cross-linker nella sequenza proteica con l'uso di linee cellulari auxotrofi, mutagenesi sito-diretta o catena laterale derivatizzazione.
I nostri precedenti studi ssHDX-MS e SSPL-MS hanno dimostrato che l'etichettatura delle proteine dipende dalla natura e la quantità degli eccipientiutilizzato 24,27,28,31-33,37,38. ssHDX-MS di Mb co-liofilizzato con guanidina cloridrato (Gdn.HCl) hanno mostrato maggiore assorbimento di deuterio di Mb co-liofilizzato a basso peso molecolare zuccheri 32. In uno studio SSPL-MS separato, Mb co-liofilizzato con Gdn.HCl ha mostrato una maggiore protezione da etichettatura fotolitico di Mb con saccarosio 33. Inoltre, le misure quantitative da ssHDX-MS sono stati fortemente correlati con la stabilità di proteine durante la conservazione a lungo termine 28. Questi studi suggeriscono che ssHDX o SSPL di proteine riflette il grado di ritenzione strutturale della proteina in polvere liofilizzata. Noi crediamo che il mantenimento della struttura secondaria in polveri liofilizzate fornisce un ambiente favorevole per l'etichettatura catena laterale con pLeu e la protezione di ammide idrogeno da cambio deuterio. Tuttavia, confronto dettagliato del contenuto informativo di questi metodi deve essere eseguita in futuro. Sebbene stabilire l'utilità di ssHDX-MS e SSPL-MScome strumento di screening formulazione alla fine richiederà che essere applicato a molte proteine, i risultati dei nostri studi recenti supportano l'adozione più ampia. Con un ulteriore sviluppo, questi metodi dovrebbero essere ampiamente utile per caratterizzare formulazioni di proteine a stato solido nel settore biofarmaceutico.
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
The authors gratefully acknowledge financial support from NIH R01 GM085293 (PI: E. M. Topp) and from the College of Pharmacy at Purdue University.
Materiali
| Name | Company | Catalog Number | Comments |
| Equine myoglobin | Sigma-Aldrich | M0630-5G | |
| D-(+)-Trehalose dihydrate | Sigma Aldrich | #T9531 | |
| D-Sorbitol | Sigma Aldrich | #240850 | |
| L-Photo-leucine | Thermo Scientific | #22610 | |
| Potassium phosphate monobasic | Sigma-Aldrich | #P0662 | |
| Potassium phosphate dibasic | Sigma-Aldrich | #P3786 | |
| Deuterium Oxide | Cambridge Isotope Laboratories | #DLM-4-PK | Alternate (Cat. No.: 151882, Sigma-Aldrich) |
| Immobilized pepsin | Applied Biosystems | #2-3132-00 | |
| Trypsin | Promega | #V511A | Chymotrypsin (Cat. No.: #V1062, Promega) can be additionally used |
| Water, Optima LC/MS grade | Fisher Chemical | #7732-18-5 | |
| Acetonitrile | Sigma-Aldrich | #34998 | |
| Formic acid | Thermo Scientific | #28905 | |
| ESI-TOF Calibrant | Agilent Technologies | #G1969-85000 | Highly flammable liquid |
| Protein microtrap | Michrom Bioresources | TR1/25108/03 | |
| Peptide microtrap | Michrom Bioresources | TR1/25109/02 | |
| Analytical column | Agilent Technologies | Zorbax 300SB-C18 | |
| Freeze dryer | VirTis AdVantage Plus | ||
| Stratalinker equipped with five 365 nm lamps | Stratagene Corp. | Stratalinker 2400 | |
| HPLC | Agilent Technologies | 1200 series LC | Refrigerated LC system for HDX-MS |
| ESI-qTOF MS | Agilent Technologies | 6520 qTOF | |
| HDExaminer (HDX-MS data analysis software) | Sierra Analytics | http://www.massspec.com/HDExaminer.html |
Riferimenti
- Lawrence, S. Billion dollar babies--biotech drugs as blockbusters. Nat. Biotechnol. 25 (4), 380-382 (2007).
- . . Global Markets and Manufacturing Technologies for Protein Drugs. , BIO021D (2013).
- Lai, M. C., Topp, E. M. Solid-state chemical stability of proteins and peptides. J. Pharm. Sci. 88 (5), 489-500 (1999).
- Carpenter, J. F., Pikal, M. J., Chang, B. S., Randolph, T. W. Rational design of stable lyophilized protein formulations: some practical advice. Pharm. Res. 14 (8), 969-975 (1997).
- Carpenter, J. F., Chang, B. S., Garzon-Rodriguez, W., Randolph, T. W. Rational design of stable lyophilized protein formulations: theory and practice. Pharm. Biotechnol. 13, 109-133 (2002).
- Wüthrich, K. Protein structure determination in solution by NMR spectroscopy. J. Biol. Chem. 265 (36), 22059-22062 (1990).
- Ilari, A., Savino, C. Protein structure determination by x-ray crystallography. Methods. Mol. Biol. 452, 63-87 (2008).
- Brunger, A. T. X-ray crystallography and NMR reveal complementary views of structure and dynamics. Nat. Struct. Biol. 4, 862-865 (1997).
- Yu, L. Amorphous pharmaceutical solids: preparation, characterization and stabilization. Adv Drug. Deliv. Rev. 48 (1), 27-42 (2001).
- Manning, M. C. Use of infrared spectroscopy to monitor protein structure and stability. Expert. Rev. Proteomics. 2 (5), 731-743 (2005).
- Grohganz, H., Gildemyn, D., Skibsted, E., Flink, J. M., Rantanen, J. Rapid solid-state analysis of freeze-dried protein formulations using NIR and Raman spectroscopies. J. Pharm. Sci. 100 (7), 2871-2875 (2011).
- Bai, S., Nayar, R., Carpenter, J. F., Manning, M. C. Noninvasive determination of protein conformation in the solid state using near infrared (NIR) spectroscopy. J. Pharm. Sci. 94 (9), 2030-2038 (2005).
- Pikal, M. J., et al. Solid state chemistry of proteins: II. The correlation of storage stability of freeze-dried human growth hormone (hGH) with structure and dynamics in the glassy solid. J. Pharm. Sci. 97 (12), 5106-5121 (2008).
- Wang, B., Tchessalov, S., Cicerone, M. T., Warne, N. W., Pikal, M. J. Impact of sucrose level on storage stability of proteins in freeze-dried solids: II. Correlation of aggregation rate with protein structure and molecular mobility. J. Pharm. Sci. 98 (9), 3145-3166 (2009).
- Schule, S., Friess, W., Bechtold-Peters, K., Garidel, P. Conformational analysis of protein secondary structure during spray-drying of antibody/mannitol formulations. Eur. J. Pharm. Biopharm. 65 (1), 1-9 (2007).
- Baerga-Ortiz, A., Hughes, C. A., Mandell, J. G., Komives, E. A. Epitope mapping of a monoclonal antibody against human thrombin by H/D-exchange mass spectrometry reveals selection of a diverse sequence in a highly conserved protein. Protein. Sci. 11 (6), 1300-1308 (2002).
- Coales, S. J., Tuske, S. J., Tomasso, J. C., Hamuro, Y. Epitope mapping by amide hydrogen/deuterium exchange coupled with immobilization of antibody, on-line proteolysis, liquid chromatography and mass spectrometry. Rapid. Commun. Mass. Spectrom. 23 (5), 639-647 (2009).
- Pacholarz, K. J., Garlish, R. A., Taylor, R. J., Barran, P. E. Mass spectrometry based tools to investigate protein-ligand interactions for drug discovery. Chem. Soc. Rev. 41 (11), 4335-4355 (2012).
- Houde, D., Peng, Y., Berkowitz, S. A., Engen, J. R. Post-translational modifications differentially affect IgG1 conformation and receptor binding. Mol. Cell. Proteomics. 9 (8), 1716-1728 (2010).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. J. Pharm. Sci. 100 (6), 2071-2086 (2011).
- Dorman, G., Prestwich, G. D. Using photolabile ligands in drug discovery and development. Trends. Biotechnol. 18 (2), 64-77 (2000).
- Robinette, D., Neamati, N., Tomer, K. B., Borchers, C. H. Photoaffinity labeling combined with mass spectrometric approaches as a tool for structural proteomics. Expert. Rev. Proteomics. 3 (4), 399-408 (2006).
- Greenspan, L. Humidity fixed points of binary saturated aqueous solutions. Journal of Research of the National Bureau of Standards. 81A (1), 8 (1977).
- Sophocleous, A. M., Zhang, J., Topp, E. M. Localized hydration in lyophilized myoglobin by hydrogen-deuterium exchange mass spectrometry. 1. Exchange mapping. Mol. Pharm. 9 (4), 718-726 (2012).
- Keppel, T. R., Jacques, M. E., Young, R. W., Ratzlaff, K. L., Weis, D. D. An efficient and inexpensive refrigerated LC system for H/D exchange mass spectrometry. J. Am. Soc. Mass. Spectrom. 22 (8), 1472-1476 (2011).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic. Acids. Res. 31 (13), 3784-3788 (2003).
- Sophocleous, A. M., Topp, E. M. Localized hydration in lyophilized myoglobin by hydrogen-deuterium exchange mass spectrometry. 2. Exchange kinetics. Mol. Pharm. 9 (4), 727-733 (2012).
- Moorthy, B. S., Schultz, S. G., Kim, S. G., Topp, E. M. Predicting Protein Aggregation during Storage in Lyophilized Solids Using Solid State Amide Hydrogen/Deuterium Exchange with Mass Spectrometric Analysis (ssHDX-MS). Mol. Pharm. 11 (6), 1869-1879 (2014).
- Wang, W. Lyophilization and development of solid protein pharmaceuticals. Int. J. Pharm. 203 (1-2), 1-60 (2000).
- Sarciaux, J. M., Mansour, S., Hageman, M. J., Nail, S. L. Effects of buffer composition and processing conditions on aggregation of bovine IgG during freeze-drying. J. Pharm. Sci. 88 (12), 1354-1361 (1999).
- Li, Y., Williams, T. D., Schowen, R. L., Topp, E. M. Characterizing protein structure in amorphous solids using hydrogen/deuterium exchange with mass spectrometry. Anal. Biochem. 366 (1), 18-28 (2007).
- Sinha, S., Li, Y., Williams, T. D., Topp, E. M. Protein conformation in amorphous solids by FTIR and by hydrogen/deuterium exchange with mass spectrometry. Biophys. J. 95 (12), 5951-5961 (2008).
- Iyer, L. K., Moorthy, B. S., Topp, E. M. Photolytic labeling to probe molecular interactions in lyophilized powders. Mol. Pharm. 10 (12), 4629-4639 (2013).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. J. Vis. Exp. (81), e50839 (2013).
- Kaltashov, I. A., Bobst, C. E., Abzalimov, R. R. H/D exchange and mass spectrometry in the studies of protein conformation and dynamics: is there a need for a top-down approach. Anal. Chem. 81 (19), 7892-7899 (2009).
- Jumper, C. C., Schriemer, D. C. Mass spectrometry of laser-initiated carbene reactions for protein topographic analysis. Anal. Chem. 83 (8), 2913-2920 (2011).
- Li, Y., Williams, T. D., Topp, E. M. Effects of excipients on protein conformation in lyophilized solids by hydrogen/deuterium exchange mass spectrometry. Pharm. Res. 25 (2), 259-267 (2008).
- Li, Y., Williams, T. D., Schowen, R. L., Topp, E. M. Trehalose and calcium exert site-specific effects on calmodulin conformation in amorphous solids. Biotechnol. Bioeng. 97 (6), 1650-1653 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon