Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Planar Sistema de Difusión de degradado para Investigar quimiotaxis en una matriz de colágeno 3D
En este artículo
Resumen
Cell migration is an important part of human development and life. In order to understand the mechanisms that can alter cell migration, we present a planar gradient diffusion system to investigate chemotaxis in a 3D collagen matrix, which allows one to overcome modern diffusion chamber limitations of existing assays.
Resumen
The importance of cell migration can be seen through the development of human life. When cells migrate, they generate forces and transfer these forces to their surrounding area, leading to cell movement and migration. In order to understand the mechanisms that can alter and/or affect cell migration, one can study these forces. In theory, understanding the fundamental mechanisms and forces underlying cell migration holds the promise of effective approaches for treating diseases and promoting cellular transplantation. Unfortunately, modern chemotaxis chambers that have been developed are usually restricted to two dimensions (2D) and have complex diffusion gradients that make the experiment difficult to interpret. To this end, we have developed, and describe in this paper, a direct-viewing chamber for chemotaxis studies, which allows one to overcome modern chemotaxis chamber obstacles able to measure cell forces and specific concentration within the chamber in a 3D environment to study cell 3D migration. More compelling, this approach allows one to successfully model diffusion through 3D collagen matrices and calculate the coefficient of diffusion of a chemoattractant through multiple different concentrations of collagen, while keeping the system simple and user friendly for traction force microscopy (TFM) and digital volume correlation (DVC) analysis.
Introducción
El movimiento preferido de las células hacia un gradiente de concentración, conocido como quimiotaxis, juega un papel importante en los procesos patológicos y fisiológicos en el cuerpo. Tales ejemplos son la piel y la mucosa cicatrización de la herida 1, 2 morfogénesis, la inflamación 3, y 4,5 el crecimiento del tumor. También se ha demostrado que las células cancerosas pueden migrar a través de ambas estrategias individuales y colectivas a la migración celular 6. Además, los mecanismos de inestabilidad difusionales pueden inducir la separación de células individuales o agrupados a partir de un cuerpo / objeto tumoral y luego puede emigrar hacia una fuente de nutrientes y por lo tanto invadir áreas 7 y tejidos más amplios.
Además, se ha demostrado que diversos mecanismos de migración pueden ser activos en 2D y en 3D, debido a las diferentes funciones de las moléculas de adhesión 8. Por lo tanto, un movimiento para fisiológicamente relevante en ensayos in vitro para investigar la motilidad celular en un measureabley manera simple es de importancia en la comprensión de los fenómenos movimiento celular 9. Desafortunadamente, la dificultad en el análisis de la migración celular, un ensayo de quimiotaxis integral cuantificable por lo general requiere un método laborioso de largo, fundada en la medición de los modelos de la motilidad celular y los fenómenos de transporte imparciales.
Enfoques experimentales anteriores para investigar la quimiotaxis de células incluyen la cámara de Boyden 10 y el que se ensaya agarosa 11. Sin embargo, dentro de estos primeros ensayos, los experimentos de migración celular no controlan el movimiento con respecto al tiempo. Más, importante, los gradientes de concentración utilizados para los experimentos fueron bien definidos o no completamente entendidas, mientras que sólo el mantenimiento de la señalización por no más de unas pocas horas. Por otra parte, los intentos de la cámara de quimiotaxis temprana restringen la migración de células de dos dimensiones y no permitieron una para supervisar la cinética de la migración 12. En cuanto a la cámara de Boyden, un ensayo de punto finalno permitiría al investigador para observar la migración visualmente y no podía diferenciar directamente quimiotaxis (movimiento direccional) de chemokinesis (movimiento aleatorio). Además, varias variables diferencias en el tamaño de poro y el grosor de las membranas hechos a la cámara muy difícil de reproducir fácilmente y ocultan la reacción migrante de las células a quimiocinas 13,14.
Con la nueva comprensión de la microfluídica, nuevas cámaras y micro-dispositivos se han investigado como un instrumento para investigar la locomoción celular bajo condiciones de flujo intersticiales o quimiotaxis 15,16. Bajo estos nuevos dispositivos, nuevas métricas de células se introdujeron e investigados, como el efecto del esfuerzo cortante en un celular 17,18. Desafortunadamente, las cámaras de quimiotaxis de microfluidos pasadas y actuales limitan estudios de la migración de células a los sustratos-2D un retroceso importante ya que muchos procesos biológicos, incluyendo la invasión celular tumoral y la metástasis, y immigración celular inmune, implica la migración 3D.
-Cámaras donde una solución quimioatrayente está en contacto con un gel que contiene células 3D también han sido reportados también observación directa 19,20. Estas cámaras tienen dos compartimentos, uno que contiene un quimioatrayente y uno que contiene células, se unió al lado de uno otro horizontalmente 21 o como anillos concéntricos 22. Estos sistemas se apuntaban en la dirección correcta, pero no mantienen un sistema de quimiotaxis por un período prolongado de tiempo.
Además, los investigadores también han examinado la difusividad a través de membranas de colágeno en las células de diálisis, así como la difusión de moléculas trazadoras a través de muestras de colágeno sometido a la presión hidrostática 23-25. Algunos experimentos de difusión en geles de colágeno se basan en modificaciones físicas y químicas del gel utilizando campos magnéticos y la incorporación química 26. Un método popular para modelar difusividad en collatejidos indíge- se basa en la imagen de fluorescencia de fotoblanqueo punto continuo. Este método ha revelado anisotropía en los coeficientes de difusión de macromoléculas en tejidos de colágeno orientadas. Sin embargo, photobleaching se ha utilizado en el cartílago articular y no colágeno matrices. Aunque similares, los experimentos de modelización necesarias deben ser realizadas a través de la comprensión específicamente el coeficiente de difusión de los geles de colágeno. Más importante aún, los sistemas no utilizan un método para medir la generación de fuerza de la célula.
Desafortunadamente, la mayoría de los sistemas parecen faltar uno o dos elementos clave para un sistema ideal: la que permite el seguimiento de la célula, una comprensión gradiente de difusión con un factor quimiotáctico través de la matriz, relativamente sencilla configuración con una facilidad de la reproducibilidad, la minimización de interacciones célula-célula, y la capacidad de medir unidades dimensionales para la cuantificación (es decir, velocidad, fuerza, concentración específica). Moghe et al. 27 propuso un sistema que cumple la mayoría de estos requisitos en el que las células se dispersan inicialmente en todo el gel en lugar de concentrados en la superficie del filtro, pero era difícil de medir las fuerzas que la célula genera.
Para este propósito, se presenta un sistema de difusión de gradiente plana para investigar la quimiotaxis en una matriz de colágeno en 3D, lo que permite superar las limitaciones modernas cámara de difusión de los ensayos existentes, que se basa en la microscopía de lapso de tiempo, junto con las técnicas de análisis de imagen para medir celular fuerzas en un entorno 3D. Este protocolo proporciona una manera simple, pero innovadora de la creación de una cámara de difusión 3D simple que se puede utilizar para investigar la quimiotaxis 3D en diferentes células.
Protocolo
Diseño de molde 1. 3D y Refacciones
- Molde
- Antes de trabajar, obtener un kit de elastómero de silicona, una cámara de imágenes de células vivas, un cubreobjetos de vidrio de 22 mm, y un cubo de metal de aluminio mecanizado con dimensiones de 10,07 mm x 3,95 mm x 5,99 mm. Prepare la cámara de imágenes de células vivas para el moldeo mediante la colocación de cubreobjetos en el soporte inferior y el montaje del resto de la cámara de la manera indicada por el vendedor.
- A continuación, utilizando unas pinzas, coloque el cubo de metal de aluminio mecanizado en el medio de la vivienda en vivo cámara de imágenes de células y en la parte superior del cubreobjetos, y luego dejar de lado.
- Mezclar las soluciones de elastómero de silicona de acuerdo con el protocolo del fabricante para hacer 5 ml de elastómero.
- Usando una espátula de laboratorio desechable, vierta la solución de elastómero de silicona en la configuración de cámara imágenes de células vivas y garantizar que no se mueva colocado cubo de metal de aluminio mecanizado. Coloque el sistema sobre la mesa del laboratorio, en un lugar seguro O / N para el curado.
- A la mañana siguiente, deconstruirla cámara de imágenes de células vivas, según lo recomendado por el fabricante y sacar del molde utilizando fórceps. Con unas pinzas, extraiga con cuidado el cubo de metal de aluminio mecanizado de molde. Ir al fregadero y enjuague el molde con agua desionizada. Coloque el molde sobre una toalla de papel para secarse.
- Una vez seco, utilizando un cuchillo manía, corte ranuras a través del molde, espaciados 2,34 mm de distancia de cada extremo longitudinal, en el interior del molde de silicona. Asegúrese de que las estancias de molde en un lugar seguro y seco hasta que esté listo para construir el sistema para un experimento.
- Hidrófilas e hidrófobas Glass Preparación Cubreobjetos
- Para permitir que la matriz de colágeno a que se adhieran a una superficie, crear cubreobjetos hidrófilos:
- Utilizando una pipeta desechable, mida 150 l de 3-aminopropil-trimetoxisilano y se vierte la solución en un tubo de 50 ml. Añadir 30 ml de etanol al 100% al tubo de 50 ml con una segunda pipeta desechable y cierre la tapa. Vortex la solución durante 2 min, asegurando un mezclado completo. Pnuestra solución en una placa de Petri de vidrio y dejar de lado.
- Verter 15 ml de 100% de alcohol etílico en una segunda placa de Petri y dejar de lado (asegúrese de etiquetar los platos).
- Usando una nueva pipeta desechable, mida 30 ml de agua desionizada y se vierte la solución en un tubo de 50 ml. A continuación, utilizando una nueva medida pipeta desechable a cabo 1.875 l de glutaraldehído y se vierte la solución en el mismo tubo de 50 ml y cierre la tapa. Vortex la solución durante 2 min, asegurando un mezclado completo. Vierta la mezcla en tercera placa de Petri, y dejar de lado.
- Con unas pinzas, saca uno 22 mm cubreobjetos de cristal redondo y enjuague ambos lados con 100% de mezcla de etanol con una pipeta desechable.
- Coloque cubreobjetos de vidrio enjuagado en 3-aminopropil-trimetoxisilano con 30 ml de etanol al 100% plato solución y deje reposar en la solución durante 5 minutos.
- Con unas pinzas, sacar cubreobjetos de vidrio y aclarar de nuevo con una solución de etanol al 100% con la pipeta desechable.
- Caída cubreobjetos enjuagados en 1875l de glutaraldehído y 30 ml de la mezcla de agua desionizada y dejar de lado durante 30 minutos.
- Después de 30 minutos, retire cubreobjetos de vidrio con unas pinzas y enjuague con agua desionizada y coloque sobre el tejido seco O / N para secar a temperatura ambiente.
- Inmersión cubreobjetos Repetir y moverse durante tantos cubreobjetos según sea necesario con las mismas soluciones generadas.
- Hacer cubreobjetos hidrofóbicos por el protocolo dado.
- Añadir 500 l de tridecafluoro-1,1,2,2-tetrahydrooctyl, 100 l de ácido acético y 19,4 ml de hexano en un tubo de 50 ml y agitar durante 2 min. Vierta la solución en vidrio placa de Petri y dejar de lado.
- Con unas pinzas limpias, sacar un cubreobjetos de vidrio y colocar en una solución mixta preparada durante 2 min. Transcurridos 2 minutos, sacar cubreobjetos de vidrio con unas pinzas y enjuague con agua desionizada con una pipeta desechable y lugar cubreobjetos sobre el tejido seco O / N para secar a temperatura ambiente. Cubreobjetos tienda en platos de plástico Petri hasta su uso.
- DIPP cubreobjetos Repitación y en movimiento para tantos cubreobjetos según sea necesario con las mismas soluciones generadas.
- Para permitir que la matriz de colágeno a que se adhieran a una superficie, crear cubreobjetos hidrófilos:
Asamblea 2. Molde
- Antes de utilizar moldes, con una pipeta desechable enjuague el molde con un 90% de alcohol etílico en un contenedor para desechos químicos. A continuación, colocar el molde en una placa de Petri llena de agua desionizada y deje reposar O / N.
- Tome el molde de la solución utilizando pinzas y colocar sobre una toalla para secar al aire antes de iniciar el montaje del molde.
- Mientras el molde es de secado al aire, cortar los cubreobjetos hidrófilos cuadrados en dos rectángulos ligeramente mayores que 3,95 mm x 5,99 mm usando una herramienta de diamante-trazado de alta precisión. Deslice cada cubreobjetos corte rectangular en las ranuras cortadas en el molde de silicona.
- Da la vuelta al molde boca abajo y aplicar grasa de vacío a lo largo de la parte inferior del molde de silicona con una pipeta desechable. A continuación, dar la vuelta al molde de vuelta del lado derecho hacia arriba y presione la parte inferior del molde sobre las cubiertas de vidrio hidrofílicos circulareslos labios para crear un sello.
- Con guantes desechables, recoger el molde montado y colocar en un plato desechable Petri y luego en un bio-capucha. Encienda la lámpara de Bio-campana UV durante 1 hora para esterilizar el molde antes de que ocurra la experimentación.
3. El colágeno mezcla y Matrix 3D
- Antes de preparar la mezcla de colágeno, manchas y preparar células para obtener imágenes de acuerdo con protocolos estándar 28.
- Utilizando técnicas de laboratorio estándar y protocolos de 29,30, en un bio-campana, mezclar los medios de comunicación celulares y quimioatrayente en un tubo de 15 ml a la concentración deseada quimioatrayente. Pipeta 5 ml de solución de concentración quimioatrayente medios celda específica en un tubo de 15 ml y mover la solución en un baño de agua caliente.
- Con una pipeta, extraer 5 ml de medio de células sólo en un segundo tubo de 15 ml y colocar en un baño de agua caliente para calentar la solución para experimentos de microscopía.
- En un bio-hood, pipeta de 30 l de 10x tampón fosfatosolución ed (PBS) en el tubo micro centrífuga. Añadir 6 l de 1 N de hidróxido de sodio (NaOH) en mismo tubo de centrífuga micro y agitar durante 15 seg.
- Obtener colágeno I solución de cola de rata de la nevera y pasar a la bio-campana utilizando técnicas de laboratorio estándar y protocolos 29. Asegúrese de que el colágeno está frío. Pipetear 168,3 l de colágeno en mismo tubo micro centrífuga.
- A continuación, añadir 18 l de color amarillo-verde microesferas modificado carboxilato fluorescentes utilizando una pipeta para el mismo tubo micro centrífuga. Vortex el tubo de centrífuga micro con todas las soluciones para 30 seg.
- Añadir 77,7 l de mezcla de células deseada de células medios de comunicación (aproximadamente 2 x 10 6 células / ml de concentración) en el tubo de micro centrífuga y mezclar usando punta de la pipeta durante 20 s. Según sea necesario, alterar la densidad de la matriz siguiendo la mezcla de colágeno de encargo adaptado para la adición de perlas fluorescentes y mesa de la viabilidad celular (Tabla 1).
- Pipetear 300 & #181; l de mezcla de células de colágeno en el centro bien (bien # 2) del conjunto de molde preparado. Colocar el molde sobre un plato Petri desechable y moverlo en una incubadora estándar durante 20 min a 37 ° C con 5% de CO 2.
- Retire sistema de incubadora y colocar de nuevo en bio-capucha. Con unas pinzas, retire los dos cubreobjetos hidrofóbicos por apriete sobre la parte superior de cada cubreobjetos y tirando hacia arriba y lejos del molde.
- Mover todo el sistema para la microscopía y la imagen deseada para asegurar lados del molde de colágeno son todavía recto para permitir un gradiente de difusión planar.
- Con el sistema aún bajo el microscopio, usando una 100 l de un solo canal pipeta, añadir 100 l de medio celular en el pocillo # 1. A continuación, utilizando un 100 l de un solo canal pipeta añadir 100 l de los medios de comunicación celular con la concentración deseada en quimioatrayente bien # 3.
- Inmediatamente después de la adición de soluciones en ambos pozos, comenzar de imágenes.
4. Imagen y DifusiónModelado
- Si uno quiere encontrar el coeficiente de difusión para la densidad de colágeno específico para calcular la concentración específica, siga el protocolo a continuación:
- Siga el protocolo dado anteriormente titulado "Mezcla Colágeno y Matrix 3D", en lugar de generar medios de comunicación celular con la concentración quimioatrayente deseada, haga rodamina 5 M en un tubo de 15 ml utilizando cálculos y protocolos 30 estándar.
- Image las matrices de colágeno 3D con un sistema confocal montados en un microscopio invertido usando un láser de argón (488 nm) para capturar la fluorescencia. Imagen cada 2 o 3 segundos para un máximo de 7 horas.
- Para Modelado Difusión: Usando el modelo matemático se muestra a continuación, escribir un código de software computacional para el procesamiento y análisis con estos pasos generales.
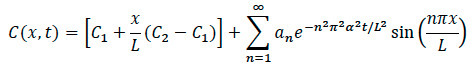
- Importación de imágenes en el software computacional para cambio de escala y la normalización de la inten máximosidad. Convertir imágenes a un color gris-mapa de la imagen para seleccionar el borde. Recogida de un eje a lo largo del centro de la imagen, a la derecha del centro y a la izquierda del centro.
- Utilizando el código de software computacional, trazar la intensidad normalizada, junto con la verdadera intensidad y la diferencia entre los dos.
- A continuación, introducir los parámetros necesarios para el montaje (concentración, tiempo y longitud).
- Trazar la concentración normalizada frente al tiempo y el perfil de concentración a través del eje x con un ajuste polinómico a los datos.
- Calcular el coeficiente de difusión utilizando el código de software computacional.
5. Las mediciones experimentales
- Haciendo referencia de nuevo a la ecuación de difusión, reescribir la ecuación para hallar la concentración específica en cualquier ubicación dentro del sistema como se muestra a continuación.
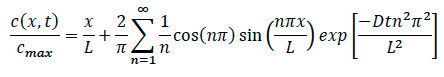
Dónde:
L = Longitud del XXmatriz de colágeno e
x = ubicación bajo investigación
D = coeficiente de difusión
t = tiempo transcurrido - Durante los experimentos de quimiotaxis, garantizar para registrar el tiempo específico que el momento de quimioquinas fue añadido al sistema. Una vez detectado el movimiento de la migración celular, asegúrese de grabar la grabación de lapso de tiempo confocal, y tenga en cuenta la hora y lugar de la orilla donde se produjo la imagen.
- Durante el análisis de datos, conecte el observado transcurrido el tiempo, la ubicación y el coeficiente de difusión para la célula específica en la ecuación para encontrar la concentración específico normalizado en cualquier punto dentro del colágeno tanto para la difusión y experimentos celulares.
6. Seguimiento de la migración de células Utilizando TFM
- Para el seguimiento de la migración de células usando técnicas de TFM / DVC siguen el protocolo a continuación:
- Después de la adición de la concentración deseada en quimioatrayente bien # 3, comience imágenes de las matrices de colágeno en 3D con un sistema confocal montado en un micr invertidaoscope para encontrar una celda para investigar.
- Una vez que se encuentra una célula en movimiento, colocar la célula en el medio del campo de vista y utilizar un motor piezoeléctrico (a la imagen en el eje z), imagen cada 1 o 2 min.
- Para el cálculo de generación de la fuerza y la deformación celular, generar un código computacional para encontrar los desplazamientos de los granos de fluorescencia siguiendo los protocolos TFM / DVC 31.
Resultados
La capacidad de este ensayo para evaluar con precisión la migración de la célula depende de una buena configuración del sistema. Por lo tanto, es fundamental para el maquillaje asegúrese de diseñar el molde sistema de difusión precisa y tener mucho cuidado en la colocación de cubreobjetos tanto hidrofóbicos e hidrofílicos, como se ilustra en la Figura 1. Si el sistema está diseñado correctamente y durante la etapa de modelado asegurando la difusión de encontrar una muy buena línea de salid...
Discusión
Los pasos más críticos para experimentos de difusión de éxito con o sin células son: establecer correctamente el conjunto de molde; el desarrollo de la destreza manual necesaria para evitar daños durante la extracción de cubreobjetos hidrófobos; asegurando que encontrar una muy buena línea de salida lineal para calcular correctamente el coeficiente de difusión; corregir los cálculos experimentales de colágeno y quimioatrayente; utilizar correctamente el sistema de imágenes de células vivas para asegurar la...
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
The authors would like to acknowledge Drs. Jonathan Reichner and Angle Byrd for cell experiment insight. The National Science Foundation Graduate Research Fellowship Program (GRFP) supported this work.
Materiales
| Name | Company | Catalog Number | Comments |
| Silicone elastomer kit | Dow Corning Corp | 182 SIL ELAST KIT .5KG | a two-part misture with a 10:1 mix |
| Live cell imaging chamber | Live Cell Instrument | CM-B18-1 | CMB for 18 mm round coverslips |
| 22 mm Glass Coverslip | Fisher Scientific | NC0180281 | Neuvitro Corp. cover slip 22 mm 1.5 |
| Machined aluminum metal cube | |||
| Hobby utility knife | X-Acto | X3201 | |
| 3-(aminopropyl) trimethoxysilane | Sigma-Aldrich | 281778-5ML | |
| Glutaraldehyde | Polysciences, Inc | 00216A-10 | Glutaraldehyde, EM Grade, 8% |
| 50 ml tube | Fisher Scientific | 14-432-22 | Standard floor model and tabletop centrifuges |
| Glass Petri dish | Fisher Scientific | 08-747A | Reusable Petri Dishes: Complete (60 x 15 mm) |
| Forceps | Fisher Scientific | 22-327-379 | Fine Point Forceps |
| Cover glasses | Fisher Scientific | 12-518-105A | Rectangle; 30 x 22 mm; Thickness No. 1 |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl | Gelest | SIT8174.0 | |
| Acetic acid | Sigma-Aldrich | 320099 | Acetic acid ACS reagent, ≥99.7% |
| Hexane | Sigma-Aldrich | 296090 | anhydrous, 95% |
| Ethyl alcohol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| High-precision diamond scribing tool | Lunzer | PV-081-3 | Straight extended tip scribe, .020" (.50 mm) diameter by .200" (5.0 mm) tip length |
| Vacuum grease | Dow Corning | 14-635-5C | High-Vacuum Grease |
| 15 ml tube | Fisher Scientific | 14-959-49D | 15 ml conical centrifuge tubes with hydrophobic, biologically inert surface |
| 10x phosphate buffered solution | Fisher Scientific | BP399-500 | 1.37 M Sodium Chloride, 0.027 M Potassium Chloride, and 0.119 M Phosphate Buffer |
| 1 N sodium hydroxide | Sigma-Aldrich | 38215 | Sodium hydroxide concentrate |
| Collagen I, rat tail | BD Biosciences | 354236 | Rat tail |
| Micro centrifuge tube | Fisher Scientific | 02-681-332 | Volume: 2.0 ml; O.D. x L: 13 x 40 mm; sterile; single-wrapped |
| [header] | |||
| Carboxylate-modified microspheres | Invitrogen | F-8813 | Carboxylate-modified microspheres, 0.5 µm, yellow-green fluorescent (505/515), 2% solids |
| Rhodamine | Sigma-Aldrich | 83689 | Rhodamine B for fluorescence |
Referencias
- Adzick, N. S. The Molecular and Cellular Biology of Wound Repair. Ann Surg. 225 (2), 236 (1997).
- Reddi, A. H. Bone morphogenetic proteins: an unconventional approach to isolation of first mammalian morphogens. Cytokine Growth F. R. 8 (1), 11-20 (1997).
- Coussens, , Werb, Z, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Vital-Lopez, F. G., Armaou, A., Hutnik, M., Maranas, C. D. Modeling the effect of chemotaxis on glioblastoma tumor progression. AIChE J. 57 (3), 778-792 (2011).
- Hughes-Alford, S. K., Lauffenburger, D. A. Quantitative analysis of gradient sensing: towards building predictive models of chemotaxis in cancer. Curr. Opin. Cell Biol. 24 (2), 284-291 (2012).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol Cell Bio. 10 (7), 445-457 (2009).
- Cristini, V. Morphologic Instability and Cancer Invasion. Clin. Cancer Res. 11 (19), 6772-6779 (2005).
- Lammermann, T., et al. Rapid leukocyte migration by integrin-independent flowing and squeezing. Nature. 453 (7191), 51-55 (2008).
- Parent, C. A., Devreotes, P. N. A cell's sense of direction. Science. 284 (5415), 765-770 (1999).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. J. Exp. Med. 115, 453-466 (1962).
- Nelson, R. D., Quie, P. G., Simmons, R. L. Chemotaxis under agarose: a new and simple method for measuring chemotaxis and spontaneous migration of human polymorphonuclear leukocytes and monocytes. J. Immunol. 115 (6), 1650-1656 (1975).
- Rosoff, W. J., McAllister, R., Esrick, M. A., Goodhill, G. J., Urbach, J. S. Generating controlled molecular gradients in 3D gels. Biotechnol. Bioeng. 91 (6), 754-759 (2005).
- Wells, A., Kassis, J., Solava, J., Turner, T., Lauffenburger, D. A. Growth factor-induced cell motility in tumor invasion. Acta Oncol. 41 (2), 124-130 (2002).
- Wilkinson, P. C. Assays of leukocyte locomotion and chemotaxis. J. Immunol. Methods. 216 (1-2), 139-153 (1998).
- Bonvin, C., Overney, J., Shieh, A. C., Dixon, J. B., Swartz, M. A. A multichamber fluidic device for 3D cultures under interstitial flow with live imaging: development, characterization, and applications. Biotechnol. Bioeng. 105 (5), 982-991 (2010).
- Noo, L. J., et al. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device. Nat. Biotechnol. 20 (8), 826-830 (2002).
- Shao, J., et al. Integrated microfluidic chip for endothelial cells culture and analysis exposed to a pulsatile and oscillatory shear stress. Lab Chip. 9 (21), 3118-3125 (2009).
- Haessler, U., Kalinin, Y., Swartz, M. A., Wu, M. An agarose-based microfluidic platform with a gradient buffer for 3D chemotaxis studies. Biomed. Microdevices. 11 (4), 827-835 (2009).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat. Rev. Cancer. 3 (5), 362-374 (2003).
- Sixt, M., Lammermann, T. In vitro analysis of chemotactic leukocyte migration in 3D environments. Methods Mol. Biol. 769, 149-165 (2011).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. J. Cell Biol. 75 (2 Pt 1), 606-616 (1977).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. J. Cell. Sci. 99 (Pt 4), 769-775 (1991).
- Gilbert, D. L. Macromolecular diffusion through collagen membranes. Int. J. Pharm. 47 (1-3), 79-88 (1988).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys. J. 83 (3), 1650-1660 (2002).
- Weadock, K., Silver, F. H., Wolff, D. Diffusivity of 125I-calmodulin through collagen membranes: effect of source concentration and membrane swelling ratio. Biomaterials. 7 (4), 263-267 (1986).
- Erikson, A., Andersen, H. N., Naess, S. N., Sikorski, P., Davies, C. d. L. Physical and chemical modifications of collagen gels: impact on diffusion. Biopolymers. 89 (2), 135-143 (2008).
- Moghe, P. V., Nelson, R. D., Tranquillo, R. T. Cytokine-stimulated chemotaxis of human neutrophils in a 3-D conjoined fibrin gel assay. J. Immunol. Methods. 180 (2), 193-211 (1995).
- Sundd, P., et al. Live cell imaging of paxillin in rolling neutrophils by dual-color quantitative dynamic footprinting. Microcirculation. 18 (5), 361-372 (2011).
- Sanders, E. R. Aseptic Laboratory Techniques: Volume Transfers with Serological Pipettes and Micropipettors. J. Vis. Exp. (63), 2754 (2012).
- Stephenson, F. H. . Calculations for Molecular Biology and Biotechnology: A Guide to Mathematics in the Laboratory 2e. , (2010).
- Bar-Kochba, E., Toyjanova, J., Andrews, E., Kim, K., Franck, C. A Fast Iterative Digital Volume Correlation Algorithm for Large Deformations. Exp. Mech. In press, (2014).
- Shenoy, V., Rosenblatt, J. Diffusion of Macromolecules in Collagen and Hyaluronic Acid, Rigid-Rod-Flexible Polymer Composite Matrixes. Macromolecules. 28 (26), 8751-8758 (1995).
- Leddy, H., Guilak, F. Site-Specific Molecular Diffusion in Articular Cartilage Measured using Fluorescence Recovery after Photobleaching. Ann. Biomed. Eng. 31 (7), 753-760 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados