このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
3Dコラーゲンマトリックス中の走化性を調査するために、平面グラデーション拡散システム
要約
Cell migration is an important part of human development and life. In order to understand the mechanisms that can alter cell migration, we present a planar gradient diffusion system to investigate chemotaxis in a 3D collagen matrix, which allows one to overcome modern diffusion chamber limitations of existing assays.
要約
The importance of cell migration can be seen through the development of human life. When cells migrate, they generate forces and transfer these forces to their surrounding area, leading to cell movement and migration. In order to understand the mechanisms that can alter and/or affect cell migration, one can study these forces. In theory, understanding the fundamental mechanisms and forces underlying cell migration holds the promise of effective approaches for treating diseases and promoting cellular transplantation. Unfortunately, modern chemotaxis chambers that have been developed are usually restricted to two dimensions (2D) and have complex diffusion gradients that make the experiment difficult to interpret. To this end, we have developed, and describe in this paper, a direct-viewing chamber for chemotaxis studies, which allows one to overcome modern chemotaxis chamber obstacles able to measure cell forces and specific concentration within the chamber in a 3D environment to study cell 3D migration. More compelling, this approach allows one to successfully model diffusion through 3D collagen matrices and calculate the coefficient of diffusion of a chemoattractant through multiple different concentrations of collagen, while keeping the system simple and user friendly for traction force microscopy (TFM) and digital volume correlation (DVC) analysis.
概要
走化性として知られている濃度勾配に向かう細胞の好ましい運動は、体内の病理学的および生理学的プロセスにおいて重要な役割を果たしています。このような例は、皮膚および粘膜創傷治癒1、形態形成2、3、炎症、および腫瘍増殖4,5です。また、癌細胞の両方の個々と集合細胞遊走戦略6を通って移動することができることが示されています。また、拡散不安定機構は腫瘍体/オブジェクトから単一またはクラスタ化された細胞の分離を誘導することができ、その後、栄養源に向かって移住し、したがって、より広い領域や組織7に侵入することができます。
また、多様な移動機構が原因で接着分子8の異なる役割に、2Dおよび3Dで活性であることが示されています。 measureablで細胞の運動性を調査するin vitroアッセイにおける生理学的に関連するので、移動e及び簡単な方法は、細胞運動現象9の理解に重要です。残念ながら、細胞の移動を分析することの困難は、総合的な定量化可能な走化性アッセイは、通常、公平な細胞の運動性と輸送現象モデルの測定に設立され、長い骨の折れる方法を、必要とします。
細胞走化性を調査する過去の実験的アプローチは、Boydenチャンバー10と下のアガロースアッセイ11を含みます。しかし、これらの初期のアッセイ内で、細胞遊走実験は、時間に対しての動きを監視していませんでした。もっと、重要なのは、実験に用いた濃度勾配が十分に定義されていないか、わずか数時間を超えないためのシグナリングを維持しつつ、完全に理解しました。また、初期の走化性チャンバーの試みは2次元に細胞移動を制限し、1つは、移行12の動態を監視することはできませんでした。 Boydenチャンバーを見ると、エンドポイントアッセイ研究者が視覚的に移行を観察することができず、直接ケモキネシス(ランダム移動)から走化性(方向性)を区別することができませんでした。また、孔径、厚さ、いくつかの変数の違いを容易に再現することは非常に困難チャンバ膜が製およびケモカイン13,14への細胞の移住反応を隠し。
マイクロフルイディクスの新しい理解して、新しいチャンバ及びマイクロデバイスは、間質流動条件または走化性15,16の下のセルの移動を調査する手段として研究されてきました。これらの新しいデバイスの下では、新しいセルのメトリクスを導入し、細胞17,18のせん断応力の影響のように、調査しました。残念ながら、過去と現在のマイクロ流体走化性チャンバー2D基板 - 腫瘍細胞の浸潤や転移を含む多くの生物学的プロセス、以来重要な後退に細胞移動の研究を制限され、イム胸細胞の移動は、3D移動を伴います。
直接観察室、化学誘引物質溶液が細胞を含む三次元ゲルと接触しても、また19,20が報告されています。これらのチャンバは、2つの区画、化学誘引物質および細胞を含むいずれかを含むものを持って、互いに水平方向に21または同心円状のリング22と横に結合されています。これらのシステムは、正しい方向に指摘されているが、長期間走化性システムを保持しません。
さらに、研究者はまた、透析細胞、ならびに静水圧23〜25にかけコラーゲンサンプルを介してトレーサー分子の拡散にコラーゲン膜を通した拡散性を検討しました。コラーゲンゲルの一部の拡散実験は、磁場および化学取込み26を用いたゲルの物理的および化学的修飾に依存しています。コーラで拡散をモデル化するための一般的な方法genous組織が連続点光退色の蛍光イメージングに依存しています。この方法は、指向膠原組織における高分子の拡散係数に異方性を明らかにしました。しかし、光退色は、関節軟骨ではなく、コラーゲンマトリックス中で使用されています。類似の間、必要なモデリング実験は、具体的にコラーゲンゲルの拡散係数を理解することを通して実施しなければなりません。さらに重要なことに、システムは、セル力発生を測定するための方法を利用しません。
マトリックスを通して細胞トラッキング、走化性因子と拡散勾配の理解を可能にする、再現性の容易さと、比較的簡単なセットアップ、の最小化:残念ながら、ほとんどのシステムでは、理想的なシステム用に1つまたは2つの重要な要素が欠けているように見えます細胞-細胞相互作用、および定量化のための寸法の単位を測定する能力( 例えば 、速度、力、特定の濃度)。 Moghe ら。 図27は 、細胞を最初にゲル全体に分散よりもむしろフィルタ表面に濃縮されたこれらの要件のほとんどを満たしシステムを提案したが、セルが生成する力を測定することは困難でした。
この目的のために、我々は細胞を測定するために、画像解析技術を用いて結合された1つは、タイムラプス顕微鏡検査に基づいて、既存のアッセイの現代の拡散室の制限を克服することを可能にする3次元コラーゲンマトリックスに走化性を調査する平面勾配拡散システムを提示します3D環境の力。このプロトコルは、異なるセル内の3D走化性を研究するために使用することができる単純な3D拡散室を作成するシンプルで革新的な方法を提供します。
プロトコル
1. 3次元金型設計と部品
- モールド
- 作業を始める前に、シリコーンエラストマーキット、ライブセルイメージング室、22ミリメートルのガラスカバースリップ、および寸法10.07ミリメートルX 3.95ミリメートルX 5.99ミリメートルで加工アルミニウム金属キューブを得ます。下のホルダーにカバースリップを配置し、ベンダーの指示どおりに、チャンバの残りの部分を組み立てることにより成形用ライブセルイメージング室を準備します。
- 次に、ピンセットを用いて、生細胞イメージング室ハウジングの中央にし、カバースリップの上に機械加工されたアルミニウム金属キューブを配置し、その後、脇に置きます。
- エラストマーを5mlにする製造業者のプロトコルに従ってシリコーンエラストマー溶液を混合します。
- 使い捨ての実験用へらを使用して、生細胞イメージング室セットアップにシリコーンエラストマー溶液を注ぎ、置か機械加工アルミニウム金属キューブを移動しないよう保証します。硬化させるための安全な場所のO / Nで、実験台の上にシステムを配置します。
- 翌朝、解体ライブセルイメージング室、メーカーが推奨する鉗子を使用して金型を引き出しています。鉗子を使用して、慎重に金型から機械加工されたアルミニウム金属キューブを抽出します。シンクに移動し、脱イオン水で金型をすすぎます。乾燥するためにペーパータオルの上に金型を配置します。
- 乾燥したら、金型を介してスリットをカットし、趣味のカッターナイフを使用して、シリコーン型の内側に、それぞれの長手方向の端部から離れて2.34ミリメートルの間隔を置いて配置。あなたは、実験のためのシステムを構築する準備が整うまで、安全で乾燥した場所で、金型の滞在を確認してください。
- 親水性および疎水性ガラスカバースリップの準備
- コラーゲンマトリックスは、表面に付着できるようにするには、親水性のカバーガラスを作成します。
- 使い捨てピペットを用いて、3-アミノプロピル - トリメトキシシラン150μlのを測定し、50mlチューブ内の溶液を注ぎます。第二の使い捨てピペットを用いて50 mlチューブに100%エタノール30mlを加え、蓋を閉じます。完全な混合を確実にする、2分間溶液をボルテックス。 Pガラスシャーレ内の当社のソリューション、脇に置きます。
- 第シャーレに100%のエチルアルコール15mlのを注ぎ、脇に置きます(料理を標識することを確認してください)。
- 新しい使い捨てピペットを用いて、脱イオン水30mlを測定し、50mlチューブに溶液を注ぎます。次に、グルタルアルデヒドの1,875μLを新しい使い捨てピペットの尺度を使用して、同じ50ミリリットルチューブに溶液を注ぎ、ふたを閉じます。完全な混合を確実にする、2分間溶液をボルテックス。第三のペトリ皿に混合物を注ぎ、脇に置き。
- 鉗子を使用して、1 22ミリメートルラウンドカバーガラスを取り出し、使い捨てピペットを用いて100%エタノール混合物で両面をすすぎます。
- 100%エタノール溶液皿30mlで3-アミノプロピルトリメトキシシランにリンスガラスカバースリップを置き、5分間、溶液中に座ってすることができます。
- ピンセットで、カバーガラスを取り出し、使い捨てピペットと100%のエタノール溶液で再度洗い流します。
- 1,875にすすぎ、カバーガラスをドロップグルタルアルデヒドのμlの脱イオン水の混合物の30ミリリットルと30分のために確保さ。
- 30分後、ピンセットでカバーガラスを除去し、脱イオン水ですすぎ、室温で乾燥させるために、乾燥組織O / Nの上に置きます。
- カバーガラスの浸漬を繰り返し、同じ生成ソリューションを必要な数のカバースリップのために移動します。
- 指定されたプロトコルによって、疎水性カバースリップを行います。
- トリデカフルオロ-1,1,2,2-テトラヒドロオクチル500μlの酢酸100μlの2分間、50mlのチューブ、ボルテックスでヘキサン19.4ミリリットルを追加します。ガラスシャーレ内の溶液を注ぎ、脇に置きます。
- きれいな鉗子を使用して、一方のガラスカバースリップを取り出し、2分間調製した混合溶液中にドロップします。 2分が経過した後、ピンセットでカバーガラスを取り出し、室温で乾燥させるために、乾燥組織O / N上に使い捨てピペットと場所カバースリップを使用して、脱イオン水ですすいでください。使用するまでプラスチックペトリ皿に保管してカバースリップ。
- 繰り返しカバースリップDIPPると同じ生成ソリューションを必要な数のカバースリップのために移動します。
- コラーゲンマトリックスは、表面に付着できるようにするには、親水性のカバーガラスを作成します。
2.金型アセンブリ
- 金型を使用する前に、使い捨てピペットを用いて、化学廃棄物容器の上に90%のエチルアルコールで金型をすすぎます。次に、脱イオン水で満たされたペトリ皿に金型を配置し、O / Nに座ることを可能にします。
- ピンセットを用いて溶液から金型を取り、型アセンブリを開始する前に、空気乾燥したタオルの上に置きます。
- 金型は、空気乾燥であるが、高精度ダイヤモンドスクライブツールを使用して3.95ミリメートルX 5.99ミリメートルよりも若干大きく2つの長方形の中に正方形の親水性のカバーガラスをカット。シリコーン型にカットスロットにそれぞれカット矩形カバースリップをスライドさせます。
- 逆さまに金型を反転し、使い捨てピペットを用いて、シリコーン型の底部に沿って真空グリースを塗布してください。次に、バック右サイドアップ型を反転し、円形の親水性のガラスカバーの上に型の底を押してくださいシールを作成するために、リップ。
- 使い捨て手袋を使用すると、組み立てられた金型をピックアップし、使い捨てペトリ皿にした後、バイオフード内に配置します。実験が発生する前に金型を滅菌するために1時間UVバイオフードランプをオンにします。
3.コラーゲン混合物と3Dマトリックス
- コラーゲン混合物、汚れを調製し、標準的なプロトコル28による撮像のための細胞を調製する前に。
- バイオフード内で、標準的な実験技術およびプロトコル29,30を使用して、所望の化学誘引物質濃度の15mlチューブに細胞培地および化学誘引物質を混ぜます。ピペット5〜15 mlのチューブに特異的な細胞メディア化学誘引物質濃度の溶液のmlおよび加熱された水浴中にソリューションを移動します。
- ピペットを使用して、顕微鏡実験のために溶液を加熱するために加熱された水浴中に第15mlチューブと場所にだけ細胞培地を5mlを抽出します。
- バイオフードでは、ピペット10×リン酸緩衝液30μlマイクロ遠心チューブにED溶液(PBS)。 15秒間同じマイクロ遠心管と渦の中に1 N水酸化ナトリウム(NaOH)の6μlを添加します。
- 冷蔵庫からコラーゲンIラット尾液を取得し、標準的な実験技術およびプロトコル29を使用して、バイオフードに移動します。コラーゲンはまだ寒いであることを確認してください。同じマイクロ遠心分離管にコラーゲンの168.3μLをピペット。
- 次に、同じマイクロ遠心チューブにピペットを用いて、黄緑色の蛍光カルボン酸で修飾された微小球の18μlを添加します。ボルテックスで30秒間、すべてのソリューションをマイクロ遠心管。
- マイクロ遠心管に所望の細胞培地-細胞混合物(約2×10 6細胞/ mlの濃度)の77.7μlを添加して、20秒間ピペットチップを使用して混合します。必要に応じて、蛍光ビーズと細胞生存率の表 ( 表1)の添加のために適合カスタムコラーゲンの混合物を、以下によりマトリックス密度を変化させます。
- ピペット300µ準備金型組立体の中央ウェルにコラーゲン - 細胞混合物のL(ウェル#2)。使い捨てペトリ皿の上に金型を置き、5%CO 2で37℃で20分間の標準的なインキュベーターに移動します。
- インキュベーターからシステムを削除し、バイオフード内に戻し入れます。鉗子を使用して、各カバースリップの上部にクランプして離れて型から引き上げるとの両方によって疎水性のカバーガラスを取り外します。
- コラーゲンモールド側が平面状の拡散勾配を可能にするためにまっすぐにまだあることを確認することが望ましい顕微鏡および画像に、システム全体を移動します。
- まだ顕微鏡システム、100μlのシングルチャンネルピペットを用いて、ウェル#1に細胞培地の100μlを添加します。次に、100μlのシングルチャンネルピペットを使用することはよく#3に所望の化学誘引物質濃度の細胞培地の100μlを添加します。
- すぐに両方のウェルにソリューションを追加した後、撮影を開始。
4.イメージングと拡散モデリング
- 一つは、特定の濃度を算出するための特定のコラーゲン密度のための拡散係数を検索したい場合は、以下の手順に従ってください。
- 代わりに、所望の化学誘引物質濃度で細胞培地を生成する、標準的な計算およびプロトコル30を使用して、15mlチューブに5μMローダミンを作る」、コラーゲン混合物と3Dマトリックス」というタイトルの上に特定のプロトコルに従ってください。
- 共焦点画像システムを用いた3次元コラーゲンマトリックスは、蛍光を捕捉するために、アルゴンレーザー(488 nm)を用いて、倒立顕微鏡に取り付けられました。画像を最大7時間毎に2または3秒。
- 拡散モデルの場合:以下に示す数学的モデルを使用して、これらの一般的な手順で処理及び分析のための計算ソフトウェアのコードを記述します。
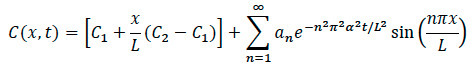
- 最大INTENによる再スケーリングおよび正規化のための計算ソフトウェアにインポートするイメージsity。エッジを選択するには、画像のグレーカラーマップに画像を変換します。中央の右、中央の左側に、画像の中心に沿って軸をピックします。
- 計算ソフトウェアコードを使用して、真の強さと両者の差とともに、規格化強度をプロットします。
- フィッティング(濃度、時間、及び長さ)のために必要な次に、入力パラメータ。
- 時間に対する正規化濃度とデータへの多項式フィットを使用して、x軸の両端の濃度プロファイルをプロットします。
- 計算ソフトウェアコードを使用して拡散係数を計算します。
5.実験測定
- 拡散方程式に戻って、以下に示すように、システム内の任意の位置での特定の濃度を見つけるために方程式を書き換えます。
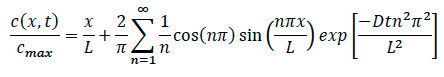
ここで、
目のL =長さEコラーゲンマトリックス
調査中のx =場所
拡散のD =係数
T =経過時間 - 走化性実験の間、瞬間ケモカインがシステムに追加された特定の時刻を記録するために確保します。細胞移動の動きが検出されると、共焦点タイムラプス撮影を記録し、撮影が発生したエッジから特定の時間と場所をメモすることを確認します。
- データ分析の間に、拡散や細胞実験の両方のためのコラーゲン内の任意の点での正規化された特定の濃度を見つけるために、式に特定のセルのために注目の経過時間、場所及び拡散係数を差し込みます。
TFMを利用6.追跡細胞の遊走
- TFM / DVC法は、以下のプロトコルに従う使用して細胞の移動を追跡します。
- よく#3への所望の化学誘引物質の濃度を添加した後、反転MICRに搭載された共焦点システムと3Dコラーゲンマトリックスを画像化開始調査するセルを見つけるoscope。
- 移動する細胞が検出されると、視野の中央にセルを配置し、(z軸における画像)が、圧電モーターを利用し、画像毎に1または2分。
- 細胞力発生と変形を計算するために、TFM / DVCプロトコル31次蛍光ビーズの変位を見つけるために、計算コードを生成します。
結果
正確に細胞の遊走を評価するために、このアッセイの能力は、システムの良いセットアップに依存しています。したがって、それは正確に拡散系の金型を設計し、 図1に示すように、疎水性および親水性の両方のカバーガラスを配置することに細心の注意を払うようにしてください非常に重要です。システムが適切に設計されており、非常に見つけることが確実に拡散モデリング...
ディスカッション
細胞の有無にかかわらず成功した拡散実験のための最も重要な手順は次のとおりです。正しくモールドアセンブリを設定します。疎水性のカバーガラスの抽出時の損傷を防止するために必要な手先の器用さを開発します。正しく拡散係数を算出するための非常に良好な直線スタートラインを見つけることを保証します。コラーゲンと化学誘引物質の両方の実験的な計算を修正。正しく乾燥し?...
開示事項
The authors declare that they have no competing financial interests.
謝辞
The authors would like to acknowledge Drs. Jonathan Reichner and Angle Byrd for cell experiment insight. The National Science Foundation Graduate Research Fellowship Program (GRFP) supported this work.
資料
| Name | Company | Catalog Number | Comments |
| Silicone elastomer kit | Dow Corning Corp | 182 SIL ELAST KIT .5KG | a two-part misture with a 10:1 mix |
| Live cell imaging chamber | Live Cell Instrument | CM-B18-1 | CMB for 18 mm round coverslips |
| 22 mm Glass Coverslip | Fisher Scientific | NC0180281 | Neuvitro Corp. cover slip 22 mm 1.5 |
| Machined aluminum metal cube | |||
| Hobby utility knife | X-Acto | X3201 | |
| 3-(aminopropyl) trimethoxysilane | Sigma-Aldrich | 281778-5ML | |
| Glutaraldehyde | Polysciences, Inc | 00216A-10 | Glutaraldehyde, EM Grade, 8% |
| 50 ml tube | Fisher Scientific | 14-432-22 | Standard floor model and tabletop centrifuges |
| Glass Petri dish | Fisher Scientific | 08-747A | Reusable Petri Dishes: Complete (60 x 15 mm) |
| Forceps | Fisher Scientific | 22-327-379 | Fine Point Forceps |
| Cover glasses | Fisher Scientific | 12-518-105A | Rectangle; 30 x 22 mm; Thickness No. 1 |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl | Gelest | SIT8174.0 | |
| Acetic acid | Sigma-Aldrich | 320099 | Acetic acid ACS reagent, ≥99.7% |
| Hexane | Sigma-Aldrich | 296090 | anhydrous, 95% |
| Ethyl alcohol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| High-precision diamond scribing tool | Lunzer | PV-081-3 | Straight extended tip scribe, .020" (.50 mm) diameter by .200" (5.0 mm) tip length |
| Vacuum grease | Dow Corning | 14-635-5C | High-Vacuum Grease |
| 15 ml tube | Fisher Scientific | 14-959-49D | 15 ml conical centrifuge tubes with hydrophobic, biologically inert surface |
| 10x phosphate buffered solution | Fisher Scientific | BP399-500 | 1.37 M Sodium Chloride, 0.027 M Potassium Chloride, and 0.119 M Phosphate Buffer |
| 1 N sodium hydroxide | Sigma-Aldrich | 38215 | Sodium hydroxide concentrate |
| Collagen I, rat tail | BD Biosciences | 354236 | Rat tail |
| Micro centrifuge tube | Fisher Scientific | 02-681-332 | Volume: 2.0 ml; O.D. x L: 13 x 40 mm; sterile; single-wrapped |
| [header] | |||
| Carboxylate-modified microspheres | Invitrogen | F-8813 | Carboxylate-modified microspheres, 0.5 µm, yellow-green fluorescent (505/515), 2% solids |
| Rhodamine | Sigma-Aldrich | 83689 | Rhodamine B for fluorescence |
参考文献
- Adzick, N. S. The Molecular and Cellular Biology of Wound Repair. Ann Surg. 225 (2), 236 (1997).
- Reddi, A. H. Bone morphogenetic proteins: an unconventional approach to isolation of first mammalian morphogens. Cytokine Growth F. R. 8 (1), 11-20 (1997).
- Coussens, , Werb, Z, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Vital-Lopez, F. G., Armaou, A., Hutnik, M., Maranas, C. D. Modeling the effect of chemotaxis on glioblastoma tumor progression. AIChE J. 57 (3), 778-792 (2011).
- Hughes-Alford, S. K., Lauffenburger, D. A. Quantitative analysis of gradient sensing: towards building predictive models of chemotaxis in cancer. Curr. Opin. Cell Biol. 24 (2), 284-291 (2012).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol Cell Bio. 10 (7), 445-457 (2009).
- Cristini, V. Morphologic Instability and Cancer Invasion. Clin. Cancer Res. 11 (19), 6772-6779 (2005).
- Lammermann, T., et al. Rapid leukocyte migration by integrin-independent flowing and squeezing. Nature. 453 (7191), 51-55 (2008).
- Parent, C. A., Devreotes, P. N. A cell's sense of direction. Science. 284 (5415), 765-770 (1999).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. J. Exp. Med. 115, 453-466 (1962).
- Nelson, R. D., Quie, P. G., Simmons, R. L. Chemotaxis under agarose: a new and simple method for measuring chemotaxis and spontaneous migration of human polymorphonuclear leukocytes and monocytes. J. Immunol. 115 (6), 1650-1656 (1975).
- Rosoff, W. J., McAllister, R., Esrick, M. A., Goodhill, G. J., Urbach, J. S. Generating controlled molecular gradients in 3D gels. Biotechnol. Bioeng. 91 (6), 754-759 (2005).
- Wells, A., Kassis, J., Solava, J., Turner, T., Lauffenburger, D. A. Growth factor-induced cell motility in tumor invasion. Acta Oncol. 41 (2), 124-130 (2002).
- Wilkinson, P. C. Assays of leukocyte locomotion and chemotaxis. J. Immunol. Methods. 216 (1-2), 139-153 (1998).
- Bonvin, C., Overney, J., Shieh, A. C., Dixon, J. B., Swartz, M. A. A multichamber fluidic device for 3D cultures under interstitial flow with live imaging: development, characterization, and applications. Biotechnol. Bioeng. 105 (5), 982-991 (2010).
- Noo, L. J., et al. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device. Nat. Biotechnol. 20 (8), 826-830 (2002).
- Shao, J., et al. Integrated microfluidic chip for endothelial cells culture and analysis exposed to a pulsatile and oscillatory shear stress. Lab Chip. 9 (21), 3118-3125 (2009).
- Haessler, U., Kalinin, Y., Swartz, M. A., Wu, M. An agarose-based microfluidic platform with a gradient buffer for 3D chemotaxis studies. Biomed. Microdevices. 11 (4), 827-835 (2009).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat. Rev. Cancer. 3 (5), 362-374 (2003).
- Sixt, M., Lammermann, T. In vitro analysis of chemotactic leukocyte migration in 3D environments. Methods Mol. Biol. 769, 149-165 (2011).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. J. Cell Biol. 75 (2 Pt 1), 606-616 (1977).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. J. Cell. Sci. 99 (Pt 4), 769-775 (1991).
- Gilbert, D. L. Macromolecular diffusion through collagen membranes. Int. J. Pharm. 47 (1-3), 79-88 (1988).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys. J. 83 (3), 1650-1660 (2002).
- Weadock, K., Silver, F. H., Wolff, D. Diffusivity of 125I-calmodulin through collagen membranes: effect of source concentration and membrane swelling ratio. Biomaterials. 7 (4), 263-267 (1986).
- Erikson, A., Andersen, H. N., Naess, S. N., Sikorski, P., Davies, C. d. L. Physical and chemical modifications of collagen gels: impact on diffusion. Biopolymers. 89 (2), 135-143 (2008).
- Moghe, P. V., Nelson, R. D., Tranquillo, R. T. Cytokine-stimulated chemotaxis of human neutrophils in a 3-D conjoined fibrin gel assay. J. Immunol. Methods. 180 (2), 193-211 (1995).
- Sundd, P., et al. Live cell imaging of paxillin in rolling neutrophils by dual-color quantitative dynamic footprinting. Microcirculation. 18 (5), 361-372 (2011).
- Sanders, E. R. Aseptic Laboratory Techniques: Volume Transfers with Serological Pipettes and Micropipettors. J. Vis. Exp. (63), 2754 (2012).
- Stephenson, F. H. . Calculations for Molecular Biology and Biotechnology: A Guide to Mathematics in the Laboratory 2e. , (2010).
- Bar-Kochba, E., Toyjanova, J., Andrews, E., Kim, K., Franck, C. A Fast Iterative Digital Volume Correlation Algorithm for Large Deformations. Exp. Mech. In press, (2014).
- Shenoy, V., Rosenblatt, J. Diffusion of Macromolecules in Collagen and Hyaluronic Acid, Rigid-Rod-Flexible Polymer Composite Matrixes. Macromolecules. 28 (26), 8751-8758 (1995).
- Leddy, H., Guilak, F. Site-Specific Molecular Diffusion in Articular Cartilage Measured using Fluorescence Recovery after Photobleaching. Ann. Biomed. Eng. 31 (7), 753-760 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved