Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fluorescencia Biomembranas Fuerza Sonda: La cuantificación simultánea de Cinética-Receptor ligando y encuadernación inducida Señalización Intracelular en una sola célula
* Estos autores han contribuido por igual
En este artículo
Resumen
We describe a technique for concurrently measuring force-regulated single receptor-ligand binding kinetics and real-time imaging of calcium signaling in a single T lymphocyte.
Resumen
Interacciones ligando-receptor de membrana median muchas funciones celulares. Cinética de unión y señalización corriente abajo provocada por estas interacciones moleculares son probablemente afectadas por el entorno mecánico en el que la unión y la señalización tienen lugar. Un estudio reciente demostró que la fuerza mecánica puede regular el reconocimiento del antígeno por y activación del receptor de células T (TCR). Esto fue posible gracias a una nueva tecnología que desarrollamos y fluorescencia denominado sonda de fuerza biomembrana (fBFP), que combina la espectroscopia de fuerza de una sola molécula con microscopía de fluorescencia. El uso de un glóbulo rojo humano ultra-suave como el sensor de fuerza sensible, una cámara de alta velocidad y en tiempo real técnicas de seguimiento de formación de imágenes, la fBFP es de ~ 1 pN (10 -12 N), ~ 3 nm y ~ 0,5 mseg en la fuerza, la resolución espacial y temporal. Con la fBFP, se puede medir con precisión la cinética individuales receptor-ligando vinculantes conforme al reglamento vigente y cal intracelular simultáneamente imagen desencadenó unión-CIUM señalización en una sola célula viva. Esta nueva tecnología puede ser utilizada para estudiar otras interacciones receptor-ligando de la membrana y de señalización en otras células bajo regulación mecánica.
Introducción
-Célula a célula y célula-matriz extracelular-a (ECM) adhesión está mediada por la unión entre receptores de la superficie celular, las proteínas ECM, y / o lípidos 1. La unión permite que las células para formar estructuras funcionales 1, así como reconocen, se comunican, y reaccionan con el medio ambiente 3.1. A diferencia de las proteínas solubles (por ejemplo, citoquinas y factores de crecimiento) que se unen a partir de una de tres dimensiones (3D) fase de fluido sobre los receptores de la superficie celular, receptores de adhesión celular forman enlaces con sus ligandos través de un hueco de la unión estrecha para puente de dos superficies opuestas que limitan molecular difusión en una dimensión (2D) de interfaz de dos 4-7. En contraste con la cinética de 3D que se mide comúnmente por ensayos de unión tradicionales (por ejemplo, resonancia de plasmón superficial o SPR), la cinética 2D han de ser cuantificados con técnicas especializadas tales como microscopía de fuerza atómica (AFM) 8-10, 11,12 cámara de flujo, micropipeta 13,14, ópticopinzas 15 y sonda de fuerza biomembrana (BFP) 16-21.
Más que simplemente proporcionar vinculación física para la cohesión celular, moléculas de adhesión son un componente importante de la maquinaria de señalización para la célula para comunicarse con sus alrededores. No ha habido un creciente interés en la comprensión de cómo el compromiso ligando de moléculas de adhesión inicia la señalización intracelular y cómo la señal inicial se transduce dentro de la célula. Intuitivamente, propiedades de unión receptor-ligando puede afectar las señales que induce. Sin embargo, es difícil de diseccionar las relaciones mecánicas entre la interacción extracelular y eventos de señalización intracelular usando conjunto tradicional de ensayos bioquímicos debido a sus muchas limitaciones, por ejemplo, una resolución temporal pobres y la completa falta de resolución espacial. Métodos que permiten tanto biofísico (2D cinética de unión al receptor-ligando) y (señalización) observaciones bioquímicas en vivo existentecélulas incluyen sustratos de rigidez sintonizable 22, matrices pilar de elastómero 23 y dispositivos de cámara de flujo / microfluidos incorporados con capacidad de fluorescencia 24-26. Sin embargo, las lecturas de la señalización y de unión al receptor-ligando tienen que obtener por separado (con mayor frecuencia por diferentes métodos), por lo que es difícil de diseccionar las relaciones temporales y espaciales de las características de los bonos con eventos de señalización.
BFP convencional es una fuerza de la espectroscopia ultrasensible con alta resolución espacio-temporal 17. Se utiliza una célula flexible de color rojo sangre (RBC) como un sensor de fuerza, lo que permite la medición de la cinética de 2D de una sola molécula, propiedades mecánicas y los cambios conformacionales 14,16,19-21,27-29. A BFP basado imágenes fluorescentes (fBFP) se correlaciona la cinética de unión al receptor-ligando con la señalización de las células activadas por la unión a escala de una sola molécula. Con esta configuración, en las actividades de señalización celular situ en el contexto de mechani superficialescal estimulación se observó en las células T 27. El fBFP es versátil y se puede utilizar para estudios de adhesión celular y la señalización mediada por otras moléculas en otras células.
Protocolo
Este protocolo sigue las directrices de y ha sido aprobado por el comité de ética de la investigación humana del Instituto de Tecnología de Georgia.
1. glóbulos rojos humano Aislamiento, biotinilación y la osmolaridad de Ajuste
Nota: Paso 1.1 debe ser realizado por un médico profesional entrenado como una enfermera, con una Junta de Revisión Institucional aprobado protocolo.
- Obtener 8-10 l (una gota) de sangre de pinchazo en el dedo y añadir a 1 ml de la tampón carbonato / bicarbonato (Tabla 1 y 2). Vórtice suavemente o pipetear la mezcla y centrifugar durante 1 min a 900 x g. Desechar el sobrenadante y lavar una vez más.
- En un pequeño vaso de precipitados pesar 3,5-4 mg de biotina-PEG3500-NHS enlazador (Tabla 1). Disolver en el tampón carbonato / bicarbonato para que la concentración final de 3 mg / ml.
- Mezclar 171 l de tampón carbonato / bicarbonato, 10 l de paquete de RBC y 1.049 l deLa biotina-PEG3500-NHS solución enlazador e incubar a TA durante 30 min. Lavar el RBC una vez con tampón de carbonato / bicarbonato y luego dos veces con N2-5% de tampón (Tabla 1 y 2).
- Mientras tanto, colocar la botella con el casquillo de engarce aflojado en un desecador de vacío de vidrio lleno de desecantes en la parte inferior y el vacío durante 5 min, y llenar el desecador con argón. Apriete la tapa y tomar la botella. Selle la botella con una película de plástico de parafina (Tabla 1), colóquelo en un recipiente lleno de desecantes en la parte inferior y guárdela en -20 ° C.
Nota: Los pasos que implican el uso de biotina-PEG3500-NHS enlazador, incluyendo 1.2 a 1.4 (excepto para la incubación y lavado en 1,3), necesario llevar a cabo lo más rápido posible. - Diluir la nistatina en N2-5 tampón% para hacer una concentración final de 40 mg / ml. Mezclar 5 l de biotina RBC con 71,4 l de nistatina (Tabla 1) solución e incubar durante 1 hora a 0 Y# 176; C. Lavar dos veces con tampón N2-5% y tienda con N2-5% de tampón + 0,5% de BSA (Tabla 1) en el refrigerador (4 ° C).
2. abalorios Silanización
- La limpieza de la superficie del grano
- Pesar 50 mg de perlas de vidrio en polvo y volver a suspender en 500 l de agua DI.
- Mezclar 0,5 ml de 30% H 2 O 2 (Tabla 1) con 9,5 ml de agua DI en un vaso de precipitados de 50 ml, a continuación, añadir 2 ml de NH4OH concentrado (Tabla 1) y llevar esta solución a una caldera en una placa caliente .
- Añadir las perlas de vidrio a la solución en ebullición y mantener la ebullición durante otros 5 min. Agite suavemente la solución cada min.
- Después de hervir, transferir ~ 5 ml de esta suspensión de perlas caliente en un tubo de 15 ml micro-centrífuga y rellene con agua RT DI. Centrifugar a 3500 xg durante 5 minutos, retirar y desechar el sobrenadante.
- Traslado otros 5 ml de suspensión de perlas caliente y añadir a las perlas lavadas, Recargar con más agua DI, mezclar bien y centrifugar de nuevo. Repita este procedimiento hasta que se utiliza alrededor de 50 ml de agua DI, que será un total de 4 a 5 veces de lavado.
- Transferir la suspensión de perlas en un vial de 1 ml. Repetir el lavado de las perlas con metanol (Tabla 1) por centrifugación a 17.000 xg durante 5 min para 3 veces, y finalmente volver a suspender las perlas en 1 ml de metanol al 100%.
- Bead Superficie Tiolación
- Para un tubo de centrífuga de 50 ml añadir 45,6 ml de metanol, 0,4 ml de ácido acético (Tabla 1), 1,85 ml de agua DI, 1,15 ml de 3-mercaptopropiltrimetoxisilano (MPTMS) (Tabla 1) y 1 ml de perlas en suspensión preparado en 2.1, luego incubar a temperatura ambiente durante 3 hr.
- Después de la reacción, eliminar todos los reactivos por lavado una vez con metanol fresco, y volver a suspender las perlas en 500 l de metanol. Divida esta suspensión de perlas de vidrio concentrado en un conjunto de 20 viales de vidrio limpios y secos contapones de rosca. Se evapora el metanol mediante el uso de un chorro de argón seco y gire lentamente los viales a fin de hacer una capa delgada de perlas secas en los lados de cada vial.
- Coloque los viales de perlas en un horno de secado precalentado a 120 ° C durante 5 minutos y luego sacar y rápidamente colocar la tapa (s) libremente en. Colocar los viales en un desecador de vacío de vidrio lleno de desecantes en la parte inferior y aspire el desecador con una bomba de vacío hasta que se enfríe.
- Purgar el desecador aspiradora con argón seco para llevar el desecador a presión atmosférica normal. Retire la tapa del desecador y rápidamente volver a apretar la tapa (s) en el vial. Selle los viales con la película de parafina plástico y almacenarlos a temperatura ambiente en una caja seca de almacenamiento oscuro.
- Tras su uso inmediato, tomar un vial de granos secos y lavar una vez con tampón fosfato (Tablas 1 y 2), vuelva a suspender en 50 l de tampón fosfato y se almacena a 4 ° C. Esta preparación concentrada del grano se hará referencia acomo "talones MPTMS" en los siguientes pasos.
Nota: Con el almacenamiento adecuado, cuentas MPTMS podrían seguir funcionando durante un máximo de tres meses.
3. Bead funcionalización
- Recubrimiento covalentemente proteínas en los granos
- Tome un cierto volumen (por ejemplo, 2,5 l) de las acciones de proteínas y mezclar con un volumen igual de tampón carbonato / bicarbonato para hacer Solución 1.
Nota: El volumen depende de la población de la concentración y la densidad de sitio final deseado de la proteína en la superficie de los granos. - En un pequeño vaso de precipitados de sopesar 2-3 mg de MAL-PEG3500-NHS enlazador (Tabla 1) y disolver con tampón carbonato / bicarbonato para llegar a una concentración final de 0,231 mg / ml.
- Mezcla de solución 1 con un volumen igual de la solución enlazador preparado en 3.1.2. Incubar la mezcla a TA durante 30 min para hacer la solución 2.
- Mientras tanto, colocar la botella con el casquillo de engarce aflojado en un desecador de vacío de vidrio filled con desecantes en la parte inferior y de vacío durante 5 min, y llenar el desecador con argón. Apriete la tapa y tomar la botella. Selle la botella con una película de parafina plástico, colóquelo en un recipiente lleno de desecantes en la parte inferior y guárdela en -20 ° C.
Nota: Los pasos que implican el uso de MAL-PEG3500-NHS enlazador, incluyendo 3.1.2-3.1.4 (a excepción de la incubación en 3.1.3), deberán realizarse lo más rápido posible. - Mezclar 5 l de perlas de MPTMS con la solución 2 y añadir tampón de fosfato (Tabla 1) para hacer un volumen final de 250 microlitros.
- Se incuban las perlas de la noche a TA, lavar 3 veces con tampón de fosfato, y volver a suspender en 100 l de tampón fosfato y se almacena a 4 ° C.
- Tome un cierto volumen (por ejemplo, 2,5 l) de las acciones de proteínas y mezclar con un volumen igual de tampón carbonato / bicarbonato para hacer Solución 1.
- Preparación de proteína / perlas de estreptavidina (SA) Revestidos
- Siga el protocolo 3.1.1-3.1.4.
- Mezclar 5 l de perlas de MPTMS con una solución de 2 y 5 l de 4 mg / ml de estreptavidina-maleimida (SA-MAL) (Tabla 1) Solución y luego añadir tampón de fosfato para hacer un volumen final de 250 microlitros.
- Incubar las perlas durante la noche a RT, lavar 3 veces con tampón de fosfato, y finalmente volver a suspender en 100 l de tampón de fosfato y se almacenan a 4 ° C.
- El recubrimiento de estreptavidina en los granos de cristal
- Mezclar 5 l de perlas de MPTMS con 5 l de 4 mg / ml solución SA y añadir 140 l de tampón de fosfato.
- Se incuban las perlas de la noche a TA, lavar 3 veces con tampón de fosfato, y volver a suspender en 50 l de tampón fosfato y se almacena a 4 ° C.
- Recubrir las perlas SA recubiertas con una proteína biotinilada
- Mezclar 5 l de perlas SA recubiertas con la proteína (volumen dependiendo de la densidad de revestimiento deseado) y se añade tampón fosfato para hacer que el volumen final sea 100 l.
- Incubar la mezcla durante la noche a 4 ° C o durante 3 horas a RT, lavar 3 veces con tampón de fosfato, y re-suspender en 50 l phosphate tampón y almacenar a 4 ° C.
4. Preparación de la célula
Nota: Para purificar las células, seguir los protocolos de purificación de células estándar correspondientes al tipo de células en uso, por ejemplo las células T 27 o ciertas líneas celulares 21,29.
- Para los experimentos de fBFP, una vez que se prepara la suspensión de células, añadir Fura2-AM (Tabla 1) disuelto en DMSO en la suspensión de células para alcanzar una concentración final de 2 mM, incubar durante 30 min a RT y luego lavar una vez. Mantenga esta suspensión celular cargado fluorescente en la oscuridad hasta su uso.
5. Preparación para Micropipetas y una Cámara de la célula
- Preparación Micropipetas
- Cortar tubos capilares de vidrio largo (Tabla 1) con un cortador de vidrio en piezas cortas de alrededor de 3 pulgadas de largo. Monte una pieza en el extractor micropipeta (Tabla 1), haga clic en el "Pull", perotonelada de modo que el centro del capilar se calienta por la máquina y el capilar se tiró de los dos extremos para hacer dos capilares con puntas afiladas (micropipetas primas).
Nota: Siguiendo la pauta de productos, la morfología deseada de la pipeta prima tiene 8.6 mm de la forma cónica y 0,1 a 0,5 micras punta. - Montar una pipeta prima en el soporte de la pipeta de la forja micropipeta (Tabla 1). El calor para fundir la esfera de cristal en la fragua. Inserte la punta de la pipeta en bruto dentro de la esfera de cristal. Enfriar la esfera de cristal y tirar la pipeta prima para romperlo desde fuera y dejar la punta dentro de la esfera. Repita este procedimiento hasta que se obtiene el orificio de la punta deseada.
Nota: Los ejemplos de un diámetro interior punta de la micropipeta: 2,0-2,4 micras para un RBC, ~ 1,5 m para una perla, ~ 2-4 micras para una célula T y -7 m para una célula de hibridoma.
- Cortar tubos capilares de vidrio largo (Tabla 1) con un cortador de vidrio en piezas cortas de alrededor de 3 pulgadas de largo. Monte una pieza en el extractor micropipeta (Tabla 1), haga clic en el "Pull", perotonelada de modo que el centro del capilar se calienta por la máquina y el capilar se tiró de los dos extremos para hacer dos capilares con puntas afiladas (micropipetas primas).
- La construcción de una Sala de la célula
Nota: la cámara de la celda está construida sobre la base de una casa-madsoporte de la cámara e, que consta de dos piezas de cuadrados de metal (cobre / aluminio) y un mango que los une (Figura 1D). - Cortar un 40 mm x 22 mm x 0,2 mm cubreobjetos utilizando un cortador de vidrio en dos piezas 40 mm x 11 mm x 0,2 mm (cubreobjetos 1 y 2). Glue cubreobjetos 1 por la grasa hacia el lado superior del soporte de cámara en la forma en que puentea los dos cuadrados de metal, y de manera similar cubreobjetos pegamento 2 para el lado inferior, que formará una cámara de la celda paralelo-cubreobjetos (Figura 1D).
- Utilizar una pipeta para inyectar 200 l de tampón experimental entre las dos cubreobjetos. Asegúrese de que la memoria intermedia se une a ambos cubreobjetos. Gire suavemente y agitar la cámara para que el búfer tocar ambos extremos de la cámara.
- Inyectar cuidadosamente aceite mineral en ambos lados de la cámara de flanqueando la sellando de este modo la memoria intermedia desde el aire abierto experimental zona de amortiguación. Inyectar suspensiones de cuentas de la sonda (por ejemplo, perlas recubiertas con pMHC), glóbulos rojos y blancos(por ejemplo, células T) en las regiones superior, media e inferior de la zona de amortiguamiento, respectivamente.
6. BFP experimento
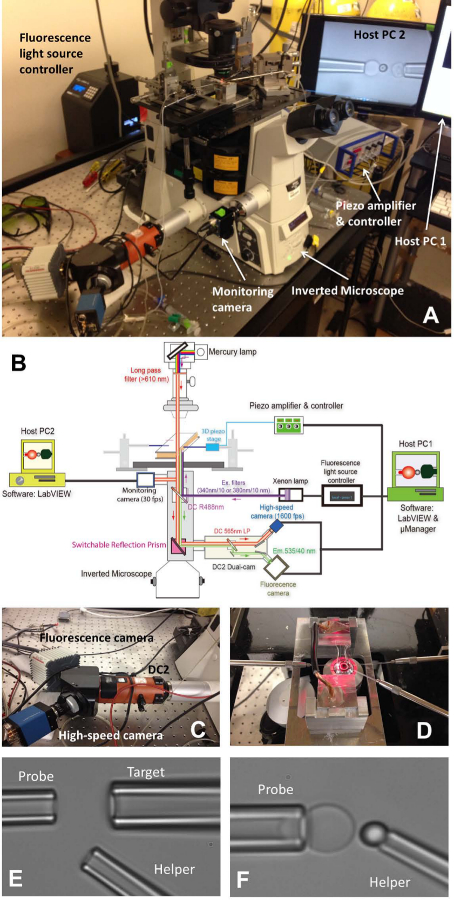
Figura 1: Montaje fBFP (A) Un cuadro visión general del sistema de hardware fBFP.. (B) Un dibujo esquemático del sistema de hardware fBFP. (C) El sistema de doble leva "DC2" (naranja) sobre el que se montan la cámara de alta velocidad (azul) y una cámara de fluorescencia (blanco). (D) La platina del microscopio que se adapta una cámara de experimentación y tres sistemas de manipulación de micropipeta. Micrografías de ajuste de BFP en una cámara experimental (E y F). (E) Micropipetas que muestra el montaje de la pipeta sonda (izquierda), una pipeta de destino (arriba a la derecha) y de la pipeta helper (menor rderecho). La colocación del talón (F) de la sonda. Un cordón de la sonda fue manipulado por una pipeta ayudante y adjunto a un ápice de RBC para formar una sonda de fuerza. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Encienda el microscopio (Tabla 1) y fuente de luz. Coloque la cámara en la platina del microscopio principal (Figura 1 A, D).
- Instale los tres micropipetas de BFP (Figura 1D Izquierda:. Sonda, para tomar un RBC, a la derecha: blanco, para tomar un celular o perla, abajo a la derecha: ayudante, para tomar un grano).
- Use un micro-inyector (Tabla 1) para rellenar una micropipeta con tampón experimental. Quítese el soporte de pipetas (Tabla 1) y manténgalo en un lugar más bajo para permitir que el agua que gotea de la punta. Inserte rápidamente la micropipeta en la punta titular y asegurarse de que nadie la burbuja de aire se mete en la micropipetadurante la inserción. Apriete el tornillo del soporte.
- Montar cada titular de la pipeta en su correspondiente micro-manipulador. Empuje la micropipeta hacia la cámara de manera que sus puntas entran en la zona de amortiguamiento de cámara. Ajuste la posición de la micropipeta y encontrarlos bajo el microscopio de campo de visión.
- Mover todo el escenario soporte de la cámara para encontrar las colonias de tres elementos (los glóbulos rojos, los objetivos y las cuentas de la sonda) uno por uno. Ajuste la posición de la micropipeta correspondiente girando las perillas de los manipuladores de dejar la punta del enfoque micropipeta una célula / perla. Aspirar el de células / perlas mediante el ajuste de la presión dentro de la micropipeta correspondiente. Los tres micropipetas capturarán sus correspondientes elementos.
- Mover todo el escenario soporte de la cámara para encontrar un espacio abierto lejos de las colonias de los elementos inyectados en la que se realiza el experimento. Cambie el modo visual microscopio para visualizar la imagen en el borradoruter programa en la pantalla de ordenador. Mueva los tres elementos de puntas de pipeta en el campo de visión del programa.
- Alinear la perla de la sonda y RBC, y maniobrar cuidadosamente el cordón de la sonda hasta el ápice de la RBC, incidir brevemente la gota sobre el RBC y suavemente retraer. Ajuste la presión de la micropipeta ayudante para soplar suavemente el cordón de distancia, por lo que se dejará pegado a la RBC ápice (Figura 1F). Aléjese la micropipeta ayudante y alinear el objetivo y cordón de la sonda (Figura 2).
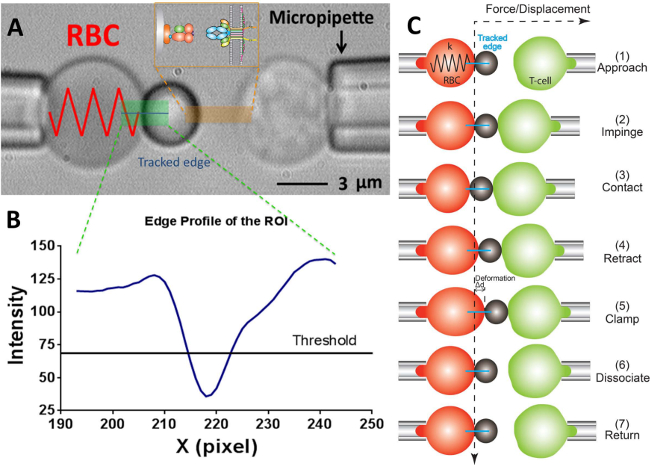
Figura 2:. Esquema de BFP y su ciclo de ensayo (A) Video-micrografía que representa una sonda de fuerza (izquierda) y un objetivo de células T (derecha) aspirado por su respectiva sonda de fuerza estacionaria pipettes.The consta de un RBC hinchado y una unida ligando de soporte de talón. El receptor-cojineteDe células T (target) está montado en un piezotranslator alineado opuesto a la sonda. El retorno de la inversión se indica en verde. El seguidor de borde se indica en una línea azul. El inserto representa el ligando (pMHC, con el lado del talón) y el receptor (TCR, lado de células T) par sobre las dos superficies opuestas en la zona marcada en naranja. (B) El perfil de intensidad del borde del talón en (A). La región de retorno de la inversión en el x -dirección se traza como eje x (en número de píxeles) y la intensidad de la luz (en valor de escala de grises) de media por hurgar en la basura 30 píxeles a lo largo de la dirección x y. (C) La deflexión de la RBC y la posición de la perla y la diana (células T) en un ciclo de prueba de ensayo de fuerza de sujeción. Las líneas de trazos verticales y horizontales indican la posición de fuerza cero del ápice RBC y el curso del tiempo, respectivamente. El seguidor borde de la línea de la deformación de RBC se muestra en azul en cada panel. Las mismas aún menos pasos que se adopten en la frecuencia de la adhesiónensayo (que carece de los pasos de "pinza" y "disociar") y ensayo de fluctuación térmica (que carece de la etapa de "disociar").
- En el programa, en la ventana de campo de visión utilizar las herramientas en el programa para medir el radios respectivos de la micropipeta sonda (R p), el RBC (R 0), el área de contacto circular entre el RBC y el talón de la sonda (R c) , que permite la estimación de la constante de muelle del RBC (k) por la siguiente ecuación 17.30,
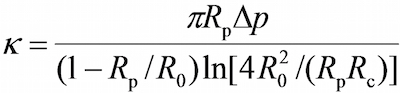
donde Δ p es la presión de aspiración en la punta de la pipeta de la sonda.
Nota: Se desprende de la ley de Hooke la que la fuerza de unión, F, puede ser cuantificada por el producto de la constante de muelle y el desplazamiento de la perla de la sonda (d), es decir, F = d (Figura 2C). - Introduzca la constante del resorte RBC deseada en el programa (Por favor refiérase a la sección Protocolo 6.6. La constante del resorte se ajusta normalmente a 0.25 o 0.3 pN / nm para el ensayo de la frecuencia de ensayo fuerza de sujeción y la adherencia y 0,1 pN / nm para el ensayo de fluctuación térmica) , que devolverá una presión de aspiración requerida en la unidad de centímetro de agua. Ajuste la altura del depósito de agua que se vincula a la pipeta de la sonda hasta que se alcance la presión de aspiración requerida.
- Dibuja una línea horizontal a través del ápice de RBC, que dará lugar a una curva en la ventana adyacente indica el brillo (valor de escala de grises) de cada píxel a lo largo de esta línea. Arrastre la línea límite de estar en torno a la mitad de la profundidad de la curva (Figura 2A, B).
Nota: El punto mínimo de la curva de brillo por debajo de la línea de umbral indica la posición del límite del grano, por lo tanto sólo se permite un mínimo local. Si dos o más mínimos locales son en la actualidad,indica que la imagen no es óptima (probablemente debido a la imagen quede fuera de foco, o una mala alineación entre el talón de la sonda y el RBC). - Seleccione el modo de experimento deseada: ensayo de fluctuación térmica, ensayo de adherencia de frecuencia o ensayo fuerza de sujeción. Establezca los parámetros como desee (por ejemplo, la fuerza de choque = 15 pN, tasa de carga = 1000 PN / s, tiempo de contacto = 1 seg, la fuerza de sujeción = 20 pN (para el ensayo de sujeción de fuerza), etc).
- Haga clic en "Inicio", que permite que el programa para mover la pipeta objetivo y conducir el destino dentro y fuera de contacto con la sonda (véase la sección Resultados representante para más detalles). La recolección de datos se realiza en paralelo, que registra la posición de la perla de la sonda en tiempo real. Detenga el programa haciendo clic en el botón "experimento Stop", momento en el cual se abrirá una ventana a cabo para permitir guardar los datos adquiridos.
7. Fluorescencia BFP (fBFP) experimento
- Para utilizar el fluoréscence función del sistema de BFP, encender la fuente de luz de excitación (Tabla 1) y la cámara de fluorescencia (Tabla 1), que son controlados por un programa separado (Tabla 1). En el programa, seleccionar los parámetros de la fluorescencia de imagen, incluyendo la ganancia, la exposición, los canales de excitación (en este caso, 340 nm y 380 nm de luz), etc. Siga todas las preparaciones en el protocolo experimento BFP, incluyendo la alineación de la sonda y el objetivo, lo que permitirá la visualización de la imagen fluorescente en vivo de células diana excitado por 340 nm o 380 nm luz de excitación.
- Utilice la herramienta de corte a más o menos la sección de la zona dentro de la cual la célula se mantendrá durante todo el período de registro.
Nota: Debido a la utilización del ciclo de aproximación-contact-retracción, la célula estará moviendo hacia adelante y hacia atrás repetidamente, por lo tanto el área de sección es mucho más grande que la propia célula. - Haga clic en "Grabar" para permitir que el un 340 nmd 380 nm de luz para excitar alternativamente el colorante de fluorescencia intracelular (Fura2), y un par de imágenes de fluorescencia correspondiente se grabará alternativamente alrededor de una vez cada segundo. Al mismo tiempo, haga clic en "Inicio" en el programa para comenzar el experimento BFP para el análisis de la interacción molecular y el experimento de imágenes de fluorescencia para controlar la señalización de calcio intracelular. El sistema producirá un archivo de datos brutos para la unión receptor-ligando (véase la Figura 6A abajo) y una serie de imágenes fluorescentes en formato .tiff para las señales de calcio.
Análisis 8. Datos
- Análisis de Datos BFP
- Análisis de los datos para el ensayo de adherencia de frecuencia
- Secuencialmente inspeccionar la señal de cada ciclo de "fuerza en función del tiempo" y simplemente grabar el que los ciclos contienen un evento adhesión y cuáles no, y resumir para producir una frecuencia media de adherencia.
- Recoger la fuerza de ruptura de cada evento de la adhesión, quees el valor de pico de la fuerza linealmente en rampa antes de la rotura de bonos. Después de recoger una cantidad suficiente de las fuerzas de ruptura en una gama de velocidades de rampa, derivar la distribución de la fuerza de ruptura en cada velocidad de rampa del que se deriva la fuerza dependiente de fuera de velocidad de disociación receptor-ligando usando análisis de espectroscopia de fuerza dinámica 18,31.
- Análisis de los datos para el ensayo de fluctuación térmica
- Secuencialmente inspeccionar la señal de fase de cierre de cada ciclo, lo que probablemente contiene múltiples eventos de la asociación y disociación de bonos. Utilice el nivel de fluctuación térmica fase de sujeción (la desviación estándar promedio de un intervalo de deslizamiento de 70 puntos de tiempo secuenciales de la posición del talón) en la "fuerza en función del tiempo" señal como una guía para distinguir los eventos de la asociación y disociación de bonos, ya que un vínculo formación corresponde a una disminución en la fluctuación térmica.
- Designar el intervalo desde el instante de disociación del enlace (cuando el Thermal fluctuación reanuda al nivel normal) para el instante de la próxima formación de un enlace con el tiempo de espera, y designar la duración de la unión de su asociación a la disociación como vínculo de por vida, que tanto se recogen durante la inspección de datos. Calcular el tiempo medio de espera y el promedio de vida útil de bonos, que, respectivamente, reflejan el recíproco de sobre-tasa y la de fuera de la tasa de bajo cero fuerza de 16,30.
- Análisis de los datos de ensayo de fuerza de sujeción
- Parámetros de registro de todos los eventos de toda la vida, incluyendo la fuerza media y la duración vínculo con el número de secuencia, así como la hora de inicio y la hora de finalización, lo que permitirá una para dibujar una curva de vida acumulada (por ejemplo, la figura 6C, curva amarilla).
- Se recoge una cantidad suficiente de eventos de toda la vida bajo una gama de fuerzas. Grupo de ellos en diferentes contenedores de fuerza, que producirá una vida media en cada bin fuerza, y el rendimiento en conjunto una "vida media vs. curva de fuerza "(Figura 4).
- Análisis de los datos para el ensayo de adherencia de frecuencia
- Análisis de fluorescencia de calcio Imaging Datos
- Ajustar el umbral de intensidad hasta que las imágenes de fluorescencia muestran un contorno claro de la célula en ambos canales 340 nm y 380 nm y sin ruido de fondo (Figura 5A, B). A continuación, revise el Ca 2+ trama de señal intracelular por marco con un pseudo-color que indica el nivel de intensidad (Figura 6B), que se deriva basa en la relación de intensidad de 340 nm / 380 nm, para generar la "intensidad normalizada Ca 2+ vs . curva de tiempo "(Figura 6C). Utilice las imágenes de fluorescencia pseudo-color para producir una película que muestra el segundo nivel de fluorescencia por segundo.
Resultados
La técnica BFP fue iniciado por el laboratorio de Evans en 1995 17. Esta herramienta picoforce se ha utilizado ampliamente para medir las interacciones de proteínas inmovilizadas sobre superficies, a fin de analizar la cinética de dos dimensiones de las moléculas de adhesión interacción con sus ligandos 16,19,20, 30, para medir la elasticidad molecular 21,29, y para determinar conformacional proteína cambia 21. Para una fBFP, un conjunto adicional de dispositivos relaci...
Discusión
Un experimento exitoso fBFP conlleva algunas consideraciones críticas. En primer lugar, para el cálculo de la fuerza para ser fiable, la micropipeta, el RBC, y el talón de la sonda deben estar alineados lo más cerca posible coaxial posible. La proyección de la RBC dentro de la pipeta debe ser alrededor de un diámetro pipeta sonda de manera que la fricción entre el RBC y la pipeta es insignificante. Para un típico RBC humano, el diámetro óptimo de la pipeta es 2,0-2,4 micras, que produce un mejor ajuste de la E...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Research related to this paper and the development of the fBFP technology in the Zhu lab were supported by NIH grants AI044902, AI077343, AI038282, HL093723, HL091020, GM096187, and TW008753. We thank Evan Evans for inventing this empowering experimental tool, and members of the Evans lab, Andrew Leung, Koji Kinoshita, Wesley Wong, and Ken Halvorsen, for helping us to build the BFP. We also thank other Zhu lab members, Fang Kong, Chenghao Ge and Kaitao Li, for their helps in the instrumentation development.
Materiales
| Name | Company | Catalog Number | Comments |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4 • H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | N2-5% buffer preparation |
| Potassium chloride (KCl) | Sigma-Aldrich | P9541 | N2-5% buffer preparation |
| Potassium phosphate monobasic (KH2PO4) | Sigma-Aldrich | P5655 | N2-5% buffer preparation |
| Sucrose | Sigma-Aldrich | S0389 | N2-5% buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Bead functionalization |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Nystatin | Sigma-Aldrich | N6261 | RBC osmolarity adjustment |
| Ammonium Hydroxide (NH4OH) | Sigma-Aldrich | A-6899 | Glass bead silanization |
| Methanol | BDH | 67-56-1 | Glass bead silanization |
| 30% Hydrogen Peroxide (H2O2) | J. T. Barker | Jan-86 | Glass bead silanization |
| Acetic Acid (Glacial) | Sigma-Aldrich | ARK2183 | Glass bead silanization |
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Borosilicate Glass beads | Distrilab Particle Technology | 9002 | Glass bead functionalization |
| Streptavidin−Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
| BSA | Sigma-Aldrich | A0336 | Ligand functionalizing |
| Fura2-AM | Life Technologies | F-1201 | Intracellular calcium fluorescence dye loading |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650 | Intracellular calcium fluorescence dye loading |
| Quantibrite PE Beads | BD Biosciences | 340495 | Density quantification |
| Flow Cytometer | BD Biosciences | BD LSR II | Density quantification |
| Capillary Tube 0.7-1.0 mm x 30 inches | Kimble Chase | 46485-1 | Micropipette making |
| Flaming/Brown Micropipette Puller | sutter instrument | P-97 | Micropipette making |
| Pipette microforce | Narishige | MF-900 | Micropipette making |
| Mineral Oil | Fisher Scientific | BP2629-1 | Chamber assembly |
| Microscope Cover Glass | Fisher Scientific | 12-544-G | Chamber assembly |
| Micro-injector | World Precision Instruments | MF34G-5 | Chamber assembly |
| 1 ml syringe | BD | 309602 | chamber assembly |
| Micropipette holder | Narishige | HI-7 | Chamber assembly |
| Home-designed mechanical parts and adaptors fabrications using CNC machining. | Biophysics Instrument | All parts are customized according to the CAD designs. | BFP system |
| Microscope (TiE inverted) | Nikon | MEA53100 | BFP system |
| Objective CFI Plan Fluor 40x (NA 0.75, WD 0.72 mm, Spg) | Nikon | MRH00401 | BFP system |
| Camera, GE680, 640 x 480, GigE, 1/3" CCD, mono | Graftek Imaging | 02-2020C | BFP system |
| Prosilica GC1290 - ICX445, 1/3", C-Mount, 1280 x 960, Mono., CCD, 12 Bit ADC | Graftek Imaging | 02-2185A | BFP system |
| Manual submicron probehead with high resolution remote control | Karl Suss | PH400 | BFP system |
| Anti-vibration table (5’ x 3’) | TMC | 77049089 | BFP system |
| 3D manual translational stage | Newport | 462-XYZ-M | |
| SolidWorks 3D CAD software | SOLIDWORKS Corp. | Version 2012 SP5 | BFP system |
| LabVIEW software | National Instruments | Version 2009 | BFP system, BFP program |
| 3D piezo translational stage | Physik Instrumente | M-105.3P | BFP system |
| Linear piezo accuator | Physik Instrumente | P-753.1CD | BFP system |
| Micromanager software | Version 1.4 | fBFP system, fluorescence imaging program | |
| Dual Cam (DC-2) | Photometrics | 77054724 | fBFP system |
| Dual Cam emission filter (T565LPXR) | Photometrics | 77054725 | fBFP system |
| Fluorescence Camera | Hamamatsu | ORCA-R2 C10600-10B | fBFP system |
| Plastic paraffin film (Parafilm) | Bemis Company, Inc | PM996 | bottle sealing |
| Carbonate/bicarbonate buffer (pH 8.5) | 8.4 g/L sodium carbonate (Na2CO3), 10.6 g/L sodium bicarbonate (NaHCO3) | ||
| Phosphate buffer (pH 6.5-6.8) | 27.6 g/L NaPhosphate monobasic (NaH2PO4 • H2O), 28.4 g/L Anhy. NaPhosphate dibasic (Na2HPO4) | ||
| N2-5% buffer (pH 7.2) | 20.77 g/L potassium chloride (KCl), 2.38 g/L sodium chloride (NaCl), 0.13 g/L potassium phosphate monobasic (KH2PO4), 0.71 g/L anhy. sodium phosphate dibasic (Na2HPO4), 9.70 g/L sucrose |
Referencias
- Aplin, A. E., Howe, A., Alahari, S. K., Juliano, R. L. Signal transduction and signal modulation by cell adhesion receptors: the role of integrins, cadherins, immunoglobulin-cell adhesion molecules, and selectins. Pharmacological reviews. 50, 197-263 (1998).
- Davis, M. M., Bjorkman, P. J. T-cell antigen receptor genes and T-cell recognition. Nature. 334, 395-402 (1988).
- Dado, D., Sagi, M., Levenberg, S., Zemel, A. Mechanical control of stem cell differentiation. Regenerative medicine. 7, 101-116 (2012).
- Edwards, L. J., Zarnitsyna, V. I., Hood, J. D., Evavold, B. D., Zhu, C. Insights into T cell recognition of antigen: significance of two-dimensional kinetic parameters. Frontiers in immunology. 3, 86 (2012).
- Zhu, C., Jiang, N., Huang, J., Zarnitsyna, V. I., Evavold, B. D. Insights from in situ analysis of TCR-pMHC recognition: response of an interaction network. Immunological reviews. 251, 49-64 (2013).
- Huang, J., Meyer, C., Zhu, C. T. T cell antigen recognition at the cell membrane. Molecular immunology. 52, 155-164 (2012).
- Zarnitsyna, V., Zhu, C. T. T cell triggering: insights from 2D kinetics analysis of molecular interactions. Physical biology. 9, 045005 (2012).
- Binnig, G., Quate, C. F., Gerber, C. Atomic Force Microscope. Physical Review Letters. 56, 930-933 (1986).
- Marshall, B. T., et al. Direct observation of catch bonds involving cell-adhesion molecules. Nature. 423, 190-193 (2003).
- Kong, F., Garcia, A. J., Mould, A. P., Humphries, M. J., Zhu, C. Demonstration of catch bonds between an integrin and its ligand. The Journal of cell biology. 185, 1275-1284 (2009).
- Yago, T., et al. Catch bonds govern adhesion through L-selectin at threshold shear. The Journal of cell biology. 166, 913-923 (2004).
- Yago, T., et al. Platelet glycoprotein Ibalpha forms catch bonds with human WT vWF but not with type 2B von Willebrand disease vWF. The Journal of clinical investigation. 118, 3195-3207 (2008).
- Chesla, S. E., Selvaraj, P., Zhu, C. Measuring two-dimensional receptor-ligand binding kinetics by micropipette. Biophysical journal. 75, 1553-1572 (1998).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464, 932-936 (2010).
- Heinrich, V., Wong, W. P., Halvorsen, K., Evans, E. Imaging biomolecular interactions by fast three-dimensional tracking of laser-confined carrier particles. Langmuir : the ACS journal of surfaces and colloids. 24, 1194-1203 (2008).
- Chen, W., Evans, E. A., McEver, R. P., Zhu, C. Monitoring receptor-ligand interactions between surfaces by thermal fluctuations. Biophysical journal. 94, 694-701 (2008).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical. 68, 2580-2587 (1995).
- Evans, E., Leung, A., Heinrich, V., Zhu, C. Mechanical switching and coupling between two dissociation pathways in a P-selectin adhesion bond. Proceedings of the National Academy of Sciences of the United States of America. 101, 11281-11286 (2004).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal Flanking Region of the A1 Domain Regulates the Force-dependent Binding of von Willebrand Factor to Platelet Glycoprotein Ib. Journal of Biological Chemistry. 288, (2013).
- Chen, W., Lou, J., Zhu, C. Forcing switch from short- to intermediate- and long-lived states of the alphaA domain generates LFA-1/ICAM-1 catch bonds. The Journal of biological chemistry. 285, 35967-35978 (2010).
- Chen, W., Lou, J., Evans, E. A., Zhu, C. Observing force-regulated conformational changes and ligand dissociation from a single integrin on cells. The Journal of cell biology. 199, 497-512 (2012).
- Judokusumo, E., Tabdanov, E., Kumari, S., Dustin, M. L., Kam, L. C. Mechanosensing in T lymphocyte activation. Biophysical journal. 102, L5-L7 (2012).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences of the United States of America. 111, 2241-2246 (2014).
- Nesbitt, W. S., et al. Distinct glycoprotein Ib/V/IX and integrin alpha IIbbeta 3-dependent calcium signals cooperatively regulate platelet adhesion under flow. The Journal of biological chemistry. 277, 2965-2972 (2002).
- Mazzucato, M., Pradella, P., Cozzi, M. R., De Marco, L., Ruggeri, Z. M. Sequential cytoplasmic calcium signals in a 2-stage platelet activation process induced by the glycoprotein Ibalpha mechanoreceptor. Blood. 100, 2793-2800 (2002).
- Lefort, C. T., Ley, K. Neutrophil arrest by LFA-1 activation. Frontiers in immunology. 3, 157 (2012).
- Liu, B., Chen, W., Evavold, B. D., Zhu, C. Accumulation of dynamic catch bonds between TCR and agonist peptide-MHC triggers T cell signaling. Cell. 157, 357-368 (2014).
- Lou, J., et al. Flow-enhanced adhesion regulated by a selectin interdomain hinge. The Journal of cell biology. 174, 1107-1117 (2006).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature communications. 5, 4886 (2014).
- Chen, W., Zarnitsyna, V. I., Sarangapani, K. K., Huang, J., Zhu, C. Measuring Receptor-Ligand Binding Kinetics on Cell Surfaces: From Adhesion Frequency to Thermal Fluctuation Methods. Cellular and molecular bioengineering. 1, 276-288 (2008).
- Marshall, B. T., Sarangapani, K. K., Lou, J., McEver, R. P., Zhu, C. Force history dependence of receptor-ligand dissociation. Biophysical. 88, 1458-1466 (2005).
- Xiang, X., et al. Structural basis and kinetics of force-induced conformational changes of an alphaA domain-containing integrin. PloS one. 6, e27946 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados