È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Fluorescenza Biomembrane Forza Probe: quantificazione simultanea di Cinetica recettore-ligando e Binding-indotta intracellulare segnalazione su una singola cella
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
We describe a technique for concurrently measuring force-regulated single receptor-ligand binding kinetics and real-time imaging of calcium signaling in a single T lymphocyte.
Abstract
Recettore-ligando membrana mediano molte funzioni cellulari. Cinetica Binding e segnalazione a valle innescato da queste interazioni molecolari sono probabilmente influenzate dall'ambiente meccanico in cui legame e segnalazione avvengono. Uno studio recente ha dimostrato che la forza meccanica può regolare il riconoscimento dell'antigene da e attivazione del recettore delle cellule T (TCR). Ciò è stato reso possibile da una nuova tecnologia che abbiamo sviluppato e fluorescenza Definito sensore di forza biomembrane (fBFP), che combina spettroscopia di forza singola molecola con microscopia a fluorescenza. L'utilizzo di un globulo rosso umano ultra-morbido come il sensore di forza sensibile, una macchina fotografica ad alta velocità e in tempo reale, le tecniche di monitoraggio delle immagini, la fBFP è di ~ 1 PN (10 -12 N), ~ 3 nm e ~ 0.5 msec a forza, risoluzione spaziale e temporale. Con il fBFP, si può misurare con precisione singoli cinetica di legame recettore-ligando ai sensi del regolamento vigente e allo stesso tempo l'immagine vincolante-attivato cal intracellularecico segnalazione su una singola cellula dal vivo. Questa nuova tecnologia può essere utilizzata per studiare altri recettore-ligando di membrana e la segnalazione in altre cellule sotto regolazione meccanica.
Introduzione
Cellula-cellula e cellula-matrice extracellulare (ECM) adesione è mediata dal legame tra i recettori della superficie cellulare, proteine ECM, e / o lipidi 1. Binding consente cellule formano strutture funzionali 1, così come riconoscono, comunicare e reagiscono all'ambiente 1-3. A differenza di proteine solubili (ad esempio, citochine e fattori di crescita) che si legano da un tridimensionale (3D) fase fluida sui recettori di superficie cellulare, i recettori di adesione cellulare formano legami con i propri ligandi attraverso una stretta spazi di giunzione per colmare due superfici contrapposte che limitano molecolare diffusione in un dimensionale interfaccia due (2D) 4-7. In contrasto con la cinetica 3D che sono comunemente misurata mediante analisi di legame tradizionali (ad esempio, di risonanza plasmonica di superficie o SPR), cinetica 2D devono essere quantificati con le tecniche specializzate, come microscopia a forza atomica (AFM) 8-10, flusso camera di 11,12, micropipetta 13,14, otticapinzette 15 e vigore biomembrane sonda (BFP) 16-21.
Più che semplicemente fornendo linkage fisico per coesione cellulare, molecole di adesione sono un componente importante della macchina di segnalazione per la cella di comunicare con l'ambiente circostante. C'è stato un crescente interesse per comprendere come l'impegno ligando di molecole di adesione avvia segnale intracellulare e come il segnale iniziale viene trasdotto all'interno della cellula. Intuitivamente, proprietà del recettore-ligando può influire i segnali che induce. Tuttavia, è difficile sezionare rapporti meccanicistici tra l'interazione extracellulare ed eventi di segnalazione intracellulare utilizzando insieme tradizionale di saggi biochimici causa delle loro molte limitazioni, ad esempio, una risoluzione temporale povero e la totale assenza di risoluzione spaziale. Metodi che permettono sia biofisica (2D cinetica legame recettore-ligando) e biochimiche osservazioni (segnalazione) su Live esistentele cellule sono substrati di rigidità sintonizzabile 22, elastomeri array pilastro 23 e dispositivi camera di flusso / microfluidica incorporato con capacità di fluorescenza 24-26. Tuttavia, letture di segnalamento e il legame al recettore-ligando devono essere ottenuti separatamente (più spesso con metodi diversi), rendendo difficile sezionare relazioni temporali e spaziali delle caratteristiche dei titoli con eventi di segnalazione.
Convenzionale BFP è una spettroscopia di forza ultrasensibile con alta risoluzione spazio-temporale 17. Esso utilizza una cella flessibile rossi del sangue (RBC) come un sensore di forza, che consente la misurazione della cinetica 2D singola molecola, le proprietà meccaniche e cambiamenti conformazionali 14,16,19-21,27-29. Un BFP a base di immagini a fluorescenza (fBFP) correla la cinetica di legame recettore-ligando con la segnalazione cellulare vincolante-attivato a scala singola molecola. Con questa impostazione, in attività di segnalazione delle cellule in situ nel contesto meccanicamente superficialistimolazione cal è stata osservata in cellule T 27. Il fBFP è versatile e può essere utilizzato per studi di adesione e segnalazione cellulare mediata da altre molecole di altre celle.
Protocollo
Questo protocollo segue le linee guida ed è stato approvato dal comitato etico della ricerca umana della Georgia Institute of Technology.
Isolamento 1. globuli rossi umani, biotinilazione e Osmolarità regolazione
Nota: I passi 1.1 deve essere effettuata da un esperto medico professionale come infermiera, con un Institutional Review Board ha approvato il protocollo.
- Ottenere 8-10 microlitri (una goccia) di sangue dal dito puntura e aggiungere 1 ml di tampone carbonato / bicarbonato (Tabella 1 e 2). Vortice delicatamente o pipetta la miscela e centrifugare per 1 min a 900 x g. Gettare il surnatante e lavare una volta di più.
- In un piccolo becher pesare 3,5-4 mg di biotina-PEG3500-NHS linker (Tabella 1). Sciogliere nel tampone carbonato / bicarbonato per rendere la concentrazione finale 3 mg / ml.
- Miscelare 171 ml di tampone carbonato / bicarbonato, 10 ml di RBC confezione e 1049 ml diBiotin-PEG3500-NHS soluzione linker ed incubare a temperatura ambiente per 30 min. Lavare la RBC una volta con tampone carbonato / bicarbonato e quindi due volte con tampone di N2-5% (Tabella 1 e 2).
- Nel frattempo, posizionare la bottiglia linker con tappo svitato in un essiccatore a vuoto di vetro riempita con essiccanti nella parte inferiore e del vuoto per 5 minuti, e riempire il essiccatore con argon. Serrare il tappo e prendere la bottiglia. Sigillare la bottiglia con una pellicola di plastica di paraffina (Tabella 1), metterlo in un contenitore riempito di diseccanti sul fondo e conservare in -20 ° C.
Nota: I passi che implicano l'uso di biotina-PEG3500-NHS linker, tra 1,2-1,4 (tranne per l'incubazione e lavare in 1.3), devono essere eseguite più velocemente possibile. - Diluire nistatina in tampone N2-5% per fare una concentrazione finale di 40 mg / ml. Mescolare 5 ml di biotinilato RBC con 71,4 ml di nistatina (Tabella 1) soluzione e incubare per 1 ora a 0 &# 176; C. Lavare due volte con N2-5% tampone e negozio con N2-5% tampone + 0,5% di BSA (Tabella 1) in frigorifero (4 ° C).
2. perle di vetro silanizzazione
- Pulizia di Bead Surface
- Pesare 50 mg di perline di vetro in polvere e li ri-sospendere in 500 ml di acqua deionizzata.
- Mescolare 0,5 ml di 30% H 2 O 2 (Tabella 1) con 9,5 ml di acqua deionizzata in un bicchiere da 50 ml, quindi aggiungere 2 ml di concentrato NH 4 OH (Tabella 1) e portare la soluzione ad una caldaia su una piastra calda .
- Aggiungere le perle di vetro per la soluzione bollente e continuare a bollire per altri 5 minuti. Agitare delicatamente la soluzione ogni min.
- Dopo la bollitura, trasferire ~ 5 ml di questa sospensione di sferette caldo in un tubo da 15 ml di micro-centrifuga e riempire con acqua deionizzata RT. Centrifugare a 3.500 g per 5 minuti, rimuovere ed eliminare il surnatante.
- Trasferire altri 5 ml di sospensione di sferette caldo e aggiungere ai talloni lavati, Rabboccare con più acqua DI, mescolare bene, e centrifugare di nuovo. Ripetere questa procedura fino a quando viene utilizzato circa 50 ml di acqua deionizzata, che sarà un totale di 4 o 5 volte di lavaggio.
- Trasferire la sospensione di sferette in una fiala da 1 ml. Ripetere il lavaggio delle perline con metanolo (Tabella 1) mediante centrifugazione a 17.000 xg per 5 minuti per 3 volte, e, infine, risospendere le sfere in 1 ml di metanolo 100%.
- Bead superficie Thiolation
- Per un tubo da centrifuga da 50 ml aggiungere 45,6 ml di metanolo, 0,4 ml di acido acetico (Tabella 1), 1,85 ml di acqua deionizzata, 1,15 ml di 3-Mercaptopropyltrimethoxysilane (MPTMS) (Tabella 1) e 1 ml di perline sospensione preparata in 2.1, poi incubare a temperatura ambiente per 3 ore.
- Dopo la reazione, rimuovere tutti i reagenti lavando una volta con metanolo fresco, e risospendere le sfere in 500 ml di metanolo. Divida anche la sospensione delle perle di vetro concentrata in una serie di 20 flaconi di vetro asciutti e puliti contappi a vite. Si evapora il metanolo utilizzando un getto di argon secco e ruotare lentamente i flaconi in modo da rendere un sottile strato di perline secche sui lati di ciascun flacone.
- Posizionare le fiale di perle in un forno di essiccazione preriscaldato a 120 ° C per 5 minuti e poi estrarre rapidamente e posizionare il tappo (s) liberamente su. Mettere le fiale in un essiccatore a vuoto di vetro riempito di essiccanti nella parte inferiore e aspirare essiccatore con una pompa a vuoto fino a raffreddamento.
- Eliminare l'essiccatore aspirato con argon asciutto per portare essiccatore sino alla normale pressione atmosferica. Togliere il coperchio essiccatore e rapidamente riavvitare il tappo (s) sul flacone. Sigillare le fiale con pellicola paraffina plastica e conservarli a temperatura ambiente in una scatola asciutta conservazione al buio.
- Dopo l'uso immediato, prendere una fiala di perline secco e lavare una volta con tampone fosfato (tabelle 1 e 2), risospendere in 50 ml di tampone fosfato e conservare a 4 ° C. Questa preparazione tallone concentrato verrà indicatocome "perle MPTMS" nelle seguenti fasi.
Nota: Con una corretta conservazione, perline MPTMS potrebbero restare in funzione per un massimo di tre mesi.
3. Bead Funzionalizzazione
- Covalentemente Rivestimento proteine su Beads
- Prendere un certo volume (ad esempio, 2,5 ml) della proteina magazzino e miscelare con un volume uguale di tampone carbonato / bicarbonato di rendere Soluzione 1.
Nota: Il volume dipende dalla concentrazione magazzino e la densità sito finale desiderato della proteina sulla superficie perline. - In un piccolo becher pesare 2-3 mg di MAL-PEG3500-NHS linker (Tabella 1) e sciogliere con tampone carbonato / bicarbonato di raggiungere una concentrazione finale di 0,231 mg / ml.
- Mescolare Soluzione 1 con un volume uguale di soluzione linker preparata in 3.1.2. Incubare la miscela a temperatura ambiente per 30 min a rendere Soluzione 2.
- Nel frattempo, posizionare la bottiglia linker con tappo svitato in un vuoto vetro essiccatore filled con essiccanti nella parte inferiore e del vuoto per 5 minuti, e riempire il essiccatore con argon. Serrare il tappo e prendere la bottiglia. Sigillare la bottiglia con una pellicola di plastica di paraffina, metterlo in un contenitore riempito di diseccanti sul fondo e conservare in -20 ° C.
Nota: I passi che implicano l'uso di MAL-PEG3500-NHS linker, tra 3.1.2-3.1.4 (tranne per l'incubazione in 3.1.3), occorrerà effettuare più velocemente possibile. - Mescolare 5 ml di MPTMS perline con soluzione 2 e aggiungere tampone fosfato (Tabella 1) per rendere un volume finale di 250 microlitri.
- Incubare le perline notte a RT, lavare 3 volte con tampone fosfato, e risospendere in 100 ml di tampone fosfato e conservare a 4 ° C.
- Prendere un certo volume (ad esempio, 2,5 ml) della proteina magazzino e miscelare con un volume uguale di tampone carbonato / bicarbonato di rendere Soluzione 1.
- Preparazione proteine Beads / Streptavidina (SA) rivestiti
- Seguire il protocollo 3.1.1-3.1.4.
- Mescolare 5 ml di perline MPTMS con Soluzione 2 e 5 ml di 4 mg / ml Streptavidina-Maleimide (SA-MAL) (Tabella 1Soluzione) e quindi aggiungere tampone fosfato per fare un volume finale di 250 microlitri.
- Incubare le perline notte a RT, lavare 3 volte con tampone fosfato, e infine risospendere in 100 ml di tampone fosfato e conservare a 4 ° C.
- Rivestimento Streptavidin su Branelli di Vetro
- Mescolare 5 ml di MPTMS perle con 5 ml di 4 mg / ml soluzione SA e aggiungere 140 ml di tampone fosfato.
- Incubare le perline notte a RT, lavare 3 volte con tampone fosfato, e risospendere in 50 ml di tampone fosfato e conservare a 4 ° C.
- Rivestimento Beads SA rivestite con una proteina Biotinylated
- Mescolare 5 ml di perline SA rivestite con la proteina (volume a seconda della densità di rivestimento desiderato) e aggiungere tampone fosfato per rendere il volume finale sia 100 microlitri.
- Incubare la miscela per una notte a 4 ° C o per 3 ore a temperatura ambiente, lavate 3 volte con tampone fosfato e risospendere in 50 microlitri phosphate buffer e conservare a 4 ° C.
4. cellulare Preparazione
Nota: Per purificare le cellule, seguire protocolli di purificazione cella di riferimento corrispondenti al tipo di cellule in uso, per esempio cellule T 27 o certe linee cellulari 21,29.
- Per gli esperimenti fBFP, una volta che la sospensione cellulare viene preparato, aggiungere Fura2-AM (Tabella 1) disciolto in DMSO nella sospensione cellulare per raggiungere una concentrazione finale di 2 mM, incubare per 30 minuti a temperatura ambiente e poi lavare una volta. Conservare la sospensione cellulare fluorescente caricata nel buio fino al momento dell'uso.
5. Preparazione per Micropipette e una Camera delle cellule
- Preparazione Micropipette
- Tubi Tagliare lungo capillari di vetro (Tabella 1) con un tagliatore di vetro in brevi pezzi di circa 3 pollici di lunghezza. Montare un pezzo sul estrattore micropipetta (Tabella 1), fare clic su "Pull", maton in modo che la parte centrale del capillare viene riscaldato dalla macchina e il capillare sarà tirato sulle due estremità per fare due capillari con punte acuminate (micropipette prime).
Nota: Seguendo le linee guida del prodotto, la morfologia desiderata della pipetta prima ha 6-8 mm cono e 0.1-0.5 micron punta. - Montare una pipetta crudo sul supporto pipetta della fucina micropipetta (Tabella 1). Calore per fondere la sfera di vetro sulla fucina. Inserire la punta della pipetta prima all'interno della sfera di vetro. Raffreddare la sfera di vetro e tirare la pipetta prima di rompere dall'esterno e lasciare la punta all'interno della sfera. Ripetere questa procedura fino a ottenere l'orifizio punta desiderata.
Nota: Esempi di un diametro interno di punta micropipetta: 2.0-2.4 micron di RBC, ~ 1.5 micron per un cordone, ~ 2-4 micron di cellule T e -7 micron per una cella ibridoma.
- Tubi Tagliare lungo capillari di vetro (Tabella 1) con un tagliatore di vetro in brevi pezzi di circa 3 pollici di lunghezza. Montare un pezzo sul estrattore micropipetta (Tabella 1), fare clic su "Pull", maton in modo che la parte centrale del capillare viene riscaldato dalla macchina e il capillare sarà tirato sulle due estremità per fare due capillari con punte acuminate (micropipette prime).
- Costruire una Camera cellulare
Nota: la camera di cella è costruito sulla base di una casa-pazzae portacamera, che consiste di due pezzi di quadrati metalli (rame / alluminio) e ha una maniglia che li unisce (Figura 1D). - Tagliare a 40 mm x 22 mm x 0,2 millimetri coprioggetto con un taglierino vetro in due pezzi 40 millimetri x 11 mm x 0,2 mm (coprivetrino 1 e 2). Colla coprioggetto 1 con grasso al lato superiore del supporto della camera in modo che collega i due quadrati di metallo, e similmente coprioggetto colla 2 alla parte inferiore, che formano una camera cellule parallelo coprioggetto (Figura 1D).
- Utilizzare una pipetta per iniettare 200 ml di tampone sperimentale tra le due lamelle. Assicurarsi che il buffer attribuisce ad entrambi i vetrini. Ruotare delicatamente e scuotere la camera di lasciare che il buffer di toccare entrambi i lati della camera.
- Iniettare cautela olio minerale in entrambi i lati della camera fiancheggiano la zona cuscinetto sperimentale sigillando il buffer dall'aria aperta. Iniettare sospensioni di perline sonda (per esempio, perle pMHC Perle), globuli rossi e gli obiettivi(per esempio, cellule T) nelle regioni superiori, medie e inferiori della zona cuscinetto rispettivamente.
6. esperimento BFP
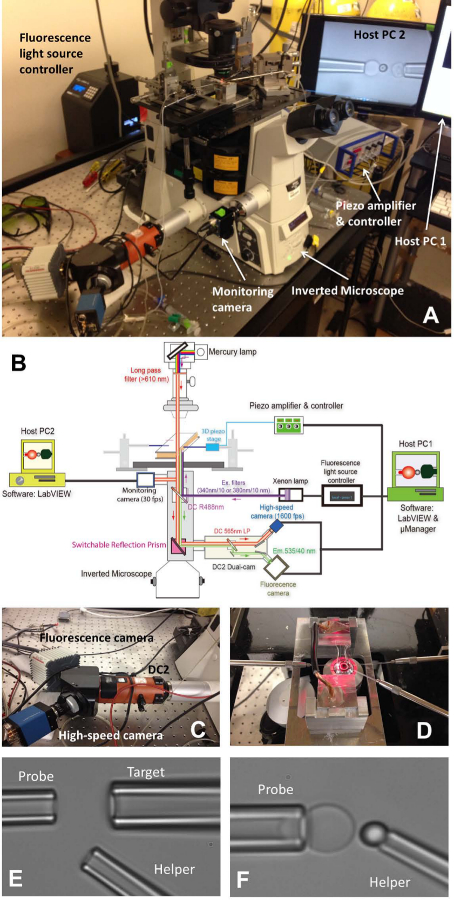
Figura 1: montaggio fBFP (A) Un quadro panoramica del sistema hardware fBFP.. (B) Un disegno schematico del sistema hardware fBFP. (C) Il sistema dual-cam "DC2" (arancione) su cui la macchina fotografica ad alta velocità (blu) e una telecamera di fluorescenza (bianco) sono stati montati. (D) La fase di microscopio che si adatta una camera esperimento e tre sistemi di manipolazione micropipetta. Micrografie impostazione BFP in una camera sperimentale (E ed F). (E) Micropipette che mostra il montaggio la pipetta della sonda (a sinistra), l'obiettivo pipetta (in alto a destra) e il suo assistente pipetta (r bassaestra). Posizionamento tallone (F) Sonda. Una goccia sonda è stata manipolata da una pipetta aiuto e attaccato ad un apice RBC per formare un sensore di forza. Clicca qui per vedere una versione più grande di questa figura.
- Accendere il microscopio (Tabella 1) e la sorgente luminosa. Posizionare la camera sul palco principale del microscopio (Figura 1A, D).
- Installare tutte le tre micropipette di BFP (Figura 1D sinistra:. Sonda, per afferrare un RBC, a destra: bersaglio, per afferrare una cella o tallone, in basso a destra: aiutante, per afferrare un tallone).
- Utilizzare un micro-iniettore (Tabella 1) per riempire una micropipetta con tampone sperimentale. Togliere il supporto pipetta (Tabella 1) e tenerlo in un luogo più basso per permettere acqua gocciola dalla punta. Inserire rapidamente il micropipetta nella punta supporto e assicurarsi che nessuna bolla d'aria entra nel micropipettadurante l'inserimento. Stringere la vite del supporto.
- Montare ciascun titolare pipetta sul suo corrispondente micro-manipolatore. Spingere la micropipetta verso la camera in modo che le loro punte entrano nell'area di buffer camera. Regolare la posizione della micropipetta e li trova sotto il campo ottico di vista.
- Spostarsi all'interno dello stadio portacamera di trovare le colonie di tre elementi (globuli rossi, gli obiettivi e le perline sonda) uno per uno. Regolare la posizione del corrispondente micropipetta ruotando le manopole dei manipolatori per far la punta della micropipetta approccio una cella / tallone. Aspirare cella / tallone regolando la pressione all'interno della corrispondente micropipetta. Tutte e tre le micropipette cattureranno i loro elementi corrispondenti.
- Spostarsi all'interno dello stadio portacamera di trovare uno spazio aperto, lontano da colonie degli elementi iniettate in cui verrà effettuata l'esperimento. Commutare la modalità visiva microscopio per visualizzare l'immagine sul compprogramma uter sullo schermo del computer. Spostare tutti i tre elementi su punte per pipette nel campo visivo del programma.
- Allineate il tallone sonda e RBC e manovrare con attenzione il tallone sonda al vertice del RBC, incidere brevemente il tallone sul RBC e delicatamente ritrattare. Regolare la pressione della micropipetta aiutante a soffiare delicatamente il tallone via, in modo che possa essere lasciato incollato sul RBC apice (Figura 1F). Allontanare il micropipetta aiutante e allineare il bersaglio e tallone sonda (Figura 2A).
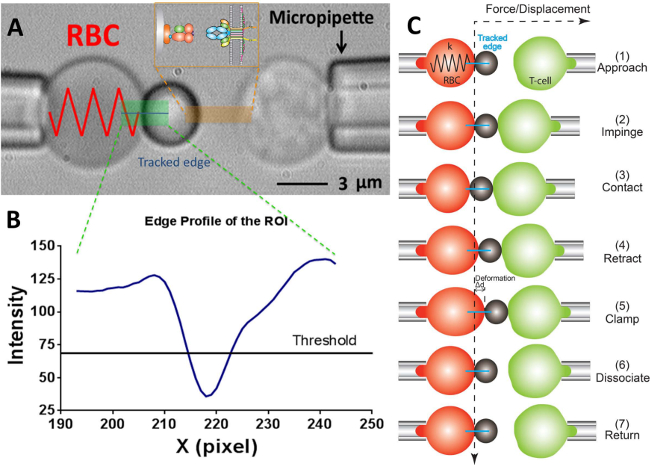
Figura 2:. Schema BFP e il suo ciclo di prova (A) Video-micrografia raffigurante un sensore di forza (a sinistra) e una T-cellula bersaglio (destra) aspirato dal rispettivo sensore di forza stazionaria pipettes.The costituito da un RBC gonfio e una allegata ligando-cuscinetto tallone. Il recettore-cuscinettoT-cell (target) è montato un piezotranslator allineato opposta alla sonda. Il ROI è indicato in verde. L'inseguitore bordo è indicato in una linea blu. L'inserto rappresenta il ligando (pMHC, lato tallone) e del recettore (TCR, lato T-cell) coppia su due superfici opposte nella zona segnata in arancione. (B) Il profilo di intensità del bordo tallone in (A). La regione ROI in x-direzione è tracciata come asse x (in numero di pixel) e l'intensità della luce (in valore scala di grigi) in media da 30 pixel binning lungo la y-direzione. (C) La deformazione del RBC e la posizione del tallone e il bersaglio (cellule T) in un ciclo di prova di dosaggio morsetto forza. Le linee tratteggiate verticali e orizzontali indicano la posizione di zero forza dell'apice RBC e l'andamento nel tempo, rispettivamente. L'inseguitore bordo linea della deformazione RBC è mostrato in blu di ciascun pannello. Le fasi stesse ancora meno sono adottati frequenza adesionetest (che manca i passi di "pinza" e "dissociarsi") e test fluttuazione termica (che manca la fase di "dissociarsi").
- In programma, nella finestra campo visivo utilizzare gli strumenti nel programma per misurare la rispettivi raggi della micropipetta sonda (R p), la RBC (R 0), l'area di contatto circolare tra RBC e sonda tallone (R c) , che consente la stima della costante elastica della RBC (k) mediante la seguente equazione 17,30,
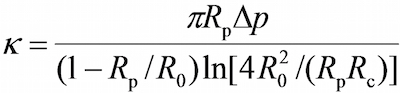
dove Δ p è la pressione di aspirazione a puntale della pipetta.
Nota: Dalle legge di Hooke che la forza vincolante, F, può essere quantificato il prodotto di costante elastica e spostamento del tallone sonda (d), cioè, F = d (Figura 2C). - Inserire la costante della molla RBC desiderato nel programma (Si prega di fare riferimento alla sezione relativa al protocollo 6.6. La costante della molla è in genere fissato a 0,25 o 0,3 PN / nm per il morsetto forza dosaggio e l'adesione saggio di frequenza e 0,1 PN / nm per il dosaggio fluttuazione termica) , che restituirà una pressione di aspirazione necessaria nell'unità di centimetro di acqua. Regolare l'altezza del serbatoio dell'acqua che si collega a pipetta della sonda fino a raggiungere la pressione di aspirazione desiderata.
- Tracciare una linea orizzontale attraverso l'apice RBC, che produrrà una curva nella finestra adiacente indica la luminosità (valore di scala di grigi) di ciascun pixel lungo questa linea. Trascinare la linea di soglia per essere a circa metà della profondità della curva (Figura 2A, B).
Nota: Il punto di minimo della curva di luminosità al di sotto della linea di soglia indica la posizione del confine tallone, quindi solo un minimo locale è permessa. Se due o più minimi locali sono allo stato attuale,indica che l'immagine non è ottimale (probabilmente dovuto all'immagine essendo fuori fuoco, o un cattivo allineamento tra tallone sonda e la RBC). - Selezionare la modalità desiderata esperimento: saggio fluttuazione termica, frequenza adesione test o test morsetto forza. Impostare i parametri come desiderato (ad esempio, la forza d'urto = 15 pN, caricando tasso = 1.000 PN / s, tempo di contatto = 1 sec, forza di serraggio = 20 PN (per forza di serraggio test), ecc).
- Istruzioni "Start", che consente al programma di spostare la pipetta bersaglio e guidare il bersaglio in e fuori dal contatto con la sonda (vedere la sezione Risultati rappresentativi per i dettagli). La raccolta dei dati sarà eseguita in parallelo, che registra la posizione del tallone della sonda in tempo reale. Arrestare il programma cliccando sul pulsante "esperimento Stop", momento in cui una finestra pop-out per consentire il salvataggio dei dati acquisiti.
7. fluorescenza BFP (fBFP) esperimento
- Per utilizzare il fluorescence funzione del sistema BFP, accendere la sorgente di luce di eccitazione (Tabella 1) e la telecamera di fluorescenza (Tabella 1), che sono controllate da un programma separato (Tabella 1). In programma, selezionare i parametri per l'imaging di fluorescenza, compreso il guadagno, l'esposizione, i canali di eccitazione (in questo caso, 340 nm e 380 nm di luce), ecc. Seguite tutti i preparativi nel protocollo dell'esperimento BFP, tra cui l'allineamento della sonda e il bersaglio, che consentirà la visualizzazione delle immagini in diretta a fluorescenza delle cellule bersaglio eccitato da 340 nm o 380 nm luce di eccitazione.
- Utilizzare lo strumento sezionamento a circa sezione l'area entro cui la cella rimarrà per tutto il periodo di registrazione.
Nota: Grazie all'uso del ciclo di accostamento contatto-retrazione, la cella sarà muovendo avanti e indietro ripetutamente, così l'area in sezione è molto più grande della cella stessa. - Clicca su "Record" per consentire l'una 340 nmd 380 nm luce per eccitare alternativamente la fluorescenza colorante intracellulare (Fura2), ed una coppia di corrispondenti immagini di fluorescenza saranno registrate alternativamente circa una volta ogni secondo. Contemporaneamente cliccare su "Start" in programma per iniziare l'esperimento BFP per l'interazione molecolare analisi e l'esperimento di imaging di fluorescenza per monitorare segnale calcio intracellulare. Il sistema produrrà un file di dati grezzi per il legame recettore-ligando (vedere Figura 6A sotto) e una serie di immagini fluorescenti in formato .tiff per i segnali di calcio.
Analisi 8. Dati
- Analisi BFP dati
- L'analisi dei dati per l'analisi di frequenza adesione
- Sequenziale ispezionare il segnale di ogni ciclo di "forza in funzione del tempo" e registrano semplicemente che i cicli contengono un evento di adesione e quali no, e riassumono per produrre una frequenza media di adesione.
- Raccogliere la forza di rottura di ogni evento di adesione, cheè il valore di picco della forza lineare rampa prima rottura legame. Dopo aver raccolto una quantità sufficiente di forze di rottura in una gamma di velocità di rampa, derivare la distribuzione della forza di rottura ad ogni velocità di rampa da cui è derivata la forza-dipendente off-rate di dissociazione recettore-ligando con forza dinamica spettroscopia analisi 18,31.
- L'analisi dei dati per l'analisi termica fluttuazione
- Sequenziale ispezionare il segnale di fase di chiusura di ciascun ciclo, che probabilmente contiene più eventi di associazione legame e di dissociazione. Utilizzare il livello fluttuazione termica fase di serraggio (la deviazione standard media di un intervallo di scorrimento di 70 punti di tempo sequenziali della posizione tallone) nella "forza vs. tempo" segnale come guida per distinguere gli eventi di associazione e di dissociazione legame, poiché un legame formazione corrisponde ad una diminuzione della fluttuazione termica.
- Designare l'intervallo dal momento di dissociazione del legame (quando il thermal fluttuazione riprende al livello normale) all'istante della successiva formazione del legame come tempo di attesa, e designare la durata del legame dalla sua associazione alla dissociazione come vita legame, che sono entrambi raccolte durante l'ispezione dei dati. Calcolare il tempo medio di attesa e la durata media legame, che riflettono, rispettivamente, il reciproco di on-rate e quello di off-rate sotto zero forza 16,30.
- L'analisi dei dati per l'analisi morsetto forza
- Parametri record di tutti gli eventi di durata compresa la forza media e la durata legame con il numero di sequenza, così come l'ora di inizio e l'ora di fine, che permetteranno di trarre una curva vita cumulativa (per esempio, la Figura 6C, curva gialla).
- Raccogliere una quantità sufficiente di vita gli eventi sotto una gamma di forze. Raggrupparli in contenitori diversi di forza, che produrrà una vita media in ogni bin vigore, e complessivamente produrre una "vita media vs. Curva forza "(Figura 4).
- L'analisi dei dati per l'analisi di frequenza adesione
- Calcio fluorescenza Analisi Imaging dati
- Regolare la soglia di intensità fino a che le immagini di fluorescenza mostrano un chiaro contorno della cella in entrambi i canali 340 nm e 380 nm senza rumore di fondo (Figura 5A, B). Poi rivedere il Ca2 + intracellulare trama di segnale per fotogramma con uno pseudo-colore che indica il livello di intensità (Figura 6B), che è ricavato in base al rapporto di intensità di 340 nm / 380 nm, per generare il "Ca 2+ intensità normalizzata vs . Curva tempo "(Figura 6C). Utilizzare le immagini pseudo-colore fluorescenza per produrre un film che mostra il secondo livello di fluorescenza per secondo.
Risultati
La tecnica BFP è stato lanciato da laboratorio Evans nel 1995 17. Questo strumento picoforce è stato ampiamente utilizzato per misurare le interazioni di proteine immobilizzate su superfici, in modo da analizzare la cinetica bidimensionali di molecole di adesione interagire con i loro ligandi 16,19,20, 30, per misurare l'elasticità molecolare 21,29, e per determinare proteine conformazionale cambia 21. Per un fBFP, un ulteriore set di dispositivi epi-fluores...
Discussione
Un esperimento di successo fBFP comporta alcune considerazioni critiche. In primo luogo, per il calcolo forza di essere affidabile, la micropipetta, l'RBC, ed il tallone della sonda devono essere allineati come vicino coassiale possibile. La proiezione del RBC all'interno della pipetta dovrebbe essere circa un diametro della sonda pipetta in modo che l'attrito tra RBC e la pipetta è trascurabile. Per un tipico RBC umana, il diametro ottimale pipetta è 2.0-2.4 micron, che produce un migliore adattamento di ...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Research related to this paper and the development of the fBFP technology in the Zhu lab were supported by NIH grants AI044902, AI077343, AI038282, HL093723, HL091020, GM096187, and TW008753. We thank Evan Evans for inventing this empowering experimental tool, and members of the Evans lab, Andrew Leung, Koji Kinoshita, Wesley Wong, and Ken Halvorsen, for helping us to build the BFP. We also thank other Zhu lab members, Fang Kong, Chenghao Ge and Kaitao Li, for their helps in the instrumentation development.
Materiali
| Name | Company | Catalog Number | Comments |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4 • H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | N2-5% buffer preparation |
| Potassium chloride (KCl) | Sigma-Aldrich | P9541 | N2-5% buffer preparation |
| Potassium phosphate monobasic (KH2PO4) | Sigma-Aldrich | P5655 | N2-5% buffer preparation |
| Sucrose | Sigma-Aldrich | S0389 | N2-5% buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Bead functionalization |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Nystatin | Sigma-Aldrich | N6261 | RBC osmolarity adjustment |
| Ammonium Hydroxide (NH4OH) | Sigma-Aldrich | A-6899 | Glass bead silanization |
| Methanol | BDH | 67-56-1 | Glass bead silanization |
| 30% Hydrogen Peroxide (H2O2) | J. T. Barker | Jan-86 | Glass bead silanization |
| Acetic Acid (Glacial) | Sigma-Aldrich | ARK2183 | Glass bead silanization |
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Borosilicate Glass beads | Distrilab Particle Technology | 9002 | Glass bead functionalization |
| Streptavidin−Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
| BSA | Sigma-Aldrich | A0336 | Ligand functionalizing |
| Fura2-AM | Life Technologies | F-1201 | Intracellular calcium fluorescence dye loading |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650 | Intracellular calcium fluorescence dye loading |
| Quantibrite PE Beads | BD Biosciences | 340495 | Density quantification |
| Flow Cytometer | BD Biosciences | BD LSR II | Density quantification |
| Capillary Tube 0.7-1.0 mm x 30 inches | Kimble Chase | 46485-1 | Micropipette making |
| Flaming/Brown Micropipette Puller | sutter instrument | P-97 | Micropipette making |
| Pipette microforce | Narishige | MF-900 | Micropipette making |
| Mineral Oil | Fisher Scientific | BP2629-1 | Chamber assembly |
| Microscope Cover Glass | Fisher Scientific | 12-544-G | Chamber assembly |
| Micro-injector | World Precision Instruments | MF34G-5 | Chamber assembly |
| 1 ml syringe | BD | 309602 | chamber assembly |
| Micropipette holder | Narishige | HI-7 | Chamber assembly |
| Home-designed mechanical parts and adaptors fabrications using CNC machining. | Biophysics Instrument | All parts are customized according to the CAD designs. | BFP system |
| Microscope (TiE inverted) | Nikon | MEA53100 | BFP system |
| Objective CFI Plan Fluor 40x (NA 0.75, WD 0.72 mm, Spg) | Nikon | MRH00401 | BFP system |
| Camera, GE680, 640 x 480, GigE, 1/3" CCD, mono | Graftek Imaging | 02-2020C | BFP system |
| Prosilica GC1290 - ICX445, 1/3", C-Mount, 1280 x 960, Mono., CCD, 12 Bit ADC | Graftek Imaging | 02-2185A | BFP system |
| Manual submicron probehead with high resolution remote control | Karl Suss | PH400 | BFP system |
| Anti-vibration table (5’ x 3’) | TMC | 77049089 | BFP system |
| 3D manual translational stage | Newport | 462-XYZ-M | |
| SolidWorks 3D CAD software | SOLIDWORKS Corp. | Version 2012 SP5 | BFP system |
| LabVIEW software | National Instruments | Version 2009 | BFP system, BFP program |
| 3D piezo translational stage | Physik Instrumente | M-105.3P | BFP system |
| Linear piezo accuator | Physik Instrumente | P-753.1CD | BFP system |
| Micromanager software | Version 1.4 | fBFP system, fluorescence imaging program | |
| Dual Cam (DC-2) | Photometrics | 77054724 | fBFP system |
| Dual Cam emission filter (T565LPXR) | Photometrics | 77054725 | fBFP system |
| Fluorescence Camera | Hamamatsu | ORCA-R2 C10600-10B | fBFP system |
| Plastic paraffin film (Parafilm) | Bemis Company, Inc | PM996 | bottle sealing |
| Carbonate/bicarbonate buffer (pH 8.5) | 8.4 g/L sodium carbonate (Na2CO3), 10.6 g/L sodium bicarbonate (NaHCO3) | ||
| Phosphate buffer (pH 6.5-6.8) | 27.6 g/L NaPhosphate monobasic (NaH2PO4 • H2O), 28.4 g/L Anhy. NaPhosphate dibasic (Na2HPO4) | ||
| N2-5% buffer (pH 7.2) | 20.77 g/L potassium chloride (KCl), 2.38 g/L sodium chloride (NaCl), 0.13 g/L potassium phosphate monobasic (KH2PO4), 0.71 g/L anhy. sodium phosphate dibasic (Na2HPO4), 9.70 g/L sucrose |
Riferimenti
- Aplin, A. E., Howe, A., Alahari, S. K., Juliano, R. L. Signal transduction and signal modulation by cell adhesion receptors: the role of integrins, cadherins, immunoglobulin-cell adhesion molecules, and selectins. Pharmacological reviews. 50, 197-263 (1998).
- Davis, M. M., Bjorkman, P. J. T-cell antigen receptor genes and T-cell recognition. Nature. 334, 395-402 (1988).
- Dado, D., Sagi, M., Levenberg, S., Zemel, A. Mechanical control of stem cell differentiation. Regenerative medicine. 7, 101-116 (2012).
- Edwards, L. J., Zarnitsyna, V. I., Hood, J. D., Evavold, B. D., Zhu, C. Insights into T cell recognition of antigen: significance of two-dimensional kinetic parameters. Frontiers in immunology. 3, 86 (2012).
- Zhu, C., Jiang, N., Huang, J., Zarnitsyna, V. I., Evavold, B. D. Insights from in situ analysis of TCR-pMHC recognition: response of an interaction network. Immunological reviews. 251, 49-64 (2013).
- Huang, J., Meyer, C., Zhu, C. T. T cell antigen recognition at the cell membrane. Molecular immunology. 52, 155-164 (2012).
- Zarnitsyna, V., Zhu, C. T. T cell triggering: insights from 2D kinetics analysis of molecular interactions. Physical biology. 9, 045005 (2012).
- Binnig, G., Quate, C. F., Gerber, C. Atomic Force Microscope. Physical Review Letters. 56, 930-933 (1986).
- Marshall, B. T., et al. Direct observation of catch bonds involving cell-adhesion molecules. Nature. 423, 190-193 (2003).
- Kong, F., Garcia, A. J., Mould, A. P., Humphries, M. J., Zhu, C. Demonstration of catch bonds between an integrin and its ligand. The Journal of cell biology. 185, 1275-1284 (2009).
- Yago, T., et al. Catch bonds govern adhesion through L-selectin at threshold shear. The Journal of cell biology. 166, 913-923 (2004).
- Yago, T., et al. Platelet glycoprotein Ibalpha forms catch bonds with human WT vWF but not with type 2B von Willebrand disease vWF. The Journal of clinical investigation. 118, 3195-3207 (2008).
- Chesla, S. E., Selvaraj, P., Zhu, C. Measuring two-dimensional receptor-ligand binding kinetics by micropipette. Biophysical journal. 75, 1553-1572 (1998).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464, 932-936 (2010).
- Heinrich, V., Wong, W. P., Halvorsen, K., Evans, E. Imaging biomolecular interactions by fast three-dimensional tracking of laser-confined carrier particles. Langmuir : the ACS journal of surfaces and colloids. 24, 1194-1203 (2008).
- Chen, W., Evans, E. A., McEver, R. P., Zhu, C. Monitoring receptor-ligand interactions between surfaces by thermal fluctuations. Biophysical journal. 94, 694-701 (2008).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical. 68, 2580-2587 (1995).
- Evans, E., Leung, A., Heinrich, V., Zhu, C. Mechanical switching and coupling between two dissociation pathways in a P-selectin adhesion bond. Proceedings of the National Academy of Sciences of the United States of America. 101, 11281-11286 (2004).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal Flanking Region of the A1 Domain Regulates the Force-dependent Binding of von Willebrand Factor to Platelet Glycoprotein Ib. Journal of Biological Chemistry. 288, (2013).
- Chen, W., Lou, J., Zhu, C. Forcing switch from short- to intermediate- and long-lived states of the alphaA domain generates LFA-1/ICAM-1 catch bonds. The Journal of biological chemistry. 285, 35967-35978 (2010).
- Chen, W., Lou, J., Evans, E. A., Zhu, C. Observing force-regulated conformational changes and ligand dissociation from a single integrin on cells. The Journal of cell biology. 199, 497-512 (2012).
- Judokusumo, E., Tabdanov, E., Kumari, S., Dustin, M. L., Kam, L. C. Mechanosensing in T lymphocyte activation. Biophysical journal. 102, L5-L7 (2012).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences of the United States of America. 111, 2241-2246 (2014).
- Nesbitt, W. S., et al. Distinct glycoprotein Ib/V/IX and integrin alpha IIbbeta 3-dependent calcium signals cooperatively regulate platelet adhesion under flow. The Journal of biological chemistry. 277, 2965-2972 (2002).
- Mazzucato, M., Pradella, P., Cozzi, M. R., De Marco, L., Ruggeri, Z. M. Sequential cytoplasmic calcium signals in a 2-stage platelet activation process induced by the glycoprotein Ibalpha mechanoreceptor. Blood. 100, 2793-2800 (2002).
- Lefort, C. T., Ley, K. Neutrophil arrest by LFA-1 activation. Frontiers in immunology. 3, 157 (2012).
- Liu, B., Chen, W., Evavold, B. D., Zhu, C. Accumulation of dynamic catch bonds between TCR and agonist peptide-MHC triggers T cell signaling. Cell. 157, 357-368 (2014).
- Lou, J., et al. Flow-enhanced adhesion regulated by a selectin interdomain hinge. The Journal of cell biology. 174, 1107-1117 (2006).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature communications. 5, 4886 (2014).
- Chen, W., Zarnitsyna, V. I., Sarangapani, K. K., Huang, J., Zhu, C. Measuring Receptor-Ligand Binding Kinetics on Cell Surfaces: From Adhesion Frequency to Thermal Fluctuation Methods. Cellular and molecular bioengineering. 1, 276-288 (2008).
- Marshall, B. T., Sarangapani, K. K., Lou, J., McEver, R. P., Zhu, C. Force history dependence of receptor-ligand dissociation. Biophysical. 88, 1458-1466 (2005).
- Xiang, X., et al. Structural basis and kinetics of force-induced conformational changes of an alphaA domain-containing integrin. PloS one. 6, e27946 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon