É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fluorescência Biomembrane Força Probe: quantificação simultânea de Kinetics receptor de ligando e induzida Encadernação-Sinalização Intracelular em uma única célula
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
We describe a technique for concurrently measuring force-regulated single receptor-ligand binding kinetics and real-time imaging of calcium signaling in a single T lymphocyte.
Resumo
Interacções ligando-receptor de membrana medeiam muitas funções celulares. Encadernação cinética e sinalização a jusante desencadeada por essas interações moleculares são provavelmente afectado pelo ambiente em que a ligação mecânica e sinalização ocorrem. Um estudo recente demonstrou que a força mecânica pode regular o reconhecimento do antigénio pelo desencadeamento e do receptor de células T (TCR). Isso foi possível graças a uma nova tecnologia que nós desenvolvemos e fluorescência denominadas sonda vigor biomembrana (fBFP), que combina força espectroscopia única molécula com microscopia de fluorescência. Usando um glóbulo vermelho humano ultra-suave como o sensor de força sensível, uma câmera de alta velocidade e em tempo real técnicas de rastreamento de imagens, o fBFP é de ~ 1 pN (10 -12 N), a 3 nm e ~ 0,5 ms em vigor, a resolução espacial e temporal. Com o fBFP, pode-se medir com precisão cinética de ligação ligando-receptor individuais no âmbito da regulamentação vigente e cal intracelular simultaneamente imagem ligação desencadeadaCium sinalização sobre uma única célula viva. Esta nova tecnologia pode ser utilizada para estudar a interacção receptor-ligando outra membrana e sinalização em outras células sob regulação mecânica.
Introdução
Célula-a-célula e célula-matriz extracelular de (ECM) adesão é mediada pela ligação entre os receptores da superfície celular, proteínas de ECM, e / ou lípidos 1. A ligação permite que as células de modo a formar uma estrutura funcional, bem como reconhecer, comunicar, e reagir ao ambiente 1-3. Ao contrário de proteínas solúveis (por exemplo, citoquinas e factores de crescimento) que se ligam de uma imagem tridimensional (3D) de fase fluida para os receptores da superfície celular, os receptores de adesão de células formar ligações com os seus ligandos através de uma das junções de hiato estreita para unir duas superfícies opostas que limitam molecular difusão numa dimensional (2D) de interface dois 4-7. Em contraste com a cinética de 3D que são vulgarmente medidos por ensaios de ligação tradicionais (por exemplo, de ressonância de plasmon de superfície ou SPR), a cinética 2D têm de ser quantificado com técnicas especializadas, tais como microscopia de força atómica (AFM), 8-10, câmara de fluxo 11,12, micropipeta 13,14, ópticopinças 15 e sonda vigor biomembrana (BFP) 16-21.
Mais do que simplesmente fornecendo ligação física para a coesão celular, moléculas de adesão são um dos principais componentes da maquinaria de sinalização para a célula comunique com o espaço envolvente. Tem havido um interesse crescente na compreensão de como ligando acoplamento de moléculas de adesão inicia a sinalização intracelular e como o sinal inicial é transduzida no interior da célula. Intuitivamente, propriedades de receptor-ligando de ligação pode ter um impacto que induz os sinais. No entanto, é difícil para dissecar as relações mecanicistas entre a interação extracelular e eventos de sinalização intracelular utilizando conjunto tradicional de ensaios bioquímicos por causa de suas muitas limitações, por exemplo, uma resolução temporal pobres e completa falta de resolução espacial. Métodos que permitem tanto biofísico (cinética de ligação ao receptor do ligando 2D) e (sinalização) observações bioquímicas em directo existentecélulas incluem substratos de rigidez ajustável 22, elastômero pilar matrizes 23 e dispositivos de câmara de fluxo / microfluídicos incorporados com capacidade de fluorescência 24-26. No entanto, as leituras de sinalização e de ligação ao receptor do ligando tem que ser obtido separadamente (na maioria das vezes através de métodos diferentes), o que torna difícil para dissecar as relações temporais e espaciais das características de títulos com eventos de sinalização.
BFP convencional é uma espectroscopia de força ultra-sensível de alta resolução espaço-temporal 17. Ele usa uma célula de sangue vermelho flexível (RBC) na forma de um sensor de força, permitindo a medição da cinética de 2D única molécula, propriedades mecânicas e alterações conformacionais 14,16,19-21,27-29. A BFP baseado imagens fluorescentes (fBFP) correlaciona-se a cinética de ligação ligando-receptor com a sinalização celular desencadeada obrigatório-a escala única molécula. Com esta configuração, em atividades de sinalização celular situ no contexto de mechani superfíciecal estimulação foi observada em células T-27. O fBFP é versátil e pode ser utilizado para estudos de adesão celular e de sinalização mediadas por outras moléculas em outras células.
Protocolo
Este protocolo segue as diretrizes da e foi aprovado pelo comitê de ética de pesquisa em humanos de Georgia Institute of Technology.
1. hemácias Humano Isolamento, Biotinilação e Ajuste osmolaridade
Nota: Passo 1.1 deve ser realizada por um profissional médico, como uma enfermeira treinada, com um Conselho de Revisão Institucional protocolo aprovado.
- Obter 8-10 ul (uma gota) de sangue a partir de picada no dedo e adicionar a 1 mL do tampão de carbonato / bicarbonato (Tabela 1 e 2). Suavemente vortex ou pipetar a mistura e centrifugar durante 1 min a 900 x g. Elimine o sobrenadante e lava-se mais uma vez.
- Em uma pequena proveta de pesar 3,5-4 mg de biotina-NHS-PEG3500 ligante (Tabela 1). Dissolve-se que no tampão de carbonato / bicarbonato a fazer a concentração final de 3 mg / ml.
- Misturar 171 ul de tampão carbonato / bicarbonato, a 10 ul de pacote de RBC e 1049 ul deA biotina-NHS-PEG3500 solução ligante e incubar à TA durante 30 min. Lava-se a RBC uma vez com tampão de carbonato / bicarbonato e em seguida duas vezes com tampão% N2-5 (Tabela 1 e 2).
- Enquanto isso, colocar a garrafa com a tampa solta ligante num exsicador de vácuo de vidro cheio de dessecantes na parte inferior e de vácuo durante 5 min, e encher o exsicador com árgon. Aperte a tampa e tomar a garrafa. Fecha-se o frasco com uma película de plástico de parafina (Tabela 1), colocá-lo dentro de um recipiente preenchido com dessecantes na parte inferior e na loja de -20 ° C.
Nota: As etapas que envolvem a utilização de biotina-NHS-PEG3500 ligante, incluindo 1,2-1,4 (excepto para a incubação e lavagem em 1,3), precisam ser realizadas tão depressa quanto possível. - Diluir nistatina% em tampão de N2-5 para fazer uma concentração final de 40 ug / ml. Misturar 5 mL de RBC biotinilados com 71,4 mL de nistatina (Tabela 1) em solução e incubar durante 1 hora a 0 &# 176; C. Lavar duas vezes com tampão e armazenar N2-5% com tampão N2-5% + 0,5% de BSA (Tabela 1) no frigorífico (4 ° C).
2. Glass Bead Silanização
- Limpeza da superfície do grânulo
- Pesar 50 mg de esferas de vidro em pó e re-suspendem, em 500 mL de água DI.
- Misturar 0,5 ml de 30% de H 2 O 2 (Tabela 1) com 9,5 ml de água desionizada numa proveta de 50 ml, em seguida, adicionar 2 ml de NH4OH concentrado (Tabela 1) e trazer esta solução a uma caldeira numa placa de aquecimento .
- Adicionar as esferas de vidro para a solução em ebulição e prosseguir a ebulição durante mais 5 min. Agite suavemente a solução a cada min.
- Depois de ferver, transferir ~ 5 ml desta suspensão de esferas quente em um tubo de micro-centrífuga de 15 ml e completar com água RT DI. Centrifugar a 3.500 xg durante 5 minutos, remover e descartar o sobrenadante.
- Transferir mais 5 ml de suspensão de esferas quente e adicionar aos esferas lavadas, Encher com mais DI água, misture bem, e centrifugar novamente. Repetir este procedimento até cerca de 50 ml de água desionizada é utilizada, o que será um total de 4 a 5 vezes de lavagem.
- Transferir a suspensão do grânulo em um frasco de 1 ml. Repetir a lavagem das pérolas com metanol (Tabela 1) por centrifugação a 17.000 xg durante 5 min para 3 vezes, e finalmente re-suspender as pérolas em 1 ml de metanol a 100%.
- Bead Superfície tiola�o
- A um tubo de centrífuga de 50 ml de adicionar 45,6 mL de metanol, 0,4 ml de ácido acético (Tabela 1), 1,85 ml de água desionizada, 1,15 ml de 3-mercaptopropiltrimetoxissilano (MPTMS) (Tabela 1) e 1 ml de suspensão de grânulos preparados de 2,1, em seguida incubar à TA durante 3 h.
- Após a reacção, remover todos os reagentes por lavagem uma vez com metanol fresco, e re-suspender as pérolas em 500 ul de metanol. Uniformemente dividir esta suspensão de esferas de vidro concentrada em um conjunto de 20 frascos de vidro secas e limpas comtampas de rosca. Evapora-se o metanol por meio de um jacto de árgon seco e lentamente rodar os frascos, de modo a fazer uma fina camada de grânulos secos sobre os lados de cada recipiente.
- Colocar os tubos de grânulos numa estufa de secagem a pré-aquecido a 120 ° C durante 5 min e, em seguida, retirar rapidamente e colocar a tampa (s) em frouxamente. Colocar os tubos num exsicador de vácuo de vidro cheio de dessecantes na parte inferior e o exsicador de vácuo com uma bomba de vácuo até que se arrefeceu.
- Purga-se o exsicador aspirado com árgon seco para trazer o exsicador até à pressão atmosférica normal. Retire a tampa exsicador e rapidamente voltar a apertar a tampa (s) no frasco. Selar os frascos com filme plástico parafina e armazená-los à temperatura ambiente em uma caixa de armazenamento escuro e seco.
- Após a utilização imediata, tomar um frasco de grânulos secos e lavar uma vez com tampão fosfato (Tabelas 1 e 2), re-suspender em 50 ul de tampão de fosfato e armazenar a 4 ° C. Esta preparação talão concentrado será referidocomo "contas" MPTMS nos seguintes passos.
Nota: Com o armazenamento adequado, contas MPTMS poderia permanecer em funcionamento por até três meses.
3. Bead funcionalização
- As proteínas de revestimento de forma covalente em grânulos
- Tomar um determinado volume (por exemplo, 2,5 ul) do banco de proteína e misturar com um volume igual de tampão carbonato / bicarbonato a fazer a solução 1.
Nota: O volume depende da concentração da solução e a densidade de local final desejada da proteína na superfície do grânulos. - Em uma pequena proveta de pesar 2-3 mg de MAL-PEG3500-NHS ligante (Tabela 1) e dissolvê-lo com tampão carbonato / bicarbonato a atingir uma concentração final de 0,231 mg / ml.
- Misturar uma solução com um volume igual da solução de ligante preparada no ponto 3.1.2. Incubar a mistura à temperatura ambiente durante 30 minutos para preparar uma solução 2.
- Enquanto isso, coloque a garrafa de ligação com tampa solta no vazio vidro exsicador filled com dessecantes na parte inferior e de vácuo durante 5 min, e encher o exsicador com árgon. Aperte a tampa e tomar a garrafa. Selar a garrafa com parafina filme plástico, coloque-o em um recipiente cheio de dessecantes na parte inferior e armazenar em -20 ° C.
Nota: As etapas que envolvem o uso de MAL-PEG3500-NHS ligante, incluindo 3.1.2-3.1.4 (excepto para a incubação no ponto 3.1.3), têm de ser realizado o mais rápido possível. - Misturar 5 ul de pérolas MPTMS com Solução 2 e adicionar tampão de fosfato (Tabela 1) para fazer um volume final de 250 ul.
- Incubar as pérolas durante a noite à temperatura ambiente, lavar 3 vezes com tampão de fosfato, e re-suspensão em 100 ul de tampão de fosfato e armazenar a 4 ° C.
- Tomar um determinado volume (por exemplo, 2,5 ul) do banco de proteína e misturar com um volume igual de tampão carbonato / bicarbonato a fazer a solução 1.
- Preparar proteína / Beads Streptavidin (SA) Revestido
- Siga o protocolo 3.1.1-3.1.4.
- Misturar 5 ul de pérolas Solução MPTMS com 2 e 5 ul de 4 mg / ml de estreptavidina-maleimida (SA-MAL) (Tabela 1Solução) e, em seguida, adicionar tampão de fosfato para fazer um volume final de 250 ul.
- Incubar as pérolas durante a noite à temperatura ambiente, lavar 3 vezes com tampão de fosfato e finalmente re-suspender-se em 100 ul de tampão de fosfato e armazenar a 4 ° C.
- Revestimento Streptavidin em grânulos de vidro
- Misturar 5 ul de pérolas MPTMS com 5 ul de solução de 4 mg / ml SA e adicionar 140 ul de tampão de fosfato.
- Incubar as pérolas durante a noite à temperatura ambiente, lavar 3 vezes com tampão de fosfato, e re-suspender em 50 ul de tampão de fosfato e armazenar a 4 ° C.
- Revestimento dos grânulos SA revestido com uma proteína Biotinilado
- Misturar 5 ul de pérolas SA revestidos com a proteína (de volume, dependendo da densidade de revestimento desejada) e adicionar tampão de fosfato para fazer um volume final de 100 ul ser.
- Incubar a mistura durante a noite a 4 ° C ou durante 3 horas à temperatura ambiente, lavar 3 vezes com tampão de fosfato, e re-suspensão em 50 uL de pHosphate tampão e armazenar a 4 ° C.
4. Preparação celular
Nota: Para a purificação das células, seguir os protocolos de purificação de células normais correspondentes ao tipo de células, em utilização, por exemplo T-27 ou células de determinadas linhagens de células 21,29.
- Para as experiências de fBFP, uma vez que a suspensão de células é preparada, adicionar Fura2-AM (Tabela 1) dissolvido em DMSO para a suspensão de células para atingir uma concentração final de 2 fiM, incubar durante 30 min à temperatura ambiente e, em seguida, lavar uma vez. Manter esta suspensão de células fluorescente carregado no escuro até o uso.
5. Preparação para Micropipetas e uma câmara de celular
- Preparando Micropipetas
- Tubos cortados longo capilar de vidro (Tabela 1) com um cortador de vidro em peças curtas de cerca de 3 polegadas de comprimento. Monte uma peça para o extrator micropipeta (Tabela 1), clique no botão "Pull", mastonelada de modo que o meio do capilar irá ser aquecido pela máquina e o capilar irá ser puxado sobre as duas extremidades para fazer dois capilares com pontas afiadas micropipetas (bruto).
Nota: Seguindo a orientação do produto, a morfologia desejada da pipeta cru tem 6-8 mm de conicidade e 0,1-0,5 uM ponta. - Montar uma pipeta em bruto no suporte da pipeta da forja micropipeta (Tabela 1). Calor para derreter a esfera de vidro na forja. Insira a ponta da pipeta cru dentro da esfera de vidro. Esfriar a esfera de vidro e puxe a pipeta cru para quebrá-lo do lado de fora e deixar a ponta dentro da esfera. Repita este procedimento até que o orifício do bico desejado é obtido.
Nota: Os exemplos de um diâmetro interior da ponta da micropipeta: 2,0-2,4 mm para um RBC, ~ 1,5 um para um cordão, ~ 2-4 mm para uma célula T e -7 mm para uma célula de hibridoma.
- Tubos cortados longo capilar de vidro (Tabela 1) com um cortador de vidro em peças curtas de cerca de 3 polegadas de comprimento. Monte uma peça para o extrator micropipeta (Tabela 1), clique no botão "Pull", mastonelada de modo que o meio do capilar irá ser aquecido pela máquina e o capilar irá ser puxado sobre as duas extremidades para fazer dois capilares com pontas afiadas micropipetas (bruto).
- A construção de uma Câmara celular
Nota: a câmara de célula é construído sobre a base de uma casa-madcâmara de suporte e, que consiste em duas peças de quadrados de metal (cobre / alumínio) e uma pega que os liga em conjunto (Figura 1D). - Corte de 40 mm x 22 mm x 0,2 milímetros lamela utilizando um cortador de vidro em duas peças 40 milímetros x 11 milímetros x 0,2 milímetros (lamela 1 e 2). Cola lamela 1 por graxa para o lado de cima do suporte da câmara da maneira que preenche os dois quadrados de metal, e de forma semelhante lamela 2 de cola ao lado de fundo, que irá formar uma câmara da célula paralelo-lamela (Figura 1D).
- Usar uma pipeta para injectar 200 ul de tampão experimental entre as duas lamelas. Verifique se o tampão atribui a ambas as lamelas. Delicadamente gire e sacuda a câmara para deixar o tampão tocar ambas as extremidades da câmara.
- Cuidadosamente injectar óleo mineral em ambos os lados da câmara de flanqueamento a zona tampão experimental selando assim o tampão do ar aberto. Injectar suspensões de pérolas de sonda (por exemplo, grânulos pMHC-revestidas), glóbulos vermelhos e alvos(por exemplo, células T) nas regiões superior, média e inferior da zona de amortecimento, respectivamente.
6. BFP experimento
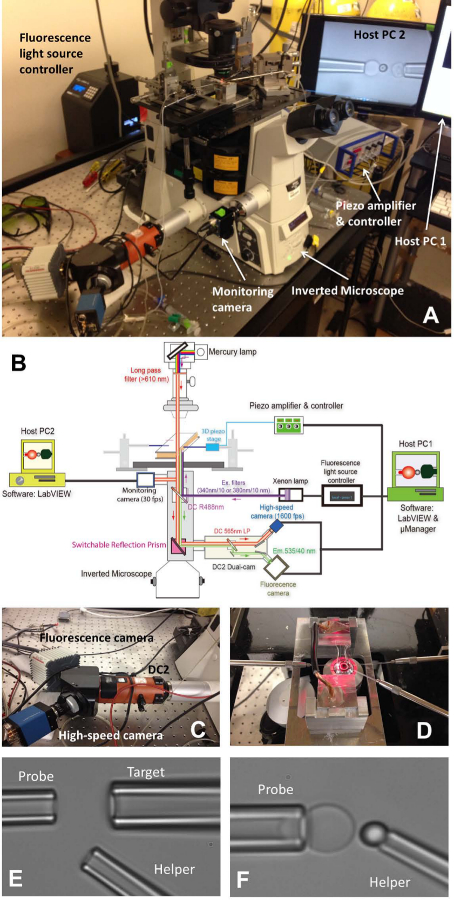
Figura 1: montagem fBFP (A) Uma imagem de visão geral do sistema de hardware fBFP.. (B) Um desenho esquemático do sistema de hardware fBFP. (C) O sistema dual-cam "DC2" (laranja) na qual a câmera de alta velocidade (azul) e uma câmera de fluorescência (branco) foram montados. (D) O estágio do microscópio que se adapta uma câmara de experiência e três sistemas de manipulação de micropipeta. Micrografias de configuração BFP em uma câmara experimental (E e F). (E) Micropipetas montagem mostrando a pipeta sonda (à esquerda), pipeta alvo (canto superior direito) e ajudante pipeta (inferior rireita). Talões (F) Probe. Uma gota sonda foi manipulado por uma pipeta ajudante e anexado a um ápice RBC para formar uma sonda de força. Por favor clique aqui para ver uma versão maior desta figura.
- Ligue o microscópio (Tabela 1) e fonte de luz. Coloque a câmara para o palco principal microscópio (Figura 1A, D).
- Instale todos os três micropipetas de BFP (Figura 1D esquerda:. Sonda, para pegar um RBC, direita: alvo, para pegar uma célula ou grânulo, inferior direito: ajudante, para pegar um talão).
- Usar um micro-injector (Tabela 1) para enchimento de uma micropipeta com tampão experimental. Retire o suporte da pipeta (Tabela 1) e mantê-lo em um lugar mais baixo para permitir que a água pingando da ponta. Inserir rapidamente a micropipeta na ponta titular e certifique-se nenhuma bolha de ar entra na micropipetadurante a inserção. Aperte o parafuso do suporte.
- Monte cada titular pipeta sua correspondente micro-manipulador. Empurre a micropipeta para a câmara de modo a que as suas dicas entrar na área do buffer da câmara. Ajuste a posição da micropipeta e encontrá-los sob o microscópio de campo de visão.
- Deslocar-se no palco titular câmara de encontrar as colônias de três elementos (as hemácias, as metas e as contas de sonda) um por um. Ajustar a posição da micropipeta correspondente rodando os botões dos manipuladores para deixar a ponta da micropipeta uma abordagem célula / conta. Aspirar a célula / conta ajustando a pressão no interior da micropipeta correspondente. Todos os três micropipetas irá capturar os seus elementos correspondentes.
- Deslocar-se no palco titular câmara para encontrar um espaço aberto longe das colônias de elementos injetados onde o experimento serão executadas. Mude o modo visual microscópio para visualizar a imagem no computer programa na tela do computador. Mova todos os três elementos sobre pontas de pipeta no campo de visão do programa.
- Alinhe o talão sonda e RBC, e manobrar cuidadosamente o talão sonda para o ápice da RBC, colidir brevemente o talão para o RBC e suavemente retrair. Ajustar a pressão da micropipeta auxiliar para soprar suavemente o talão de distância, de modo que ela ser deixada colada sobre o vértice de RBC (Figura 1F). Afaste-se da micropipeta ajudante e alinhar o alvo e talão de sonda (Figura 2A).
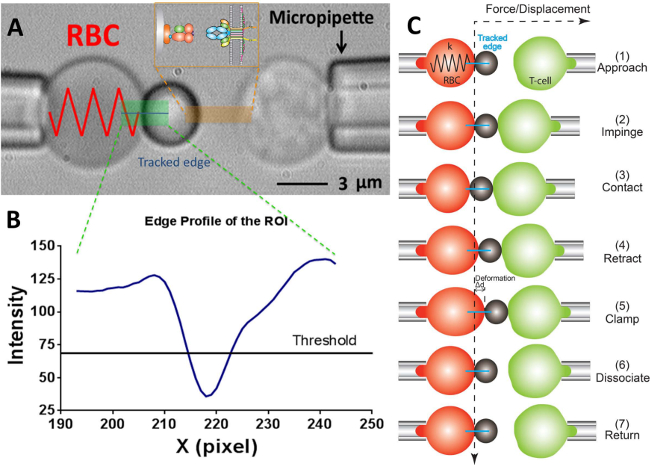
Figura 2:. Regime de PBF e o seu ciclo de teste (A) Vídeo-micrografia que descreve uma sonda de força (esquerda) e um T para a célula alvo (direita) aspirada pela respectiva sonda força estacionária pipettes.The consiste de um RBC inchado e um ligado grânulo de rolamento ligando. O receptor-bearingDe células T (alvo) está montado a uma piezotranslator alinhado oposto ao da sonda. O ROI é indicada em verde. O rastreador de borda é indicado em uma linha azul. A inserção mostra o ligando (pMHC, lado talão) e receptor (TCR, lado a célula T) par, nas duas faces opostas na área marcada em cor de laranja. (B) O perfil de intensidade do bordo do grânulo em (A). A região ROI no x -Direção é plotado como eixo x (em número de pixels) ea intensidade da luz (no valor de escala de cinza) em média por binning 30 pixels ao longo do y -Direção. (C) A deformação dos RBC e a posição do grânulo e o alvo (células T) num ciclo de teste de ensaio de força da braçadeira. As linhas tracejadas verticais e horizontais indicam a posição-zero da força do ápice de RBC e a evolução no tempo, respectivamente. O rastreador borda linha da deformação RBC é mostrado em azul em cada painel. Os passos mesmos ainda menos são adoptadas de freqüência de adesãoensaio (a que falta os passos de "grampo" e "dissociar") e o ensaio de flutuação térmica (que não possui o passo de "dissociar").
- No programa, na janela do campo de visão usar as ferramentas no programa para medir o respectivo raios da micropipeta sonda (R p), o RBC (R 0), a área de contacto circular entre a RBC e talão de sonda (R c) , o qual permite a estimativa da constante de RBC (k) de mola através da seguinte equação 17,30,
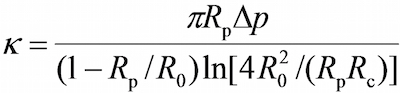
onde Δ p é a pressão de aspiração na ponta da pipeta sonda.
Nota: Resulta da lei de Hooke que a força de ligação, F, pode ser quantificada pelo produto da constante de mola e deslocamento do cordão de sonda (d), isto é, F = d (Figura 2C). - Digite a constante da mola RBC desejado para o programa (Por favor, consulte Protocolo secção 6.6. A constante da mola é normalmente fixado em 0,25 ou 0,3 PN / nm para o ensaio freqüência ensaio força da braçadeira e adesão e 0,1 PN / nm para o ensaio de flutuação térmica) , que irá retornar uma pressão de aspiração necessária na unidade de centímetros de água. Ajuste a altura do tanque de água que liga a pipeta da sonda até que a pressão de aspiração necessária seja alcançada.
- Desenhar uma linha horizontal através do ápice de RBC, a qual irá produzir uma curva na janela adjacente indicando o brilho (valor de escala de cinzentos) de cada pixel ao longo desta linha. Arrastar a linha de limiar para ser de cerca de metade da profundidade da curva (Figura 2A, B).
Nota: O ponto mínimo na curva de brilho abaixo da linha limiar indica a posição do limite de grânulo, assim, apenas um mínimo local é permitido. Se dois ou mais mínimos locais são no presente,indica que a imagem não é o ideal (provavelmente devido à imagem de estar fora de foco, ou um alinhamento pobre entre o talão sonda eo RBC). - Selecione o modo de experiência desejada: ensaio de flutuação térmica, ensaio de freqüência de adesão ou ensaio de força de fechamento. Defina os parâmetros conforme desejado (por exemplo, a força do impacto = 15 pN, taxa de carregamento = 1.000 PN / s, tempo de contato = 1 seg, força de aperto = 20 pN (para o ensaio braçadeira vigor), etc).
- Clique em "Iniciar", que permite que o programa para mover a pipeta alvo e direcionar o alvo dentro e fora de contato com a sonda (ver secção Resultados representativos para detalhes). A recolha de dados vai ser realizados em paralelo, que regista a posição do cordão de sonda em tempo real. Pare o programa clicando no botão "Stop experimento", momento em que uma janela irá aparecer para permitir salvar os dados adquiridos.
7. Fluorescência BFP (fBFP) experimento
- Para usar o fluoréscence função do sistema de BFP, ligar a fonte de luz de excitação (Tabela 1) e a câmara de fluorescência (Tabela 1), que são controladas por um programa separado (Tabela 1). No programa, selecione os parâmetros para a imagem de fluorescência, incluindo o ganho, a exposição, os canais de excitação (neste caso, 340 nm e 380 nm de luz), etc. Siga todas as preparações no protocolo experimental BFP, incluindo o alinhamento da sonda e o alvo, que irá permitir a visualização da célula alvo de imagem fluorescente vivo excitado por 340 nm ou 380 nm de luz de excitação.
- Use a ferramenta de corte para cerca de seção na zona em que a célula irá ficar durante todo o período de gravação.
Nota: Devido à utilização do ciclo de abordagem-contacto de retracção, a célula irá ser mover para a frente e para trás de maneira repetitiva, assim, a área seccionada é muito maior do que a própria célula. - Clique em "Record" para permitir a uma 340 nmd 380 nm de luz para excitar o corante fluorescente alternadamente intracelular (Fura2), e um par correspondente de imagens de fluorescência será gravado alternadamente cerca de uma vez em cada segundo. Simultaneamente clique em "Iniciar" no programa para iniciar o experimento BFP para interacção molecular e análise da experiência por imagem de fluorescência para monitorizar a sinalização intracelular de cálcio. O sistema irá produzir um ficheiro de dados em bruto para a ligação ao receptor-ligando (ver Figura 6A abaixo) e uma série de imagens fluorescentes em formato .Tiff para os sinais de cálcio.
Análise 8. Dados
- Análise de Dados BFP
- A análise dos dados para o ensaio de freqüência de adesão
- Sequencialmente inspecionar sinal de cada ciclo de "força vs tempo" e que simplesmente gravar ciclos conter um evento de adesão e quais não, e resumir para produzir uma frequência média de adesão.
- Recolha da força de ruptura de cada evento adesão, queé o valor de pico da força linearmente rampa antes da ruptura bond. Depois de recolher uma quantidade suficiente de forças de ruptura na gama de taxas de rampa, derivar a distribuição da força de ruptura em cada taxa de rampa a partir da qual a taxa de dissociação de dissociação do receptor dependente de ligando-força é derivado utilizando a força dinâmica análise de espectroscopia de 18,31.
- A análise dos dados para o ensaio de flutuação térmica
- Sequencialmente inspecionar o sinal da fase de fixação de cada ciclo, o que provavelmente contém vários eventos da associação de obrigações e de dissociação. Usar o nível de flutuação térmica fase de aperto (o desvio padrão médio de um intervalo de deslizamento de 70 pontos de tempo sequenciais do grânulo) na posição de "força em função do tempo" sinal como um guia para distinguir os eventos de associação e de dissociação ligação, uma vez que uma ligação formação corresponde a uma redução na flutuação térmica.
- Designar o intervalo a partir do instante de ligação de dissociação (quando o therMal flutuação retoma ao nível normal) ao instante da próxima formação de ligação como o tempo de espera, e estabelecer a duração da ligação da sua associação para dissociação como vida ligação, os quais são ambos recolhidos durante a inspecção dos dados. Calcule o tempo médio de espera eo tempo de vida médio de títulos, que, respectivamente, refletem o recíproco da taxa on-e off-a de taxa abaixo de zero-força 16,30.
- A análise dos dados para o ensaio de força de fechamento
- Parâmetros de registro de todos os eventos da vida, incluindo a força média e tempo de vida vínculo com o número sequencial, bem como a hora de início ea hora de término, o que permitirá um para desenhar uma curva cumulativo (por exemplo, a Figura 6C, curva amarela).
- Recolher uma quantidade suficiente de acontecimentos da vida sob uma gama de forças. Agrupá-los em diferentes caixas de força, que irá produzir um tempo de vida médio de cada força bin, e completamente render uma "vida média vs. força curva "(Figura 4).
- A análise dos dados para o ensaio de freqüência de adesão
- Análise de fluorescência de cálcio dados de imagem
- Ajustar o limiar de intensidade até que as imagens de fluorescência mostram um claro contorno da célula em ambos os canais 340 nm e 380 nm sem o ruído de fundo (Figura 5A, B). Em seguida, analisar o Ca2 + intracelular quadro de sinal por frame com uma pseudo-cor que indica o nível de intensidade (Figura 6B), o qual é determinado com base no rácio de intensidade de 340 nm / 380 nm, para gerar o "normalizado Ca 2+ intensidade vs . tempo curva "(Figura 6C). Use as imagens pseudo-cor de fluorescência para produzir um filme que mostra o nível de fluorescência segundo a segundo.
Resultados
A técnica BFP foi iniciada pelo laboratório Evans em 1995 17. Esta ferramenta picoforce tem sido extensivamente utilizado para medir as interacções de proteínas imobilizadas em superfícies, de modo a analisar a cinética bidimensionais de moléculas de adesão que interage com os seus ligandos 16,19,20, 30, para medir a elasticidade molecular 21,29, e para determinar conformacional proteína muda 21. Para uma fBFP, um conjunto adicional de dispositivos relacionados-epi-f...
Discussão
A experiência bem sucedida fBFP implica algumas considerações críticas. Em primeiro lugar, para o cálculo de força para ser confiável, micropipeta, a RBC, eo talão sonda deve ser alinhado como perto de coaxial possível. A projecção do RBC dentro da pipeta deve ser de cerca de um diâmetro de sonda de pipeta de modo que o atrito entre a RBC e a pipeta é negligenciável. Para um RBC humano típico, o diâmetro ideal pipeta é 2,0-2,4 mm, que produz um melhor ajuste da Equação 1 17,30. Em segundo l...
Divulgações
The authors have nothing to disclose.
Agradecimentos
Research related to this paper and the development of the fBFP technology in the Zhu lab were supported by NIH grants AI044902, AI077343, AI038282, HL093723, HL091020, GM096187, and TW008753. We thank Evan Evans for inventing this empowering experimental tool, and members of the Evans lab, Andrew Leung, Koji Kinoshita, Wesley Wong, and Ken Halvorsen, for helping us to build the BFP. We also thank other Zhu lab members, Fang Kong, Chenghao Ge and Kaitao Li, for their helps in the instrumentation development.
Materiais
| Name | Company | Catalog Number | Comments |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4 • H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | N2-5% buffer preparation |
| Potassium chloride (KCl) | Sigma-Aldrich | P9541 | N2-5% buffer preparation |
| Potassium phosphate monobasic (KH2PO4) | Sigma-Aldrich | P5655 | N2-5% buffer preparation |
| Sucrose | Sigma-Aldrich | S0389 | N2-5% buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Bead functionalization |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Nystatin | Sigma-Aldrich | N6261 | RBC osmolarity adjustment |
| Ammonium Hydroxide (NH4OH) | Sigma-Aldrich | A-6899 | Glass bead silanization |
| Methanol | BDH | 67-56-1 | Glass bead silanization |
| 30% Hydrogen Peroxide (H2O2) | J. T. Barker | Jan-86 | Glass bead silanization |
| Acetic Acid (Glacial) | Sigma-Aldrich | ARK2183 | Glass bead silanization |
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Borosilicate Glass beads | Distrilab Particle Technology | 9002 | Glass bead functionalization |
| Streptavidin−Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
| BSA | Sigma-Aldrich | A0336 | Ligand functionalizing |
| Fura2-AM | Life Technologies | F-1201 | Intracellular calcium fluorescence dye loading |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650 | Intracellular calcium fluorescence dye loading |
| Quantibrite PE Beads | BD Biosciences | 340495 | Density quantification |
| Flow Cytometer | BD Biosciences | BD LSR II | Density quantification |
| Capillary Tube 0.7-1.0 mm x 30 inches | Kimble Chase | 46485-1 | Micropipette making |
| Flaming/Brown Micropipette Puller | sutter instrument | P-97 | Micropipette making |
| Pipette microforce | Narishige | MF-900 | Micropipette making |
| Mineral Oil | Fisher Scientific | BP2629-1 | Chamber assembly |
| Microscope Cover Glass | Fisher Scientific | 12-544-G | Chamber assembly |
| Micro-injector | World Precision Instruments | MF34G-5 | Chamber assembly |
| 1 ml syringe | BD | 309602 | chamber assembly |
| Micropipette holder | Narishige | HI-7 | Chamber assembly |
| Home-designed mechanical parts and adaptors fabrications using CNC machining. | Biophysics Instrument | All parts are customized according to the CAD designs. | BFP system |
| Microscope (TiE inverted) | Nikon | MEA53100 | BFP system |
| Objective CFI Plan Fluor 40x (NA 0.75, WD 0.72 mm, Spg) | Nikon | MRH00401 | BFP system |
| Camera, GE680, 640 x 480, GigE, 1/3" CCD, mono | Graftek Imaging | 02-2020C | BFP system |
| Prosilica GC1290 - ICX445, 1/3", C-Mount, 1280 x 960, Mono., CCD, 12 Bit ADC | Graftek Imaging | 02-2185A | BFP system |
| Manual submicron probehead with high resolution remote control | Karl Suss | PH400 | BFP system |
| Anti-vibration table (5’ x 3’) | TMC | 77049089 | BFP system |
| 3D manual translational stage | Newport | 462-XYZ-M | |
| SolidWorks 3D CAD software | SOLIDWORKS Corp. | Version 2012 SP5 | BFP system |
| LabVIEW software | National Instruments | Version 2009 | BFP system, BFP program |
| 3D piezo translational stage | Physik Instrumente | M-105.3P | BFP system |
| Linear piezo accuator | Physik Instrumente | P-753.1CD | BFP system |
| Micromanager software | Version 1.4 | fBFP system, fluorescence imaging program | |
| Dual Cam (DC-2) | Photometrics | 77054724 | fBFP system |
| Dual Cam emission filter (T565LPXR) | Photometrics | 77054725 | fBFP system |
| Fluorescence Camera | Hamamatsu | ORCA-R2 C10600-10B | fBFP system |
| Plastic paraffin film (Parafilm) | Bemis Company, Inc | PM996 | bottle sealing |
| Carbonate/bicarbonate buffer (pH 8.5) | 8.4 g/L sodium carbonate (Na2CO3), 10.6 g/L sodium bicarbonate (NaHCO3) | ||
| Phosphate buffer (pH 6.5-6.8) | 27.6 g/L NaPhosphate monobasic (NaH2PO4 • H2O), 28.4 g/L Anhy. NaPhosphate dibasic (Na2HPO4) | ||
| N2-5% buffer (pH 7.2) | 20.77 g/L potassium chloride (KCl), 2.38 g/L sodium chloride (NaCl), 0.13 g/L potassium phosphate monobasic (KH2PO4), 0.71 g/L anhy. sodium phosphate dibasic (Na2HPO4), 9.70 g/L sucrose |
Referências
- Aplin, A. E., Howe, A., Alahari, S. K., Juliano, R. L. Signal transduction and signal modulation by cell adhesion receptors: the role of integrins, cadherins, immunoglobulin-cell adhesion molecules, and selectins. Pharmacological reviews. 50, 197-263 (1998).
- Davis, M. M., Bjorkman, P. J. T-cell antigen receptor genes and T-cell recognition. Nature. 334, 395-402 (1988).
- Dado, D., Sagi, M., Levenberg, S., Zemel, A. Mechanical control of stem cell differentiation. Regenerative medicine. 7, 101-116 (2012).
- Edwards, L. J., Zarnitsyna, V. I., Hood, J. D., Evavold, B. D., Zhu, C. Insights into T cell recognition of antigen: significance of two-dimensional kinetic parameters. Frontiers in immunology. 3, 86 (2012).
- Zhu, C., Jiang, N., Huang, J., Zarnitsyna, V. I., Evavold, B. D. Insights from in situ analysis of TCR-pMHC recognition: response of an interaction network. Immunological reviews. 251, 49-64 (2013).
- Huang, J., Meyer, C., Zhu, C. T. T cell antigen recognition at the cell membrane. Molecular immunology. 52, 155-164 (2012).
- Zarnitsyna, V., Zhu, C. T. T cell triggering: insights from 2D kinetics analysis of molecular interactions. Physical biology. 9, 045005 (2012).
- Binnig, G., Quate, C. F., Gerber, C. Atomic Force Microscope. Physical Review Letters. 56, 930-933 (1986).
- Marshall, B. T., et al. Direct observation of catch bonds involving cell-adhesion molecules. Nature. 423, 190-193 (2003).
- Kong, F., Garcia, A. J., Mould, A. P., Humphries, M. J., Zhu, C. Demonstration of catch bonds between an integrin and its ligand. The Journal of cell biology. 185, 1275-1284 (2009).
- Yago, T., et al. Catch bonds govern adhesion through L-selectin at threshold shear. The Journal of cell biology. 166, 913-923 (2004).
- Yago, T., et al. Platelet glycoprotein Ibalpha forms catch bonds with human WT vWF but not with type 2B von Willebrand disease vWF. The Journal of clinical investigation. 118, 3195-3207 (2008).
- Chesla, S. E., Selvaraj, P., Zhu, C. Measuring two-dimensional receptor-ligand binding kinetics by micropipette. Biophysical journal. 75, 1553-1572 (1998).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464, 932-936 (2010).
- Heinrich, V., Wong, W. P., Halvorsen, K., Evans, E. Imaging biomolecular interactions by fast three-dimensional tracking of laser-confined carrier particles. Langmuir : the ACS journal of surfaces and colloids. 24, 1194-1203 (2008).
- Chen, W., Evans, E. A., McEver, R. P., Zhu, C. Monitoring receptor-ligand interactions between surfaces by thermal fluctuations. Biophysical journal. 94, 694-701 (2008).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical. 68, 2580-2587 (1995).
- Evans, E., Leung, A., Heinrich, V., Zhu, C. Mechanical switching and coupling between two dissociation pathways in a P-selectin adhesion bond. Proceedings of the National Academy of Sciences of the United States of America. 101, 11281-11286 (2004).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal Flanking Region of the A1 Domain Regulates the Force-dependent Binding of von Willebrand Factor to Platelet Glycoprotein Ib. Journal of Biological Chemistry. 288, (2013).
- Chen, W., Lou, J., Zhu, C. Forcing switch from short- to intermediate- and long-lived states of the alphaA domain generates LFA-1/ICAM-1 catch bonds. The Journal of biological chemistry. 285, 35967-35978 (2010).
- Chen, W., Lou, J., Evans, E. A., Zhu, C. Observing force-regulated conformational changes and ligand dissociation from a single integrin on cells. The Journal of cell biology. 199, 497-512 (2012).
- Judokusumo, E., Tabdanov, E., Kumari, S., Dustin, M. L., Kam, L. C. Mechanosensing in T lymphocyte activation. Biophysical journal. 102, L5-L7 (2012).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences of the United States of America. 111, 2241-2246 (2014).
- Nesbitt, W. S., et al. Distinct glycoprotein Ib/V/IX and integrin alpha IIbbeta 3-dependent calcium signals cooperatively regulate platelet adhesion under flow. The Journal of biological chemistry. 277, 2965-2972 (2002).
- Mazzucato, M., Pradella, P., Cozzi, M. R., De Marco, L., Ruggeri, Z. M. Sequential cytoplasmic calcium signals in a 2-stage platelet activation process induced by the glycoprotein Ibalpha mechanoreceptor. Blood. 100, 2793-2800 (2002).
- Lefort, C. T., Ley, K. Neutrophil arrest by LFA-1 activation. Frontiers in immunology. 3, 157 (2012).
- Liu, B., Chen, W., Evavold, B. D., Zhu, C. Accumulation of dynamic catch bonds between TCR and agonist peptide-MHC triggers T cell signaling. Cell. 157, 357-368 (2014).
- Lou, J., et al. Flow-enhanced adhesion regulated by a selectin interdomain hinge. The Journal of cell biology. 174, 1107-1117 (2006).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature communications. 5, 4886 (2014).
- Chen, W., Zarnitsyna, V. I., Sarangapani, K. K., Huang, J., Zhu, C. Measuring Receptor-Ligand Binding Kinetics on Cell Surfaces: From Adhesion Frequency to Thermal Fluctuation Methods. Cellular and molecular bioengineering. 1, 276-288 (2008).
- Marshall, B. T., Sarangapani, K. K., Lou, J., McEver, R. P., Zhu, C. Force history dependence of receptor-ligand dissociation. Biophysical. 88, 1458-1466 (2005).
- Xiang, X., et al. Structural basis and kinetics of force-induced conformational changes of an alphaA domain-containing integrin. PloS one. 6, e27946 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados