Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayo basado en HPLC para supervisar extracelular de nucleótidos / nucleósidos de metabolismo en las células de la leucemia crónica humana linfocítica
En este artículo
Resumen
The protocol described here represents an easy and reproducible method that employs reverse phase high-performance liquid chromatography (RP-HPLC) to measure purine metabolism on chronic lymphocytic leukemia (CLL) cells cultured under different conditions.
Resumen
Este método describe un cromatografía sensible, específico, fiable y reproducible de fase inversa de alto rendimiento líquido (RP-HPLC) ensayo desarrollado y validado para la cuantificación de nucleótidos extracelulares de purina y nucleósidos producidos por la leucemia linfocítica crónica purificada (CLL) las células bajo diferentes condiciones de cultivo . La separación cromatográfica de la adenosina 5'-monofosfato (AMP), adenosina (ADO) e inosina (INO) se lleva a cabo a temperatura ambiente en una columna a base de sílice, de fase inversa que se utiliza para la retención de compuesto polar. El método incluye una fase móvil binaria, que consiste en acetato de amonio 7 mM y acetonitrilo con un caudal de 1,00 ml / min. Los eluatos se controlan mediante un detector de UV fotodiodo matriz de conjuntos a 260 nm. Una curva de calibración estándar se genera para calcular la ecuación para la cuantificación de análisis de cada compuesto purina. se llevan a cabo entonces el control del sistema, adquisición de datos y análisis. La aplicación de este protocolo, AMP, INO y ADO eluyen a los 7, 11 y 11,9 min, respectivamente, y el tiempo total de funcionamiento para cada muestra es de 20 min. Este protocolo se puede aplicar a diferentes tipos de células y líneas celulares (tanto de suspensión y adherentes), utilizando medios de cultivo como matriz. Las ventajas son fáciles y rápidos de preparación de muestras y la exigencia de una pequeña cantidad de sobrenadante para el análisis. Además, el uso de un medio libre de suero permite omitir el paso de la precipitación de proteínas con acetonitrilo que afecta a la concentración final de los compuestos de purina. Una de las limitaciones del método es el requisito de la columna de equilibrado ejecutar antes de cada muestra de ejecución única, haciendo que el tiempo total de funcionamiento del experimento más largo y la prevención de las aplicaciones de cribado de alto rendimiento.
Introducción
La adenosina (ADO) es un nucleósido de purina con una molécula de adenina unido a un resto de azúcar ribosa molécula a través de un enlace glucosídico. Cuando está presente en el medio extracelular, que protege las células del daño excesivo por la acción del sistema inmune. Este papel se ha puesto de relieve el uso de diferentes modelos de enfermedades, tales como la colitis 1, diabetes tipo 2, asma 3, 4 sepsis, lesión isquémica y 5. Una de las principales funciones de ADO es la inhibición de las respuestas inmunes en el microambiente tumoral, lo que contribuye a la evasión inmune tumor 6. Por esta razón, los mecanismos implicados en la formación de ADO y la señalización son de considerable interés terapéutico 7.
los niveles de ADO en el microambiente del tejido son relativamente bajos en condiciones fisiológicas normales y ciertamente por debajo del umbral de sensibilidad de las células inmunes. Sin embargo, durante la hipoxia, isquemia, inflamación, infección, metabólicasel estrés y la transformación tumoral que aumentan rápidamente 8. Los niveles elevados de ADO extracelular en respuesta a las señales perturbando tejidos tienen una doble función: reportar la lesión del tejido de una manera autocrina y paracrina y generar respuestas de los tejidos que pueden ser generalmente visto como citoprotector.
Extracelular ADO se puede formar a través de una variedad de mecanismos, que incluyen la liberación de compartimentos intracelulares mediados por transportadores de nucleósidos 9 o acumulación debido a la degradación deteriorado operado por la adenosina desaminasa. La principal vía que conduce a un aumento de los niveles extracelulares de ADO implica la acción de una cascada de ectonucleotidasas, que están asociadas a la membrana ectoenzimas generando ADO phosphohydrolysis de nucleótidos liberadas de las células muertas o moribundas. Esta vía procede a través de la acción secuencial de CD39 (trifosfato de ectonucleoside diphosphohydrolase-1) que convierte extracelular de adenosina 5'-trifosfato (ATP) o adenosina 5'-difosfato (ADP) a la adenosina 5'-monofosfato (AMP) y de CD73 (5'-nucleotidasa), que convierte AMP a ADO 10.
ADO extracelular provoca sus respuestas fisiológicas mediante la unión a receptores de cuatro transmembrana ADO, es decir, A1, A2A, A2B y A3. Cada receptor tiene diferentes afinidades por ADO y distribución de tejidos específicos. Todos los receptores tienen siete dominios transmembrana y son acoplados a proteína G a las proteínas intracelulares de unión a GTP (proteínas G), que puede inducir (proteína Gs) o inhibir (proteína Gi) la actividad de la adenilato ciclasa y, posteriormente, la producción de cAMP intracelular. Por lo tanto, los cambios en los niveles de cAMP citoplasmática impacto sobre la actividad de la proteína quinasa intracelular durante 11 respuestas fisiológicas. En condiciones fisiológicas ADO extracelular está por debajo de 1 m, que pueden activar indiscriminadamente A1, A2A y receptores A3. Sin embargo, la activación de subtipo A2B requiere considerablemente más altoconcentraciones de nucleósido, tales como los generados en condiciones fisiopatológicas. Alternativamente, ADO extracelular puede degradarse a inosina (INO) por la adenosina desaminasa (ADA) y CD26, una proteína de ADA complejante localización de ADA en la superficie celular. Otra posibilidad es que ADO es internalizado por la célula a través de los transportadores de nucleósidos equilibrador (ENT) y fosforilado a AMP por ADO proteína quinasa 12,13.
El objetivo de este protocolo es describir un método analítico de cromatografía líquida de alta resolución en fase inversa (RP-HPLC) para cuantificar en una sola ejecución del AMP sustrato y el ADO y productos INO, tal como se genera por los linfocitos humanos. Nuestra experiencia se obtuvo inicialmente utilizando células de pacientes crónicos leucemia linfocítica (CLL), que se caracterizan por la expansión de una población madura de linfocitos CD19 + / CD5 + B constitutivamente expresan CD39 14,15. Demostramos aproximadamente el 30%de los pacientes CLL expresan la CD73 ectoenzyme y que este fenotipo se correlaciona con un mal pronóstico 16. Esta subpoblación de células leucémicas co-expresan CD39 y CD73 puede producir activamente ADO extracelular a partir de ADP y / o AMP. La preincubación de las células CD73 + CLL con α, la síntesis de ADO extracelular β-metilen-ADP (APCP), un conocido inhibidor de la actividad enzimática de CD73, bloquea completamente confirmando que CD73 representa la enzima de cuello de botella de la que caen en cascada 16.
las células de LLC también expresan ADA y la ADA proteína CD26 de complejos, que son responsables de la conversión de ADO en INO. Mediante el uso de inhibidores específicos de la ADA, como eritro-9- (2-hidroxi-3-nonil) I wiadenine (EHNA) y clorhidrato de desoxicoformicina (dCF), es posible bloquear la degradación ADO extracelular en INO. Además, el pretratamiento con un inhibidor de ADA en combinación con dipiridamol, que bloquea los transportadores de nucleósidos, aumenta la acumulación de células en ADOsobrenadantes.
a continuación, hemos ampliado este protocolo para las células derivadas de otros linajes, incluyendo linfocitos T y células mieloides, lo que confirma la producción de ADO CD73-dependiente. Estos hallazgos sugieren que este protocolo HPLC es muy versátil y que puede ser aplicado a diferentes linajes de células y para diferentes condiciones de cultivo (Figura 1).
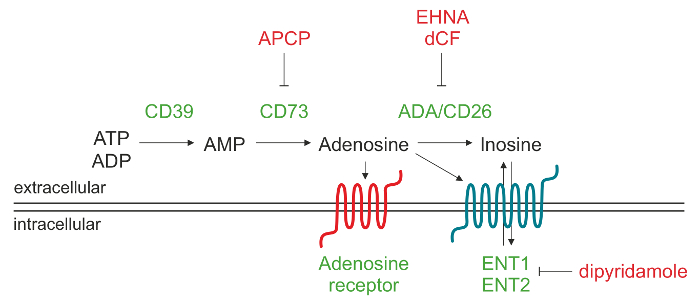
Figura 1. Representación esquemática de la maquinaria enzimática responsable de la producción ADO extracelular. La adenosina 5'-trifosfato (ATP) y / o adenosina 5'-difosfato (ADP) puede ser degradado por CD39 a la adenosina 5'-monofosfato (AMP), el cual a su vez, se convierte por CD73 en el nucleósido adenosina (ADO). Una vez ADO se produce en el espacio extracelular, se puede volver a entrar en la célula a través de los transportadores de nucleósidos (ENT), se convierte en inosina (INO) ose unen a diferentes tipos de receptores P1 ADO. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
CLL muestras de sangre se obtienen de acuerdo con las directrices institucionales y Declaración de Helsinki.
1. Aislamiento de leucémica linfocitos de muestras de sangre de pacientes con LLC
- Tomar la muestra de sangre en tubo de heparina sódica (tapón verde) 17.
- Hacer una dilución 1: 3 de sangre entera con RT 1x solución salina tamponada con fosfato (PBS).
- Purificar células mononucleares de sangre periférica (PBMC) a partir de muestras de sangre mediante centrifugación en gradiente de densidad.
- Underlay 5 ml de medio de centrifugación de densidad (por ejemplo, Ficoll) en un tubo de centrífuga de 15 ml y cuidadosamente transferir 10 ml de sangre diluida. Inmediatamente centrifugar a 1500 xg durante 20 min a TA.
- Aspirar parte del sobrenadante con una pipeta Pasteur de vidrio conectado a una bomba de vacío y recoger el anillo de PBMC en la interfase entre los medios de centrifugación de densidad y plasma diluido con una pipeta de 5 ml. Transferir a un tubo de centrífuga de 50 ml y se lava con PBS (50 ml de volumen final). Centrifugar a 500 xg durante 5 min a 4 ° C.
- Aspirar el sobrenadante con una pipeta Pasteur de vidrio, resuspender el precipitado en 50 ml de PBS y contar las células usando un hemocitómetro.
- PBMCs se tiñen con anti-CD19 y anticuerpos -CD5 para citometría de flujo siguiendo un protocolo estándar para la inmunotinción y comprobar si la composición subpoblación CMSP 18.
Nota: Los linfocitos de la LLC son CD19 + / CD5 +. - Compruebe el% de células CD19 + / CD5 + células utilizando un citómetro de flujo 18. Purificar los linfocitos B de pacientes con ≥95% de las células leucémicas siguiendo las instrucciones detalladas en el paso 2 para el aislamiento negativo de células B CD19 + 19 / + CD5.
2. Purificación de las células leucémicas B por Aislamiento Negativo
- Resuspender 10 7 PBMCs / ml en PBS 0,1% de albúmina de suero bovino (BSA) EDTA 2 mM, pH 7,4 (tampón de aislamiento).
- Añadir 101; g de anticuerpo monoclonal de ratón anti-CD3, -CD14 y -CD16 anticuerpos primarios durante 10 7 células y se incuba durante 30 min a 4 ° C.
- Lavar las células con el tampón de aislamiento y se centrifuga a 500 xg durante 5 min a 4 ° C.
- Resuspender las células en el tampón de aislamiento en 10 7 PBMCs / ml.
- Antes de la incubación con las células, resuspender de oveja anti-IgG de ratón perlas magnéticas en el vial. Transferir 100 l de perlas por 10 7 células a un tubo.
- Añadir 1 ml de tampón de aislamiento y se coloca el tubo en un soporte de imán para 1 min. Descartar el sobrenadante con una pipeta de 5 ml.
- Retire el tubo del imán y volver a suspender las perlas lavadas en 100 l de tampón de aislamiento.
- Incubar 10 7 células con 100 l de perlas magnéticas de 30 min a 4 ° C con inclinación suave y rotación.
- Se coloca el tubo en el soporte de imán para los 2 min y transferir el sobrenadante con las células no unidas CD19 + / CD5 +a un tubo fresco con una pipeta de 5 ml.
- Al final del protocolo de aislamiento, manchar 100 l de células con anti-CD19 y -CD5 anticuerpos para citometría de flujo y se incuba 30 min a 4 ° C. Se lavan las células con 3 ml de PBS 1% de BSA, desechar el exceso de sobrenadante y comprobar la pureza de células B de nuevo por citometría de flujo 19.
Nota: Realizar una etapa de aislamiento negativo adicional si la pureza de las células B es menor que 95%.
3. Preparación de Inhibidores de archivo estándar y Soluciones
- Para preparar AMP, pesar 0,00345 g de AMP y se disuelven en 5 ml de medio libre de suero para tener una solución estándar 2 mM de AMP.
- Para preparar ADO, pesar 0.00265 g de ADO y se disuelven en 5 ml de medio libre de suero para tener una solución estándar de 2 mm de ADO.
- Para preparar INO, pesar 0.00268 g de INO y se disuelven en 5 ml de medio libre de suero para tener una solución estándar de 2 mm de INO.
- Preparar una solución 400 mM de AMP, ADO yINO mezclando 1 ml de la solución madre 2 mM en 4 ml de (5 ml de volumen final) medio libre de suero.
- Hacer una dilución 1: 4 de cada solución estándar de 400 mM para obtener una concentración 100 mM pipeteando 500 l de la solución 400 mM en 1.500 l de medio libre de suero (2 ml de volumen final).
- Preparar diluciones en serie de cada compuesto en medio libre de suero para obtener las siguientes concentraciones: 100 mM - 50 mM - 25 mM - 10 mM - 5 M - 2,5 M - 1 M - 0,5 M - 0,25 M.
Nota: Por ejemplo, pipeta de 1 ml de la solución 100 mM en 1 ml de medio libre de suero (dilución 1: 2) para obtener una concentración de 50 mM y proceder con las diluciones restantes.
- Preparar una solución madre 10 mM de APCP disuelto en PBS; alícuota y el inventario en - 30 ° C.
- Preparar soluciones madre 10 mM de clorhidrato de EHNA, DCF y dipiridamol disuelto en sulfóxido de dimetilo (DMSO); alícuota y el inventario en -30DO.
4. Programa el método HPLC
- Preparar un tampón de acetato de amonio 7 mM disolviendo 0,770 g de acetato de amonio en 2 L de agua doblemente desionizada (Tampón A). Ajustar a pH 3,0 con ácido clorhídrico.
- Preparar una botella de vidrio que contiene al menos 2 L de acetonitrilo ultrapura para HPLC (Tampón B) y conectar la precolumna y la columna a la HPLC.
Nota: Las columnas y los tampones son a TA mientras está en uso. - Iniciar sesión en el software de HPLC y seleccione el botón "proyecto de exploración". Ir al menú "Archivo" y seleccione "nuevo método" y luego "método de instrumento".
- Programa el método de equilibrado de la columna según la Tabla 1. Establecer un caudal de 1,00 ml / min y programar el detector de UV para leer a 260 nm. Guardar el método instrumento y seleccione de nuevo "nuevo método" del menú "Archivo" y luego "método set". Elija el método de instrumentos previamente guardadoy guardar el método actual establecido con el mismo nombre.
| Hora | Velocidad de flujo (ml / min) | %UN | %SEGUNDO |
| 1.00 | 100 | 0 | |
| 1.24 | 1.00 | 100 | 0 |
| 6.22 | 1.00 | 2 | 98 |
| 18.65 | 1.00 | 2 | 98 |
Tabla 1: método de equilibrado de la columna Representación esquemática de los cambios de disolvente para el equilibrado de la columna.. Tampón A: acetato de amonio 7 mM, pH 3,0. Tampón B: acetonitrilo.
- Repita el paso 4.3 para programar el método de ejecución de la muestra se indica en la Tabla 2. Establecer un caudal de 1,00 mL / min y el programa de detector de UV a leer a 260 nm. Guardar el método instrumento y repita el mismo procedimiento que se describe en el paso 4.4 para guardar el método set ..
| Hora | Velocidad de flujo (ml / min) | %UN | %SEGUNDO |
| 1.00 | 0 | 100 | |
| 3.74 | 1.00 | 0 | 100 |
| 13.71 | 1.00 | 15 | 85 |
| 17.00 | 1.00 | 100 | 0 |
| 20.00 | 1.00 | 100 | 0 |
Tabla 2: Ejecutar método de muestreo. Representación esquemática de los cambios de disolvente para la medición de HPLC de los compuestos de purina. Pulirer A: acetato de amonio 7 mM, pH 3,0. Tampón B: acetonitrilo.
- Seleccione el botón "ejecutar muestras", elegir la opción "nuevo método conjunto de muestras" en el menú "archivo".
- Seleccionar la opción "vacío" e introduzca el número del vial (en el inyector automático), el nombre de la muestra, el volumen de inyección (50 l). Seleccione el método de configuración guardado antes de la columna "método set" y entrar en el tiempo de ejecución total de (min).
Nota: Introduzca el método de equilibrado de la columna (Tabla 1) en el "método set" columna antes de cada corrida de muestras y entrar en el tiempo de ejecución total de (min).
5. Generación de una curva de calibración estándar para cada compuesto
- Inyectar 50 l de (medio libre de suero específico, que no contiene adenosina desaminasa) en blanco y de cada muestra estándar siguiendo los métodos descritos en la Tabla 1 y la Tabla 2.
- Use la columna de equilibradométodo (Tabla 1) antes de cada muestra de ejecución y establecer las condiciones de gradiente descritos en la Tabla 2 para obtener la separación pico adecuado. Consulte la Tabla 3 para tiempos de retención reportados de AMP, ADO y INO bajo estas condiciones de HPLC.
| Tiempo de retención | λ max | |
| AMPERIO | 8.00 min | 260 |
| INO | 11.00 min | 260 |
| ALHARACA | 11.90 min | 260 |
Tabla 3: Los tiempos de retención de los compuestos de purina tiempos de retención típicos observados para AMP, ADO y INO.. El detector de UV está programado para leer a 260 nm.
- Determinar el área del pico del AMP, ADO y INO a diferentes concentraciones de elegir el botón "proceso" en el software de HPLC y al lado de la opción "integrar". Alternativamente seleccionar manualmente los puntos de inicio y final de cada pico.
- Trazar las áreas de pico frente a la concentración nominal de cada estándar para obtener la curva de calibración de nueve puntos (100, 50, 25, 10, 5, 2,5, 1, 0,5, 0,25 M).
- Calcular la ecuación de una línea recta de AMP, ADO y INO: y = mx + b,
donde x es la concentración de M e y corresponden a el área del pico.
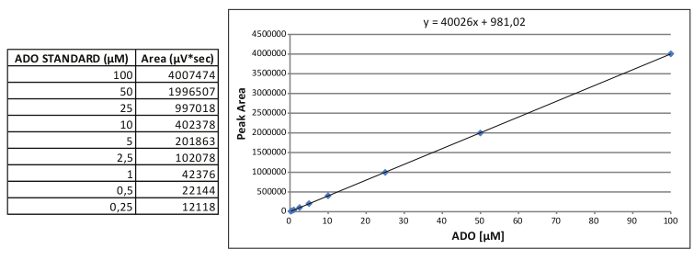
Figura 2. Generación de una curva patrón interno. Curva estándar de calibración Representante de ADO y la ecuación relativa obtenida. Por favor, haga clic en ellae para ver una versión más grande de esta figura.
6. El tratamiento previo con inhibidores de la e incubación con el sustrato (AMP)
- Para probar AMP consumo, ADO y la generación de INO y sin inhibición de CD73, ADA y los transportadores de nucleósidos, volver a suspender + / células CD5 + CLL 2 x 10 6 CD19 (aislado y purificado en los pasos 1 y 2) en 250 l de medio libre de suero y células de la placa en una placa de 48 pocillos (o en un tubo de microcentrífuga de 1,5 ml).
- Para bloquear la actividad enzimática de CD73, se diluye 1: 1000 la solución madre de APCP (10 mM) en el medio de cultivo para obtener una concentración final 10 mM. Resuspender 2 x 10 6 células de LLC en 250 l de medio de cultivo que contiene APCP y pretratar 60 min a 37 ° C en una incubadora de cultivo celular.
- Para bloquear la conversión de ADO en INO, diluir 1: 1000 la solución madre de hidrocloruro de EHNA y / o DCF (tanto 10 mM) en medio libre de suero para tener una concentración final 10 mM.Resuspender 2 x 10 6 células de LLC en 250 l de medio de cultivo que contiene EHNA y / o DCF. Incubar durante 30 min a 37 ° C en la incubadora de cultivo celular.
- Para bloquear la captación de ADO a través de los transportadores de nucleósidos, diluir 1: 1000 la solución madre de dipiridamol (10 mM) en medio libre de suero para tener una concentración final 10 mM. Resuspender 2 x 10 6 células de LLC en 250 l de medio de cultivo que contiene dipiridamol y se incuba durante 30 min a 37 ° C en la incubadora de cultivo celular.
- Preparar soluciones individuales de APCP, EHNA, DCF y dipiridamol diluido 1: 1.000 en 400 mM de AMP.
- Añadir 250 l de 400 mM AMP o AMP más inhibidores para obtener una concentración final de 200 mM AMP. Incluir una condición en ausencia del sustrato a ser utilizado como muestra en blanco.
- Incubar 30-60 min a 37 ° C.
Nota: La concentración óptima de AMP y el tiempo de incubación se determinó experimentalmente. - en tél final del tiempo de incubación, recoger 500 l de los sobrenadantes en tubos de microcentrífuga fríos (en hielo) y se centrifuga inmediatamente a 17.000 xga 4 ° C durante 5 min.
- La transferencia de los sobrenadantes en tubos nuevos de 1.5 ml y almacenar inmediatamente a -80 ° C o proceder a la preparación de muestras para corre el HPLC.
7. Preparación Las muestras para HPLC
- Filtrar los sobrenadantes en tubos nuevos microcentrífuga de 1,5 ml con los 0,2 micras filtros de jeringa. Utilizar jeringas de 1 ml para el filtrado.
- Si el sistema de HPLC se proporciona con un muestreador automático, preparar los viales de vidrio para HPLC y utilizar los 0,1 ml micro-insertos para pequeños volúmenes de muestra.
- Transferencia al menos 100 l de muestra en el frasco de vidrio con una micropipeta o una pipeta Pasteur de vidrio. Tenga cuidado para transferir sin burbujas y cerrar el vial con el tapón de rosca.
- Las muestras están ahora listas para el análisis por RP-HPLC.
8. HPLC Measurequeri- de purinas
- Seleccione el botón "muestras de ejecución"; elija "nuevo método conjunto de muestras" en el menú "archivo".
- Seleccionar la opción "vacío" e indicar: el número del vial (en el inyector automático), el nombre de la muestra, el volumen de inyección (50 l).
- Seleccione el método de equilibrado de la columna y el método de ejemplo de ejecución descrito en la Tabla 1 y la Tabla 2, respectivamente, para todas las pistas en blanco y de muestra que siguen.
Nota: Establecer el método de equilibrado de la columna (Tabla 1) antes de cada análisis de la muestra. - Inyectar 50 l de (medio libre de suero) en blanco y de cada muestra.
- Determinar la concentración de purinas en cada muestra, cuantificar el AMP, ADO y INO mediante la obtención de las áreas de los picos en los tiempos de retención descritos en la Tabla 3.
- Seleccione el botón "proceso" y al lado de la opción "integrar". Alternativamente elegir manualmente el comenzar unand puntos finales de cada pico individual para obtener la medida del área. Esto se hace mediante el trazado de una línea entre los puntos de inicio y final del pico.
- Determinar la concentración de M aplicando la ecuación obtenida para la curva estándar: y = mx + b, donde x representa la concentración de M e Y es el área del pico medida a partir de la muestra desconocida.
Nota: Un ejemplo de la curva de calibración estándar ADO se informa en la Figura 2, donde: x = (y área - 981,02) / 40026
- Teniendo en cuenta que 2 x 10 6 células se volvieron a suspender en 0.500 ml de medio, calcular los moles de AMP, ADO y INO consumidos y / o producidos por 10 6 células de la LLC que aplican la siguiente proporción:
micromoles: 1.000 ml = x moles: 0,250 ml
Nota: Los moles en 1.000 ml (número de moles en 1 L) de son equivalentes a la concentración de M y X son los moles de nucleotide o nucleósido producidos por 10 6 células.- Por último, convertir los moles en nmoles consumidos y / o producidos por 10 6 células de LLC:
nmoles = x 1.000 micromoles
- Por último, convertir los moles en nmoles consumidos y / o producidos por 10 6 células de LLC:
Resultados
Para evaluar el porcentaje (%) de las células leucémicas en PBMCs recién purificada a partir de un paciente con CLL representante, las células se marcaron con anticuerpos anti-CD5 anti-CD19 y. El panel izquierdo de la Figura 3 representa un gráfico de puntos cytofluorimetric con una puerta selectivo en células vivas. La figura 3 muestra un ejemplo de PBMC de un paciente antes de CLL (panel medio) y después de la purificación de células B (panel ...
Discusión
El protocolo descrito aquí permite evaluar la actividad de la CD39 / CD73 maquinaria adenosinérgico en medios de cultivo celular a partir de células leucémicas humanas purificadas. A través de este método HPLC podemos seguir y medir cuantitativamente la generación enzimática de ADO (CD73-dependiente) y su consiguiente degradación a INO (CD26 / ADA dependiente). El uso de inhibidores de la enzima permite controlar el protocolo y tener controles internos. Las ventajas y las novedades de este protocolo son que: i)...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Este trabajo es apoyado por Associazione Italiana Ricerca Cancro (IG # 12754).
Materiales
| Name | Company | Catalog Number | Comments |
| Human blood | |||
| Milli-Q water | Millipore | double deionised water | |
| Ficoll-Paque Plus | GE-Healthcare | 17-1440-03 | |
| purified anti-CD3, -CD14, -CD16 | made in-house | mouse monoclonal | |
| PE-labeled anti-CD19 | Miltenyi Biotec | 120-014-229 | |
| FITC-labeled anti-CD5 | Miltenyi Biotec | 130-096-574 | |
| Dynabeads sheep anti-mouse IgG | Invitrogen | 11031 | |
| Phosphate-buffered saline (PBS) | Amresco | E404-200TABS | tablets |
| bovine serum albumin (BSA) | ID bio | 1000-70 | standard grade |
| isolation buffer | PBS 0.1% BSA 2 mM EDTA, pH 7.4 | ||
| AIM V serum free medium | GIBCO | 12055-091 | liquid (research grade) |
| adenosine 5’-diphosphate (ADP) | Sigma-Aldrich | A2754 | |
| adenosine 5’-monosphate (AMP) | Sigma-Aldrich | A1752 | |
| adenosine (ADO) | Sigma-Aldrich | A9251 | |
| inosine (INO) | Sigma-Aldrich | I4125 | |
| α,β-methylene-ADP (APCP) | Sigma-Aldrich | M8386 | CD73 inhibitor |
| EHNA hydrochloride | Sigma-Aldrich | E114 | adenosine deaminase inhibitor |
| Deoxycoformycin (dCF) | Tocris | 2033 | adenosine deaminase inhibitor |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650 | |
| Dipyridamole | Sigma-Aldrich | D9766 | nucleoside transporter inhibitor |
| acetonitrile (CHROMASOLV Plus) | Sigma-Aldrich | 34998 | HPLC-grade |
| ammonium acetate | Sigma-Aldrich | 9688 | 7 mM, pH 3.0 |
| hydrochloric acid | Sigma-Aldrich | 30721-1L | min. 37% |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Bürker cell counter | VWR | 631-0920 | hemocytometer |
| DynaMag-15 Magnet | Invitrogen | 12301D | Dynal magnetic bead separator |
| microcentrifuge safe-lock tubes | Eppendorf | 030-120-0086 | 1.5 ml |
| PET centrifuge tubes | Corning | 430053/430304 | 15 – 50 ml |
| Minisart RC4 syringe filters | Sartorius Stedim Biotech | 17821 | membrane 0.2 µm |
| short thread vials | VWR | 548-0029 | 1.5 ml/glass |
| micro-inserts | VWR | 548-0006 | 0.1 ml/glass |
| screw caps | VWR | 548-0085 | 9 mm/PP blue |
| Atlantis dC18 Column | Waters | 186001344 | 5 µm, 4.6 mm x 150 mm |
| Atlantis dC18 Guard Column | Waters | 186001323 | 5 µm, 4.6 mm x 20 mm |
| Waters Alliance 2965 Separations Module | Waters | HPLC separation module | |
| Waters 2998 Photodiode Array (PDA) Detector | Waters | UV detector | |
| Waters Empower2 software | Waters |
Referencias
- Naganuma, M., Wiznerowicz, E. B., Lappas, C. M., Linden, J., Worthington, M. T., Ernst, P. B. Cutting edge: Critical role for A2A adenosine receptors in the T cell-mediated regulation of colitis. J Immunology. 177 (5), 2765-2769 (2006).
- Nemeth, Z. H., et al. Adenosine receptor activation ameliorates type 1 diabetes. FASEB J. 21 (10), 2379-2388 (2007).
- Fan, M., Jamal Mustafa, S. Role of adenosine in airway inflammation in an allergic mouse model of asthma. Int Immunopharmacol. 6 (1), 36-45 (2006).
- Csoka, B., et al. A2B adenosine receptors protect against sepsis-induced mortality by dampening excessive inflammation. J Immunol. 185 (1), 542-550 (2010).
- Peart, J. N., Headrick, J. P. Adenosinergic cardioprotection: multiple receptors, multiple pathways. Pharmacol Ther. 114 (2), 208-221 (2007).
- Ohta, A., et al. A2A adenosine receptor protects tumors from antitumor T cells. Proc Natl Acad Sci U S A. 103 (35), 13132-13137 (2006).
- Hasko, G., Linden, J., Cronstein, B., Pacher, P. Adenosine receptors: therapeutic aspects for inflammatory and immune diseases. Nat Rev Drug Discov. 7 (9), 759-770 (2008).
- Cronstein, B. N. Adenosine, an endogenous anti-inflammatory agent. J Appl Physiol (1985). 76 (1), 5-13 (1994).
- Molina-Arcas, M., Casado, F. J., Pastor-Anglada, M. Nucleoside transporter proteins. Curr Vasc Pharmacol. 7 (4), 426-434 (2009).
- Deaglio, S., et al. Adenosine generation catalyzed by CD39 and CD73 expressed on regulatory T cells mediates immune suppression. J Exp Med. 204 (6), 1257-1265 (2007).
- Linden, J. Regulation of leukocyte function by adenosine receptors. Adv Pharmacol. 61, 95-114 (2011).
- Antonioli, L., Blandizzi, C., Pacher, P., Hasko, G. Immunity, inflammation and cancer: a leading role for adenosine. Nat Rev Cancer. 13 (12), 842-857 (2013).
- Antonioli, L., Csoka, B., Fornai, M., et al. Adenosine and inflammation: what's new on the horizon. Drug Discov Today. 19 (8), 1051-1068 (1051).
- Chiorazzi, N., Rai, K. R., Ferrarini, M. Chronic lymphocytic leukemia. N Engl J Med. 352 (8), 804-815 (2005).
- Abousamra, N. K., Salah El-Din, M., Hamza Elzahaf, E., Esmael, M. E. Ectonucleoside triphosphate diphosphohydrolase-1 (E-NTPDase1/CD39) as a new prognostic marker in chronic lymphocytic leukemia. Leuk Lymphoma. 56 (1), 113-119 (2015).
- Serra, S., et al. CD73-generated extracellular adenosine in chronic lymphocytic leukemia creates local conditions counteracting drug-induced cell death. Blood. 118 (23), 6141-6152 (2011).
- Chen, L. S., Keating, M. J., Gandhi, V. Blood collection methods affect cellular protein integrity: implications for clinical trial biomarkers and ZAP-70 inn CLL. Blood. 124 (7), 1192-1195 (2014).
- Kalina, T., et al. EuroFlow standardization of flow cytometer instrument settings and immunophenotyping protocols. Leukemia. 26 (9), 1986-2010 (2012).
- Deaglio, S., et al. CD38 and ZAP-70 are functionally linked and mark CLL cells with high migratory potential. Blood. 110 (12), 4012-4021 (2007).
- Sachsenmeier, K. F., et al. Development of a novel ectonucleotidase assay suitable for high-throughput screening. J Biomol Screen. 17 (7), 993-998 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados