Method Article
La incorporación de residuos específicos de no canónicos aminoácidos en las proteínas modelo usando una
En este artículo
Resumen
An easy-to-use, cell-free expression protocol for the residue-specific incorporation of noncanonical amino acid analogs into proteins, including downstream analysis, is presented for medical, pharmaceutic, structural and functional studies.
Resumen
El conjunto canónico de aminoácidos da lugar a una gama excepcionalmente amplia de la funcionalidad de la proteína. Sin embargo, el conjunto de residuos sigue imponiendo limitaciones a las aplicaciones potenciales de proteína. La incorporación de aminoácidos no canónicos puede ampliar este ámbito. Hay dos enfoques complementarios para la incorporación de aminoácidos no canónicos. Para la incorporación específica de sitio, además de los mecanismos de traslación canónicas endógenos, un par ortogonal aminoacil-tRNA-sintetasa-ARNt debe ser siempre que no interactúa con los canónicos. En consecuencia, un codón que no está asignado a un ácido amino canónica, por lo general un codón de parada, también se requiere. Esta expansión código genético permite la incorporación de un aminoácido no canónico en un único, sitio dado dentro de la proteína. El trabajo aquí presentado describe la incorporación de residuos específico en el que el código genético es reasignado dentro del sistema de traslación endógeno. La maquinaria de traducción de unaccepts el aminoácido no canónica como un sustituto para incorporarlo en lugares prescritos canónicamente, es decir, todas las ocurrencias de un aminoácido en la proteína canónica se sustituyen por la no canónica. La incorporación de aminoácidos no canónicos puede cambiar la estructura de la proteína, haciendo que las propiedades físicas y químicas modificadas considerablemente. Análogos de aminoácidos no canónicos a menudo actúan como inhibidores del crecimiento celular para huéspedes de expresión, ya que modifican las proteínas endógenas, lo que limita en la producción de proteínas in vivo. In vivo la incorporación de aminoácidos no canónicos tóxicos en proteínas sigue siendo particularmente difícil. A continuación, se presenta un enfoque libre de células para un reemplazo completo de la L-arginina por la no canónica aminoácido L-canavanina. Elude las dificultades inherentes a la expresión in vivo. Además, se incluye un protocolo para preparar proteínas diana para el análisis espectral de masas. Se muestra que la L-lisina se puede sustituir por hidroxi-L-lisina,aunque con menor eficiencia. En principio, cualquier análogo de aminoácido no canónica puede incorporarse utilizando el método presentado, siempre y cuando el sistema de traducción in vitro endógena reconoce.
Introducción
El código genético es universal a la biosfera. Codifica para un conjunto de 20 aminoácidos canónicos, que a veces se extiende por seleniocisteína 1 o 2 pyrrolysine. Es el ribosoma que traduce el código genético con la ayuda de tRNAs en cadenas de aminoácidos que se pliegan en proteínas. Los grupos funcionales de los aminoácidos canónicos, en combinación con modificaciones postraduccionales, contribuyen a una gama excepcionalmente amplia de la función de proteínas 3,4. En principio, las limitaciones funcionales debido a la limitada serie de aminoácidos canónicos se pueden superar mediante la incorporación de más, aminoácidos canónicos (NCAA) que permiten nuevas químicas y nuevas funcionalidades 3,4.
Hay dos enfoques complementarios para la incorporación de la NCAA: sitio- o la incorporación de residuos específicos. El primer método implica considerables dificultades técnicas, ya que el conjunto canónico de aminoacil-ARNt-sintetizadoretases (aaRS) y ARNt deben ampliarse por un par ortogonal aaRs-ARNt que no debe interactuar con la maquinaria de traducción endógena. Sobre la base de la ingeniería cuidadosa, este enfoque incorpora las NCAA como mutaciones puntuales en los sitios de proteínas deseadas. La incorporación específica de sitio de NCAA está genéticamente codificado por un codón que no está asignado a un ácido amino canónica (CAA), por lo general un codón de 5-9. Este método implica cambios en la función en un centro determinado en lugar de a través de toda la proteína 10-13.
En contraste, la incorporación de residuos específica se basa en el reconocimiento erróneo del aminoácido no canónico por la maquinaria de traducción canónica. La incorporación se produce debido a la falta de especificidad de sustrato de los IAA. La incorporación de residuos específicos de la NCAA, basada en la obra de Cohen y compañeros de trabajo 14, ha dado lugar a importantes aplicaciones 3,10, entre ellos etiquetado bioortogonal 15-17 de proteínaso elucidación de la estructura de las proteínas en cristalografía de rayos X 18.
Como aaRs naturales generalmente prefieren su aminoácido relacionado sobre un NCAA isoestructural, eficiente en la incorporación vivo en residuos específicos por lo general requiere un huésped de expresión auxotrófico no es capaz de sintetizar el análogo canónica de la NCAA. Las células huésped se cultivan en medio de crecimiento que sólo entrega una baja concentración del CAA análoga. Su agotamiento en combinación con la administración de suplementos consecutiva con la NCAA obliga al huésped de expresión para incorporar la NCAA en la proteína modelo en múltiples sitios, canónicamente prescritos. En contraste con el enfoque específico del sitio, esto generalmente tiene un profundo impacto en toda la estructura de la proteína, lo que lleva a modificar considerablemente las propiedades físicas y químicas de las proteínas 19,20. Sin embargo, la mayoría de la NCAA son inhibidores de crecimiento para el huésped de expresión 3, ya que se incorporan en muchos otros proteins, además de los de interés durante la expresión de genes recombinantes. Esto limita claramente el enfoque in vivo. La incorporación in vivo de los aminoácidos que son tóxicos o que tienen fuerte influencia en la estructura de la proteína sigue siendo particularmente difícil. Sin embargo, estas moléculas se encuentran entre los más prometedores para diseñar proteínas con funciones extraordinarias.
Un ejemplo es el,, de origen natural L-canavanina noncanonical tóxico (Can), un análogo de la L-arginina (Arg). Afecta y bloques Arg caminos de reacción reguladoras y catalíticas, y su presencia en la célula viva puede conducir a la muerte inmediata 3,21-23. Su incorporación en proteínas en las posiciones de arginina puede reducir la estabilidad de la proteína 21-23. Debido a la toxicidad resultante, la expresión de canavanina que contiene proteínas en Escherichia coli (E. coli) y otros huéspedes de expresión común sigue siendo un reto. Por estas razones, completa in vivo iNCORPORACIÓN de Can en todas las posiciones Arg apropiadamente se ha confirmado una sola vez 24, utilizando un sistema de producción de una sola proteína elaborada. Sin embargo, Can ha sido propuesta como un agente anti-cáncer de 25 a 27, y como un estimulador para las enfermedades autoinmunes en humanos 28. Además, es objeto de diversos estudios sobre su anti-metabólico, antibacterianos, antifúngicos y antivirales 25. Estas propiedades plantean una demanda de eficiente y fácil de llevar a cabo los métodos de expresar Puede que contiene proteínas de farmacéuticos, médicos y estudios funcionales.
Aunque muchos de los problemas que están conectados a la producción in vivo pueden ser eludidas mediante sistemas de expresión de células libres, en los enfoques in vitro para residuos específicos solamente han sido poco explorado. Se ha informado de la incorporación de residuos específicos libre de células de un análogo de L-triptófano 29 y 30 NCAA múltiple. Estos métodos se basan en la altamente efficient T7 RNA polimerasa. La ARN polimerasa de T7 implica la transcripción del bacteriófago similar, reduciendo así la funcionalidad genética en comparación con la transcripción endógena.
La incorporación de residuos específica completa de Can en una proteína modelo en todas las posiciones Arg se informó recientemente de 31, utilizando un sistema de expresión libre de células 32. Una ligera modificación del mismo sistema permitió la incorporación específica de sitio de diferentes análogos de pyrrolysine en un modelo de proteína a través de la supresión codón de parada 33. El sistema 31 de células libres de empleado - 33 se basa en un todo E. sistema de transcripción-traducción coli. Sin embargo, se permite la expresión de proteínas tan eficientemente como en los sistemas de bacteriófagos de corriente (0,5 a 1 mg / ml de proteína recombinante) 32, al tiempo que conserva gran parte de la modularidad de transcripción-traducción original.
En este trabajo, un protocolo detallado se proporciona en la forma en la resid-ue la incorporación específica de NCAA se puede realizar, utilizando todo esto E. sistema libre de células coli 32. Además, se proponen nuevas medidas para preparar las proteínas expresadas para una evaluación apropiada a través de espectroscopia de masas HPLC-ESI. Para expandir las propiedades de este sistema libre de células, este trabajo no sólo se refiere a la incorporación de Can publicada el 31 sino que también presenta nuevos datos relacionados con la no canónica de L-lisina análogo L-hidroxi-lisina.
El siguiente protocolo para la incorporación de residuos específicos de la NCAA es una adaptación de un protocolo publicado recientemente en JoVe 34. Este último protocolo describe cómo llevar a cabo la expresión libre de células altamente eficiente con aminoácidos estándar. Además, se presenta la preparación del extracto libre de células en bruto, la solución de aminoácidos, la solución de energía stock y la memoria intermedia de energía usada en este enfoque. El siguiente protocolo se centra en pasos modificados en comparación con el anterior protocolo con el fin de permitir la incorporación de residuos específicos de la NCAA. Se recomiendan pipetas calibradas, puntas de pipeta bajo vinculante y tubos de microcentrífuga para la preparación. En lo que sigue, se usan las abreviaturas de la IUPAC para los aminoácidos.
Protocolo
Precaución Por favor, consulte a todas las hojas de datos de seguridad de materiales pertinentes (MSDS) antes de usar. Varios de los productos químicos utilizados son tóxicos agudos. Equipo de protección personal que se requiere (protector de ojos, máscara contra el polvo, guantes, bata de laboratorio, pantalones largos, cerrados-dedo del pie zapatos), así como trabajar en una campana de humos.
1. Amino Acid Solution Preparación
- La preparación de la solución de la NCAA (168 mM)
NOTA: La preparación de la solución madre de la NCAA se describió para el análogo Arg CAN como un ejemplo. De acuerdo con ello adaptar los valores de otra NCAA.- Colocar un tubo de 1,5 ml de reacción sobre una microbalanza. Pesar 46,1 mg de Can dentro del tubo de reacción para la preparación de 1 ml de una solución 168 mM. Use un microespátula estéril. Para una mezcla racémica de la NCAA, el doble de la concentración de la solución madre.
- Añadir 977 l de ddH 2 O. estéril Minuciosamente agitar hasta Can i esdisolución n completa.
NOTA: Para un volumen total de la solución de 1 ml, el volumen físico del aminoácido disuelto tiene que ser compensado. Para cualquiera de los aminoácidos, la estimación de la mitad de la masa sólida en mg como el aumento de volumen correspondiente en l (100 mg sólido tendrá un volumen de 50 l de la solución) 35. La mayoría de los aminoácidos se pueden disolver a esta concentración. Si no, reducir la concentración hasta la disolución completa. - Utilizar directamente la solución madre de la NCAA para la preparación de las soluciones de aminoácidos en la sección 1.2 o el flash congela en nitrógeno líquido y almacenarlo a -20 ° C. PRECAUCIÓN! Por razones de seguridad, usar un protector de ojos y crio-guantes para protegerse del contacto con líquidos salpicaduras de nitrógeno.
- Preparación de las soluciones de aminoácidos
NOTA: Para la preparación de las soluciones de aminoácidos, utilice la toma de muestras de aminoácido que proporciona los isómeros L de los 20 Caas en stock separadasoluciones (1,5 ml, tamponado con HEPES / KOH, <0,1% NaN 3, pH 7,5), cada uno a una concentración de 168 mM, a excepción de L-leucina (140 mM). Para una preparación casera de estas soluciones madre (tamponada con KOH), siga este protocolo 35.- Descongelar las soluciones madre de las 20 Caas (muestreador de aminoácidos o preparado de acuerdo con 35) y de la NCAA (preparado en el apartado 1.1) a TA.
- Después de la descongelación, con frecuencia las soluciones madre vórtice para volver a disolver cualquier aminoácido precipitados. Como algunos aminoácidos son más difíciles de disolver, se incuban en un bloque de calentamiento a 37 ° C hasta disolución completa. Cys no puede disolverse completamente. Coloque todos los aminoácidos en el hielo, a excepción de Asn, Phe y Cys - guardar éstos a temperatura ambiente para evitar la precipitación.
- Utilice los siguientes valores para utilizar una séptima parte del kit completo.
NOTA: Escala hacia abajo apropiada para trabajar con volúmenes más pequeños y para salvar partes del kit para experimentos adicionales. Escala para Incorporation de NCAA en proteínas modelo a gran escala. Para evitar la descongelación frecuente, que es probable que reducir la estabilidad de los aminoácidos, alicuotar las soluciones madre de aminoácidos individuales en volúmenes de 200 l. Este volumen alícuota de 200 l y los volúmenes alícuotas utilizadas en el paso 1.2.4.1 cuenta las pérdidas debidas a la pipeta. - En primer lugar, preparar una solución de mezcla patrón de aminoácidos que se divide en el paso 1.2.4.3 para finalizar la preparación de 3 soluciones de aminoácidos diferente compuestas (secciones 1.2.5 - 1.2.7). En estas soluciones, se concentran todos los aminoácidos a 6 mM, a excepción de Leu (5 mM).
- Transferencia de 1,4 ml de ddH 2 O estéril en un tubo de centrífuga de 15 ml. Ponerlo en hielo. Añadir 175 l de cada solución de aminoácidos de valores. Añadir una tras otra, a excepción de la solución madre de la CAA (por ejemplo, Arg) para ser sustituido por la NCAA (por ejemplo, CAN). Agite bien en vórtex después de cada adición y poner la solución de nuevo en hielo.
NOTA:Leu es a 5 mM en las 3 soluciones de aminoácidos diferente compuesta, en comparación con 6 mM para los otros aminoácidos. La concentración reducida no reduce la eficacia de la expresión. La ampliación de hasta 6 mm es adecuado también. - Transferencia de las soluciones de aminoácidos de acciones en el siguiente orden para evitar la precipitación: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, y Cys. Recuerde no añadir la solución madre de la CAA (por ejemplo, Arg), que es análoga a la NCAA (por ejemplo, CAN). Por último, a fondo vórtice. Se incuba a 37 ° C hasta que la solución sea lo más claro posible.
- Dividir esta solución maestro de la mezcla de aminoácidos en tres volúmenes iguales de 1,35 ml. Transferir cada uno de los volúmenes divididos en 1,5 ml tubos de reacción. Mantenerlos en hielo.
- Transferencia de 1,4 ml de ddH 2 O estéril en un tubo de centrífuga de 15 ml. Ponerlo en hielo. Añadir 175 l de cada solución de aminoácidos de valores. Añadir una tras otra, a excepción de la solución madre de la CAA (por ejemplo, Arg) para ser sustituido por la NCAA (por ejemplo, CAN). Agite bien en vórtex después de cada adición y poner la solución de nuevo en hielo.
- Preparar una solución de aminoácidos que consiste en los 20 Caas a una concentración de 6 mM de cada uno, a excepción de Leu que es de 5 mM. Para el primer volumen of 1,35 ml, como resultado de la escisión en el paso 1.2.4.3, se añaden 50 l de la solución 168 mM de la CAA (por ejemplo, Arg), que es análoga a la NCAA (por ejemplo, CAN). Fondo de vórtice.
- Poner de nuevo en hielo. Alícuota de esta solución 1,4 ml en volúmenes de 16 l en tubos de reacción. Tenga en cuenta que este volumen de la solución de aproximadamente 85 conduce a alícuotas. Etiquetar estas alícuotas "+ CAA" (por ejemplo, + Arg).
- Flash congelar las alícuotas en nitrógeno líquido y se almacena a -80 ° C. PRECAUCIÓN! Para mayor seguridad, use un protector de ojos y crio-guantes para protegerse de las salpicaduras de nitrógeno líquido.
- Preparar una solución de aminoácidos que se compone de 19 Caas a excepción de la CAA (por ejemplo, Arg), que es análoga a la NCAA (por ejemplo, CAN). Añadir cada aminoácido a una concentración de 6 mM, a excepción de Leu (5 mM).
- Añadir 50 l de ddH estéril 2 O al segundo volumen de 1,35 ml, como resultado de la escisiónen el paso 1.2.4.3. Agite bien en vórtex y volver a poner en hielo. Alícuota de esta solución 1,4 ml en volúmenes de 16 l en tubos de reacción. Tenga en cuenta que este volumen de la solución de aproximadamente 85 conduce a alícuotas. Etiquetar estas alícuotas "- Caa" (por ejemplo, - Arg).
- Flash congelar las alícuotas en nitrógeno líquido y se almacena a -80 ° C. PRECAUCIÓN! Para mayor seguridad, use un protector de ojos y crio-guantes para protegerse de las salpicaduras de nitrógeno.
- Preparar una mezcla de aminoácidos que contiene 19 CAA y la NCAA (por ejemplo, Can) que sustituye al canónica (por ejemplo, Arg). Añadir cada aminoácido a una concentración de 6 mM, a excepción de Leu (5 mM). Para el último volumen de 1,35 ml, como resultado de la escisión en el paso 1.2.4.3, se añaden 50 l de la solución madre de 168 mM de la NCAA (por ejemplo, CAN). Etiquetarlo "+ NCAA" (por ejemplo, + CAN). Agite bien en vórtex y volver a poner en hielo.
- Alícuota de esta solución 1,4 ml en volúmenes de 16 & #181; l en tubos de reacción. Tenga en cuenta que este volumen de la solución de aproximadamente 85 conduce a alícuotas. Etiquetar estas alícuotas "+ NCAA" (por ejemplo, + CAN).
- Flash congelar las alícuotas en nitrógeno líquido y se almacena a -80 ° C. PRECAUCIÓN! Para mayor seguridad, use un protector de ojos y crio-guantes para protegerse de las salpicaduras de nitrógeno.
NOTA: Los 16 volúmenes alícuotas l utilizados en los pasos 1.2.5.1, 1.2.6.1 y 1.2.7.1 son ligeramente mayores que las necesarias para tener en cuenta las pérdidas por pipeteo.
2. Energía tampón de preparación de
NOTA: Cada lote de extracto crudo es único y requiere concentraciones de Mg y K-glutamato 34 optimizado. El volumen de la alícuota de extracto bruto depende de la concentración de proteínas 34. Utilice la plantilla de cálculo previsto (Supplemental material 1) para diferentes valores. Encuentra más instrucciones en el Adicional de materiales leyenda de la figura 1, se explIning cómo emplear esta plantilla.
- Preparar y almacenar a -80 ° C, la solución 14x energía y alícuotas de extracto crudo de acuerdo con el protocolo modificado 34. Calibrar el extracto bruto dependiendo de las concentraciones de Mg y K-glutamato para la eficiencia de expresión optimizada 34.
NOTA: La composición final de la solución de energía 14x es: mM HEPES 700 (pH 8), ATP mM 21, GTP mM 21, CTP mM 12.6, UTP 12,6 mM, 2,8 mg / ml tRNA, CoA 3,64 mM, NAD 4,62 mM, 10,5 cAMP mM, 0,95 mM de ácido folínico, espermidina 14 mM, 420 mM de 3-PGA. - Descongelar en el hielo la solución 100 mM Mg-glutamato de valores, solución de 3 M K-glutamato, 14x solución de energía y el 40% de PEG-8000 para preparar la mezcla maestra. Mantenerlos en hielo.
- Mezclar 9,18 l de solución madre 100 mM Mg-glutamato, 3,06 l de solución madre 3 M K-glutamato, 21,86 l de solución de energía 14x, 15,3 l de 40% de PEG-8000 y 1,6 l estéril ddH2O en un tubo de reacción. Vórtice a fondo este mástiler la mezcla después de cada adición, y mantener en hielo.
- Alícuota de la mezcla maestra (51 l) en volúmenes de 16 l (3 alícuotas) en los tubos de reacción. vórtice la mezcla maestra con frecuencia durante las alícuotas. Flash congelar las alícuotas en nitrógeno líquido.
NOTA: El volumen de la alícuota de 16 l, así como el volumen de mezcla maestra son un poco mayores que las necesarias para tener en cuenta las pérdidas por pipeteo. - Use un colador para recoger los tubos de protección de energía. Guarde los tubos a -80 ° C. PRECAUCIÓN! Utilice un protector de ojos y crio-guantes para protegerse de las salpicaduras de nitrógeno.
3. Preparación y Ejecución de las reacciones libres de células para la incorporación de residuos específicos de la NCAA
- En primer lugar, preparar la solución de ADN del vector en ddH2O
- Para la expresión de proteínas altamente eficiente, utiliza el vector de expresión pbest-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonar el gen que codifica para la proteína modelo en este vector 36,37 </ Sup>.
NOTA: También puede utilizar otros promotores que son reconocidos por σ 70 también, pero tenga en cuenta que la eficacia de expresión puede ser reducida. - Transformar 37,38 vector en E. coli cepa KL 740 32 (Yale CGCS #: 4382), purificar el ADN amplificado vector de 37,39 y cuantificar la concentración de la solución de ADN 40-42. Almacenar la solución de ADN a -20 ° C o directamente utilizarlo para la preparación reacción de la pila de pago (pasos 3.4.1, 3.4.2 y 3.4.3).
- Para la expresión de proteínas altamente eficiente, utiliza el vector de expresión pbest-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonar el gen que codifica para la proteína modelo en este vector 36,37 </ Sup>.
- Calibre la eficacia de la expresión libre de células dependiendo de la concentración vector constructo usado de acuerdo con el protocolo modificado 34. Usar la concentración óptima que conduce a rendimientos más altos de proteínas para la preparación de reacción libre de células (pasos 3.4.1, 3.4.2 y 3.4.3).
NOTA: La preparación de las reacciones libres de células se ejemplifica utilizando una nM solución de ADN vector stock 90 que conduce a una concentración vector final de 10 nM en el libre de células-reacción y sigue los valores óptimos anteriores de Mg y K-glutamato, así como el volumen de extracto de alícuota. Utilice la plantilla de cálculo para diferentes valores. - Descongelar en hielo 3 partes alícuotas de extracto crudo de cada uno de 30 volumen l (preparado de acuerdo con el protocolo modificado 34), 1 amino solución de ácido alícuota marcado "+ CAA" (por ejemplo, + Arg), 1 amino solución de ácido alícuota etiqueta "- CAA" (por ejemplo, , - Arg) y 1 amino solución de ácido alícuota del signo "+ NCAA" (por ejemplo, + CAN) (preparado en las secciones 1.2.5 - 1.2.7), 3 tampones de energía alícuotas (preparado en el apartado 2) y la solución de ADN del vector ( preparado en el apartado 3.1).
NOTA: El extracto crudo es ligeramente viscosa y que puede contener burbujas de aire. Eliminar las burbujas de aire por centrifugación a 10.000 xg durante 30 segundos a 4 ° C. Ponga alícuotas de extracto crudo de nuevo en hielo. - Preparar 3 reacciones libres de células composición diferente (cada 90 l de volumen final) mediante la mezcla de extracto crudo (33,33%), la energíatampón (16,67%), 1 de los 3 alícuotas diferente compuestas de aminoácidos de solución (16,67%) y solución de ADN vector. Opcionalmente, añadir más biomoléculas (ADN, proteínas, tRNA, etc.), pero apropiadamente reducir el volumen de ddH 2 O.
- Preparar la reacción libre de células de referencia (90 l) que expresan la proteína modelo sin modificar.
- Añadir los 15 l de tampón de energía, 15 l de la parte alícuota de solución de aminoácidos del signo "+ CAA" (por ejemplo, + Arg), 10 l de 90 solución de ADN del vector nM y 20 l de ddH 2 O estéril a los 30 l de crudo extraer. Mezclar pipeteando arriba y abajo y suavemente vortex después de la adición de cada ingrediente.
- Alícuota de los 90 l de reacción libre de células en 15 volúmenes iguales de 6 l. Transferir cada uno de los 15 volúmenes en un tubo de reacción separado. Cierre los tubos y volver a ponerlos en hielo. Etiquetar todos los tubos de reacción como "CFR (+ CAA)" (por ejemplo, CFR (+ Arg)).
- Preparar la reacción negativa de control libre de células (90 l), donde ni el CAA (por ejemplo, Arg), ni el análogo no canónica (por ejemplo, CAN) se añaden.
- Añadir los 15 l de tampón de energía, 15 l de la parte alícuota de solución de aminoácidos etiquetados "- Caa" (por ejemplo, - Arg), 10 l de 90 solución de ADN del vector nM y 20 l de ddH 2 O estéril a los 30 l de crudo extraer. Mezclar pipeteando arriba y abajo y suavemente vortex después de la adición de cada ingrediente.
- Alícuota de los 90 l de reacción libre de células en 15 volúmenes iguales de 6 l. Transferir cada uno de los 15 volúmenes en un tubo de reacción separado. Cierre los tubos y volver a ponerlos en hielo. Etiquetar todos los tubos de reacción como "CFR (-cAA)" (por ejemplo, CFR (Arg)).
- Preparar la reacción libre de células (90 l) que se supone que el residuo específicamente incorporar el NCAA (por ejemplo, Can) en el modelo de proteína expresada.
- Añadir la 15l de tampón de energía, 15 l de la parte alícuota de solución de aminoácidos del signo "+ NCAA" (por ejemplo, + CAN), 10 l de solución de ADN 90 nM y 20 l de ddH 2 O estéril a los 30 l de extracto crudo. Mezclar pipeteando arriba y abajo y suavemente vortex después de la adición de cada ingrediente.
NOTA: La eficacia de la expresión puede ser reducida en comparación con la expresión con aminoácidos estándar. Si es necesario, simplemente ampliar el volumen de reacción libre de células. - Alícuota de los 90 l de reacción libre de células en 15 volúmenes iguales de 6 l. Transferir cada uno de los 15 volúmenes en un tubo de reacción separado. Cierre los tubos y volver a ponerlos en hielo. Para el aumento de los volúmenes de reacción, en consecuencia alícuota en otros volúmenes de 6 l. Etiquetar todos los tubos de reacción como "CFR (+ NCAA)" (por ejemplo, CFR (+ Can)).
- Añadir la 15l de tampón de energía, 15 l de la parte alícuota de solución de aminoácidos del signo "+ NCAA" (por ejemplo, + CAN), 10 l de solución de ADN 90 nM y 20 l de ddH 2 O estéril a los 30 l de extracto crudo. Mezclar pipeteando arriba y abajo y suavemente vortex después de la adición de cada ingrediente.
- Preparar la reacción libre de células de referencia (90 l) que expresan la proteína modelo sin modificar.
- Incubar todos los tubos a 29 ° CO / N.
NOTA: Sólo pequeños volúmenes de reacción adecuadas de oxígeno permiten diffusion en la reacción que es crucial para la expresión de proteínas altamente eficiente. Los volúmenes de reacción superiores a 10 l requieren oxigenación activa a través de la agitación 34. Para grandes volúmenes, dividir la reacción en volúmenes más pequeños de 15 l. - Después de la expresión libre de células, piscina todo idénticamente compuesto reacciones libres de células divididas de 6 l. En primer lugar, la piscina todas las 15 reacciones libres de células divididas de 6 l etiqueta "CFR (+ CAA)" (por ejemplo, CFR (+ Arg)). Entonces, la piscina todas las 15 reacciones libres de células divididas de 6 l etiqueta "CFR (-cAA)" (por ejemplo, CFR (Arg)). Por último, la piscina todas las 15 reacciones libres de células divididas de 6 l etiqueta "CFR (+ NCAA)" (por ejemplo, CFR (+ Can)). Para el aumento de los volúmenes de reacción, por lo tanto la piscina más volúmenes de 6 l.
- Comprobar el nivel de expresión de la proteína modelo en los tres agrupada, de otro compuesto reacciones libres de células mediante la realización de desnaturalización SDS-PAGE 43,44 (sección 4.1).
NOTA: Utilice esta metho d para una evaluación preliminar del experimento incorporación. - Preparar las proteínas expresadas modelo libres de células para un análisis apropiado mediante espectroscopia de masas HPLC-ESI (sección 4.4). En primer lugar, se purifican 45 de ellos (sección 4.2). Por último, el intercambio de la memoria intermedia (sección 4.3) para evitar el alto ruido de fondo durante la espectroscopia de masas.
NOTA: Las secciones 3.4.1, 3.4.2 y 3.4.3 de plomo a condiciones de reacción libres de células típicas 34: 8,9 - proteína 9,9 mg / ml (a partir de extracto crudo), 4.5 a 10.5 mM Mg-glutamato, 40-160 mM K-glutamato, 1 mM de cada aminoácido excepto leucina, leucina 0,83 mM, HEPES 50 mM,, cAMP 1,5 mM ATP mM y GTP, CTP 0,9 mM y UTP, 0,2 mg / ml tRNA, CoA mM 0,26, NAD 0,33 mM 0,75 , 0,068 mM de ácido folínico, espermidina 1 mM, 30 mM 3-PGA, 2% de PEG-8000 y 10 nM pbest-OR2-OR1-Pr-UTR1-gene_of_model_protein-T500. Si se desea, un procedimiento diferente de la preparación de reacción libre de células puede llevarse a cabo que conduce a las condiciones de reacción anteriores.
- SDS-PAGE de las reacciones libres de células
NOTA: Realice desnaturalizante SDS-PAGE para un análisis rápido y preliminar de las proteínas que se expresan sin ninguna purificación adicional o extracción, y ejecutar los siguientes pasos.- Descongelar el estándar de proteína en hielo. Asegúrese de que consiste en proteínas con pesos moleculares similares a la proteína modelo expresado por su localización en el gel (paso 4.1.13).
NOTA: Aquí, el estándar utilizado proporciona proteínas en un amplio intervalo de pesos moleculares (miosina: 212 kDa, de unión a maltosa-proteína-β-galactosidasa: 158 kDa, β-galactosidasa: 116 kDa, fosforilasa b: 97 kDa, albúmina de suero : 66 kDa, deshidrogenasa glutámico: 56 kDa, maltosa-proteína de unión: 43 kDa, la tiorredoxina reductasa: 35 kDa, triosafosfato isomerasa: 27 kDa, inhibidor de tripsina: 20 kDa, LYsozyme: 14 kDa, aprotinina: 7 kDa, la insulina A: 3 kDa, cadena B: 2 kDa). - Como las reacciones libres de células son ligeramente viscoso debido a la alta concentración de proteínas, se diluye con ddH 2 O estéril 5-10 veces antes de SDS-PAGE para asegurar la migración de gel apropiado y para evitar la saturación después de la tinción. Con el fin de no perder demasiado de la muestra, diluir 1 l de reacción libre de células en 4 l de ddH 2 O. estéril Preparar la dilución en tubos de reacción, a fondo vórtice y poco girar el líquido hacia abajo con un mini centrifugar durante 2 - 3 segundos a 2.000 x g.
- Añadir 5 l de tampón de carga 2x a 5 l de reacción libre de células diluida. Agite bien en vórtex y centrifugar con una mini centrifugar durante 2 - 3 segundos a 2.000 x g.
NOTA: Aquí, después de la adición, las biomoléculas se disuelven en 62,5 mM Tris / Cl -, 10% de glicerol, 2% SDS, y 0,0025% de azul de bromofenol a pH 6,8, una composición típica. El uso de otros colorantes de carga que llevan a ligeramente diferente dilución conditions pueden ser adecuadas también. - A fondo el vórtex con la estándar de la proteína y la transferencia de 15 l de la misma en un tubo de reacción.
NOTA: Dependiendo del tamaño del gel y para otros estándares de proteína, el volumen recomendado puede diferir de lo anterior. - Colocar los tubos de reacción en un bloque de calentamiento. Compruebe si las tapas de los tubos están bien cerradas. Se calienta a 95-100 ° C durante 3-5 min. Hacer esto para desnaturalizar las proteínas y para permitir SDS-envoltura alrededor del esqueleto de la proteína.
NOTA: Algunos patrones de proteínas no deben calentarse, consulte las instrucciones del proveedor. - Mientras tanto, la transferencia de la gestión de amortiguación en la cámara de electroforesis en gel. El contenido de tampón de 1x es Tris 25 mM, glicina 192 mM, 0,1% de SDS a pH 8,3 (con HCl).
- Fijar el prefabricado 4 - 20% gradiente de Tris-glicina-SDS gel (10 cm x 10 cm x 1 mm) a la cámara de electroforesis. Retire el peine y enjuague los pocillos con una jeringa cargada con tampón de desarrollo.
NOTA: La concentración de gel strongly depende del tamaño y la naturaleza de la proteína modelo expresado. La concentración superior separa la E. proteínas de E. coli de extracto crudo, así como proteínas modelo de bajo peso molecular. geles autolanzamiento son adecuados también. - Retire los tubos del bloque de calefacción. Girar el líquido hacia abajo con un mini centrifugar durante 2 - 3 segundos a 2.000 x g. Poco vórtice y repetir girando hacia abajo por 2 - 3 segundos a 2.000 x g.
- La transferencia de los 15 l de estándar de proteína (24 - 48 mg de proteína) y 10 l (11 a 22 g de proteína) de cada muestra en los pocillos y empezar a electroforesis. Aquí, SDS-PAGE se realiza a 125 V y 20 - 40 mA durante aproximadamente 90 min, un protocolo típico.
- extraer con cuidado el gel de la cassette. Transferirlo a la solución de fijación (metanol al 50%, ácido acético al 10%, 40% ddH2O) durante 30 min. ¡CUIDADO! El metanol es tóxico por inhalación y en contacto con la piel. Use guantes de protección y trabajar bajo una campana de humos.
- Transferir el gel en solución de tinción (0,025% azul brillante de Coomassie G-250, 10% de ácido acético, 90% ddH 2 O). Manchar durante 60 minutos.
- Transferir el gel en solución decolorante (ácido acético 10%, 90% ddH 2 O) para 60 a 120 min.
NOTA: La fijación, tinción y la decoloración fuertemente dependen del tamaño y la naturaleza de la proteína expresada. Este protocolo se aplica a una amplia gama de proteínas de pesos moleculares más bien pequeñas 46. - Plegar el gel sobre una transparencia mate sobre un fondo blanco que genera un contraste apropiado para las bandas de proteínas teñidas.
- Descongelar el estándar de proteína en hielo. Asegúrese de que consiste en proteínas con pesos moleculares similares a la proteína modelo expresado por su localización en el gel (paso 4.1.13).
- La purificación de los 45 con cola de histidina proteínas modelo de las reacciones libres de células
NOTA: Para la purificación de proteínas, existen varios métodos que ofrecen resultados similares. Este protocolo purifica proteínas expresadas modelo libres de células que tienen una polihistidina-etiqueta C-terminal (His-tag). Se utiliza un kit de purificación adecuada para proteínas que se Expressed en los pequeños volúmenes de reacción de la expresión de proteínas libre de células. Es idéntico para la purificación de proteínas modelo nativos o modificados. Por lo tanto, se describe en general sobre la base de una reacción libre de células única.- Para un análisis espectroscópico de masas HPLC-ESI su caso, extraer y purificar las proteínas modelo de la reacción libre de células y ejecutar los siguientes pasos.
- Mezclar volúmenes iguales de 90 - 150 l de tampón de unión y Su-90 - 150 l de reacción libre de células. Pipetear arriba y hacia abajo, seguido por agitación suave.
NOTA: El uso de Su-tampón de unión se recomienda. Sin embargo, medio de reacción libre de células puede ser un material de partida, siempre y cuando la proteína modelo es soluble, el pH es entre 7,5 a 8, imidazol / Su concentración es <10 mM, la concentración de agentes reductores fuertes es <15 mM y no metal- agentes quelantes están presentes. Si el volumen de la mezcla es superior a 300 l, dividirlo en volúmenes iguales y alícuota Tesis volúmenes en diferentes rtubos eaction para las siguientes etapas de purificación. - Preparar el sistema de columna. A fondo el vórtex con la solución madre de la gel Su afinidad hasta que la resina gel se disuelva completamente. Transferir 250 l de resina de gel en la columna. Utilice una punta de pipeta de 1 ml de la resina gel viscoso. Colocar la columna en un tubo de recogida.
- Centrifugar el tubo de columna / recogida durante 5 - 10 seg a 13.000 - 15.000 x g. Asegúrese de que la resina de gel está completamente agotada. Si no es así, extender el tiempo de centrifugación por adicional de 5 - 10 seg, pero preste atención a que el gel de afinidad no se seca sobre. De acuerdo con ello extender el tiempo de centrifugación en los pasos 4.2.1.5, 4.2.1.7 y 4.2.1.9.
NOTA: La resina de gel está completamente agotada, si se vuelve rígido y sin sobrenadante se mantiene en la parte superior. - Transferencia de 150 - 300 l de tampón de reacción libre de células / His de unión a la columna. Si el volumen de la mezcla excede el volumen recomendado, dividir en varios columnas de centrifugación. Volver a suspender la resina de gel por frequent tocando y suave agitación en vórtex durante un tiempo de incubación de al menos 2 minutos. Para volúmenes mayores de 200 l, se incuba durante otros 1 - 2 min.
NOTA: El tiempo de incubación suficiente es crucial para la unión de las proteínas modelo a la resina de gel. - Centrifugar el tubo de columna / recogida durante 5 - 10 seg a 13.000 - 15.000 x g. Desechar el flujo de filtrado y coloque la columna de nuevo en el tubo de recogida.
- Añadir 250 l de Su-tampón de lavado. Volver a suspender la resina de gel de golpecitos y de vórtex suave.
- Centrifugar el tubo de columna / recogida durante 5 - 10 seg a 13.000 - 15.000 x g. Desechar el flujo de filtrado y coloque la columna de nuevo en el tubo de recogida. Repita el paso 4.2.1.6 y centrifugar de nuevo durante 5 - 10 seg a 13.000 - 15.000 x g.
- Colocar la columna en un tubo de reacción estándar 1,5 ml. Añadir 150 ml de tampón de elución y volver a suspender la resina de gel de golpecitos y de vórtex suave. Reducir el volumen a 100 l de mayor contenido de proteína purificada con modeloconcentraciones después de la elución en la siguiente etapa 4.2.1.9.
NOTA: La abundancia total de la proteína purificada modelo puede reducirse debido a la posible elución incompleta de todas las proteínas unidas a resina de gel, si se reduce el volumen de tampón de elución. - Centrifugar el tubo de columna / reacción durante 5 - 10 seg a 13.000 - 15.000 xg para diluir la proteína purificada en el tampón de elución. Si se divide antes, aunar todas las soluciones en una solución de reserva.
- Antes de ejecutar el intercambio de tampón, determinar la concentración de proteína usando métodos estándar, por ejemplo, el Ensayo de Bradford 47,48.
NOTA: Para una espectroscopia de masa apropiada HPLC-ESI (sección 4.4), utilizar concentraciones óptimas de proteínas que son normalmente de aproximadamente 0,5 mg / ml con volúmenes requeridos de 20 - 50 l. Si la concentración es inferior a 0,2 mg / ml, concentrar la solución de proteína después del intercambio de tampón utilizando un concentrador de hilado de vacío. En este caso, primero lea la sección 4.3.8 para adaptar adecuadamente el aficionado intercambioer.
- Mezclar volúmenes iguales de 90 - 150 l de tampón de unión y Su-90 - 150 l de reacción libre de células. Pipetear arriba y hacia abajo, seguido por agitación suave.
- Para un análisis espectroscópico de masas HPLC-ESI su caso, extraer y purificar las proteínas modelo de la reacción libre de células y ejecutar los siguientes pasos.
- El cambio de tampón para la espectroscopia de masas HPLC-ESI
NOTA: el intercambio de El tampón de elución His-tag para evitar el alto ruido de fondo en análisis de espectroscopia de masas debido a la alta concentración de imidazol (> 150 mM) y otras sales tales como NaH 2 PO 4 (> 300 mm) o NaCl (> 50 mM) 49. El siguiente protocolo de intercambio de tampón utiliza un sistema de columna de centrifugación de filtración en gel prehidratado. Se ejecuta de forma idéntica para proteínas modelo nativos o modificados. Por lo tanto, se describe en general sobre la base de una reacción libre de células única. Tenga en cuenta que existen otros métodos de intercambio de tampón fáciles de realizar, por ejemplo, cartuchos de mini-diálisis.- Preparar 100 ml de tampón de almacenamiento de proteínas (50 mM Tris-Cl pH 8, NaCl 100 mM, y 10% de glicerol). Pesar en un autoclave de 100 ml botella 605,7 mg Tris-Base, 584,4 mg de NaCl y añadir 10 ml de glicerol. Llenar hasta aproximadamente 80 ml y ajustar el valor de pH a 8 por unaNaOH dding. De manera constante el valor de pH con un medidor de pH. Por último, llenar hasta la marca de 100 ml.
NOTA: Utilice este tampón para estabilizar una amplia gama de proteínas modelo y de no molestar a análisis espectroscópico de masas, siempre y cuando HPLC se lleva a cabo antes de la medición espectroscópica de masas. Sin embargo, dependiendo de la naturaleza de la proteína expresada modelo, utilizar otros tampones que podrían ser mejor adecuado. Si su proteína diana no es estable a pH 8, ajustar el pH en consecuencia. No utilizar concentraciones de glicerol más altas. - Calentar las columnas de spin de filtración en gel durante al menos 15 min a RT.
- Volver a suspender el gel prehidratado con unos golpecitos suaves o vórtex y eliminar las burbujas de aire.
- Primero quite la tapa inferior, y luego tomar la tapa superior hacia fuera. Colocar la columna en un tubo de lavado (al menos 2 ml). Transferir el tubo de la columna / lavado en la centrífuga. Cada columna posee una marca de orientación. Centrifugar a 1000 xg durante 2 minutos para eliminar el tampón de almacenamiento gel. DesctARD El flujo continuo.
NOTA: Tener en cuenta para proceder en este orden. Asegúrese de que la columna tiene la misma orientación en todas las etapas adicionales de centrifugación. - Añadir hasta 400 l del tampón de almacenamiento de proteínas. Centrifugar durante 2 min a 1.000 x g. Repita esto para cargar completamente el tampón de almacenamiento de proteínas en gel. Añadir con cuidado hasta 100 l de la solución de la proteína purificada modelo. Pipeta directamente en el centro de la cama gel.
NOTA: Los volúmenes de soluciones de proteínas purificadas, en particular de proteínas modificadas, podrían ser mayores. Dividir tales soluciones más de varias columnas de intercambio de tampón. Las proteínas deben tener pesos moleculares de más de 5 kDa para este tipo de intercambio de tampón. - Colocar la columna en un tubo de recogida y se centrifuga durante 2 min a 1.000 x g.
Nota: Este paso lleva a la dilución de la proteína modelo purificada en el tampón de almacenamiento de proteínas. Si se divide antes, aunar todas las soluciones en una solución de reserva. - º congelación de flashsolución de proteína de correo en nitrógeno líquido y se almacena a -80 ° C o directamente analizan mediante espectroscopia de masas HPLC-ESI 50 (sección 4.4). PRECAUCIÓN! Utilice un protector de ojos y crio-guantes para protegerse de las salpicaduras de nitrógeno.
- Llevar a cabo las siguientes etapas del protocolo de concentrar la solución de proteína utilizando un concentrador de vacío de hilado que se evapora el disolvente cuidadosamente 51.
NOTA: Estos pasos son necesarios si la concentración de proteína es demasiado baja para la espectroscopia de masas HPLC-ESI (<0,2 mg / ml). La concentración de proteína volumen de la solución y el modelo es aproximadamente el mismo antes y después de intercambio de tampón. El proceso de concentración se describe mediante el siguiente ejemplo: Después de la purificación His-tag, la proteína modelo se disuelve en 100 l Su tampón de elución (etapa 4.2.1.9) y tiene una concentración de 0,07 mg / ml (etapa 4.2.1.10) .- Para asegurarse de que la concentración de proteína y el volumen de la solución después de la evaporación son enmenos 0,2 mg / ml y 20 l, respectivamente, diluir, antes de intercambio de tampón, el tampón de almacenamiento de proteína preparada en la etapa 4.3.1 al menos tres veces, pero a más de cinco veces. Igualmente se evapore en el siguiente, al menos, dos tercios (66,67 l) oa lo sumo cuatro quintas partes (80 l).
NOTA: La dilución de la proteína de tampón de almacenamiento antes de que el intercambio de tampón es crucial tener en cuenta que los valores de concentración de este tampón (etapa 4.3.1) todavía se realizan a pesar de la evaporación. Después de la dilución del tampón de almacenamiento de proteínas, realice primero el intercambio de tampón (pasos 4.3.2 - 4.3.6) antes de ejecutar los siguientes pasos 4.3.8.2 - 4.3.8.4. - Tras el intercambio de tampón, poner las completas 100 l de solución de proteína modelo en un tubo abierto en el concentrador de hilado de vacío.
NOTA: La proteína modelo se disuelve en el tampón de almacenamiento de proteína diluida. Incline hacia el centro de rotación para evitar que el líquido de la pérdida durante la rotación. Asegurar que un blanco abierto del mismo volumen deH2O se coloca simétricamente para equilibrar el sistema de hilatura. - Cierre la tapa del concentrador y comenzar la rotación. Para asegurar la estabilidad de proteínas, se concentran a TA o a bajas temperaturas de hasta 30 ° C.
NOTA: El concentrador utilizado acelera automáticamente a 170 xg y establece el vacío. En el siguiente se acelera a su velocidad máxima de 240 x g. - interrumpir el proceso de concentración con frecuencia para examinar el volumen de líquido. Detener la concentración, cuando el volumen de solución que queda es de entre 20 l y 33 l.
NOTA: De acuerdo con ello adaptar los pasos 4.3.8.1 - 4.3.8.4 para otros volúmenes de solución (paso 4.2.1.9) y otras concentraciones de proteína (paso 4.2.1.10). Flash congelar la solución de proteína concentrada en nitrógeno líquido y se almacena a -80 ° C, o directamente a través de analizarlo espectroscopia de masas HPLC-ESI 50. ¡PRECAUCIÓN! Use un protector ocular y crio-guantes para protegerse de las salpicaduras de nitrógeno.
- Para asegurarse de que la concentración de proteína y el volumen de la solución después de la evaporación son enmenos 0,2 mg / ml y 20 l, respectivamente, diluir, antes de intercambio de tampón, el tampón de almacenamiento de proteína preparada en la etapa 4.3.1 al menos tres veces, pero a más de cinco veces. Igualmente se evapore en el siguiente, al menos, dos tercios (66,67 l) oa lo sumo cuatro quintas partes (80 l).
- Preparar 100 ml de tampón de almacenamiento de proteínas (50 mM Tris-Cl pH 8, NaCl 100 mM, y 10% de glicerol). Pesar en un autoclave de 100 ml botella 605,7 mg Tris-Base, 584,4 mg de NaCl y añadir 10 ml de glicerol. Llenar hasta aproximadamente 80 ml y ajustar el valor de pH a 8 por unaNaOH dding. De manera constante el valor de pH con un medidor de pH. Por último, llenar hasta la marca de 100 ml.
- HPLC-ESI análisis de espectrometría de masa 50 de proteínas modelo
- Efectuar la separación de HPLC de 5 - solución de 15 l de proteína (preparado en la sección 4.3) en una columna de fase inversa C5 (3 m, 100 x 2,1 mm) con un gradiente de 20% - 90% de acetonitrilo y 0,1% de ácido fórmico durante 25 min y posterior espectrometría de masas ESI con detección en un Analysers de masas de tiempo de vuelo en el intervalo de 300 - 3000 m / z.
NOTA: Dependiendo de la naturaleza de la proteína expresada modelo, utilizar otros disolventes o columnas que podrían ser mucho más adecuado. - Deconvolute medir los espectros de masas usando el software apropiado 52 para el cálculo de masas de carga cero.
- Efectuar la separación de HPLC de 5 - solución de 15 l de proteína (preparado en la sección 4.3) en una columna de fase inversa C5 (3 m, 100 x 2,1 mm) con un gradiente de 20% - 90% de acetonitrilo y 0,1% de ácido fórmico durante 25 min y posterior espectrometría de masas ESI con detección en un Analysers de masas de tiempo de vuelo en el intervalo de 300 - 3000 m / z.
Resultados
Este protocolo de guía a través de la incorporación de residuos específicos de células libres de la NCAA en proteínas modelo. Propone SDS-PAGE para una evaluación preliminar del experimento incorporación y nuevas medidas para preparar las proteínas modelo para un análisis espectroscópico de masas HPLC-ESI apropiado.
Aquí, se presentan resultados representativos de la incorporación de residuos específica libre de células del análogo Arg Puede, así como la Lys analógico L-hidroxi-lisina (Hyl). Las soluciones de aminoácidos diferentes, la memoria intermedia de energía, el ADN que codifica para la proteína de vector modelo y las reacciones libres de células se preparan como se ha descrito anteriormente. La reacción libre de células de referencia está provisto de la solución de aminoácidos que consiste en los 20 Caas. Para cada experimento, una reacción libre de células de control negativo se suministra con la solución de aminoácidos que carece de la analógica canónica de la NCAA en cuestión. Para cada aproach, una reacción libre de células expresa la proteína modelo en presencia de la solución de aminoácidos, en el que el CAA se sustituye por el análogo no canónica. Su-etiqueta de purificación, intercambio de tampón y el análisis de espectrometría de masas HPLC-ESI se ejecutan de acuerdo con el protocolo descrito anteriormente.
La proteína es el modelo C-terminal marcado con His deGFP 32, una versión truncada de EGFP 53. Su secuencia de aminoácidos de una letra se puede encontrar en (material suplementario 2). Esta proteína modelo contiene 6 Arg y Lys 18 posiciones, respectivamente. El vector de expresión es pbest-OR2-OR1-Pr-UTR1-deGFP-T500.
En el caso de la incorporación completa de la NCAA, se puede suponer que la reacción del control negativo no expresa deGFP, ya que uno de los 20 Caas no se encuentra. Contrariamente, deGFP debe ser detectable en los otros dos reacciones: la nativa en el referencia de reacción libre de células y la proteína modificada en la reacción libre de células que se proporciona con la NCAA.
La Figura 1A muestra la evaluación preliminar SDS-PAGE del experimento incorporación Can. La reacción libre de células de referencia tiene el nivel más alto de expresión deGFP. En la reacción libre de células que se proporciona con Can, deGFP se expresa a una concentración ligeramente inferior. No expresión deGFP puede ser detectado en el control negativo. Este resultado SDS-PAGE es una buena indicación para una incorporación exitosa de Can en el deGFP proteína diana.
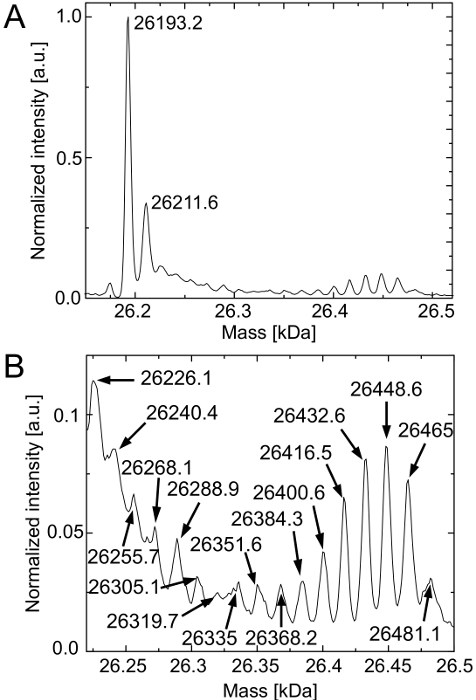
Para probar la hipótesis de la incorporación completa de Can en deGFP, ambas proteínas purificadas modelo, visualizadas en la Figura 1B, se analizó a través de espectroscopía de masas HPLC-ESI. La Figura 1C muestra el espectro de masas de las moléculas deconvoluted deGFP purificados. La masa de deconvoluted DEGF P que se expresa en la reacción libre de células de referencia es 26,192.8 Da. Para deGFP expresado en el que puede contener reacción libre de células aparece una masa de 26.202,5 Da. Las masas esperadas para la deGFP6Arg nativa y la deGFP6Can modificado con Arg ser totalmente reemplazados por Can son 26.193 Da y 26.204 Da, respectivamente. La diferencia de masa de 1,5 Da para deGFP6Can está dentro del error de la deconvolución del espectro. Por lo tanto, se confirma la plena incorporación de lata en deGFP en todas las posiciones 6 Arg.
Los dos picos de intensidad reducida corresponden a la deGFP6Arg nativos y modificados deGFP6Can que no alcanzan su madurez fluoróforo. El fluoróforo se genera autocatalíticamente por la eliminación de una molécula de H 2 O, seguido de oxidación. Esto conduce a un aumento de la masa de 20 Da si este proceso no continúa.
pg "/>
Figura 1. Evaluación de SDS-PAGE del experimento puede incorporación y espectroscopia de masas HPLC-ESI de las moléculas deGFP expresadas y purificadas libres de células. (A) Evaluación preliminar del experimento puede incorporación utilizando SDS-PAGE. De izquierda a derecha: estándar de proteínas, reacción libre de células de referencia, control negativo y la reacción libre de células proporcionando puede en lugar de Arg. (B) SDS-PAGE después de Su-etiqueta de purificación y de intercambio de tampón de las moléculas expresadas deGFP. De izquierda a derecha: estándar de proteínas, deGFP purificada a partir de la reacción de referencia, purificado deGFP de la reacción que contiene Can. (C) La confirmación de la plena incorporación de Can mediante espectroscopía de masas HPLC-ESI. Las masas esperadas del deGFP nativa y la deGFP modificado con Arg totalmente reemplazados por Can son 26.193 Da y 26.204 Da, respectivamente. Cada espectro se normalizó a su más alta intensidad (recuento). posiciones de los picos se indicanen Da. Para fines de visualización, los carriles de gel se extraen de las imágenes de gel, unidos entre sí, se convierten en formato de escala de grises, el tamaño está optimizado y el contraste, así como el brillo se mejoran. Líneas de gel originales se presentan en el material suplementario 3. Haga clic aquí para ver una versión más grande de esta figura.
La Figura 2A muestra la evaluación preliminar SDS-PAGE del experimento incorporación Hyl. En la reacción libre de células que se proporciona con Hyl, deGFP se expresa. Contrariamente a la primera experimento, una banda deGFP débil se puede observar en la reacción de control negativo. Esto podría ser debido a los residuos de Lys en las reacciones libres de células. Esto permite una expresión deGFP débil en la reacción de control negativo, donde se añaden ni Lys ni Hyl.
Para H Espectroscopía de masas ESI-PLC, las moléculas deGFP de la reacción libre de células provistas de Hyl se purifican y se intercambia el tampón (Figura 2B).
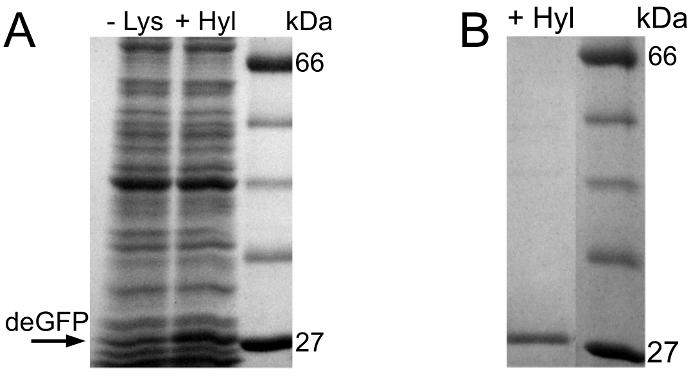
Figura 2. Evaluación de SDS-PAGE del experimento de incorporación Hyl (A) De izquierda a derecha:. Reacción libre de células de control negativo, la reacción libre de células que contiene Hyl y el nivel de proteínas. (B) SDS-PAGE después de Su-etiqueta de purificación y de intercambio de tampón de las moléculas expresadas deGFP. De izquierda a derecha: deGFP purificada de la Hyl reacción y que contiene proteína estándar. Para fines de visualización, los carriles de gel se extraen de las imágenes de gel, unidos entre sí, se convierten en formato de escala de grises, el tamaño está optimizado y el contraste, así como el brillo se mejoran. líneas de gel originales se presentan en el material complementario 3.f = "https://www.jove.com/files/ftp_upload/54273/54273fig2large.jpg" target = "_ blank"> Haga clic aquí para ver una versión más grande de esta figura.
La Figura 3 muestra el espectro de masas de moléculas deconvoluted deGFP purificados. Figura 3A confirma la hipótesis de que ya están presentes residuos de Lys en las reacciones libres de células. El pico predominante del espectro corresponde a la nativa (masa esperada: 26193 Da) deGFP. Una vez más, las moléculas de deGFP de un 20 Da mayor masa que no desarrollan su fluoróforo pueden ser detectados. Los residuos de Lys se cargan preferentemente en el tRNA Lys por lisil-tRNA-sintetasa que conduce a un alto nivel de expresión de los deGFP18Lys nativas.
La diferencia de masa entre Hyl y Lys es 16 Da. Debido a la presencia de Hyl que está en competencia a los residuos de Lys se generan todas las posibles especies deGFP (deGFP18Hyl,deGFP17Hly + 1Lys, ..., deGFP16Hyl + 2Lys) (Figura 3B). Es cierto, el pico de deGFP1Hyl + 17Lys se solapa con el pico de la deGFP nativo que no produjo su fluoróforo (Figura 3A) y la masa de algunos picos difiere más de 2 Da de la masa esperada. Estas diferencias de masa se pueden atribuir a la alta ruido debido a pequeñas cantidades de las especies deGFP. Sin embargo, Hyl generalmente se incorpora en el sistema libre de células. Otras mejoras tienen que hacer para abolir residuos de Lys en las reacciones libres de células.
. Figura 3. espectroscopia HPLC-ESI masa de las moléculas deGFP purificadas de la reacción libre de células que contiene Hyl (A) Los deGFP18Lys nativos se detecta predominantemente (masas esperados: con fluoróforo 26.193 Da, sin fluoróforo madura 26.213 Da). (B)Magnificación revela la existencia de todas las posibles especies deGFP (deGFP18Hyl, deGFP17Hly + 1Lys, deGFP16Hyl + 2Lys, ..., deGFP1Hyl + 17Lys). Sus masas esperados son 26.193 Da + N x 16 Da (N = 1, ..., 18). El espectro se normalizó a su más alta intensidad (recuento). Posiciones de los picos se indican en Da. Por favor, haga clic aquí para ver una versión más grande de esta figura.

1. Material de la plantilla suplementaria de cálculo. En la sección 2, la preparación de la mezcla maestra de amortiguación de energía se ejemplifica usando alícuotas de extracto crudo de 30 l y óptimo de Mg y K-concentraciones de glutamato de respectivamente 3 y 30 mm. En este ejemplo se lleva a un volumen de mezcla maestra que produce 3 alícuotas de amortiguamiento de energía. en secla 3, la preparación de la reacción libre de células se ejemplifica el uso de una solución 90 nM de ADN social que lleva a una concentración de ADN del vector óptimo de 10 nM en la reacción libre de células. Por favor, haga clic aquí para descargar este archivo.
Para una trazabilidad adecuada, el uso de la plantilla de cálculo se ejemplifica mediante la inserción de estos valores típicos que difieren del ejemplo anterior: crudo de volumen de extracto: 28 l, óptima Mg-glutamato: 2 mm, óptimo K-glutamato: 40 mM, número de alícuotas de amortiguación deseadas: 100, concentración óptima de vectores en células libres de reacción: 8 nM, el ADN del vector stock solución: 150 nm.
En primer lugar entrar en 28 l de volumen como extracto crudo en el campo de naranja de la primera sección de la plantilla. A continuación, entrar en la segunda sección de la plantilla de 2 mm de unad 40 mM como óptima de Mg y concentraciones de K-glutamato en los campos de color naranja. Teniendo en cuenta el Mg óptima y K-concentraciones, la composición de un tampón de energía 15 l, así como un correspondiente, ampliado alícuota 16 l se calcula. A continuación, en consecuencia introducir 100 como número deseado de alícuotas de amortiguación de energía (16 l). La plantilla se adapta a los volúmenes de los diferentes componentes del tampón para la mezcla maestra 1700 l como sigue: 204 l de solución madre 100 mM Mg-glutamato, 136 l de solución madre 3 M K-glutamato, 728.73 l de solución de energía 14x, 510 l de 40% de PEG-8000 y 121.27 l estéril ddH2O Por último, en la sección tercera plantilla, introduzca 8 nM y 150 nM como concentración óptima vector en la reacción libre de células y, respectivamente, el ADN vector concentración de la solución de stock. La plantilla se adapta a los volúmenes de los diferentes componentes que deben añadirse a los 28 l de extracto crudo para finalizar la preparación de un 90 lreacción libre de células como sigue: 15 l de tampón de energía, 15 l de una de las 3 soluciones de aminoácidos diferente compuestas, 4,80 l de solución de ADN del vector (150 nM) y 27.20 l de ddH estéril 2 O.
El material suplementario 2. Una carta secuencia de aminoácidos de la proteína modelo deGFP. Esta proteína modelo contiene 6 Arg y Lys 18 posiciones. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Material suplementario 3. longitud y carriles de gel no modificados que corresponden a las imágenes de gel presentado completa la Figura 1 y 2. Las bandas de gel se presenta en cada uno s individuales ubfigure se extraen de la misma en gel de poliacrilamida SDS. En la figura 1 y 2 estos carriles fueron unidas entre sí a efectos de presentación. Los pesos moleculares de las bandas de proteína estándar se indican al lado de las figuras. (A.1) carriles de gel Uncropped de la Figura 1A. De izquierda a derecha: estándar de proteínas, reacción libre de células de referencia, control negativo y la reacción libre de células proporcionando puede en lugar de Arg. (A.2) Uncropped líneas de gel de la figura 1B. De izquierda a derecha: estándar de proteínas, deGFP purificada a partir de la reacción de referencia, purificado deGFP de la reacción que contiene Can. (B.1) carriles de gel Uncropped de la Figura 2A. De izquierda a derecha: la reacción libre de células de control negativo, la reacción libre de células que contiene Hyl y el nivel de proteínas. (B.2) Uncropped líneas de gel de la Figura 2B. De izquierda a derecha: deGFP purificada de la Hyl reacción y que contiene proteína estándar.d / 54273 / 54273supfig3large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Una herramienta fácil de usar sistema de expresión libre de células como una estrategia viable hasta el residuo específicamente incorporar NCAA en proteínas, se presentó. Con este fin, el extracto crudo se complementa con la codificación de ADN del vector para la proteína de interés, la memoria intermedia de energía y los aminoácidos correspondientes. Tenga en cuenta que el volumen de la alícuota extracto crudo depende de la concentración de proteína de extracto bruto 34. La eficacia de la expresión libre de células se optimiza en función de la concentración de la construcción de ADN vector. Los volúmenes de los componentes del tampón de energía varían en función de la optimizado Mg y las concentraciones de K-glutamato con el fin de permitir a altos rendimientos de la proteína expresada modelo libre de células.
Una evaluación preliminar del experimento incorporación se puede obtener mediante la realización de SDS-PAGE del medio de reacción libre de células no purificado. Para un análisis más detallado, espectroscopia de masas HPLC-ESI se propone como un medio para comprobar si hay incor completa, en residuos específicosporación de la NCAA. Como preparación para este último, los sistemas de columnas de centrifugado se utilizan para permitir la purificación His-tag y el intercambio de tampón con los pequeños volúmenes que utilizamos en este protocolo.
Incluyendo espectroscopia de masas HPLC-ESI, todo el protocolo se puede realizar dentro de los 2 días. No incluye ninguna medida particularmente críticas. Sin embargo, las optimizaciones de concentración de Mg y K-glutamato, así como de vector de ADN son cruciales para expresar altos rendimientos de la proteína modelo. El uso del vector de expresión altamente eficiente gene_of_model_protein-pbest-OR2-OR1-Pr-UTR1-T500 es muy recomendable. La elución de Su-etiquetados proteínas es por lo general debido a la alta concentración de imidazol (> 150 mM) y otras sales tales como NaH 2 PO 4 (> 300 mm) o NaCl (> 50 mM) que generan mucho ruido de fondo en análisis de espectroscopia de masas 49 . Intercambio de dichos tampones de elución con un tampón de almacenamiento adecuado proteína estabiliza el modelo protey reduce drásticamente el ruido de fondo durante el análisis espectroscópico de masas.
Como resultado, se puede sustituye Arg en las seis posiciones dentro de la proteína modelo. En el sistema de expresión, no hay residuos de Arg puede ser detectado. Esto simplifica la incorporación de residuos específicos de análogos de Arg en comparación con otros sistemas de expresión que requieren nuevas estrategias de agotamiento 29,30. El enfoque libre de células presentado elude las limitaciones inherentes de los métodos in vivo que se deben a la toxicidad puede, o la fuerte dependencia de la secuencia de ARNm en las estrategias de producción única de proteínas 24,31. Contrariamente a la empleada en el sistema in vitro, la escisión in vivo de Can a homoserina y hidroxiguanidina se produce 31.
Sin embargo, el sistema libre de células retiene una cantidad suficiente de Lys para competir con análogos tales como Hyl. El análisis HPLC-ESI espectroscópico de masas muestra que la proteína modelo contiene tanto, el Canonical así como el análogo no canónico en diferentes proporciones. La incorporación de residuos específica de Lys es posible en general, pero para la sustitución completa, más estrategias de agotamiento, o aaRs especialmente diseñados y optimizados ARNt para el reconocimiento de la NCAA necesita ser desarrollado.
Hemos logrado excelentes rendimientos de las proteínas expresadas, modelo modificados libres de células mediante la adición de la NCAA en la misma concentración que los canónicos. La eficacia de incorporación depende de la naturaleza de la NCAA a ser incorporados. Incluso los mayores rendimientos todavía podrían ser realizable mediante la optimización de la concentración de la NCAA.
Los resultados presentados demuestran la aplicabilidad del sistema empleado para la incorporación de residuos específicos de la NCAA, siempre que sean aceptados por el sistema de traslación endógena canónica. Para la incorporación de residuos específicos de la NCAA específicos, uno más tiene que comprobar si los residuos de la distur CAA análogaB, el sistema de expresión.
Sistemas de transcripción-traducción libres de células se pueden diseñar de diferentes organismos para responder a diferentes demandas 54. El totalmente E. coli mecanismos de transcripción-traducción del sistema libre de células que aquí se presenta active el uso de bacteriófago y E. promotores de E. coli, y pueden actuar en paralelo o de forma consecutiva en cascadas 55. La aplicabilidad general y la facilidad de uso hacen que el método de una herramienta potente para la investigación adicional en la toxicidad de aminoácidos y la aplicación terapéutica.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
E.G. Worst and A. Ott acknowledge financial support by the Deutsche Forschungsgemeinschaft (DFG) within the collaborative research center SFB 1027 as well as Saarland University. E.G. Worst, A. Ott and V. Noireaux further acknowledge financial aid by the Human Frontiers Science Program Organization (HFSPO). The authors thank Tobias Baumann and Stefan Oehm (Institute of Chemistry, Technische Universität Berlin) for critical reading.
Materiales
| Name | Company | Catalog Number | Comments |
| Protective eyewear | Sigma-Aldrich, St. Louis, USA | Z758841 | |
| Nitrile gloves (size S) | Sigma-Aldrich, St. Louis, USA | Z768960 | Catalog numbers other sizes: Z768979 for M, Z768987 for L and Z768995 for XL |
| Eppendorf Safe-Lock Tube 1.5 ml (PCR clean) | Eppendorf, Hamburg, Germany | 30123.328 | |
| Microbalance Discovery DV114CM | Ohaus, Greifensee, Switzerland | 80104140 | |
| Microspatula (L 6 5/8 in., stainless steel, rod diam. 0.09 in.) | Sigma-Aldrich, St. Louis, USA | Z243213 | |
| L-Canavanine | Sigma-Aldrich, St. Louis, USA | C9758 | Acute toxicity: wear eyeshields, dust mask, protective gloves |
| Hydroxylysine (racemic mixture) | Sigma-Aldrich, St. Louis, USA | H0377 | |
| Cryo-gloves (size S, water resistent) | Sigma-Aldrich, St. Louis, USA | Z183490 | Catalog numbers other sizes: Z183512 for M, Z183520 for L and Z183539 for XL |
| RTS Amino Acid Sampler | Biotechrabbit, Hennigsdorf, Germany | BR1401801 | For homemade preparation of amino acid stock solutions, follow this protocol35 and use the solid amino acid kit LAA21-1KT, L-proline (81709-25G), L-cysteine (30089-25G), L-histidine (53319-25G) and L-lysine (L5501-5G) (all from Sigma-Aldrich, St. Louis, USA) |
| HEPES | Sigma-Aldrich, St. Louis, USA | H6147 | |
| ATP | Sigma-Aldrich, St. Louis, USA | A8937 | |
| CTP | Affymetrix, Santa Clara, USA | 14121 | |
| GTP | Affymetrix, Santa Clara, USA | 16800 | |
| UTP | Affymetrix, Santa Clara, USA | 23160 | |
| tRNA (from E. coli, pack size 100 mg) | Sigma-Aldrich, St. Louis, USA | 10109541001 | Catalog number for pack size of 500 mg is 10109550001 |
| CoA | Sigma-Aldrich, St. Louis, USA | C4282 | |
| NAD (from yeast ) | Sigma-Aldrich, St. Louis, USA | N6522 | |
| cAMP | Sigma-Aldrich, St. Louis, USA | A9501 | |
| Folinic acid | Sigma-Aldrich, St. Louis, USA | F7878 | |
| 3-PGA | Sigma-Aldrich, St. Louis, USA | P8877 | |
| Mg-glutamate | Sigma-Aldrich, St. Louis, USA | 49605 | |
| K-glutamate | Sigma-Aldrich, St. Louis, USA | G1149 | |
| pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 | Addgene, Cambridge, USA | Plasmid #40019 | |
| 4-20% precast Tris-Glycine Gels (10 cm x 10 cm x 1 mm, 10 courses) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 81610 | |
| SDS running buffer (10 x concentrate, 5,000 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 50001 | |
| SDS loading buffer (2 x concentrate, 50 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 05002 | |
| Unstained protein marker, broad range (2-212 kDa) | New England Biolabs, Ipswich, USA | P7702S | |
| Methanol | Merck, Darmstadt, Germany | 1060091011 | Toxic by inhalation, in contact with skin and if swallowed: wear protective gloves and work under fume hood |
| Acetic acid (99.8%) | VWR International, Darmstadt, Germany | 20104.447 | |
| Coomassie Blue G-250 (10 g) | Biozym Scientific, Hessisch Oldendorf, Germany | 902120 | |
| His-Spin Protein Miniprep kit | Zymo Research Europe, Freiburg, Germany | P2002 | Product also distributed by Zymo Research Corporation, Irvine, USA |
| Trizma Base | Sigma-Aldrich, St. Louis, USA | T1503 | |
| Hydrochloric acid | Sigma-Aldrich, St. Louis, USA | H1758 | |
| Glycerol, 99% | VWR International, Darmstadt, Germany | 24397.296DB | |
| CentriPure Z25 mini spin columns | Genaxxon bioscience, Ulm, Germany | CP-0205-Z100 | |
| Sodium chloride | Sigma-Aldrich, St. Louis, USA | S9888 | |
| Concentrator 5301 | Eppendorf, Hamburg, Germany | 5301 000.210 | |
| 2xYT | MP biomedicals, Santa Ana, USA | 113012032 | |
| Bacto-Agar | BD Diagnostics, Franklin Lakes, USA | 214010 | |
| Bead-beating tubes (polypropylene microvials) | Biospec, Bartlesville, USA | 522S | |
| Beads, 0.1 mm dia. | Biospec, Bartlesville, USA | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Merck, Darmstadt, Germany | 71402 | |
| Bradford BSA protein assay Kit | Bio-Rad, München, Germany | 500-0201 | |
| Chloramphenicol | Sigma-Aldrich, St. Louis, USA | C1919 | |
| Cuvettes, 1.5 ml | Thermo Fisher Scientific, Waltham, USA | 14-955-127 | |
| DTT | Sigma-Aldrich, St. Louis, USA | D0632 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad, München, Germany | 732-6204 | |
| Nunc 384-well optical bottom plates | Thermo Fisher Scientific, Waltham, USA | 142761 | |
| Nunc sealing tape | Thermo Fisher Scientific, Waltham, USA | 232701 | |
| PEG-8000 | Promega, Madison, USA | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich, St. Louis, USA | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich, St. Louis, USA | P8709 | |
| Slide-A-Lyzer dialysis cassettes, 10k MWCO (Kit) | Thermo Fisher Scientific, Waltham, USA | 66382 | |
| Spermidine | Sigma-Aldrich, St. Louis, USA | 85558 | |
| 1 L centrifuge bottle | Beckman-Coulter, Brea, USA | A98813 | |
| 4 L Erlenmeyer flask | Kimble Chase, Vineland (NJ), USA | 26500-4000 | |
| Avanti J-26XP centrifuge | Beckman-Coulter, Brea, USA | 393127 | Or equivalent centrifuge able to centrifuge 1 L bottles. |
| Forma 480 orbital shaker | Thermo Fisher Scientific, Waltham, USA | 480 | Or equivalent shaker able to shake chest-size 6 x 4 L . |
| JLA-8.1000 rotor | Beckman-Coulter, Brea, USA | 363688 | Or equivalent 5,000 x g rotor for the centrifuge above, able to centrifuge 1 L bottles. |
| Mini-Beadbeater-1 | Biospec, Bartlesville, USA | 3110BX | |
| Microfuge 22R refrigerated microcentrifuge | Beckman-Coulter, Brea, USA | 368831 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| Heating block HLC HBT 130 | Labexchange, Burladingen, Germany | 24465 | Or equivalent heating block able to heat samples in reaction tubes up to 100 °C. |
| Eppendorf MiniSpin centrifuge | Eppendorf, Hamburg, Germany | 5452000018 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| IKA Vortex 3 (4 mm orbital shaker diameter, 0 - 2,500 rpm) | Sigma-Aldrich, St. Louis, USA | Z654760 | Or equivalent vortex. |
| Scotsman AF103 ice flaker machine | Kälte-Berlin, Berlin, Germany | AF103 | Or equivalent ice flaker machine. |
| MyTemp mini digital incubator | Sigma-Aldrich, St. Louis, USA | Z763314 | Or equivalent incubator able to heat samples at 29 °C. |
| EcoCell electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12005 | Or equivalent electrophoresis chamber able to perform vertical gel electrophoresis with the above precast gels or other gels used. |
| Power-phor power supply for electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12001 | Or equivalent power supply able to supply above used electrophoresis cell / chamber with power |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 1 | VWR International, Darmstadt, Germany | 613-5278 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 2 | VWR International, Darmstadt, Germany | 613-5279 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D10ST (0.1 - 10 µl) | Gilson, Middleton, USA | F171101 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D200ST (2 - 200 µl) | Gilson, Middleton, USA | F171301 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D1000ST (100 - 1,000 µl) | Gilson, Middleton, USA | F171501 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| 50 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 50-0156 | |
| 15 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 525-0150 | |
| 14 ml polypropylene tubes (round bottom, two-position vent stopper, sterile) | Greiner Bio-One, Frickenhausen, Germany | 187262 | |
| Discovery BIO Wide Pore C5 HPLC Column (3 µm particle size, L x I.D. 10 cm x 2.1 mm) | Sigma-Aldrich, St. Louis, USA | 567227-U | |
| Agilent 1260 HPLC machine | Agilent Technologies, Santa Clara, USA | G1312B | |
| 6500 Series Accurate-Mass Quadrupole Time-of-Flight (Q-TOF) LC/MS | Agilent Technologies, Santa Clara, USA | G6530BA | |
| Acetonitrile | Sigma-Aldrich, St. Louis, USA | 270717 | |
| FLUOstar Omega microplate reader | BMG Labtech, Ortenberg, Germany | 415-101 | Or equivalent microplate reader able to measure the fluorescence of the expressed model protein |
| Hanna Checker pH meter | Sigma-Aldrich, St. Louis, USA | Z351091 | |
| Formic acid eluent additive for LC-MS | Sigma-Aldrich, St. Louis, USA | 56302 |
Referencias
- Böck, A., et al. Selenocysteine: the 21st amino acid. Mol. Microbiol. 5 (3), 515-520 (1991).
- Srinivasan, G., James, C. M., Krzycki, J. A. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA. Science. 296 (5572), 1459-1462 (2002).
- Budisa, N. Prolegomena to Future Experimental Efforts on Genetic Code Engineering by Expanding Its Amino Acid Repertoire. Angew. Chem. Int. Ed. Engl. 43, 6426-6463 (2004).
- Wang, L., Xie, J., Schultz, P. G. Expanding the Genetic Code. Annu. Rev. Biophys. Biomol. Struct. 35, 225-249 (2006).
- Chin, J. W. Expanding and Reprogramming the Genetic Code of Cells and Animals. Annu. Rev. Biochem. 83, 379-408 (2014).
- Neumann, H. Rewiring translation - Genetic code expansion and its applications. FEBS Lett. 586 (15), 2057-2064 (2012).
- Liu, C. C., Schultz, P. G. Adding New Chemistries to the Genetic Code. Annu. Rev. Biochem. 79, 413-444 (2010).
- Goerke, A. R., Swartz, J. R. High-Level Cell-Free Synthesis Yields of Proteins Containing Site-Specific Non-Natural Amino Acids. Biotechnol. Bioeng. 102 (2), 400-416 (2009).
- Albayrak, C., Swartz, J. R. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation. Nucleic Acids Res. 41 (11), 5949-5963 (2013).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr. Opin. Chem. Biol. 14 (6), 774-780 (2010).
- Xiu, X., Puskar, N. L., Shanata, J. A. P., Lester, H. A., Dougherty, D. A. Nicotine binding to brain receptors requires a strong cation-pi interaction. Nature. 458 (7237), 534-537 (2009).
- Grünewald, J., et al. Mechanistic studies of the immunochemical termination of self-tolerance with unnatural amino acids. Proc. Natl. Acad. Sci. USA. 106 (11), 4337-4342 (2009).
- Nikić, I., Lemke, E. A. Genetic code expansion enabled site-specific dual-color protein labeling: superresolution microscopy and beyond. Curr. Opin. Chem. Bio. 28, 164-173 (2015).
- Munier, R., Cohen, G. N. Incorporation d'analogues structuraux d'aminoacides dans les protéines bactériennes. Biochim. Biophys. Acta. 21 (3), 592-593 (1956).
- Lepthien, S., Merkel, L., Budisa, N. In Vivo Double and Triple Labeling of Proteins Using Synthetic Amino Acids. Angew. Chem. Int. Ed. 49 (32), 5446-5450 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). Proc. Natl. Acad. Sci. USA. 103 (25), 9482-9487 (2006).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat. Neurosci. 13 (7), 897-905 (2011).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Hoesl, M. G., et al. Lipase Congeners Designed by Genetic Code Engineering. ChemCatChem. 3 (1), 213-221 (2011).
- Bae, J. H., et al. Expansion of the Genetic Code Enables Design of a Novel "Gold" Class of Green Fluorescent Proteins. J. Mol. Biol. 328 (5), 1071-1081 (2003).
- Schachtele, C. F., Rogers, P. Canavanine death in Escherichia coli. J. Mol. Biol. 14 (2), 474-489 (1965).
- Rosenthal, G. A. The biological effects and mode of action of L-canavanine, a structural analogue of L-arginine. Q. Rev. Biol. 52 (2), 155-178 (1977).
- Rosenthal, G. A., Dahlman, D. L. Incorporation of L-Canavanine into Proteins and the Expression of Its Antimetabolic Effects. J. Agric. Food Chem. 39 (5), 987-990 (1991).
- Ishida, Y., Park, J. H., Mao, L., Yamaguchi, Y., Inouye, M. Replacement of All Arginine Residues with Canavanine in MazF-bs mRNA Interferase Changes Its Specificity. J. Biol. Chem. 288 (11), 7564-7571 (2013).
- Thomas, D. A., Rosenthal, G. A., Gold, D. V., Dickey, K. Growth Inhibition of a Rat Colon Tumor by L-Canavanine. Cancer Res. 46 (6), 2898-2903 (1986).
- Bence, A. K., Worthen, D. R., Adams, V. R., Crooks, P. A. The antiproliferative and immunotoxic effects of L-canavanine and L-canaline. Anticancer Drugs. 13 (3), 313-320 (2002).
- Bence, A. K., Crooks, P. A. The Mechanism of L-Canavanine Cytotoxicity: Arginyl tRNA Synthetase as a Novel Target for Anticancer Drug Discovery. J. Enzyme Inhib. Med. Chem. 18 (5), 383-394 (2003).
- Akaogi, J., et al. Role of non-protein amino acid L-canavanine in autoimmunity. Autoimmun. Rev. 5 (6), 429-435 (2006).
- Singh-Blom, A., Hughes, R. A., Ellington, A. D. An amino acid depleted cell-free protein synthesis system for the incorporation of non-canonical amino acid analogs into proteins. J. Biotechnol. 178, 12-22 (2014).
- Oh, S. -. J., Lee, K. -. H., Kim, H. -. C., Catherine, C., Yun, H., Kim, D. -. M. Translational Incorporation of Multiple Unnatural Amino Acids in a Cell-free Protein Synthesis System. Bioprocess. Eng. 19 (3), 426-432 (2014).
- Worst, E. G., Exner, M. P., De Simone, A., Schenkelberger, M., Noireaux, V., Budisa, N., Ott, A. Cell-free expression with the toxic amino acid canavanine. Bioorg. Med. Chem. Lett. 25 (17), 3658-3660 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. J. Biol. Eng. 4 (8), (2010).
- Chemla, Y., Ozer, E., Schlesinger, O., Noireaux, V., Alfonta, L. Genetically expanded cell-free protein synthesis using endogenous pyrrolysyl orthogonal translation system. Biotechnol. Bioeng. 112 (8), 1663-1672 (2015).
- Sun, Z. Z., Hayes, C. A., Shin, J., Caschera, F., Murray, R. M., Noireaux, V. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. J. Vis. Exp. (79), e50762 (2013).
- Caschera, F., Noireaux, V. Preparation of amino acid mixtures for cell-free expression systems. Biotechniques. 58 (1), 40-43 (2015).
- Green, M. R., Sambrook, J. . Molecular Cloning: A Laboratory Manual. , (2012).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. J. Vis. Exp. (6), e253 (2007).
- Zhang, S., Cahalan, M. D. Purifying Plasmid DNA from Bacterial Colonies Using the Qiagen Miniprep Kit. J. Vis. Exp. (6), e247 (2007).
- Moreno, L. A., Cox, K. L. Quantification of dsDNA using the Hitachi F-7000 Fluorescence Spectrophotometer and PicoGreen Dye. J. Vis. Exp. (45), e2465 (2010).
- Desjardins, P., Conklin, D. NanoDrop Microvolume Quantitation of Nucleic Acids. J. Vis. Exp. (45), e2699 (2010).
- Sukumaran, S. Concentration Determination of Nucleic Acids and Proteins Using the Micro-volume Bio-spec Nano Spectrophotometer. J. Vis. Exp. (48), e2699 (2011).
- Laemmli, U. K. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Hochuli, E., Bannwarth, W., Döbeli, H., Gentz, R., Stüber, D. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nature Biotechnology. 6 (11), 1321-1325 (1988).
- Schägger, H., Aquila, H., Von Jagow, G. Coomassie blue-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for direct visualization of polypeptides during electrophoresis. Anal. Biochem. 173 (1), 201-205 (1988).
- Bradford, M. M. A Rapid Method for the Quantitation of Microgram Quantities of Protein Utilizing the Principle of Protein-Dye Binding. Anal Biochem. 72, 248-254 (1976).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. J. Vis. Exp. (38), e1918 (2010).
- Banerjee, S., Mazumdar, S. Electrospray Ionization Mass Spectrometry: A Technique to Access the Information beyond the Molecular Weight of the Analyte. Int. J. Anal. Chem. 2012, 1-40 (2012).
- Fujiwara, K., Nomura, S. M. Condensation of an additive-free cell extract to mimic the conditions of live cells. PLoS One. 8 (1), e54155 (2013).
- Zhang, Z., Marshall, A. G. A universal algorithm for fast and automated charge state deconvolution of electrospray mass-to-charge ratio spectra. J. Am. Soc. Mass. Spectrom. 9 (3), 225-233 (1998).
- Li, X., Zhang, G., Ngo, N., Zhao, X., Kain, S. R., Huang, C. C. Deletions of the Aequorea victoria Green Fluorescent Protein Define the Minimal Domain Required for Fluorescence. J. Biol. Chem. 272 (45), 28545-28549 (1997).
- Gagoski, D., Polinkovsky, M. E., Mureev, S., Kunert, A., Johnston, W., Gambin, Y., Alexandrov, K. Performance benchmarking of four cell-free protein expression systems. Biotechnol. Bioeng. 113 (2), 292-300 (2016).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. ACS Synth. Biol. 1 (1), 29-41 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados