Method Article
Incorporação específico de resíduos de não-canônicos Aminoácidos em proteínas modelo Usando um
Neste Artigo
Resumo
An easy-to-use, cell-free expression protocol for the residue-specific incorporation of noncanonical amino acid analogs into proteins, including downstream analysis, is presented for medical, pharmaceutic, structural and functional studies.
Resumo
O conjunto canónico de aminoácidos leva a uma excepcionalmente ampla gama de funcionalidades proteína. No entanto, o conjunto de resíduos ainda impõe limitações sobre os pedidos de proteínas potenciais. A incorporação de aminoácidos noncanonical pode ampliar este âmbito. Existem duas abordagens complementares para a incorporação de aminoácidos não-canónicos. Para a incorporação específica do local, para além dos mecanismos endógenos de translação canónicos, um par aminoacil-ARNt-sintetase-ARNt ortogonal deve ser desde que não interage com os canónicas. Por conseguinte, um codão que não é atribuído a um aminoácido canónica, geralmente um codão de terminação, também é necessário. Esta expansão código genético permite a incorporação de um aminoácido não-canônico a uma única, dado local no interior da proteína. O trabalho aqui apresentado descreve a incorporação específica do resíduo onde o código genético é transferido dentro do sistema translacional endógeno. A maquinaria de tradução umccepts o aminoácido não-canônico como um substituto para incorporá-lo em locais canonicamente prescrita, ou seja, todas as ocorrências de um aminoácido na proteína canónica são substituídos por um não-canônico. A incorporação de aminoácidos noncanonical pode alterar a estrutura da proteína, fazendo com que as propriedades físicas e químicas consideravelmente modificadas. Análogos de aminoácidos noncanonical muitas vezes actuam como inibidores de crescimento de células de hospedeiros de expressão, uma vez que modificar proteínas endógenas, o que limita na produção de proteínas in vivo. In vivo, a incorporação de aminoácidos em proteínas tóxicas noncanonical permanece um desafio especial. Aqui, é apresentada uma abordagem isenta de células para uma completa substituição de L-arginina por o aminoácido L-canavanina não-canônico. Isso evita as dificuldades inerentes de expressão in vivo. Além disso, um protocolo para a preparação de proteínas alvo para a análise de espectro de massa está incluído. Mostra-se que a L-lisina pode ser substituída por L-hidroxi-lisina,embora com menor eficiência. Em princípio, qualquer análogo de aminoácido não-canônico podem ser incorporados utilizando o método apresentado enquanto o endógena in vitro sistema de tradução que reconhece.
Introdução
O código genético é universal para a biosfera. Ele codifica para um conjunto de 20 aminoácidos canónicos, que às vezes é prorrogado por selenocisteína 1 ou 2 pyrrolysine. É o que se traduz ribossoma do código genético, com a ajuda de ARNt em cadeias de aminoácidos que se dobram em proteínas. Os grupos funcionais dos aminoácidos canónicos, em combinação com modificações pós-tradução, contribuem para uma gama excepcionalmente ampla de 3,4 a função da proteína. Em princípio, as limitações funcionais devido ao conjunto limitado de aminoácidos canónicos pode ser superado mediante a incorporação de mais, aminoácidos não-canónicos (NCAAs) que permitem novos produtos químicos e novas funcionalidades 3,4.
Existem duas abordagens complementares para a incorporação de: a NCAAs site- ou a incorporação específica do resíduo. O primeiro método implica dificuldades técnicas consideráveis, uma vez que o conjunto de canônica de aminoacil-tRNA-synthetases (RAA) e tRNAs deve ser expandido por um par RAA-ARNt ortogonal que não deve interagir com o endógena máquinas de tradução. Com base em engenharia cuidadosa, esta abordagem incorpora os NCAAs como mutações pontuais nos locais de proteína desejada. Incorporação específica do local de NCAAs é geneticamente codificado por um codão que não é atribuído a um ácido amino canónica (CAA), normalmente um codão de terminação a 5-9. Este método implica alterações na função em um determinado site, em vez de ao longo de toda a proteína 10-13.
Em contraste, a incorporação específica do resíduo baseia-se no reconhecimento erróneo do aminoácido não-canônico pela maquinaria de tradução canónica. A incorporação ocorre devido à falta de especificidade para o substrato das RAA. A incorporação específica do resíduo de NCAAs, construída sobre o trabalho de Cohen e colegas de trabalho de 14, levou a aplicações importantes 3,10, entre eles rotulagem bio-ortogonal 15-17 de proteínasou elucidação da estrutura de proteínas em cristalografia de raios X 18.
Como RAA naturais geralmente preferem sua aminoácido cognato mais de uma NCAA isoestrutural, eficientes na incorporação específica do resíduo vivo geralmente requer um hospedeiro de expressão auxotr�ica não é capaz de sintetizar o analógico canônica da NCAA. As células hospedeiras são cultivadas em meio de crescimento, que proporciona apenas uma baixa concentração do análogo CAA. Seu esgotamento em combinação com a suplementação consecutivo, com a NCAA força o hospedeiro de expressão para incorporar o NCAA na proteína modelo em vários locais, canonicamente prescritos. Em contraste com a abordagem de local específico, este geralmente tem um profundo impacto sobre toda a estrutura da proteína, conduzindo a modificado consideravelmente as propriedades físicas e químicas de proteínas 19,20. No entanto, a maior parte do NCAAs são inibidores do crescimento para o hospedeiro de expressão a 3, em que são incorporados em muitos outros Proteins, além daqueles de interesse durante a expressão do gene recombinante. Isto limita claramente a abordagem in vivo. A incorporação in vivo de aminoácidos que são tóxicos ou têm uma forte influência sobre a estrutura da proteína permanece um desafio especial. No entanto, estas moléculas estão entre os mais promissores para a engenharia de proteínas com funções extraordinárias.
Um exemplo é o tóxico, não-canônico, que ocorrem naturalmente L-canavanina (CAN), um análogo da L-arginina (Arg). Ela afeta e blocos Arg caminhos de reação de regulação e catalíticos associados, e sua presença na célula viva pode levar a morte imediata 3,21-23. A sua incorporação em proteínas nas posições de arginina pode reduzir a estabilidade da proteína 21-23. Devido à toxicidade resultante, a expressão de proteínas contendo canavanina em Escherichia coli (E. coli) e outros hospedeiros de expressão comum continua a ser um desafio. Por estas razões, completo in vivo, incorporation de Can em todas as posições Arg foi adequadamente confirmada apenas uma vez 24, utilizando um sistema de produção de single-proteína elaborado. No entanto, pode tem sido proposta como um agente anti-cancro 25-27, e como um estimulador de doenças autoimunes em seres humanos 28. Além disso, é objecto de vários estudos sobre a sua actividade anti-metabólica, antibacteriano, antifúngico e as propriedades antivirais 25. Estas propriedades levantar uma demanda por eficiente e fácil de executar métodos para expressar Pode contendo proteínas para farmácias e estudos funcionais.
Apesar de muitos problemas que estão ligados à produção in vivo pode ser contornado usando sistemas de expressão livre de células, nas abordagens específicas de resíduos in vitro só foram pouco explorados. A incorporação específica do resíduo isento de células de um análogo de L-triptofano e 29 múltiplos NCAAs 30 têm sido relatados. Estes métodos baseiam-se no altamente effictário polimerase de ARN de T7. A polimerase de ARN T7 de bacteriófago implica a transcrição semelhante, reduzindo assim a funcionalidade genético em comparação com a transcrição endógena.
A incorporação específica do resíduo completo de lata em um modelo de proteína em todas as posições Arg Recentemente, foi relatado 31, utilizando um sistema de expressão isento de células 32. Uma ligeira modificação do mesmo sistema permitiu a incorporação específica do local de diferentes análogos de pirrolisina em um modelo de proteína através de codão de terminação de supressão de 33. O sistema livre de células empregadas 31-33 é baseado em um todo E. coli sistema de transcrição-tradução. No entanto, permite a expressão da proteína de forma tão eficiente como nos sistemas actuais de bacteriófagos (0,5 - 1 mg / ml de proteína recombinante) 32, enquanto retém a maior parte do original modularidade de transcrição-tradução.
Neste trabalho, um protocolo detalhada é fornecida sobre a forma como o residincorporação de UE específicos de NCAAs pode ser realizado, utilizando tudo isto E. sistema livre de células coli 32. Além disso, são propostas novas medidas para preparar as proteínas expressas para avaliação adequada via espectroscopia de massa HPLC-ESI. Para expandir as propriedades deste sistema isento de células, este trabalho não se refere apenas à incorporação publicada de Can 31, mas também apresenta novos dados relacionados com a não-canônico L-lisina analógico L-hidroxi-lisina.
O protocolo seguinte para a incorporação específica do resíduo de NCAAs é uma adaptação de um protocolo recentemente publicado em 34 JoVE. O último protocolo descreve como realizar a expressão isento de células altamente eficiente com aminoácidos padrão. Além disso, apresenta-se a preparação do extracto em bruto livre de células, a solução de amino ácido, a solução de reserva de energia e o tampão de energia utilizada nesta abordagem. O protocolo seguinte concentra-se em passos modificados em comparação com o anterior Protocolo, a fim de permitir a incorporação específica do resíduo de NCAAs. pipetas calibradas, dicas de ligação baixas pipetas e tubos de micro-centrífuga são recomendados para a preparação. No que se segue, as abreviaturas IUPAC para os aminoácidos são utilizados.
Protocolo
Atenção: É favor consultar todas as fichas de dados de segurança do material relevantes (MSDS) antes do uso. Muitos dos produtos químicos usados são altamente tóxicos. equipamento de protecção pessoal é necessária (eyeshield, máscara de pó, luvas, jaleco, calça de corpo inteiro, fechou-toe sapatos), bem como trabalhar em um exaustor.
1. Preparação de Soluções de Aminoácidos
- Preparação da solução do NCAA (168 mM)
NOTA: A preparação solução-mãe da NCAA é descrito para o análogo Arg pode como um exemplo. Por conseguinte adaptar os valores para outra NCAAs.- Coloque um tubo de 1,5 ml de reacção sobre uma microbalança. Pesar 46,1 mg de lata no interior do tubo de reacção para a preparação de 1 ml de uma solução 168 mM. Use um microspatula estéril. Para uma mistura racémica do NCAA, o dobro da concentração da solução mãe.
- Adicionar 977 ul de ddH 2 O. estéril Agitar bem em vórtice até Can é idissolução n completa.
NOTA: Para um volume de solução total de 1 ml, o volume física do aminoácido dissolvido tem que ser compensada. Para qualquer um dos aminoácidos, estimativa metade da massa em mg sólido como o aumento de volume em ul correspondente (100 mg de sólido terá um volume de 50 ul da solução de 35). A maioria dos aminoácidos pode ser dissolvido a esta concentração. Se não for, reduzir a concentração até à dissolução completa. - Usar diretamente a solução de NCAA para a preparação das soluções de aminoácidos na secção 1.2 ou o flash congelá-lo em azoto líquido e armazená-lo a -20 ° C. ATENÇÃO! Por segurança, use uma eyeshield e Cryo-luvas para ser protegido contra líquidos salpicos de azoto.
- Preparação das soluções de aminoácidos
NOTA: Para a preparação das soluções de aminoácidos, usar o amostrador de aminoácidos que proporciona a L-isómeros dos 20 CAA no stock separadaAs soluções (1,5 ml, tamponado com HEPES / KOH, <0,1% de NaN3, pH 7,5), cada um a uma concentração de 168 mM, excepto para a L-leucina (140 mM). Para uma preparação caseira destas soluções stock (tamponado com KOH), siga este protocolo 35.- Descongelar as soluções de estoque dos 20 CaaS (amostrador de aminoácidos ou preparados de acordo com 35) e da NCAA (preparada no ponto 1.1) à temperatura ambiente.
- Após o descongelamento, muitas vezes vortex as soluções estoque para dissolver quaisquer aminoácidos precipitados. Como alguns aminoácidos são mais difíceis de dissolver, incubar-los num bloco de aquecimento a 37 ° C até à dissolução completa. Cys pode não se dissolvem totalmente. Coloque todos os aminoácidos no gelo, exceto para Asn, Phe e Cys - mantê-los à temperatura ambiente para evitar a precipitação.
- Use os seguintes valores para usar um sétimo do kit completo.
NOTA: Escala para baixo de forma adequada para trabalhar com volumes menores e salvar partes do kit para outras experiências. Escala para incorporatião de NCAAs em proteínas modelo em grande escala. Para evitar que a descongelação frequente, o que é susceptível de reduzir a estabilidade dos aminoácidos, alíquota as soluções estoque individuais de aminoácidos em volumes de 200 ul. Este volume alíquota de 200 ul e os volumes alíquota utilizada na conta do passo 1.2.4.1 para perdas devido a pipetagem. - Em primeiro lugar, se preparar uma solução de mistura mestre de aminoácidos que vai ser dividido no passo 1.2.4.3 para finalizar a preparação de 3 soluções de aminoácidos compostas de maneira diferente (secções 1.2.5 - 1.2.7). Nestas soluções, concentrar todos os aminoácidos a 6 mM, excepto para a Leu (5 mM).
- Transferir 1,4 ml de ddH 2 O estéril para um tubo de centrífuga de 15 ml. Coloque-o em gelo. Adicionar 175 ul de cada solução de aminoácido. Adicionar uma após a outra, excepto para a solução stock de CAA (por exemplo, Arg) para ser substituída pela NCAA (por exemplo, é possível). Agitar bem em vórtice após cada adição e colocar a solução de volta no gelo.
NOTA:Leu é a 5 mm, no 3 soluções de aminoácidos compostas de maneira diferente, em comparação com 6 mm para os outros aminoácidos. A concentração reduzida não reduzir a eficiência de expressão. Dimensionamento até 6 mm é adequada também. - Transferir as soluções de aminoácidos, na seguinte ordem para evitar a precipitação: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, lie, Lys, Met, Phe, Pro, Ser, Thr, Vai, Trp, Tyr, Leu, e Cys. Lembre-se de não adicionar a solução-mãe da CAA (por exemplo, Arg), que é análogo ao NCAA (por exemplo, é possível). Finalmente, completamente vórtice. Incubar a 37 ° C para tornar a solução mais clara possível.
- Dividir esta solução mestre mistura de aminoácidos em três volumes iguais de 1,35 ml. Transferir cada um dos volumes de divisão em tubos de 1,5 ml de reacção. Mantenha-os no gelo.
- Transferir 1,4 ml de ddH 2 O estéril para um tubo de centrífuga de 15 ml. Coloque-o em gelo. Adicionar 175 ul de cada solução de aminoácido. Adicionar uma após a outra, excepto para a solução stock de CAA (por exemplo, Arg) para ser substituída pela NCAA (por exemplo, é possível). Agitar bem em vórtice após cada adição e colocar a solução de volta no gelo.
- Prepara-se uma solução de aminoácidos que consiste em todos os 20 AACs a uma concentração de 6 mM de cada, com excepção de que Leu é de 5 mm. Para o primeiro volume óF 1,35 ml, como um resultado do desdobramento no passo 1.2.4.3, adicionar 50 ul da solução de 168 mM de estoque de CAA (por exemplo, Arg) que é análogo ao NCAA (por exemplo, é possível). Completamente vortex.
- Coloque de volta no gelo. Alíquota esta solução 1,4 mL em volumes de 16 uL para dentro de tubos de reacção. Note-se que este volume de solução a cerca de 85 leva a alíquotas. Rotular essas alíquotas "+ CAA" (por exemplo, + Arg).
- Flash congelar as alíquotas em nitrogênio líquido e armazenar a -80 ° C. ATENÇÃO! Por segurança, use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de azoto líquido.
- Prepara-se uma solução de aminoácidos que é composta por 19 CAA CAA excepto para o (por exemplo, Arg) que é análogo ao NCAA (por exemplo, Can). Adicionar cada aminoácido a uma concentração de 6 mM, excepto para a Leu (5 mM).
- Adicionar 50 ul de DDH estéril 2 O para o segundo volume de 1,35 ml, como um resultado do desdobramentono passo 1.2.4.3. Agitar bem em vórtice e colocar de volta no gelo. Alíquota esta solução 1,4 mL em volumes de 16 uL para dentro de tubos de reacção. Note-se que este volume de solução a cerca de 85 leva a alíquotas. Rotular essas alíquotas "- CAA" (por exemplo, - Arg).
- Flash congelar as alíquotas em nitrogênio líquido e armazenar a -80 ° C. ATENÇÃO! Por segurança, use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de nitrogênio.
- Prepare uma mistura de aminoácidos que contém 19 CAAS eo NCAA (por exemplo, Can) que substitui o canônico (por exemplo, Arg). Adicionar cada aminoácido a uma concentração de 6 mM, excepto para a Leu (5 mM). Para o último de 1,35 ml de volume, como um resultado do desdobramento no passo 1.2.4.3, adicionar 50 ul da solução estoque 168 mM do NCAA (por exemplo, Can). Rotulá-la "+ NCAA" (por exemplo, + CAN). Agitar bem em vórtice e colocar de volta no gelo.
- Alíquota esta solução 1,4 mL em volumes de 16 & #181; G em tubos de reacção. Note-se que este volume de solução a cerca de 85 leva a alíquotas. Rotular essas alíquotas "+ NCAA" (por exemplo, + CAN).
- Flash congelar as alíquotas em nitrogênio líquido e armazenar a -80 ° C. ATENÇÃO! Por segurança, use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de nitrogênio.
NOTA: Os 16 volumes alíquotas ul utilizados nas etapas 1.2.5.1, 1.2.6.1 e 1.2.7.1 são ligeiramente mais elevados do que o necessário para compensar as perdas devido à pipetagem.
2. Preparação acumulador de energia
NOTA: Cada lote de extrato bruto é único e requer concentrações de Mg e K-glutamato 34 otimizado. O volume da alíquota de extracto em bruto depende da concentração de proteína 34. Use o modelo de cálculo prevista (Material Suplementar 1) para valores diferentes. Encontre mais instruções em Material Suplementar 1 legenda da figura, EXPOSIÇÃOining como empregar esse modelo.
- Preparar e armazenar a -80 ° C a solução 14x energia e alíquotas extrato bruto de acordo com o protocolo não modificada 34. Calibra-se o extracto em bruto, dependendo das concentrações de Mg e K-glutamato de eficiência de expressão optimizada 34.
NOTA: A composição final de solução de energia 14x é: HEPES 700 mM (pH 8), ATP 21 mM, GTP 21 mM, CTP 12,6 mM, UTP 12,6 mM, 2,8 mg / ml de ARNt, CoA 3,64 mM, NAD 4,62 mM, 10,5 cAMP mM, ácido folínico 0,95 mM, espermidina 14 mM, 420 mM de 3-PGA. - Thaw no gelo a solução 100 mM Mg-glutamato estoque, solução de 3 M K-glutamato, solução 14x de energia e 40% de PEG-8000 para preparar a mistura principal. Mantenha-os no gelo.
- Misture 9,18 mL de uma solução de estoque de 100 mM de Mg-glutamato, 3,06 uL de solução mãe de 3 M K-glutamato, 21,86 ml de solução de energia 14x, 15,3 ul de 40% de PEG-8000 e 1,6 ul estéreis ddH2O num tubo de reacção. Agitar bem em vórtice este mastroer misturar após cada adição, e mantê-lo em gelo.
- Alíquota da mistura principal (51 ul) em volumes de 16 ul (3 aliquotas) em tubos de reacção. vortex com frequência mistura principal durante alíquotas. Flash congelar as alíquotas em nitrogênio líquido.
NOTA: O volume da alíquota de 16 ul, bem como o volume de mistura principal são ligeiramente mais elevados do que o necessário para compensar perdas devidas a pipetagem. - Use um filtro para recolher os tubos amortecedores de energia. Armazenar os tubos a -80 ° C. CUIDADO! Use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de nitrogênio.
3. Preparação e Execução de reações sem células de Incorporação de Resíduos específicos do de NCAAs
- Em primeiro lugar, preparar a solução de vector de DNA em DDH 2 O.
- Para a expressão da proteína é altamente eficiente, utilizar o vector de expressão pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonar o gene que codifica para a proteína modelo para este vector 36,37 </ Sup>.
NOTA: Como alternativa, utilizar outros promotores que são reconhecidos pelo σ 70, bem como, mas note que a eficiência de expressão pode ser reduzida. - Transformar 37,38 o vetor em E. estirpe de E. coli KL 740 32 (Yale MDPS #: 4382), Purifica-se o ADN do vector amplificado 37,39 e quantificar a concentração da solução de ADN 40-42. Guardar a solução de ADN a -20 ° C ou diretamente usá-lo para a preparação de reação celular-fee (etapas 3.4.1, 3.4.2 e 3.4.3).
- Para a expressão da proteína é altamente eficiente, utilizar o vector de expressão pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonar o gene que codifica para a proteína modelo para este vector 36,37 </ Sup>.
- Calibrar a eficiência de expressão isento de células, dependendo da concentração utilizada construção de vector de acordo com o protocolo não modificada 34. Use a melhor concentração que leva a rendimentos mais elevados de proteína para a preparação de reacção livre de células (passos 3.4.1, 3.4.2 e 3.4.3).
NOTA: A preparação de reacções livre de células é exemplificado usando uma solução de estoque nM ADN do vector 90 que leva a uma concentração final de 10 nM vetor na isento de células-reacção seguinte e os valores óptimos acima de Mg e K-glutamato, bem como o volume de extracto alíquota. Use o modelo de cálculo para valores diferentes. - Thaw no gelo 3 alíquotas extrato bruto cada um dos 30 volumes ul (preparada de acordo com o protocolo não modificada 34), um aminoácido solução de ácido alíquota rotulados como "+ CAA" (por exemplo, + Arg), 1 aminoácidos solução de ácido alíquota rotulados "- CAA" (por exemplo, , - Arg) e 1 amino solução de ácido alíquota rotulados como "+ NCAA" (por exemplo, + Can) (preparado nas seções 1.2.5 - 1.2.7), 3 alíquotas buffers de energia (preparado na seção 2) e a solução de DNA vetor ( preparada na secção 3.1).
NOTA: O extracto bruto é ligeiramente viscosa e pode conter bolhas de ar. Retirar as bolhas de ar através de centrifugação a 10000 xg durante 30 seg a 4 ° C. Coloque alíquotas extrato bruto de volta no gelo. - Prepare 3 reações livre de células de forma diferente compostas (cada uma com 90 mL de volume final) através da mistura de extrato bruto (33,33%), energiatampão (16,67%), um dos 3 aliquotas de forma diferente de aminoácidos compostas de solução (16,67%) e solução de ADN do vector. Opcionalmente, adicionar outras biomoléculas (ADN, proteínas, ARNt, etc.), mas adequadamente reduzir o volume de ddH 2 O.
- Preparar a reacção livre de célula de referência (90 ul) expressando a proteína modelo não modificado.
- Adicionar 15 ul de tampão de energia, 15 ul da solução alíquota de aminoácido marcado com "+ CAA" (por exemplo, + Arg), 10 ul de 90 solução de DNA do vector nM e 20 ul de DDH estéril 2 O para os 30 ul de bruto extrair. Misturar por pipetagem para cima e para baixo e suavemente vortex depois da adição de cada ingrediente.
- Alíquota os 90 ul de reacção livre de células em 15 volumes iguais de 6 mL. Transferir cada um dos volumes de 15 para um tubo de reacção separada. Feche os tubos e colocá-los de volta no gelo. Rotular todos os tubos de reacção como "CFR (+ CAA)" (por exemplo, CFR (+ Arg)).
- Prepare a reação negativa de controle livre de células (90 ul), onde nem a CAA (por exemplo, Arg), nem o analógico não-canônico (por exemplo, é possível) são adicionados.
- Adicionar 15 ul de tampão de energia, 15 mL da solução alíquota aminoácido identificado como "- CAA" (por exemplo, - Arg), 10 mL de 90 solução de DNA vector nm e 20 l de DDH estéril 2 O para os 30 mL de crude extrair. Misturar por pipetagem para cima e para baixo e suavemente vortex depois da adição de cada ingrediente.
- Alíquota os 90 ul de reacção livre de células em 15 volumes iguais de 6 mL. Transferir cada um dos volumes de 15 para um tubo de reacção separada. Feche os tubos e colocá-los de volta no gelo. Rotular todos os tubos de reacção como "CFR (-cAA)" (por exemplo, CFR (Arg)).
- Prepare a reação livre de células (90 mL) que é suposto ao resíduo especificamente incorporar a NCAA (por exemplo, Can) na proteína modelo expresso.
- Adicionar o 15ul de tampão de energia, 15 ul da solução alíquota de aminoácido marcado com "+ NCAA" (por exemplo, pode +), 10 ul de solução de ADN 90 nM e 20 ul de ddH 2 O esterilizada para os 30 ul de extracto bruto. Misturar por pipetagem para cima e para baixo e suavemente vortex depois da adição de cada ingrediente.
NOTA: A eficiência de expressão pode ser reduzida em comparação com a expressão com aminoácidos padrão. Se necessário, simplesmente dimensionar-se o volume de reacção isento de células. - Alíquota os 90 ul de reacção livre de células em 15 volumes iguais de 6 mL. Transferir cada um dos volumes de 15 para um tubo de reacção separada. Feche os tubos e colocá-los de volta no gelo. Para maiores volumes de reação, de acordo alíquota em outros volumes de 6 mL. Rotular todos os tubos de reacção como "CFR (+ NCAA)" (por exemplo, CFR (+ Can)).
- Adicionar o 15ul de tampão de energia, 15 ul da solução alíquota de aminoácido marcado com "+ NCAA" (por exemplo, pode +), 10 ul de solução de ADN 90 nM e 20 ul de ddH 2 O esterilizada para os 30 ul de extracto bruto. Misturar por pipetagem para cima e para baixo e suavemente vortex depois da adição de cada ingrediente.
- Preparar a reacção livre de célula de referência (90 ul) expressando a proteína modelo não modificado.
- Incubar todos os tubos a 29 ° CO / N.
NOTA: Apenas pequenos volumes de reacção de permitir diff adequados de oxigêniousion para a reacção que é crucial para a expressão da proteína é altamente eficiente. Os volumes de reacção superiores a 10 jul exigem oxigenação activa através da agitação 34. No caso de grandes volumes, dividir a reacção em volumes menores do que 15 jil. - Depois de expressão livre de células, piscina todos identicamente composta reações livres de células de divisão de 6 mL. Primeiro, piscina todos os 15 reações livres de células de divisão de 6 ul rotulados como "CFR (+ CAA)" (por exemplo, CFR (+ Arg)). Então, piscina todos os 15 reações livres de células de divisão de 6 ul rotulados como "CFR (-cAA)" (por exemplo, CFR (Arg)). Finalmente, piscina todos os 15 reações livres de células de divisão de 6 ul rotulados como "CFR (+ NCAA)" (por exemplo, CFR (+ Can)). Para maiores volumes de reação, de acordo reunir mais volumes de 6 mL.
- Verificar o nível da proteína modelo de expressão em todos os três reunidas, de maneira diferente reacções composto livre de células através da realização de SDS-PAGE 43,44 (secção 4.1) desnaturantes.
NOTA: Utilize este meto d para uma avaliação preliminar da experiência de incorporação. - Prepare as proteínas expressas modelo livre de células para uma análise adequada através de espectroscopia de massa HPLC-ESI (secção 4.4). Em primeiro lugar, purificar 45 deles (secção 4.2). Finalmente, trocar o tampão (4.3) para evitar a alta ruído de fundo durante a espectroscopia de massa.
NOTA: As seções 3.4.1, 3.4.2 e 3.4.3 levar a condições de reacção livre de células típicas 34: 8,9 - 9,9 mg / ml de proteína (de extrato bruto), 4,5 - mM Mg-glutamato 10,5, 40 - 160 mM K-glutamato, 1 mM de cada aminoácido excepto leucina, leucina 0,83 mM, HEPES 50 mM, 1,5 mM de ATP e GTP, CTP 0,9 mM e UTP, 0,2 mg / ml de ARNt, CoA 0,26 mM, NAD 0,33 mM, AMPc 0,75 mM , ácido folínico 0,068 mM, espermidina 1 mM, 30 mM de 3-PGA, 2% de PEG-8000 e 10 nM pBEST-OR2-OR1-Pr-UTR1 gene_of_model_protein-T500. Se desejado, um procedimento de preparação diferente da reacção isento de células pode ser realizada que conduz às condições de reacção anteriores.
- SDS-PAGE das reacções isentos de células
NOTA: Execute desnaturação SDS-PAGE para uma análise rápida e preliminar das proteínas expressas sem qualquer purificação adicional ou extracção, e execute os seguintes passos.- Descongelar o padrão de proteína em gelo. Certifique-se de que ele é composto por proteínas com pesos moleculares semelhantes ao modelo de proteína expressa pela sua localização no gel (passo 4.1.13).
NOTA: Aqui, o padrão usado fornece proteínas sobre uma ampla gama de pesos moleculares (miosina: 212 kDa, de ligação de maltose-proteína-β-galactosidase: 158 kDa, β-galactosidase: 116 kDa, fosforilase b: 97 kDa, albumina de soro : 66 kDa, glutâmico desidrogenase: 56 kDa, maltose da proteína de ligação: 43 kDa, tioredoxina redutase: 35 kDa, triosefosfato isomerase: 27 kDa, do inibidor de tripsina: 20 kDa, LYsozyme: 14 kDa, a aprotinina: 7 kDa, a insulina A: 3 kDa, cadeia B: 2 kDa). - Desde reações livres de células são ligeiramente viscoso devido à alta concentração de proteína, dilui-los com DDH estéril 2 O 5 - 10 vezes antes de SDS-PAGE para garantir a migração gel apropriada e para evitar a saturação após a coloração. A fim de não perder muito amostra, diluir 1 ml de reacção livre de células em 4 ul de DDH estéril 2 O. Preparar uma diluição em tubos de reacção, completamente vortex e logo girar o líquido para baixo com um mini centrifugar por 2-3 seg a 2.000 x g.
- Adicionar 5 uL de tampão de carregamento 2 x 5 mL de reacção isento de células diluídas. Agitar bem em vórtice e girar para baixo com um mini centrifugar por 2-3 seg a 2.000 x g.
NOTA: Aqui, depois disso, as biomoléculas são dissolvidos em 62,5 mM de Tris / Cl -, 10% de glicerol, 2% de SDS, e 0,0025% de azul de bromofenol, a pH 6,8, uma composição típica. A utilização de outros corantes de carga que levam a um pouco diferente de diluição conditions pode ser adequada também. - vortex completamente o padrão de proteína, e transferir 15 ul do mesmo em um tubo de reacção.
NOTA: Dependendo do tamanho do gel e para outras normas de proteína, o volume recomendado pode ser diferente do acima. - Coloque os tubos de reacção em um bloco de aquecimento. Verifique se as tampas dos tubos estão devidamente fechadas. Aquece-se a 95-100 ° C durante 3-5 min. Faça isso para desnaturar as proteínas e permitir SDS-embrulho ao redor do esqueleto da proteína.
NOTA: Alguns padrões de proteínas não devem ser aquecidos, consulte as instruções do fornecedor. - Ao mesmo tempo, transferir o tampão de corrida para a câmara de electroforese em gel. O teor de tampão de corrida 1x é Tris 25 mM, glicina 192 mM, SDS a 0,1% a pH 8,3 (com HCl).
- Corrigir o prefabricados de 4-20% de gradiente de gel de Tris-Glicina SDS-(10 cm x 10 cm x 1 mm) para a câmara de electroforese. Remover o pente e lave os poços com uma seringa carregada com o tampão de corrida.
NOTA: O stro concentração do gelngly depende do tamanho e da natureza da proteína expressa modelo. A concentração acima separa a E. coli proteínas extrato bruto, bem como proteínas de baixo peso molecular. modelo géis auto-lançados são adequados também. - Retire os tubos do bloco de aquecimento. Girar o líquido para baixo com um mini centrifugar por 2-3 seg a 2.000 x g. Pouco vortex e repetir girando para baixo por 2-3 seg a 2.000 x g.
- Transferir os 15 mL de padrão de proteína (24 - 48 jig de proteína) e 10 ul (11-22 ug de proteína) de cada amostra para os poços e comece a electroforese. Aqui, SDS-PAGE é realizada a 125 V e 20 - 40 mA durante cerca de 90 min, um protocolo típico.
- Cuidadosamente extrair o gel a partir da cassete. Transferi-lo para a solução de fixação (metanol a 50%, 10% de ácido acético, 40% ddH2O) durante 30 min. CUIDADO! O metanol é tóxico por inalação e em contacto com a pele. Usar luvas de protecção e trabalho sob uma coifa.
- Transferir o gel em solução de coloração (0,025% de azul brilhante de Coomassie G-250, 10% de ácido acético, 90% ddH2O). Mancha por 60 min.
- Transferir o gel em solução de descoloração (10% de ácido acético, 90% ddH2O) para 60-120 min.
NOTA: A fixação, coloração e descoloração fortemente depender do tamanho e da natureza da proteína expressa. Este protocolo é aplicável a uma ampla gama de proteínas de pesos moleculares relativamente pequenas 46. - Deslocar o gel sobre uma transparência mate sobre um fundo branco, que gera um contraste adequado para as bandas de proteínas coradas.
- Descongelar o padrão de proteína em gelo. Certifique-se de que ele é composto por proteínas com pesos moleculares semelhantes ao modelo de proteína expressa pela sua localização no gel (passo 4.1.13).
- A purificação das 45 proteínas marcadas no seu modelo de as reacções de células livres
NOTA: Para a purificação de proteínas, existem vários métodos que entregam resultados semelhantes. Este protocolo purifica proteínas expressas modelo livre de células que têm uma etiqueta de poli-histidina C-terminal (His-tag). Ele usa um kit de purificação adequado para proteínas que são expressed nos pequenos volumes de reacção de expressão da proteína isenta de células. Ele é idêntico para a purificação de proteínas modelo nativos ou modificados. Assim, é geralmente descrito na base de uma reacção livre de uma única célula.- Para uma análise por espectroscopia de massa de HPLC-ESI apropriado, extrair e purificar as proteínas modelo a partir da reacção isento de células e executar as seguintes etapas.
- Mistura de volumes iguais de 90 - 150 ul de His-tampão de ligação e 90 - 150 ul de reacção isento de células. Pipeta cima e para baixo, seguido por vórtex suave.
NOTA: Usando His-tampão de ligação é recomendado. No entanto, o meio reaccional isento de células pode ser um material de partida, desde que a proteína é solúvel modelo, o pH é entre 7,5 - 8, imidazole / Sua concentração é <10 mM, a concentração de agentes redutores fortes é <15 mM e nenhum metal- agentes quelantes estão presentes. Se o seu volume de mistura excederem 300 ul, dividi-lo em volumes iguais e alíquota teses volumes em r diferentetubos eaction para as seguintes etapas de purificação. - Prepara-se o sistema de coluna. Agitar bem em vórtice a solução estoque do gel His-afinidade até que a resina de gel é completamente dissolvido. Transferir 250 ul de gel de resina na coluna. Use um 1 ml ponta da pipeta para a resina gel viscoso. Colocar a coluna num tubo de recolha.
- Centrífuga tubo de coluna / coleta para 5 - 10 segundos a 13.000 - 15.000 x g. Certifique-se que a resina gel está completamente descarregada. Se não, estender o tempo de centrifugação por adicional de 5 - 10 segundos, mas preste atenção que o gel de afinidade não é sobre-seco. Consequentemente estender o tempo de centrifugação em passos 4.2.1.5, 4.2.1.7 e 4.2.1.9.
NOTA: A resina gel está completamente descarregada se torna-se duro e não sobrenadante permanece no topo. - Transferir 150 - 300 ul de tampão de reacção isento de células / His-se ligar à coluna. Se o volume de mistura excederem o volume recomendado, dividida em várias colunas de spin. Ressuspender a resina gel por frequent tocando e suave vórtex durante um tempo de incubação de pelo menos 2 min. Para volumes maiores que 200 mL, incubar adicional 1-2 min.
NOTA: O tempo de incubação suficiente é crucial para a ligação das proteínas modelo para a resina em gel. - Centrífuga tubo de coluna / coleta para 5 - 10 segundos a 13.000 - 15.000 x g. Descartar o fluxo de passagem e colocar a coluna de volta para o tubo de recolha.
- Adicionar 250 mL de His-tampão de lavagem. Ressuspender a resina gel tocando e vórtex suave.
- Centrífuga tubo de coluna / coleta para 5 - 10 segundos a 13.000 - 15.000 x g. Descartar o fluxo de passagem e colocar a coluna de volta para o tubo de recolha. Repita o passo 4.2.1.6 e centrifugar novamente por 5 - 10 segundos a 13.000 - 15.000 x g.
- Colocar a coluna para um tubo padrão de reacção de 1,5 ml. Adicionar 150 mL de tampão de eluição e ressuspender a resina gel tocando e vórtex suave. Reduzir o volume de 100 ul para maior teor de proteína con modelo purificadacentrações após eluição no passo seguinte 4.2.1.9.
NOTA: A abundância de proteína total de modelo purificado pode ser reduzido devido ao possível eluição incompleta de todas as proteínas ligadas a resina de gel, se o volume do tampão de eluição é reduzida. - Centrifugar tubo de coluna / reacção durante 5 - 10 segundos a 13.000 - 15.000 xg para diluir a proteína purificada no tampão de eluição. Se dividir antes, reunir todas as soluções em uma solução estoque.
- Antes de efectuar a permuta de tampão, determinar a concentração de proteína usando métodos padrão, por exemplo, o Ensaio de Bradford 47,48.
NOTA: Para obter uma espectroscopia de massa de HPLC-ESI apropriado (secção 4.4), utilizar concentrações de proteína óptimas que são geralmente cerca de 0,5 mg / ml, com volumes necessários de 20 - 50 mL. Se a concentração é inferior a 0,2 mg / ml, concentra-se a solução de proteína após a troca de tampão usando um concentrador de vácuo de fiar. Neste caso, primeiro leia a seção 4.3.8 para se adaptar adequadamente o lustre trocaer.
- Mistura de volumes iguais de 90 - 150 ul de His-tampão de ligação e 90 - 150 ul de reacção isento de células. Pipeta cima e para baixo, seguido por vórtex suave.
- Para uma análise por espectroscopia de massa de HPLC-ESI apropriado, extrair e purificar as proteínas modelo a partir da reacção isento de células e executar as seguintes etapas.
- A troca de tampão para a espectroscopia de massa de HPLC-ESI
NOTA: Substituir o tampão de eluição de His para evitar elevado ruído de fundo na análise por espectroscopia de massa devido à elevada concentração de imidazole (> 150 mm) e outros sais, tais como NaH 2 PO 4 (> 300 mm) ou NaCl (> 50 mM) 49. O seguinte protocolo de troca de tampão utiliza um sistema de coluna de centrifugação de filtração em gel pré-hidratada. É executada de forma idêntica para proteínas modelo nativos ou modificados. Assim, é geralmente descrito na base de uma reacção livre de uma única célula. Note-se que existem outros métodos de troca de tampão fáceis de executar, por exemplo, cartuchos de mini-diálise.- Preparação de 100 ml de tampão de armazenagem da proteína (50 mM Tris-Cl pH 8, NaCl 100 mM e glicerol a 10%). Pesar em uma autoclave de 100 ml Frasco 605,7 mg de Tris-Base, 584,4 mg de NaCl e adicione 10 ml de glicerol. Encha até aproximadamente 80 ml e ajusta o valor de pH a 8 por umNaOH dding. Constantemente verificar o valor do pH com um medidor de pH. Finalmente, encha até à marca de 100 ml.
NOTA: Use este tampão para estabilizar uma ampla gama de proteínas de modelo e para não perturbar análise de espectroscopia de massa, desde que HPLC é realizada antes da medição espectroscópica de massa. No entanto, dependendo da natureza da proteína expressa modelo, utilizar outros tampões que podem ser melhor adequados. Se a sua proteína alvo não é estável a pH 8, ajustar o pH de acordo. Não use concentrações de glicerol superiores. - Aquecer as colunas de centrifugação de filtração em gel, durante pelo menos 15 min à TA.
- Ressuspender o gel pré-hidratado agitando ligeiramente ou vórtex e remover as bolhas de ar.
- Primeiro retire a tampa do fundo, e em seguida, tomar a tampa superior de distância. Colocar a coluna no tubo de lavagem (um, pelo menos, 2 ml). Transferir o tubo de coluna / lavagem na centrifugadora. Cada coluna possui uma marca de orientação. Centrifuga-se a 1000 xg durante 2 minutos para remover o tampão de gel de armazenamento. Discoard o fluxo de passagem.
NOTA: Preste atenção para prosseguir nesta ordem. Assegure-se que a coluna tem a mesma orientação em todas as etapas de centrifugação. - Adicione até 400 l do tampão de armazenamento de proteína. Centrifugar durante 2 minutos a 1000 x g. Repetir este para carregar completamente o tampão de armazenamento de proteínas em gel. Cuidadosamente adicionar até 100 ul da solução da proteína purificada modelo. Pipeta directamente para o centro do leito de gel.
NOTA: os volumes de solução de proteínas purificadas, particularmente de proteínas modificadas, poderão ser mais elevadas. Dividir tais soluções ao longo de várias colunas de permuta de tampão. As proteínas devem ter pesos moleculares mais elevados do que 5 kDa para este tipo de troca de tampão. - Colocar a coluna para um tubo de recolha e centrifugação durante 2 min a 1000 x g.
NOTA: Este passo leva à diluição da proteína purificada modelo no tampão de armazenagem da proteína. Se dividir antes, reunir todas as soluções em uma solução estoque. - th congelamento de flashsolução de proteína e em nitrogênio líquido e armazenar a -80 ° C ou diretamente analisá-lo via HPLC-ESI espectroscopia de massa 50 (secção 4.4). CUIDADO! Use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de nitrogênio.
- Execute os seguintes passos do protocolo de concentrar a solução de proteína usando um concentrador de vácuo de fiar que cuidadosamente evapora o solvente 51.
NOTA: Estes passos são necessários se a concentração de proteína é demasiado baixo para a espectroscopia de massa de HPLC-ESI (<0,2 mg / ml). A concentração de proteína volume de solução e modelo é praticamente o mesmo antes e depois da troca de tampão. O processo de concentração é descrito por meio de exemplo, o seguinte: Depois da purificação His-tag, a proteína modelo é dissolvido em 100 ul de tampão de eluição A (passo 4.2.1.9) e tem uma concentração de 0,07 mg / ml (passo 4.2.1.10) .- Para assegurar que a concentração de proteína e o volume da solução é depois evaporada apelo menos 0,2 mg / mL e 20 uL, respectivamente, dilui-se, antes da troca de tampão, o tampão de armazenamento proteína preparada no passo 4.3.1, pelo menos, três vezes, mas, no máximo, cinco vezes. Igualmente evaporar na seguinte, pelo menos, dois terços (66,67 mL) ou, no máximo, quatro quintos (80 mL).
NOTA: A diluição do tampão de armazenagem da proteína antes da troca de tampão é crucial para ter em conta que os valores de concentração deste tampão (passo 4.3.1) são ainda realizado, apesar de evaporação. Após a diluição do tampão de armazenamento de proteínas, primeiro realize a troca de tampão (passos 4.3.2 - 4.3.6) antes de executar os seguintes passos 4.3.8.2 - 4.3.8.4. - Após a troca de tampão, colocar as completos 100 ml de solução de proteína modelo em um tubo aberto para o concentrador de vácuo de fiar.
NOTA: O modelo de proteína é dissolvida na tampão de armazenagem da proteína diluída. Incline-o para o centro de rotação para evitar a perda de líquido durante a rotação. Assegure-se que uma placa aberta do mesmo volume deH 2 O está colocado simetricamente para equilibrar o sistema de fiação. - Fechar a tampa do concentrador e começar a rotação. Para assegurar a estabilidade da proteína, concentrado à RT ou a baixas temperaturas até 30 ° C.
NOTA: O concentrador usado acelera automaticamente a 170 xg e estabelece a vácuo. No seguinte ele acelera a sua velocidade máxima de 240 x g. - interromper freqüentemente o processo de concentração para o levantamento do volume de líquido. Pare a concentração, quando o volume da solução remanescente é entre 20 ul e 33 ul.
NOTA: Assim adaptar os passos 4.3.8.1 - 4.3.8.4 para outros volumes da solução (passo 4.2.1.9) e outras concentrações de proteína (passo 4.2.1.10). Flash congelar a solução concentrada de proteína em azoto líquido e armazenar a -80 ° C, ou directamente analisá-la por meio de HPLC-ESI espectroscopia de massa 50. CUIDADO! Use uma eyeshield e Cryo-luvas para ser protegido contra salpicos de nitrogênio.
- Para assegurar que a concentração de proteína e o volume da solução é depois evaporada apelo menos 0,2 mg / mL e 20 uL, respectivamente, dilui-se, antes da troca de tampão, o tampão de armazenamento proteína preparada no passo 4.3.1, pelo menos, três vezes, mas, no máximo, cinco vezes. Igualmente evaporar na seguinte, pelo menos, dois terços (66,67 mL) ou, no máximo, quatro quintos (80 mL).
- Preparação de 100 ml de tampão de armazenagem da proteína (50 mM Tris-Cl pH 8, NaCl 100 mM e glicerol a 10%). Pesar em uma autoclave de 100 ml Frasco 605,7 mg de Tris-Base, 584,4 mg de NaCl e adicione 10 ml de glicerol. Encha até aproximadamente 80 ml e ajusta o valor de pH a 8 por umNaOH dding. Constantemente verificar o valor do pH com um medidor de pH. Finalmente, encha até à marca de 100 ml.
- HPLC-ESI análise por espectrometria de massa 50 de proteínas modelo
- Realizar a separação de HPLC de 5 - solução de 15 ul de proteína (preparada na secção 4.3) numa coluna C5 de fase reversa (3 um, 100 x 2,1 mm) com um gradiente de 20% - 90% de acetonitrilo e 0,1% de ácido fórmico ao longo do 25 min e subsequente espectrometria de massa de ESI com detecção de um analysator de massa de tempo-de-voo na gama de 300 - 3000 m / z.
NOTA: Dependendo da natureza da proteína expressa modelo, usar outros solventes ou colunas que podem ser melhor adequados. - Deconvolute medida espectros de massa usando software apropriado 52 para calcular massas-carga zero.
- Realizar a separação de HPLC de 5 - solução de 15 ul de proteína (preparada na secção 4.3) numa coluna C5 de fase reversa (3 um, 100 x 2,1 mm) com um gradiente de 20% - 90% de acetonitrilo e 0,1% de ácido fórmico ao longo do 25 min e subsequente espectrometria de massa de ESI com detecção de um analysator de massa de tempo-de-voo na gama de 300 - 3000 m / z.
Resultados
Este protocolo orienta através da incorporação específica do resíduo livre de células de NCAAs em proteínas modelo. Propõe SDS-PAGE para uma avaliação preliminar do experimento constituição e novas medidas para preparar as proteínas modelo para uma análise espectroscópica de massa HPLC-ESI adequada.
Aqui, os resultados representativos da incorporação específica do resíduo isento de células do análogo Arg Can, bem como o análogo de L-Lys hidroxi-lisina (Hyl) são apresentados. As soluções de aminoácidos diferentes, o acumulador de energia, o ADN que codifica para o vector de proteína modelo e reacções livres de células são preparados como descrito acima. A reacção isento de células de referência é fornecida com a solução de amino ácidos consistindo de 20 CAA. Para cada experiência, uma reacção isento de células de controlo negativo é fornecido com a solução de aminoácido que não possui o análogo canónica do NCAA em questão. Para cada aproxoach, uma reacção isento de células exprime a proteína modelo na presença da solução de aminoácidos, em que a CAA é substituído por o análogo não-canônico. His-marcador de purificação, a troca de tampão e a análise por espectrometria de massa de HPLC-ESI são executadas de acordo com o protocolo descrito acima.
A proteína é o modelo do terminal C marcada com His deGFP 32, uma versão truncada da EGFP 53. A sua carta uma sequência de aminoácidos podem ser encontradas em (material suplementar 2). Esta proteína contém 6 modelo Arg e Lys 18 posições, respectivamente. O vector de expressão é pBEST-OR2-OR1-Pr-UTR1-deGFP-T500.
No caso de incorporação completa da NCAA, pode-se supor que a reacção de controlo negativo não expressa deGFP, uma vez que um dos 20 CaaS está faltando. Contrariamente, deve ser deGFP detectável nas outras duas reacções: a uma nativa na referência de reacção isento de células e a proteína modificada na reacção isento de células que é fornecido com o NCAA.
A Figura 1A mostra a avaliação preliminar de SDS-PAGE da experiência de incorporação pode. A reacção isento de células de referência tem o nível de expressão mais elevado deGFP. Na reacção isento de células que é fornecido com Can, deGFP é expresso na concentração ligeiramente inferior. Nenhuma expressão deGFP pode ser detectado no controlo negativo. Este resultado SDS-PAGE é uma boa indicação para uma incorporação bem sucedida de Can no deGFP proteína alvo.
Para provar a incorporação completa hipótese de lata em deGFP, ambas as proteínas purificadas modelo, visualizados na Figura 1B, são analisadas através de espectroscopia de massa HPLC-ESI. Figura 1C mostra o espectro de massa deconvoluídos das moléculas deGFP purificados. A massa deconvoluídos de degF P, que é expresso na reacção isento de células de referência é 26,192.8 Da. Para deGFP expresso na lata contendo reacção livre de células de uma massa de 26202,5 Da aparece. As massas esperado para o deGFP6Arg nativo eo deGFP6Can modificado com Arg ser totalmente substituída por Can são 26.193 Da e 26204 Da, respectivamente. A diferença de massa de 1,5 Da para deGFP6Can está dentro do erro de desconvolução espectro. Assim, a incorporação plena da lata em deGFP em todas as posições 6 Arg é confirmada.
Os dois picos de intensidade reduzida correspondem ao deGFP6Arg nativa e deGFP6Can modificadas que não atingiram os seus fluoróforo maduro. O fluoróforo é autocataliticamente gerado por eliminação de uma molécula de H2O, seguido de oxidação. Isto leva a uma massa aumentada por 20 Da Se este processo não prossiga.
pg "/>
Figura 1. A avaliação de SDS-PAGE da experiência é possível a incorporação e espectroscopia de massa de HPLC-ESI das moléculas deGFP expressos e purificados isentos de células. (A) Avaliação Preliminar do experimento pode incorporação utilizando SDS-PAGE. Da esquerda para a direita: padrão de proteína, reação livre de células de referência, de controlo negativo e reação livre de células fornecer pode, em vez de Arg. (B) SDS-PAGE após His-tag purificação e troca de tampão das moléculas deGFP expressas. Da esquerda para a direita: padrão de proteína, deGFP purificada a partir da reação de referência, purificado deGFP a partir da reação Can contendo. (C) A confirmação da plena incorporação de Can por espectroscopia de massa HPLC-ESI. As massas esperados do deGFP nativo eo deGFP modificado com Arg totalmente substituída por Can são 26.193 Da e 26204 Da, respectivamente. Cada espectro é normalizado para a sua maior intensidade (contagem). as posições dos picos são indicadasem Da. Para efeitos de visualização, as pistas de gel são extraídos a partir das imagens do gel, unidas entre si, são convertidos em formato de escala cinzenta, tamanho é optimizada e de contraste, assim como brilho são reforçadas. Pistas de gel originais são apresentados no material suplementar 3. Por favor, clique aqui para ver uma versão maior desta figura.
A Figura 2A mostra a avaliação preliminar de SDS-PAGE da experiência incorporação Hyl. Na reacção isento de células que é fornecido com Hyl, deGFP é expresso. Contrariamente ao primeiro experimento, uma banda deGFP fraco pode ser observado na reacção de controlo negativo. Isto pode ser devido à presença de resíduos Lys nas reacções isentos de células. Isso permite que uma expressão deGFP fraco na reacção de controlo negativo, onde nem Lys nem Hyl são adicionados.
para H Espectroscopia de massa-ESI PLC, as moléculas deGFP da reacção isento de células fornecidos com Hyl são purificadas e o tampão é trocado (Figura 2B).
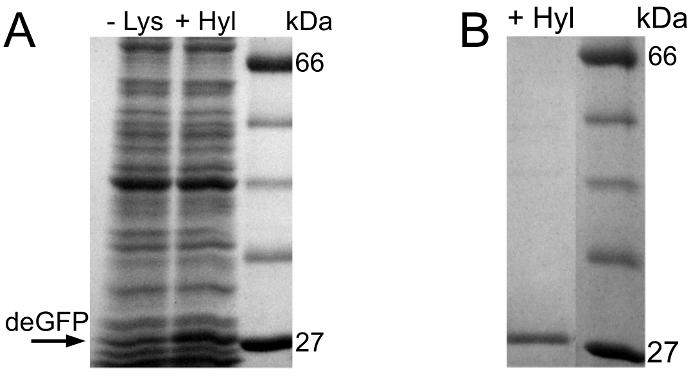
Figura avaliação 2. SDS-PAGE do experimento incorporação Hyl (A) Da esquerda para a direita:. Reacção livre de células de controlo negativo, a reação livre de células contendo Hyl e padrão de proteína. (B) SDS-PAGE após His-tag purificação e troca de tampão das moléculas deGFP expressas. Da esquerda para a direita: Purified deGFP do Hyl contendo reação e padrão de proteína. Para efeitos de visualização, as pistas de gel são extraídos a partir das imagens do gel, unidas entre si, são convertidos em formato de escala cinzenta, tamanho é optimizada e de contraste, assim como brilho são reforçadas. pistas de gel originais são apresentados no material suplementar 3.f = "https://www.jove.com/files/ftp_upload/54273/54273fig2large.jpg" target = "_ blank"> Clique aqui para ver uma versão maior desta figura.
A Figura 3 mostra o espectro de massa de moléculas deconvoluídos deGFP purificadas. A Figura 3A confirma a hipótese de os resíduos de Lys já presentes nas reacções isentos de células. O pico predominante do espectro corresponde ao nativo (massa esperada: 26,193 Da) deGFP. Mais uma vez, as moléculas de um deGFP Da maior massa 20 que não desenvolvem a sua fluoróforo pode ser detectada. Os resíduos Lys são preferencialmente carregadas no ARNt Lys pela lisil-ARNt-sintetase que conduz a um nível de expressão elevado dos deGFP18Lys nativas.
A diferença de massa entre Hyl e Lys é de 16 Da. Devido à presença de Hyl que está em competição com os resíduos de Lys todas as espécies deGFP possíveis são gerados (deGFP18Hyl,deGFP17Hly + 1Lys, ..., deGFP16Hyl 2Lys +) (Figura 3B). É certo que, o pico de deGFP1Hyl + 17Lys sobrepõe-se com o pico do deGFP nativo que não produzem a sua fluoróforo (Figura 3A) e a massa de alguns picos difere mais do que 2 Da da massa esperada. Estas diferenças de massa pode ser atribuída à alta ruído devido aos baixos valores da espécie deGFP. No entanto, Hyl é geralmente incorporado por o sistema isento de células. Outras melhorias têm de ser feitas para abolir resíduos Lys nas reações livre de células.
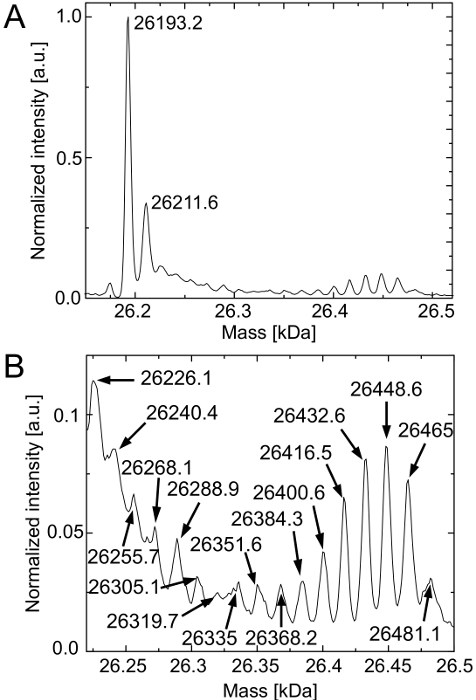
. Figura 3. A HPLC-ESI espectroscopia de massa das moléculas deGFP purificadas da reacção isento de células contendo Hyl (A) Os deGFP18Lys nativas é detectada predominantemente (massas esperadas com fluoróforo: 26193 Da, sem fluoróforo madura 26213 Da). (B)Ampliação revela a existência de todas as espécies deGFP possíveis (deGFP18Hyl, deGFP17Hly + 1Lys, deGFP16Hyl + 2Lys, ..., deGFP1Hyl + 17Lys). Suas massas são esperados 26.193 Da + N x 16 Da (N = 1, ..., 18). O espectro é normalizado para a sua maior intensidade (contagem). As posições dos picos são indicadas em Da. Por favor clique aqui para ver uma versão maior desta figura.

Material Suplementar 1. modelo de cálculo. Na seção 2, a preparação da mistura principal tampão de energia é exemplificado utilizando alíquotas de extrato bruto de 30 ul e óptima MG- e concentrações K-glutamato de, respectivamente, 3 mm e 30 mm. Este exemplo conduz a um volume de mistura principal que produz 3 aliquotas acumulador de energia. no secção 3, a preparação da reacção livre de células é exemplificado usando uma solução de 90 estoque DNA nM levando a uma concentração de DNA vector ideal de 10 nM na reacção livre de células. Por favor clique aqui para baixar esse arquivo.
Para obter uma rastreabilidade adequado, a utilização do modelo de cálculo é exemplificado por inserção destes valores típicos que diferem do exemplo acima: em bruto de volume de extracto: 28? L, óptima de Mg-glutamato: 2 mm, óptima K-glutamato: 40 mm, Número de alíquotas de amortecimento desejadas: 100, concentração ideal do vetor em reação livre de células: 8 nM, DNA solução de stock vector: 150 nM.
Primeiro entre 28 ul como o volume de extrato bruto no campo de laranja da primeira seção do modelo. Em seguida, entrar na segunda seção do modelo 2 mM umd 40 mM como ideal Mg e concentrações de K-glutamato nos campos de laranja. Tendo em conta o óptimo Mg e K-as concentrações, a composição de um acumulador de energia 15 ul, bem como um correspondente, ampliados alíquota de 16 uL é calculado. A seguir, em conformidade entrar 100 como número desejado de alíquotas de acumulador de energia (16 ul). O molde adapta-se os volumes dos diferentes componentes do tampão para a mistura principal de 1700 uL como se segue: 204 uL de solução de estoque de 100 mM de Mg-glutamato, 136 ul de solução de reserva 3 M K-glutamato, 728.73 ul de solução de energia 14x, 510 ul de 40% de PEG-8000 e 121.27 ul estéril DDH 2 O. Finalmente, na terceira secção de molde, entre 8 nM e 150 nM de concentração óptima no vector de reacção isento de células e, respectivamente, o ADN do vector de concentração da solução. O molde adapta-se os volumes dos diferentes componentes que devem ser adicionados aos 28 ul de extracto bruto para finalizar a preparao de um 90 ulreacção isento de células como se segue: 15 uL de tampão de energia, 15 ul de uma das 3 soluções de aminoácidos compostas de maneira diferente, 4,80 ml de solução de ADN do vector (150 nM), e 27.20 ul de ddH 2 O. estéril
Material suplementar 2. Uma carta sequência de aminoácidos da proteína modelo deGFP. Esta proteína modelo contém 6 Arg e 18 posições de Lys. Por favor clique aqui para ver uma versão maior desta figura.
Material suplementar 3. comprimento e pistas de gel não modificados que correspondem às imagens gel apresentou completa Figura 1 e 2. As pistas de gel apresentados em cada s individuais ubfigure são extraídos a partir do mesmo gel de poliacrilamida SDS. Na Figura 1 e 2 estas pistas foram unidas para fins de apresentação. Os pesos moleculares das bandas de proteínas padrão está indicado ao lado das figuras. (A.1) pistas de gel não cortadas da figura 1A. Da esquerda para a direita: padrão de proteína, reação livre de células de referência, de controlo negativo e reação livre de células fornecer pode, em vez de Arg. (A.2) pistas de gel não cortadas da Figura 1B. Da esquerda para a direita: padrão de proteína, deGFP purificada a partir da reação de referência, purificado deGFP a partir da reação Can contendo. (B.1) pistas de gel não cortadas da Figura 2A. Da esquerda para a direita: reacção livre de células de controlo negativo, a reação livre de células contendo Hyl e padrão de proteína. (B.2) pistas de gel não cortadas da figura 2B. Da esquerda para a direita: Purified deGFP do Hyl contendo reação e padrão de proteína.d 54273 / 54273supfig3large.jpg "target =" / _ blank "> Clique aqui para ver uma versão maior desta figura.
Discussão
An-fácil de usar sistema de expressão livre de células como uma estratégia viável ao resíduo especificamente incorporar NCAAs em proteínas, é apresentado. Para este fim, o extracto em bruto é suplementado com codificação de ADN do vector para a proteína de interesse, o acumulador de energia e os aminoácidos correspondentes. Note-se que o volume da alíquota de extracto em bruto depende da concentração de proteína extracto em bruto 34. A eficiência de expressão isento de células é optimizado dependendo da concentração construção de ADN vector. Os volumes dos componentes do tampão de energia variam em função da optimizado Mg- e concentrações de K-glutamato a fim de permitir rendimentos elevados da proteína expressa modelo livre de células.
Uma avaliação preliminar da experiência de incorporação pode ser obtida através da realização de SDS-PAGE do meio reaccional isento de células não purificado. Para uma análise mais detalhada, espectroscopia de massa HPLC-ESI é proposto como um meio para verificar se há incor completa, específica do resíduoporação da NCAA. Como preparação para o último, sistemas de colunas de spin são utilizados para permitir a purificação His-tag e troca de tampão com os pequenos volumes que usamos neste protocolo.
Incluindo espectroscopia de massa de HPLC-ESI, todo o protocolo pode ser realizado no prazo de 2 dias. Ele não inclui quaisquer medidas particularmente críticos. No entanto, optimizações concentração de Mg e K-glutamato, bem como de um vector de ADN são cruciais, de modo a expressar altos rendimentos de proteína modelo. A utilização do vector de expressão altamente eficiente pBEST-OR2-OR1-Pr-UTR1-gene_of_model_protein-T500 é fortemente recomendado. Eluição de marcadas com His proteínas é geralmente devido à alta concentração de imidazol (> 150 mm) e outros sais, tais como NaH 2 PO 4 (> 300 mm) ou NaCl (> 50 mM) que geram alto ruído de fundo na análise espectroscópica de massa 49 . Intercâmbio desses buffers de eluição com um tampão de armazenamento de proteína adequado estabiliza o modelo protee reduz drasticamente o ruído de fundo durante a análise espectroscópica de massa.
Como resultado, é possível substituir Arg na todas as seis posições dentro do modelo de proteína. No sistema de expressão, não há resíduos de Arg pode ser detectada. Isto simplifica a incorporação específica do resíduo de Arg análogos em comparação com outros sistemas de expressão que necessitam de novas estratégias de depleção 29,30. A abordagem livre de células apresentou contorna as limitações inerentes in vivo abordagens que são devido à toxicidade Can, ou a forte dependência da sequência de mRNA em estratégias de produção única de proteínas 24,31. Contrariamente à empregada no sistema in vitro, a clivagem in vivo de Can para homosserina e hydroxyguanidine ocorre 31.
No entanto, o sistema isento de células retém uma quantidade suficiente de Lys para competir com análogos tais como Hyl. A análise de espectroscopia de massa de HPLC-ESI mostra que a proteína modelo contém ambos, a Canonical, bem como o análogo não-canônico em diferentes proporções. A incorporação específica do resíduo de Lys é possível em geral, mas por substituição completa, novas estratégias de esgotamento, ou RAA especialmente projetados e ARNt optimizado para o reconhecimento de NCAAs precisa ser desenvolvida.
Obtivemos excelentes rendimentos de proteínas expressas, modelo modificados isentos de células por adição do NCAA na mesma concentração que os canónicas. A eficácia de incorporação depende da natureza do NCAA a ser incorporados. Mesmo rendimentos mais elevados ainda pode ser realizável através da optimização da concentração da NCAA.
Os resultados apresentados demonstram a aplicabilidade do sistema utilizado para a incorporação específica do resíduo de NCAAs enquanto eles são aceitos pelo sistema translacional endógena canônico. Para a incorporação específica do resíduo de NCAAs específicos, mais um necessidades para verificar se os resíduos da distur CAA análogaB, o sistema de expressão.
Sistemas de transcrição-tradução isentos de células podem ser manipuladas de diferentes organismos que respondem a diferentes exigências 54. O toda E. coli mecanismos de transcrição-tradução do sistema isento de células aqui apresentadas permitir a utilização do bacteriófago e E. os promotores de E. coli, e que pode actuar em paralelo ou consecutivamente nas cascatas de 55. A aplicabilidade geral e usabilidade tornar o método uma potente ferramenta para futuras pesquisas em toxicidade de aminoácidos e aplicação terapêutica.
Divulgações
The authors have nothing to disclose.
Agradecimentos
E.G. Worst and A. Ott acknowledge financial support by the Deutsche Forschungsgemeinschaft (DFG) within the collaborative research center SFB 1027 as well as Saarland University. E.G. Worst, A. Ott and V. Noireaux further acknowledge financial aid by the Human Frontiers Science Program Organization (HFSPO). The authors thank Tobias Baumann and Stefan Oehm (Institute of Chemistry, Technische Universität Berlin) for critical reading.
Materiais
| Name | Company | Catalog Number | Comments |
| Protective eyewear | Sigma-Aldrich, St. Louis, USA | Z758841 | |
| Nitrile gloves (size S) | Sigma-Aldrich, St. Louis, USA | Z768960 | Catalog numbers other sizes: Z768979 for M, Z768987 for L and Z768995 for XL |
| Eppendorf Safe-Lock Tube 1.5 ml (PCR clean) | Eppendorf, Hamburg, Germany | 30123.328 | |
| Microbalance Discovery DV114CM | Ohaus, Greifensee, Switzerland | 80104140 | |
| Microspatula (L 6 5/8 in., stainless steel, rod diam. 0.09 in.) | Sigma-Aldrich, St. Louis, USA | Z243213 | |
| L-Canavanine | Sigma-Aldrich, St. Louis, USA | C9758 | Acute toxicity: wear eyeshields, dust mask, protective gloves |
| Hydroxylysine (racemic mixture) | Sigma-Aldrich, St. Louis, USA | H0377 | |
| Cryo-gloves (size S, water resistent) | Sigma-Aldrich, St. Louis, USA | Z183490 | Catalog numbers other sizes: Z183512 for M, Z183520 for L and Z183539 for XL |
| RTS Amino Acid Sampler | Biotechrabbit, Hennigsdorf, Germany | BR1401801 | For homemade preparation of amino acid stock solutions, follow this protocol35 and use the solid amino acid kit LAA21-1KT, L-proline (81709-25G), L-cysteine (30089-25G), L-histidine (53319-25G) and L-lysine (L5501-5G) (all from Sigma-Aldrich, St. Louis, USA) |
| HEPES | Sigma-Aldrich, St. Louis, USA | H6147 | |
| ATP | Sigma-Aldrich, St. Louis, USA | A8937 | |
| CTP | Affymetrix, Santa Clara, USA | 14121 | |
| GTP | Affymetrix, Santa Clara, USA | 16800 | |
| UTP | Affymetrix, Santa Clara, USA | 23160 | |
| tRNA (from E. coli, pack size 100 mg) | Sigma-Aldrich, St. Louis, USA | 10109541001 | Catalog number for pack size of 500 mg is 10109550001 |
| CoA | Sigma-Aldrich, St. Louis, USA | C4282 | |
| NAD (from yeast ) | Sigma-Aldrich, St. Louis, USA | N6522 | |
| cAMP | Sigma-Aldrich, St. Louis, USA | A9501 | |
| Folinic acid | Sigma-Aldrich, St. Louis, USA | F7878 | |
| 3-PGA | Sigma-Aldrich, St. Louis, USA | P8877 | |
| Mg-glutamate | Sigma-Aldrich, St. Louis, USA | 49605 | |
| K-glutamate | Sigma-Aldrich, St. Louis, USA | G1149 | |
| pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 | Addgene, Cambridge, USA | Plasmid #40019 | |
| 4-20% precast Tris-Glycine Gels (10 cm x 10 cm x 1 mm, 10 courses) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 81610 | |
| SDS running buffer (10 x concentrate, 5,000 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 50001 | |
| SDS loading buffer (2 x concentrate, 50 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 05002 | |
| Unstained protein marker, broad range (2-212 kDa) | New England Biolabs, Ipswich, USA | P7702S | |
| Methanol | Merck, Darmstadt, Germany | 1060091011 | Toxic by inhalation, in contact with skin and if swallowed: wear protective gloves and work under fume hood |
| Acetic acid (99.8%) | VWR International, Darmstadt, Germany | 20104.447 | |
| Coomassie Blue G-250 (10 g) | Biozym Scientific, Hessisch Oldendorf, Germany | 902120 | |
| His-Spin Protein Miniprep kit | Zymo Research Europe, Freiburg, Germany | P2002 | Product also distributed by Zymo Research Corporation, Irvine, USA |
| Trizma Base | Sigma-Aldrich, St. Louis, USA | T1503 | |
| Hydrochloric acid | Sigma-Aldrich, St. Louis, USA | H1758 | |
| Glycerol, 99% | VWR International, Darmstadt, Germany | 24397.296DB | |
| CentriPure Z25 mini spin columns | Genaxxon bioscience, Ulm, Germany | CP-0205-Z100 | |
| Sodium chloride | Sigma-Aldrich, St. Louis, USA | S9888 | |
| Concentrator 5301 | Eppendorf, Hamburg, Germany | 5301 000.210 | |
| 2xYT | MP biomedicals, Santa Ana, USA | 113012032 | |
| Bacto-Agar | BD Diagnostics, Franklin Lakes, USA | 214010 | |
| Bead-beating tubes (polypropylene microvials) | Biospec, Bartlesville, USA | 522S | |
| Beads, 0.1 mm dia. | Biospec, Bartlesville, USA | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Merck, Darmstadt, Germany | 71402 | |
| Bradford BSA protein assay Kit | Bio-Rad, München, Germany | 500-0201 | |
| Chloramphenicol | Sigma-Aldrich, St. Louis, USA | C1919 | |
| Cuvettes, 1.5 ml | Thermo Fisher Scientific, Waltham, USA | 14-955-127 | |
| DTT | Sigma-Aldrich, St. Louis, USA | D0632 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad, München, Germany | 732-6204 | |
| Nunc 384-well optical bottom plates | Thermo Fisher Scientific, Waltham, USA | 142761 | |
| Nunc sealing tape | Thermo Fisher Scientific, Waltham, USA | 232701 | |
| PEG-8000 | Promega, Madison, USA | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich, St. Louis, USA | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich, St. Louis, USA | P8709 | |
| Slide-A-Lyzer dialysis cassettes, 10k MWCO (Kit) | Thermo Fisher Scientific, Waltham, USA | 66382 | |
| Spermidine | Sigma-Aldrich, St. Louis, USA | 85558 | |
| 1 L centrifuge bottle | Beckman-Coulter, Brea, USA | A98813 | |
| 4 L Erlenmeyer flask | Kimble Chase, Vineland (NJ), USA | 26500-4000 | |
| Avanti J-26XP centrifuge | Beckman-Coulter, Brea, USA | 393127 | Or equivalent centrifuge able to centrifuge 1 L bottles. |
| Forma 480 orbital shaker | Thermo Fisher Scientific, Waltham, USA | 480 | Or equivalent shaker able to shake chest-size 6 x 4 L . |
| JLA-8.1000 rotor | Beckman-Coulter, Brea, USA | 363688 | Or equivalent 5,000 x g rotor for the centrifuge above, able to centrifuge 1 L bottles. |
| Mini-Beadbeater-1 | Biospec, Bartlesville, USA | 3110BX | |
| Microfuge 22R refrigerated microcentrifuge | Beckman-Coulter, Brea, USA | 368831 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| Heating block HLC HBT 130 | Labexchange, Burladingen, Germany | 24465 | Or equivalent heating block able to heat samples in reaction tubes up to 100 °C. |
| Eppendorf MiniSpin centrifuge | Eppendorf, Hamburg, Germany | 5452000018 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| IKA Vortex 3 (4 mm orbital shaker diameter, 0 - 2,500 rpm) | Sigma-Aldrich, St. Louis, USA | Z654760 | Or equivalent vortex. |
| Scotsman AF103 ice flaker machine | Kälte-Berlin, Berlin, Germany | AF103 | Or equivalent ice flaker machine. |
| MyTemp mini digital incubator | Sigma-Aldrich, St. Louis, USA | Z763314 | Or equivalent incubator able to heat samples at 29 °C. |
| EcoCell electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12005 | Or equivalent electrophoresis chamber able to perform vertical gel electrophoresis with the above precast gels or other gels used. |
| Power-phor power supply for electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12001 | Or equivalent power supply able to supply above used electrophoresis cell / chamber with power |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 1 | VWR International, Darmstadt, Germany | 613-5278 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 2 | VWR International, Darmstadt, Germany | 613-5279 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D10ST (0.1 - 10 µl) | Gilson, Middleton, USA | F171101 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D200ST (2 - 200 µl) | Gilson, Middleton, USA | F171301 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D1000ST (100 - 1,000 µl) | Gilson, Middleton, USA | F171501 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| 50 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 50-0156 | |
| 15 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 525-0150 | |
| 14 ml polypropylene tubes (round bottom, two-position vent stopper, sterile) | Greiner Bio-One, Frickenhausen, Germany | 187262 | |
| Discovery BIO Wide Pore C5 HPLC Column (3 µm particle size, L x I.D. 10 cm x 2.1 mm) | Sigma-Aldrich, St. Louis, USA | 567227-U | |
| Agilent 1260 HPLC machine | Agilent Technologies, Santa Clara, USA | G1312B | |
| 6500 Series Accurate-Mass Quadrupole Time-of-Flight (Q-TOF) LC/MS | Agilent Technologies, Santa Clara, USA | G6530BA | |
| Acetonitrile | Sigma-Aldrich, St. Louis, USA | 270717 | |
| FLUOstar Omega microplate reader | BMG Labtech, Ortenberg, Germany | 415-101 | Or equivalent microplate reader able to measure the fluorescence of the expressed model protein |
| Hanna Checker pH meter | Sigma-Aldrich, St. Louis, USA | Z351091 | |
| Formic acid eluent additive for LC-MS | Sigma-Aldrich, St. Louis, USA | 56302 |
Referências
- Böck, A., et al. Selenocysteine: the 21st amino acid. Mol. Microbiol. 5 (3), 515-520 (1991).
- Srinivasan, G., James, C. M., Krzycki, J. A. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA. Science. 296 (5572), 1459-1462 (2002).
- Budisa, N. Prolegomena to Future Experimental Efforts on Genetic Code Engineering by Expanding Its Amino Acid Repertoire. Angew. Chem. Int. Ed. Engl. 43, 6426-6463 (2004).
- Wang, L., Xie, J., Schultz, P. G. Expanding the Genetic Code. Annu. Rev. Biophys. Biomol. Struct. 35, 225-249 (2006).
- Chin, J. W. Expanding and Reprogramming the Genetic Code of Cells and Animals. Annu. Rev. Biochem. 83, 379-408 (2014).
- Neumann, H. Rewiring translation - Genetic code expansion and its applications. FEBS Lett. 586 (15), 2057-2064 (2012).
- Liu, C. C., Schultz, P. G. Adding New Chemistries to the Genetic Code. Annu. Rev. Biochem. 79, 413-444 (2010).
- Goerke, A. R., Swartz, J. R. High-Level Cell-Free Synthesis Yields of Proteins Containing Site-Specific Non-Natural Amino Acids. Biotechnol. Bioeng. 102 (2), 400-416 (2009).
- Albayrak, C., Swartz, J. R. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation. Nucleic Acids Res. 41 (11), 5949-5963 (2013).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr. Opin. Chem. Biol. 14 (6), 774-780 (2010).
- Xiu, X., Puskar, N. L., Shanata, J. A. P., Lester, H. A., Dougherty, D. A. Nicotine binding to brain receptors requires a strong cation-pi interaction. Nature. 458 (7237), 534-537 (2009).
- Grünewald, J., et al. Mechanistic studies of the immunochemical termination of self-tolerance with unnatural amino acids. Proc. Natl. Acad. Sci. USA. 106 (11), 4337-4342 (2009).
- Nikić, I., Lemke, E. A. Genetic code expansion enabled site-specific dual-color protein labeling: superresolution microscopy and beyond. Curr. Opin. Chem. Bio. 28, 164-173 (2015).
- Munier, R., Cohen, G. N. Incorporation d'analogues structuraux d'aminoacides dans les protéines bactériennes. Biochim. Biophys. Acta. 21 (3), 592-593 (1956).
- Lepthien, S., Merkel, L., Budisa, N. In Vivo Double and Triple Labeling of Proteins Using Synthetic Amino Acids. Angew. Chem. Int. Ed. 49 (32), 5446-5450 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). Proc. Natl. Acad. Sci. USA. 103 (25), 9482-9487 (2006).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat. Neurosci. 13 (7), 897-905 (2011).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Hoesl, M. G., et al. Lipase Congeners Designed by Genetic Code Engineering. ChemCatChem. 3 (1), 213-221 (2011).
- Bae, J. H., et al. Expansion of the Genetic Code Enables Design of a Novel "Gold" Class of Green Fluorescent Proteins. J. Mol. Biol. 328 (5), 1071-1081 (2003).
- Schachtele, C. F., Rogers, P. Canavanine death in Escherichia coli. J. Mol. Biol. 14 (2), 474-489 (1965).
- Rosenthal, G. A. The biological effects and mode of action of L-canavanine, a structural analogue of L-arginine. Q. Rev. Biol. 52 (2), 155-178 (1977).
- Rosenthal, G. A., Dahlman, D. L. Incorporation of L-Canavanine into Proteins and the Expression of Its Antimetabolic Effects. J. Agric. Food Chem. 39 (5), 987-990 (1991).
- Ishida, Y., Park, J. H., Mao, L., Yamaguchi, Y., Inouye, M. Replacement of All Arginine Residues with Canavanine in MazF-bs mRNA Interferase Changes Its Specificity. J. Biol. Chem. 288 (11), 7564-7571 (2013).
- Thomas, D. A., Rosenthal, G. A., Gold, D. V., Dickey, K. Growth Inhibition of a Rat Colon Tumor by L-Canavanine. Cancer Res. 46 (6), 2898-2903 (1986).
- Bence, A. K., Worthen, D. R., Adams, V. R., Crooks, P. A. The antiproliferative and immunotoxic effects of L-canavanine and L-canaline. Anticancer Drugs. 13 (3), 313-320 (2002).
- Bence, A. K., Crooks, P. A. The Mechanism of L-Canavanine Cytotoxicity: Arginyl tRNA Synthetase as a Novel Target for Anticancer Drug Discovery. J. Enzyme Inhib. Med. Chem. 18 (5), 383-394 (2003).
- Akaogi, J., et al. Role of non-protein amino acid L-canavanine in autoimmunity. Autoimmun. Rev. 5 (6), 429-435 (2006).
- Singh-Blom, A., Hughes, R. A., Ellington, A. D. An amino acid depleted cell-free protein synthesis system for the incorporation of non-canonical amino acid analogs into proteins. J. Biotechnol. 178, 12-22 (2014).
- Oh, S. -. J., Lee, K. -. H., Kim, H. -. C., Catherine, C., Yun, H., Kim, D. -. M. Translational Incorporation of Multiple Unnatural Amino Acids in a Cell-free Protein Synthesis System. Bioprocess. Eng. 19 (3), 426-432 (2014).
- Worst, E. G., Exner, M. P., De Simone, A., Schenkelberger, M., Noireaux, V., Budisa, N., Ott, A. Cell-free expression with the toxic amino acid canavanine. Bioorg. Med. Chem. Lett. 25 (17), 3658-3660 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. J. Biol. Eng. 4 (8), (2010).
- Chemla, Y., Ozer, E., Schlesinger, O., Noireaux, V., Alfonta, L. Genetically expanded cell-free protein synthesis using endogenous pyrrolysyl orthogonal translation system. Biotechnol. Bioeng. 112 (8), 1663-1672 (2015).
- Sun, Z. Z., Hayes, C. A., Shin, J., Caschera, F., Murray, R. M., Noireaux, V. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. J. Vis. Exp. (79), e50762 (2013).
- Caschera, F., Noireaux, V. Preparation of amino acid mixtures for cell-free expression systems. Biotechniques. 58 (1), 40-43 (2015).
- Green, M. R., Sambrook, J. . Molecular Cloning: A Laboratory Manual. , (2012).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. J. Vis. Exp. (6), e253 (2007).
- Zhang, S., Cahalan, M. D. Purifying Plasmid DNA from Bacterial Colonies Using the Qiagen Miniprep Kit. J. Vis. Exp. (6), e247 (2007).
- Moreno, L. A., Cox, K. L. Quantification of dsDNA using the Hitachi F-7000 Fluorescence Spectrophotometer and PicoGreen Dye. J. Vis. Exp. (45), e2465 (2010).
- Desjardins, P., Conklin, D. NanoDrop Microvolume Quantitation of Nucleic Acids. J. Vis. Exp. (45), e2699 (2010).
- Sukumaran, S. Concentration Determination of Nucleic Acids and Proteins Using the Micro-volume Bio-spec Nano Spectrophotometer. J. Vis. Exp. (48), e2699 (2011).
- Laemmli, U. K. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Hochuli, E., Bannwarth, W., Döbeli, H., Gentz, R., Stüber, D. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nature Biotechnology. 6 (11), 1321-1325 (1988).
- Schägger, H., Aquila, H., Von Jagow, G. Coomassie blue-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for direct visualization of polypeptides during electrophoresis. Anal. Biochem. 173 (1), 201-205 (1988).
- Bradford, M. M. A Rapid Method for the Quantitation of Microgram Quantities of Protein Utilizing the Principle of Protein-Dye Binding. Anal Biochem. 72, 248-254 (1976).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. J. Vis. Exp. (38), e1918 (2010).
- Banerjee, S., Mazumdar, S. Electrospray Ionization Mass Spectrometry: A Technique to Access the Information beyond the Molecular Weight of the Analyte. Int. J. Anal. Chem. 2012, 1-40 (2012).
- Fujiwara, K., Nomura, S. M. Condensation of an additive-free cell extract to mimic the conditions of live cells. PLoS One. 8 (1), e54155 (2013).
- Zhang, Z., Marshall, A. G. A universal algorithm for fast and automated charge state deconvolution of electrospray mass-to-charge ratio spectra. J. Am. Soc. Mass. Spectrom. 9 (3), 225-233 (1998).
- Li, X., Zhang, G., Ngo, N., Zhao, X., Kain, S. R., Huang, C. C. Deletions of the Aequorea victoria Green Fluorescent Protein Define the Minimal Domain Required for Fluorescence. J. Biol. Chem. 272 (45), 28545-28549 (1997).
- Gagoski, D., Polinkovsky, M. E., Mureev, S., Kunert, A., Johnston, W., Gambin, Y., Alexandrov, K. Performance benchmarking of four cell-free protein expression systems. Biotechnol. Bioeng. 113 (2), 292-300 (2016).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. ACS Synth. Biol. 1 (1), 29-41 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados