Method Article
Formación luz mediada y el patrón de hidrogeles para aplicaciones de cultivo celular
En este artículo
Resumen
We describe a sequential process for light-mediated formation and subsequent biochemical patterning of synthetic hydrogel matrices for three-dimensional cell culture applications. The construction and modification of hydrogels with cytocompatible photoclick chemistry is demonstrated. Additionally, facile techniques to quantify and observe patterns and determine cell viability within these hydrogels are presented.
Resumen
Instrucciones químicas han sido investigados para su uso en numerosas aplicaciones de biomateriales, incluyendo la administración de fármacos, la ingeniería de tejidos, y cultivo celular. En particular, las reacciones de click-mediada luz, tales como reacciones tiol-eno y tiol-ino fotoiniciadas, permitir el control espacio-temporal sobre las propiedades del material y permitir el diseño de sistemas con un alto grado de control de la propiedad dirigida por los usuarios. Fabricación y modificación de biomateriales a base de hidrogel utilizando la precisión proporcionada por la luz y la versatilidad que ofrece estas químicas tiol-X Photoclick son de interés creciente, en particular para el cultivo de células dentro, microambientes biomiméticos bien definidos. A continuación, se describen los métodos para la photoencapsulation de las células y la posterior photopatterning de señales bioquímicas dentro de matrices de hidrogel utilizando bloques de construcción modulares y versátiles polimerizados por una reacción Photoclick tiol-eno. Específicamente, un enfoque se presenta para constructing hidrogeles de aliloxicarbonilo (Alloc) reticulaciones de péptidos -functionalized y restos peptídicos colgantes y poli tiol funcionalizado (etilenglicol) (PEG) que se polimerizan rápidamente en presencia de fotoiniciador acylphosphinate litio y dosis citocompatible de larga longitud de onda ultravioleta (UV). técnicas fáciles de visualizar y cuantificar photopatterning la concentración de péptidos añadidos se describen. Además, se establecen métodos para las células de encapsulación, específicamente las células madre mesenquimatosas humanas, y la determinación de su viabilidad y actividad. Mientras que la formación y el patrón inicial de hidrogeles tiol-Alloc se muestran aquí, estas técnicas se pueden aplicar ampliamente a una serie de sistemas de materiales otra luz y radical iniciados (por ejemplo, tiol-norborneno, tiol-acrilato) para generar sustratos estampados.
Introducción
Haga clic en las químicas se utilizan cada vez más en el diseño de materiales para numerosas aplicaciones biomédicas, incluyendo la administración de fármacos, la ingeniería de tejidos, y el cultivo celular controlada, debido a su reactividad citocompatible selectivos y eficientes, y con frecuencia. 1-3 químicas Photoclick que utilizan la luz para activar o iniciar reacciones (por ejemplo, azida-alquino, 4 tiol-eno, 5 y tetrazol-alqueno 6) son de particular interés para la formación o modificación de biomateriales. Un ritmo rápido en condiciones suaves y control de cuándo y dónde se llevan a cabo con la luz hacen que estas reacciones muy adecuados para el control dirigido por el usuario de las propiedades de biomateriales en la presencia de células de 7,8. En particular, las químicas tiol-eno Photoclick se han utilizado para generar biomateriales a base de hidrogel con robustas propiedades mecánicas 5,9 y para la encapsulación de una amplia variedad de tipos de células, incluyendo, pero not limitado a, células madre mesenquimales humanas (hMSC), fibroblastos, condrocitos, y células pancreáticas, con promesa para el cultivo celular y la entrega. 10,11 Además, estas sustancias químicas se han utilizado para el patrón espacial de las señales bioquímicas para imitar aspectos clave de microambientes de células nativas y facilitar las interacciones célula-matriz apropiados, incluyendo la adhesión, diferenciación, y la invasión. 3,12
Para la construcción de hidrogeles tiol-eno con luz, péptidos que contienen cisteína (tiol) se hacen reaccionar con polímeros funcionalizados con acrilatos o norbornenos ( 'eno') para una rápida polimerización, fotoiniciada en condiciones citocompatible. 13 La expansión de esta caja de herramientas, hemos tratado de establecer métodos para la formación del hidrogel con nuevos bloques de construcción versátil y accesible que requiere un procesamiento mínimo sintético o estaban disponibles comercialmente hacia su amplio uso como matrices extracelulares sintéticas.14 En concreto, los péptidos se modifican con aliloxicarbonilo (Alloc) -Protegido lisinas: uno para el colgante, grupos de unión a integrinas para promover la adhesión celular [K (alloc) GWGRGDS = Pep1Alloc] o dos de enlaces cruzados no degradables o de células degradable [K ( alloc) RGKGRKGK (alloc) G o KK (alloc) GGPQGIWGQGK (alloc) K = Pep2Alloc, respectivamente]. Con estas secuencias, se establecieron las condiciones para la reacción rápida (1-5 min) con cuatro brazos poli modificado con tiol (etilenglicol) (PEG4SH) utilizando dosis citocompatible de luz UV de longitud de onda larga (10 mW / cm2 a 365 nm) y el fotoiniciador de litio fenil-2,4,6-trimethylbenzoylphosphinate (LAP). Los hidrogeles resultantes eran estables bajo condiciones de cultivo celular durante semanas. Para habilitar la degradación impulsado por células y remodelación, un péptido enzimáticamente escindible se incorporó dentro de las reticulaciones de gel (es decir, GPQGIWGQ), 15 y una célula primaria modelo, las células madre mesenquimatosas humanas (hMSC), se mantuvo altamente viable después de la encapsulación y during cultura dentro de estas matrices. Además, los péptidos se han modelado espacialmente dentro de estos materiales, y hMSCs siendo viable y metabólicamente activa en condiciones photopatterning. Secuencias de péptidos colgante alternativas, no mostrados aquí (por ejemplo, IKVAV, YIGSR, GFOGER, etc.), también se puede incorporar dentro de matrices para sondear las interacciones de células adicionales con el microambiente circundante. Estos resultados son prometedores para la aplicación de estos materiales a base de hidrogel para (3D) de cultivo de células en tres dimensiones y la entrega a estudiar y las interacciones célula-matriz directos para una variedad de tipos de células.
En esto, los métodos para photoencapsulate células y señales bioquímicas posteriormente photopattern dentro del sistema de hidrogel propuesta se presentan (Figura 1). Las técnicas para observar y cuantificar estos photopatterns también se demuestran: en particular, i) el uso cuantitativo y cualitativo de ensayo de Ellman para determinar la modifición de tioles libres dentro de sustratos estructurados y ii) la utilización cualitativa complementaria de péptidos fluorescentes (AF488Pep1Alloc) para observar estos patrones en tres dimensiones. Además, los ensayos para determinar la viabilidad (viabilidad / citotoxicidad tinción Live / muerto) y la actividad metabólica (MTS; 3- (4,5-dimetiltiazol-2-il) -5- (3-carboximetoxifenil) -2- (4-sulfofenil) 2H-tetrazolio) se presentan de modo que los usuarios pueden determinar la citocompatibilidad de las condiciones photoencapsulation y photopatterning para diferentes líneas celulares dentro de matrices de hidrogel. Mientras que el protocolo se demuestra para un sistema de Photoclick hidrogel basada en la luz Facile, las técnicas pueden aplicarse a muchos otros sistemas de hidrogel iniciadas por radicales para photoencapsulation y photopatterning en presencia de células.
Protocolo
1. Preparación de Materiales para la Formación de hidrogel
- Sintetizar colgante (Pep1Alloc, AF488Pep1Alloc) y péptidos de reticulación (Pep2Alloc) mediante síntesis de péptidos en fase sólida estándar (SPPS) y técnicas de tiol funcionalizado polímero por procedimiento de tres pasos para la modificación de grupos terminales (PEG4SH). 14,16 Alternativamente, comprar PEG4SH (M n ~ 20 kDa), Pep1Alloc, y Pep2Alloc comercialmente.
- Sintetizar el fotoiniciador (LAP) por la reacción de dos etapas se describe a continuación. 14,17 Realice los pasos de síntesis (1.2.1-1.2.11) en una campana de humos y tenga cuidado al manipular productos químicos (use guantes, prendas y gafas de protección) . LAP también se puede adquirir comercialmente.
- Seco durante todo el material de vidrio en el horno (> 2 h, 80 ° C).
- Añadir una barra de agitación a una sola boca frasco vacío de fondo redondo (100 ml) y la tapa con un tapón.
- Ajustar el matraz sobre una placa de agitación magnética caliente con un soporte universal y la abrazadera.
- yonsert una aguja (18 G) a través del septo y dejar el extremo exterior abierto a la atmósfera. Inserte una segunda aguja conectada a una tubería de gas inerte. Abra la línea de gas inerte (por ejemplo, argón o nitrógeno) y purgar el matraz durante 10-15 min.
NOTA: El sistema se purgó continuamente con gas inerte, de argón o nitrógeno, durante toda la reacción. - Transferencia de 1,5 g (~ 1,4 ml) phenylphosphonite de dimetilo (PRECAUCIÓN) al matraz, utilizando una jeringa con la aguja para perforar a través del septo. Encienda la placa de agitación (velocidad media) y tener cuidado de que el contenido no se derramen sobre las paredes del matraz.
- Añadir 1,6 g (~ 1,46 ml) 2,4,6-trimetilbenzoil cloruro de (PRECAUCIÓN) gota a gota al matraz que contiene phenylphosphonite de dimetilo, utilizando una jeringa con una aguja para perforar el tabique.
- Cubrir el recipiente de reacción con papel de aluminio para protegerlo de la luz y se agita durante 18 horas en atmósfera de argón o nitrógeno.
- Al día siguiente, elevar la altura del frasco, coloque un baño de aceite en el agitador,nd bajar con cuidado el frasco en el baño de aceite. Calentar el baño y el matraz a 50 ° C mientras se mantiene agitación magnética.
- Disolver 3,05 g de bromuro de litio en 50 ml de 2-butanona. Elevar el matraz del baño de aceite y añadir la solución de bromuro de litio al matraz de fondo redondo, eliminando brevemente el tabique para verter en el frasco.
- Sellar el matraz de nuevo con el tabique (PRECAUCIÓN: Septum todavía tendrá una aguja que lleva a las líneas de argón y una aguja para ventilar), bajar el matraz de nuevo en baño de aceite calentado, y permitir que la reacción transcurra durante 10 min. se forma un precipitado sólido.
- Después de 10 minutos, apagar el argón, retirar el matraz del calor, y permita reposar la mezcla durante 4 horas (cubierto con papel de aluminio para protegerlo de la luz como iniciador sensible a la luz se ha producido. Mantenga el lugar de la aguja de ventilación.
- Verter el producto en un embudo de frita de vidrio o un embudo forrado con papel de filtro apropiado. Enjuague de filtrado 3 veces con 50 ml de 2-butanona para rquítelo cualquier bromuro de litio sin reaccionar.
- Seco (por primera vez en sobremesa y luego en un desecador de vacío) y analizar el producto mediante RMN de 1H en D2O picos característicos a 1.8 a 1.9 (6H, s), 2.1 a 2.2 (3H, s), 6.7 a 6.8 (2H, s), 7.3 a 7.4 (2H, m), 7.4 a 7.5 (1H, m), y 7,5 -7,7 (2H, m). 14,17
- Use una hoja de cálculo para calcular el volumen y la concentración de cada solución madre (PEG4SH, Pep1Alloc, Pep2Alloc, LAP) que deben estar preparados para hacer hidrogeles (Tabla 1). Para hacer geles no modelada, asegúrese de que [SH] = [Alloc] para que todos los grupos reactivos se consumen durante la polimerización. Si se planea photopatterning de geles, incluir una cantidad en exceso de grupos funcionales tiol durante la formación de gel sobre la base de la estequiometría (por ejemplo, 0,2 a 5 mM, [SH]> [Alloc]) para la reacción posterior con Pep1Alloc.
NOTA: Los geles debe contener más de 5 por ciento en peso (% en peso) PEG4SH para asegurar una rápida polimerización (a menos de 5 min). Sin embargo, en peso inferior rango%s se pueden explorar si la aplicación requiere de hidrogeles con módulos inferiores (por ejemplo, <0,5 kPa); polimerización debe comprobarse y ajustarse en consecuencia para estas composiciones% en peso de bajas. Del mismo modo, la concentración Pep1Alloc se puede ajustar para diferentes aplicaciones (por ejemplo, 0,2 a 5 mM) como se informa en la literatura para péptidos tiol funcionalizado. Se recomienda 18-21 concentración vuelta alrededor de 0,067% en peso (2,2 mM) o menos, como se describe, como concentraciones más altas pueden reducir la viabilidad celular. - Preparar soluciones madre de Pep1Alloc, Pep2Alloc, PEG4SH, y LAP bajo condiciones estériles para el cultivo de células en base a los cálculos de la Tabla 1.
- Para Pep1Alloc y Pep2Alloc, pesan individualmente la masa total de Pep1Alloc y Pep2Alloc de la síntesis de péptidos y se disuelven en solución salina estéril tamponada con fosfato (PBS) que contiene 1% de penicilina / estreptomicina (PS) y 0,5 mg / ml de Fungizone (FZ). Las concentraciones típicas de preparados gama de soluciones madre20-100 mg / ml de péptido. Mezclar estas poblaciones para lograr geles con módulos firme relacionados para aplicaciones de tejidos blandos (módulo de Young ~ 0,5-5 kPa). 22,23 Alícuota y almacenar a -20 ° C hasta su uso.
- Para PEG4SH, pesar PEG4SH en un tubo estéril de microcentrífuga y se disuelven en PBS + PS + FZ. Las concentraciones típicas de esta gama de solución de reserva de 250 a 430 mg / ml (20-30% en peso PEG4SH).
- Para LAP, LAP pesan en un tubo estéril de microcentrífuga y se disuelve en PBS + PS + FZ a una concentración final de 7,5 mg / ml.
NOTA: Preparación de soluciones frescas de PEG4SH y LAP se recomienda para todos los encapsulación o el experimento de gel para garantizar tiempos de polimerización consistentes.
- Preparar estériles jeringa con punta Moldes para la formación de hidrogeles.
- Corte cuidadosamente las puntas fuera de jeringas de 1 ml utilizando una hoja de afeitar y posteriormente eliminar los émbolos de las jeringas ejes.
- Sumergir los ejes y émbolos de jeringas en etanol al 70% durante 15 minutos unad lugar en una campana de cultivo celular estéril para secar (30 min). Si el exceso de gotas de etanol permanecen en el interior del eje de la jeringa, use el émbolo para empujar hacia fuera.
- Prepare estéril portaobjetos de vidrio Moldes para la formación de hidrogeles.
- Remojar portaobjetos de vidrio (múltiplos de 2) y las juntas de goma (0,254 mm de espesor; 2 piezas pequeñas usadas como cuñas, un rectángulo con los discos troquela, o forma de la montura cuadrada) en etanol al 70% durante 15 minutos, y el lugar en campana de cultivo celular estéril Secar.
- Escudo medio de los portaobjetos esterilizados con revestimiento anti-adhesivo según las instrucciones del fabricante para evitar la adherencia de la parte superior del gel a la superficie de deslizamiento (por ejemplo, si hay 4 diapositivas, 2 toboganes serán recubiertas con anti-adhesivo). Estos servirán como la parte superior del molde portaobjetos de vidrio.
- Calibre la lámpara para moldes de jeringa o portaobjetos de vidrio.
NOTA: Para estos experimentos, se utilizó una lámpara de arco de mercurio con un conjunto de adaptador externo de filtros y 365 nm filtro externo. otra lámparas que producen intensidades apropiadas de luz UV de longitud de onda larga se puede utilizar según lo informado por otros. 24-27- Adjuntar una lente de colimación hasta el final de la guía de luz lleno de líquido para asegurarse de intensidad relativamente uniforme de la luz en todas las muestras. Si es necesario, ajustar la distancia de la guía de luz de la muestra (s) para lograr un tamaño de punto que cubrirá las muestras de interés.
- Colocar un molde de corte de la jeringa en un soporte para tubos de microcentrífuga (para mantener el molde jeringa en posición vertical). Mantenga el radiómetro a la altura de la punta de la jeringa donde se llevarán a cabo las muestras y ajuste del obturador (% abierto) para lograr una intensidad luminosa de 10 mW / cm2 a 365 nm. Grabar los ajustes establecidos para su uso posterior.
- Colocar un molde de lámina de vidrio en la parte superior de una superficie estéril (por ejemplo, la parte superior de una caja de punta de la pipeta dentro de una cabina de bioseguridad). Mantenga el detector radiómetro a la altura del molde portaobjetos de vidrio y ajuste del obturador (% abierto) para lograr una intensidad de luz de 10 mW / cm2 a 365 nm. Grabar los ajustes establecidos para su uso posterior.
2. Formación de hidrogel y photopatterning
- Preparación de hidrogeles con dibujos-no.
NOTA: En este punto en el protocolo, si los hidrogeles son para ser utilizados en aplicaciones de cultivo celular, todas las etapas posteriores se deben realizar en condiciones estériles en una cabina estéril o campana.- Mezclar las soluciones madre de PEG4SH, Pep2Alloc, Pep1Alloc, LAP, y PBS + PS + FZ según el cálculo hoja de cálculo (véase la Tabla 1 ejemplo). Pipetear la solución de precursor de gel resultante vigorosamente para asegurar una mezcla uniforme de la solución.
- Preparar hidrogeles gruesas moldeadas en puntas de jeringa con la pipeta 10-20 l de la solución de precursor del gel (PEG / Pep / LAP / PBS) en la punta de una jeringa estéril de corte. Hacer un solo gel por jeringa, aproximadamente 0,5-1,5 mm de espesor basan en el volumen y el diámetro de la jeringa.
NOTA: Los volúmenes más grandes pueden ser explorado basan enel tamaño de gel deseada en la que los límites superiores puedan existir sobre la base de las limitaciones difusionales para Pep1Alloc durante photopatterning y / o nutrientes / desechos a / a partir de células encapsuladas durante el cultivo celular. - Preparar hidrogeles delgadas en moldes portaobjetos de vidrio mediante la colocación de la junta de goma (0,254 mm de espesor) alrededor de los bordes de la lámina de vidrio no revestida. Pipetear 5 a 10 l solución de precursor de gel (5-10 geles individuales o múltiples mu l se puede hacer pipeteando uno o más puntos de la solución en una sola diapositiva) sobre el portaobjetos de vidrio no revestido y colocar el portaobjetos de vidrio recubierto con anti-adhesivo en la parte superior de la solución de gel (geles de mayor tamaño, hasta el tamaño de la lámina de vidrio, se pueden hacer dependiendo de la aplicación final). Asegurar los portaobjetos de vidrio con pequeños clips de la carpeta para estabilizar.
- Coloque los moldes bajo la lámpara y ajustar la intensidad de la lámpara (por ejemplo,% obturador abierto) para conseguir 10 mW / cm2 a la superficie del gel para moldes punta de la jeringa o moldes deslizantes de vidrio basado en mediciones en el paso 1.7.2 y1.7.3, respectivamente.
- Aplicar la luz durante 1 a 5 minutos para permitir la completa polimerización de geles. Utilice un tiempo de polimerización más corto para los geles con mayor contenido PEG4SH (8% en peso o mayor, 1 min) y más largo para geles con menor contenido de PEG4SH (5-8% en peso, 3-5 min) para producir hidrogeles polimerizados completamente con módulos dentro de la gama de los tejidos blandos (0,5 a 5 kPa).
- Coloque los geles de moldes punta de la jeringa en una placa estéril, no tratados, de 48 pocillos, y las diapositivas de vidrio en lugar de un plato estéril.
- Enjuague 3 x 15 min con medio de cultivo celular o tampón apropiado (por ejemplo, PBS + PS + FZ, tampón de reacción de Ellman), basado en experimentos planificados, como se detalla a continuación.
- Preparación de hidrogeles con dibujos.
- Mezclar las soluciones madre de PEG4SH, Pep2Alloc, LAP, y PBS + PS + FZ según el cálculo de hoja de cálculo, dejando tioles libres (0,2-5 mm) para una posterior reacción con Pep1Alloc. Pipetear la solución de precursor vigorosamente para asegurar una mezcla uniforme de la solución.
- Preparar hidrogeles gruesas y delgadas, según constan en los pasos 2.1.2 a 2.1.6.
NOTA: No coloque geles en medio de crecimiento antes de la photopatterning. Tioles libres pueden ser consumidos por especies en el medio de crecimiento (por ejemplo, la formación de disulfuro con las proteínas que contienen tiol) y no permitirá que el patrón del gel sin etapas de procesamiento adicionales (por ejemplo, la reducción de los enlaces disulfuro). - Se preparan soluciones de Pep1Alloc (concentración final ~ 3-20 mg / ml) y LAP 2,2 mM.
- Cubra geles preformados con solución / LAP Pep1Alloc y se incuba durante 1 hora a 37 ° C.
- Retire la solución Pep1Alloc / LAP exceso. Si geles se moldearon en una punta de la jeringa, utilizar una espátula para transferir cuidadosamente los geles de la placa de 48 pocillos en la que se están incubando a un portaobjetos de vidrio estéril para patrones.
- Colocar una fotomáscara con el patrón deseado directamente en la parte superior de los geles de diapositivas-moldeado con jeringa y de vidrio. Asegúrese de que la parte impresa de la máscara toca el gel para Patt óptimafidelidad ern (es decir, la máscara debe leer bien y sea lado de la emulsión hacia abajo). Colocar las muestras bajo la lámpara y irradian durante 1 minuto con la configuración de lámpara utilizadas para portaobjetos de vidrio (paso 1.7.3).
- Después de modelar, geles lugar jeringa-moldeado en una placa tratada, la cultura no-tejidos estériles de 48 pocillos, y el lugar de vidrio geles que se adhieren a las placas de vidrio en una placa estéril deslizable moldeado.
- Enjuague 3 x 15 min con medio de cultivo celular o tampón apropiado (por ejemplo, PBS + PS + FZ, tampón de reacción de Ellman), basado en experimentos planificados.
3. La visualización y cuantificación de photopatterning
- Ensayo de Ellman para detectar y cuantificar los tioles libres en Photopatterned hidrogeles.
- Preparar tampón de reacción de Ellman, solución de trabajo de cisteína, normas, y el reactivo de Ellman, como se describe a continuación.
- Para de Ellman tampón de reacción, se disuelven 2,4 g de fosfato dibásico de sodio (Na 2 HPO4) y 74,4 mg de ácido etilendiaminotetraacético (EDTA) en 200 ml DIH 2 O (0,1 M Na 2 HPO 4, EDTA 1 mM). Ajustar el pH a 7,5-8 con soluciones de hidróxido de sodio (NaOH) o ácido fosfórico (H 3 PO 4).
- Para la solución de trabajo cisteína, disolver 5,27 mg de cisteína en 15 ml de tampón de reacción (cisteína 2 mM).
- Para Normas de cisteína, diluir la solución de trabajo de cisteína en tampón de reacción a una concentración final de 2, 1,5, 1,25, 1, 0,75, 0,5, 0,25, y 0 cisteína mM.
- Para el reactivo de Ellman, disolver 4 mg de reactivo de Ellman en tampón de reacción de 1 ml. Someter a ultrasonidos para disolver completamente el reactivo de Ellman.
- Cuantificar la concentración de tioles libres en geles con el ensayo de Ellman (Figura 2).
NOTA: 'Thin' 5 l hidrogeles, moldeado entre portaobjetos de vidrio, se describen en el siguiente procedimiento y recomendó para permitir la difusión rápida del reactivo de Ellman tediante el gel (es decir, se necesita el reactivo de más tiempo para difundirse a través de geles de espesor).- Determinar el volumen de gel hinchado de geles 'finas' 5 l (V S) en base a la relación volumétrica de hinchamiento, P.
- Hacer tres 20 'gruesas' geles l utilizando moldes de jeringa. Coloque los geles en tampón de reacción de Ellman durante 24 horas y posteriormente pesan (equilibrio de masa hinchada, H S). Liofilizar los geles y, posteriormente, se tara (peso en seco, M D).
- Uso de las masas medidas de geles espesos, calcular la relación de hinchamiento volumétrico de 28 por Q = 1 + ρ polímero / disolvente ρ (M S / M D -1) en la que polímero ρ = 1,07 g / cm 3 de PEG, 29 ρ disolvente = 1,0 g / cm3 para PBS / H2O
- Calcular la masa seca teórica del gel que se utiliza en el ensayo de Ellman (aquí, geles de 5 l 'delgadas' son típicamente uSED), sumando las masas de los componentes individuales PEG4SH, Pep1Alloc, y Pep2Alloc (M D = M + M PEG4SH Pep1Alloc + M Pep2Alloc). Por ejemplo, un gel de 5 l que contiene 10% en peso PEG4SH contiene aproximadamente 0,000535 g PEG calculado por M PEG4SH = 0.005 cm 3 x 1.07 g / cm 3 x 0,10. Masas Pep1Alloc y Pep2Alloc se pueden calcular de una manera similar sobre la base de% en peso en solución (véase la hoja de cálculo para los valores), suponiendo ρ ≈ 1,0 g / cm 3 de estas soluciones acuosas.
NOTA: Los geles también puede ser secado y pesado en lugar de calcular la masa seca teórico. Sin embargo, puede ser difícil de medir constantemente la masa seca de los finos, geles 5 l. - Sobre la base de la masa seca predicho y el valor Q determinada a partir de geles 'gruesas', calcula la masa hinchada predicho M S para el gel 'delgada' (ecuación en el paso 3.1.2.1.1). Supongamos que ρ ≈ 1,0 g / cm 3, entonces M S V S ≈. Utilice esta V S calculada para llevar a cabo el ensayo de Ellman cuantitativa.
NOTA: Se recomienda el método anterior para determinar V s para geles hinchados como los geles de 5 l 'finas' son difíciles de transferir para el pesaje.
- Formar hidrogeles delgadas para el patrón como se describe en la sección 2.2 (5 l de volumen). En concreto, hacer geles con exceso de tioles libres y la mitad "patrón" de estas muestras con Pep1Alloc utilizando un cubreobjetos clara para inundar exponer todo el gel con luz (por ejemplo, 6 geles totales con exceso de tiol: 3 sin modificar no'patterned 'y 3' estampado').
NOTA: Para este ensayo, 'patrón' geles son expuestos a la luz de inundación sin una fotomáscara. Este método se utiliza para determinar el número total de tioles libres se consumen en toda la muestra de gel y para demostrar la eficiencia con tioles libres puede ser modificado con el péptido. A partir de esta información,el número de tioles libres consumidos durante modelando con una fotomáscara se puede predecir sobre la base del espesor de gel y área de patrón (regiones expuestas a la luz). - Place 'modelada "y no'patterned' geles en pocillos de una placa de 48 pocillos y enjuague 3 x 15 min con tampón de reacción de Ellman para permitir la difusión de las especies que no han reaccionado fuera del gel y el equilibrio de hinchamiento que se produzca.
- Añadir tampón de reacción de Ellman extra para los geles se enjuagaron de manera que V s con tampón de reacción es un múltiplo de 20 l. Por ejemplo, si el V S predicho es 15 l, añadir tampón de reacción 5 l (20 l), y si el V S predicho es 30 l, añadir 10 l de tampón de reacción (40 l).
- Pipetear los estándares de cisteína en los pocillos individuales (que no contienen geles) de una placa de 96 pocillos en múltiplos de 20 l en función del volumen utilizado en la etapa anterior (es decir, si el paso anterior tenía V S + extra reacción bUffer = 40 l, añadir 40 l de cada estándar a los pocillos vacíos individuales).
- Diluir el reactivo de Ellman en tampón de reacción (múltiplos de 180 l de tampón de reacción + 3,6 l de reactivo de Ellman; por ejemplo, para el 20 183,6 367,2 l o l para 40 l en el paso 3.1.2.5).
- Añadir el reactivo de Ellman diluido a los pocillos que contienen los estándares y muestras. Para 20 muestras mu l, añadir 183,6 l solución a cada pocillo. Doble de esta cantidad de reactivo de Ellman diluido para 40 muestras mu l (o escalar en consecuencia en función del tamaño de la muestra).
- Incubar o lugar en un agitador durante 1 hr y 30 min (a temperatura ambiente) o hasta que el color de la solución coincide con el color de los geles por inspección visual para asegurar difusión suficiente de la amarilla dianión 2-nitro-5-tiobenzoato (TNB 2-), que se genera en la reacción del reactivo de Ellman con tioles libres, a partir del gel.
- Tomar 100 l de solución a partir de muestras y sas normas y colocan en pocillos de una placa de 96 pocillos.
- Leer la absorbancia a 405 nm en un lector de placas.
- Para procesar los datos, trazar la curva estándar (muestras procedentes de la etapa 3.1.1.3) (absorbancia frente a la concentración) y adaptada a una función lineal. Uso de la función lineal, la concentración de tioles libres en la solución de 'gel diluido' (gel + tampón de reacción) se puede calcular sobre la base de las lecturas de absorbancia. Por último, teniendo en cuenta la "dilución" con tampón de reacción, determinar la concentración de tiol libre en los geles sin tampón de reacción. (Por ejemplo, si el 15 de gel l se diluyó con tampón de reacción de 5 l, se multiplica por 20/15).
- Determinar el volumen de gel hinchado de geles 'finas' 5 l (V S) en base a la relación volumétrica de hinchamiento, P.
- Visualización de photopatterns con el reactivo de Ellman (Figura 3A).
- Remojar hidrogeles finas estampadas con Pep1Alloc en tampón de reacción de Ellman para 1 hr.
- Eliminar el exceso de tampón de reacción limpiando suavemente alrededor de los bordes del hidrogel con un pañuelo de papel.
- el reactivo de Ellman pipeta directamente sobre la superficie del gel. Inmediatamente imagen en un microscopio de luz a 10 veces o microscopio estereoscópico con una cámara a color.
Nota: Los geles deben obtenerse imágenes inmediatamente porque los patrones de visualización se disipará dentro de 5 min como el TNB amarilla 2- ion producido mediante la escisión del reactivo de Ellman tras la reacción con tioles en la región no modelado se difunde por todo el gel. regiones no estampadas-parecen ligeramente amarillo a simple vista; para una mejor resolución y los patrones más pequeños, ampliación (por ejemplo, el uso de un microscopio) se requiere.
- Preparar tampón de reacción de Ellman, solución de trabajo de cisteína, normas, y el reactivo de Ellman, como se describe a continuación.
- Visualización de Photopatterns con fluorescentes péptidos (Figura 3B).
- Para la visualización de los patrones con una resolución más alta o en tres dimensiones, photopattern un Pep1Alloc modificada-AF488 (o similar péptido fluorescente) a los geles en el paso 2.2.
- Imagen en un microscopio de fluorescencia. Un microscopio confocal se puede usar para tomar z-stimágenes ACK del gel de manera que el patrón puede ser observado en la x, y, y z-planos.
NOTA: Con la fluorescencia, geles deben ser protegidos de la luz para asegurar la estabilidad del fluoróforo AF488 y la fluorescencia máxima. Los geles no necesitan ser fotografiado inmediatamente, ya que el péptido fluorescente es bastante estable si se protege de la luz (la tienda en un recipiente envuelto en papel a 4 ° C).
4. La encapsulación celular en hidrogeles y photopatterning
- La recogida y preparación de células para la encapsulación.
- Siguiendo los procedimientos de cultivo de células de mamíferos estériles estándar, trypsinize y recoger las células de interés a partir de placas. Se inactiva la tripsina con medio de crecimiento después de que ocurra la separación (por ejemplo, para un matraz T-75, 5 ml de tripsina, se inactiva con 5 ml de medio, enjuague placa con 5 ml de medio para un total de 15 ml).
NOTA: Los tiempos de tripsinización pueden variar entre los tipos de células pero típicamente ocurrir entre 5 y 15 min. detachmen celulares alternativosagentes T (por ejemplo, Versene) se pueden usar para recoger las células si se desea. - Recuento de células (un mínimo de 100 o según el protocolo del fabricante) a partir de alícuotas de la suspensión celular utilizando un hemocitómetro tripsinizada u otro dispositivo de recuento de células, mientras que la centrifugación de la suspensión de células a granel (90 a 110 xg, 5 min). Vuelva a suspender el sedimento celular después de la centrifugación en un volumen mínimo de PBS o medio de crecimiento y recuento si la suspensión inicial de células tripsinizada está demasiado diluida.
- células en un volumen mínimo de PBS y las alícuotas para cada condición gel Vuelva a suspender en tubos de microcentrífuga de modo que habrá 5.000 células / l cuando se mezcla con la solución de gel (antes de la polimerización).
NOTA: alícuotas de células típicas contienen 300.000-500.000 células y son suficientes para hacer 60 a 100 l de suspensión de gel / célula que puede ser utilizado por 3-5 x 20 geles mu l con 5.000 células / l. densidades celulares superiores o inferiores pueden ser utilizados para la encapsulación, dependiendo de la cantidad de célula-célulavs. contacto célula-matriz deseada, respectivamente, y debe determinarse para cada aplicación. - Centrifugar las células / alícuotas de PBS (90 a 110 xg, 5 min).
- Con cuidado aspirar PBS de sedimento celular en tubo de microcentrífuga justo antes de la encapsulación.
NOTA: Si las células son sensibles a fuerzas de cizallamiento, la segunda etapa de centrifugación se puede eliminar mediante el recuento (por ejemplo, hemocitómetro) y posteriormente alícuotas la suspensión de células tripsinizada (tripsina + medios + células) en porciones necesarias para cada condición de gel. Estas alícuotas se centrifugan una vez (90 a 110 xg, 5 min) y la tripsina + medio se aspira, dejando un sedimento celular para la encapsulación.
- Siguiendo los procedimientos de cultivo de células de mamíferos estériles estándar, trypsinize y recoger las células de interés a partir de placas. Se inactiva la tripsina con medio de crecimiento después de que ocurra la separación (por ejemplo, para un matraz T-75, 5 ml de tripsina, se inactiva con 5 ml de medio, enjuague placa con 5 ml de medio para un total de 15 ml).
- Las células que encapsulan en hidrogeles con dibujos-no.
- Inmediatamente después de aspirar PBS, suspender las células sedimentadas en PEG4SH, Pep2Alloc, Pep1Alloc, LAP, y PBS + PS + FZ según lo calculado en la hoja de cálculo a una concentración final de 5.000 células / l.
- Molde y polimerizarse hidrogeles como described en los pasos 2.1.2 a 2.1.6.
NOTA: Al encapsular células en moldes de portaobjetos de vidrio, se debe tener cuidado al retirar la corredera de post-polimerización superior para evitar el cizallamiento del gel, que puede resultar en la muerte celular. Para ayudar con la retirada del gel del molde, moldes deslizantes pueden ser colocados en PBS estéril o medio de crecimiento después de la polimerización para mojar la junta y de hidrogel, por lo que es más fácil quitar el carro superior. Un método para evitar este cizallamiento completo es mediante el uso de moldes de jeringa. - geles Enjuague 3 x 10 min en medio de crecimiento para eliminar las especies sin reaccionar y el exceso de LAP.
- Incubar geles en medio de crecimiento a 37 ° C y 5% de CO 2 hasta el punto de tiempo deseado para su posterior análisis. Reponer medio cada 48 horas o como determinado por la aplicación (por ejemplo, el intervalo de la alimentación típica para el tipo específico de células y medio).
- Encapsular células y photopatterning en la presencia de células.
- Inmediatamente después de la aspiración de PBS,suspender las células sedimentadas en PEG4SH, Pep2Alloc, LAP, y PBS + PS + FZ según lo calculado en la hoja de cálculo a una concentración final de 5.000 células / l. Deja una concentración apropiada de tioles libres (por ejemplo, 2 mm) para la reacción posterior con Pep1Alloc.
- Molde, se polimeriza, e hidrogeles patrón como se describe en los pasos 2.2.2 a 2.2.8.
- geles Enjuague 3 x 10 min en medio de crecimiento después de modelando para eliminar las especies sin reaccionar y el exceso de LAP.
- Incubar geles en medio de crecimiento a 37 ° C y 5% de CO 2 hasta el punto de tiempo deseado para su posterior análisis. Reponer medio cada 48 horas o como determinado por la aplicación.
5. Determinación de la Actividad Viabilidad y metabólica de las células encapsuladas
- Realizar Ensayo de citotoxicidad Live / Dead para determinar la viabilidad celular encapsulado (Figura 4A).
- Retirar el medio de crecimiento a partir de geles y enjuague 3 x 15 min con PBS para permitir la difusión del medio de gels.
- Descongelar las soluciones vivas / muertas ensayo de citotoxicidad (homodímero de etidio-1 y calceína AM).
- Añadir 2 l de las etidio 2 mM homodímero-1 y 0,5 l de las soluciones de calceína AM 4 mm a 1 ml de PBS estéril. Vortex para asegurar una mezcla completa.
- Añadir 300 l solución de tinción para cada gel molde jeringa en placas de 48 pocillos, o lo suficiente para cubrir la superficie del gel en un portaobjetos de vidrio en un plato estéril, y se incuba durante 45 min.
- Eliminar el exceso de solución de tinción y enjuague para eliminar el exceso de tinte de los geles (3 x 15 min con PBS).
- Imagen en un microscopio confocal (imágenes z-stack) o en un microscopio de epifluorescencia con capacidad de z-pila a 10X y 488/525 nm Ex / Em. Cuantificar el número de células viables mediante la proyección de los z-pilas y contando el número de vivo (verde en todo el cuerpo celular) y muertos (núcleos rojos) células con software de imágenes.
- Realizar el ensayo de actividad metabólica para determinar la actividad de células después de la encapsulación (Figure 4B).
- 24 h antes de ensayo, las células de alimentación de encapsulado con medio de cultivo fresco.
NOTA: Se añade medio de unos pozos adicionales (que no contienen geles o geles sin células) como control (lecturas de fondo). - En el lugar de interés del tiempo, descongelar solución de reactivo MTS.
NOTA: Con el fin de determinar la actividad metabólica con el tiempo, un punto de tiempo inicial (12-24 horas después de la encapsulación) se recomienda como una referencia para la comparación de los puntos más adelante. - Añadir reactivo MTS a cada pocillo (20 l por 100 l medio de crecimiento).
- Incubar las muestras durante 1-4 horas a 37 ° C y CO 2 al 5%, según las instrucciones del fabricante.
NOTA: Los tiempos de incubación debería ser suficiente para que las células reducen MTS y permitir la difusión del producto formazan fuera del gel. En consecuencia, los geles más gruesas tales como geles punta de la jeringa pueden requerir tiempos de incubación más largos (~ hr 4) para una reducción suficiente y difusión MTS. - Pipetear 100 l reducida MTS / Medioen pocillos de una placa limpia y anotar la absorbancia de 96 pocillos a 490 nm en un lector de placas.
- Restar la absorbancia de fondo de los pozos sin células a partir de los valores de absorbancia de la muestra para generar valores de absorbancia baselined.
- 24 h antes de ensayo, las células de alimentación de encapsulado con medio de cultivo fresco.
Resultados
La configuración y el procedimiento para photoencapsulate células y geles posteriormente photopattern que contienen células encapsuladas se representa en la Figura 1, y un ejemplo para la preparación de soluciones madre para formar un% de gel de 10 en peso se proporciona en la Tabla 1. Usando la Tabla 1, la cantidad de monómeros (PEG4SH , Pep2Alloc, ± Pep1Alloc) y fotoiniciador (LAP) que se requieren para polimerizar hidrogeles se calcula. Sobre la base de estos cálculos, las soluciones madre de PEG4SH, Pep1Alloc, Pep2Alloc, y LAP se preparan y se mezclan con y sin Pep1Alloc para formar y photopatterning geles, respectivamente (Figura 1A). Posteriormente, las células se recogen de placas, se contaron, y se centrifugaron en cantidades adecuadas para la encapsulación (Figura 1B). El sedimento celular se resuspendió en la solución gelificante de (péptido / polímero / LAP en PBS), y mezclas de células / monómero se transfieren al vidrio o jeringa molds. Las células se encapsulan dentro del hidrogel después de la aplicación de la luz (1-5 minutos de 10 mW / cm 2 a 365 nm) (Figura 1C). Para photopatterning (Figura 1D), los geles se empaparon con Pep1Alloc y LAP durante 30 min a 1 hr y 30 min, lo que permite la difusión del péptido y el iniciador en la matriz polimerizada. Estos geles de péptidos cargados se cubren con fotomáscaras con patrones deseados y se expusieron a una segunda dosis de la luz (1 min) para conjugar los péptidos a tioles libres dentro de la matriz. Correas de suspensión sólo se une covalentemente al gel en las regiones expuestas a la luz, lo que facilita las interacciones célula-matriz adecuados y que imitan las propiedades mecánicas y bioquímicas clave del microambiente celular nativo hacia sondeo función de las células y el destino in vitro.
ensayo de Ellman proporciona un método fácil y barato para cuantificar la modificación gel y la incorporación de péptidos dentro de photopatterned sustratos. Estampadas y geles sin patrón (es decir, geles con o sin modificación péptido) se sumergen en tampón de reacción post-polimerización de Ellman. A continuación, las normas de cisteína y los geles empapados en tampón se colocan en placas de 48 pocillos y se incubaron con reactivo de Ellman (Figura 2A, izquierda). Después de 1 hora y 30 minutos, partes alícuotas de muestras se colocan en pocillos individuales de una placa de 96 pocillos, y la absorbancia se registra a 405 nm. Una curva de calibración (Figura 2A, derecha) de las normas de cisteína se representa (absorbancia frente a la concentración, ajuste lineal), y la cantidad de tioles libres en geles puede ser determinado en base a su factor de dilución. Estas concentraciones tiol libres para diferentes condiciones de polimerización y photopatterning de un gel de 10% en peso se muestran en la Figura 2B. Los geles polimerizados para 1 o 5 min sin Pep1Alloc (azul bares, I = 1 min y II = 5 min) tienen concentraciones de tiol libres estadísticamente similares, indicando tsombrero de una reacción rápida se produce la gelificación y se completa en 1 min (prueba t de dos colas, p> 0,05). Por lo tanto, la exposición adicional a la luz (2-5 minutos) no se traduce en una mayor conversión de los grupos funcionales. Los geles polimerizados por 1 min sin Pep1Alloc se empaparon con Pep1Alloc (3 mg / ml) y LAP (2,2 mM) durante 30 y 90 min (barras verdes, II = 30 min y IV = 90 min) y se expusieron a una segunda dosis de luz para 1 min. La disminución de los tioles libres (60 a 80% con respecto a la condición 1 min) indica la modificación eficiente de los geles con señales colgante en estas condiciones. Si se desea una mayor modificación, aumento de las concentraciones de solución Pep1Alloc se pueden utilizar como la accesibilidad de Pep1Alloc a tioles libres en soluciones más diluidas de péptidos pueden limitar la conversión; por ejemplo, se ha encontrado que concentraciones de hasta 20 mg / ml Pep1Alloc producen> 90% de conversión de tioles libres.
Excepcionalmente, los patrones de péptidos añadidos para el hidrogel puedeobtener imágenes rápidamente con el reactivo de Ellman (Figura 3A, en 5 min). Sin embargo, la visualización del patrón se pierde con el tiempo (mayor de 5 min) debido a la difusión del amarillo TNB 2- dianión a través del gel. Para mejorar la resolución de imagen y observar los patrones en tres dimensiones (x, y, y z-planos) y en presencia de las células, la adición de péptido fluorescente (AF488Pep1Alloc) se puede utilizar. En la Figura 3B x, y, z proyecciones de pilas de imágenes tomadas con un microscopio confocal se muestran, lo que demuestra el patrón de resolución (escala de micras).
Aproximadamente (94 ± 2)% y (94 ± 1)% de hMSCs encapsulados dentro de geles degradables (Pep2Alloc = GPQG ↓ WGQ) se mantienen (cuerpos celulares verdes) viables 1 y 6 días después de la encapsulación, respectivamente, con pocas células muertas (núcleos rojos ) observado (Figura 4A). Además, se observa hMSC propagando a 6 días después de la encapsulación ( rong> Figura 4A, recuadro), lo que indica que las células pueden remodelar e interactuar con estas matrices MMP-degradables modificados con RGDS de unión a integrinas. Los ensayos de actividad metabólicos realizados sobre células encapsuladas en geles no degradables (Pep2Alloc = RGKGRK) 1 y 3 días después de la encapsulación (Figura 4B) proporcionar una segunda medida de la viabilidad celular y demuestran que las células permanecen activas durante las diversas condiciones photoencapsulation y photopatterning probados con ensayo de Ellman (Figura 2B). En particular, no hay ninguna diferencia significativa en la actividad metabólica entre 30 min y 90 min incubaciones con Pep1Alloc + LAP (condiciones III y IV) y la encapsulación inicial (condiciones I y II), que indica que el procedimiento es apropiado para aplicaciones de photopatterning en la presencia de células encapsuladas (t-test, p> 0,05 de dos colas).
2fig1.jpg "/>
Figura 1:. Configuración para encapsular las células dentro de los hidrogeles y posteriormente photopatterning con señal bioquímica (A) Las soluciones madre de macrómeros y fotoiniciador se preparan y se mezcla (PEG4SH = espina dorsal, Pep2Alloc = reticulación, Pep1Alloc = pendiente del resto de adhesivo (MFG), LAP = fotoiniciador ). (B) Las células se recogieron para la encapsulación. (C) Las células se mezclan con soluciones de macrómero sin o con péptidos de unión a integrinas (PEG4SH / Pep2Alloc / LAP o PEG4SH / Pep2Alloc / Pep1Alloc / LAP, respectivamente) y se encapsulan después de la exposición a la luz. (D) Los geles que contienen grupos tiol en exceso durante la formación de gel (en este caso, PEG4SH / Pep2Alloc / LAP, formado con 2 mM exceso de tiol) puede tener un patrón con señales bioquímicas por la posterior adición de péptidos funcionalizadas con un solo grupo alloc (Pep1Alloc, por ejemplo, RGDS, IKVAV, etc.) para promover la adhesión celulardentro de regiones específicas del gel. A continuación, se muestra el patrón de geles con MFG con fluorescencia marcado. Haga clic aquí para ver una versión más grande de esta figura.
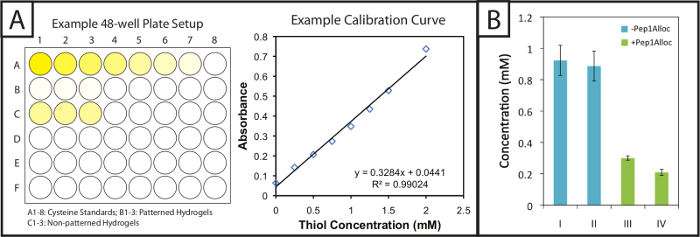
Figura 2:. Configuración y resultados del ensayo cuantitativo de Ellman para evaluar la modificación de hidrogeles estampadas Gels (A) y las normas de cisteína se incuban en placas de 48 pocillos con reactivo de Ellman. Una curva estándar lineal se representa para determinar la concentración de cisteína en las muestras de gel. (B) El exceso de tioles libres se incorporan dentro de los hidrogeles durante la formación de gel y se consumen en el patrón con un colgante alloc-péptido (min polimerización I = 1; II = 5 min de polimerización; III = 30 min de incubación con Pep1Alloc; IV = 90 min de incubación wi º Pep1Alloc; tanto III y IV polimerizan y se modela con la luz durante 1 min). Los datos mostrados ilustran la media (n = 3) con barras de error muestran el error estándar. Haga clic aquí para ver una versión más grande de esta figura.
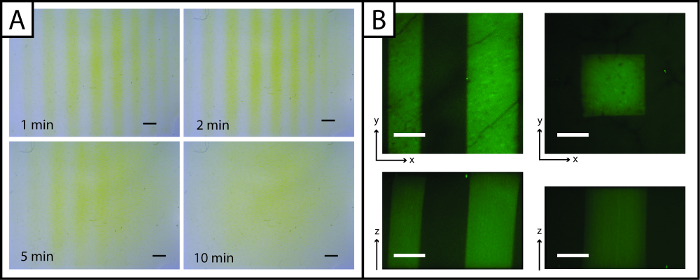
Figura 3:. Ensayo de Ellman y las imágenes fluorescentes para visualizar hidrogeles photopatterned reactivo (A) de Ellman se puede usar para detectar rápidamente los patrones (líneas) en las direcciones x y y-planos (amarillo = región sin patrón, barra de escala = 1 mm). (B) péptidos fluorescentes se pueden usar para observar los patrones en los ejes X, Y y Z. planos (verde = región modelada; 200 Barra de escala m; 10X objetivo de inmersión en agua; Ex / Em 488/525 nm).large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
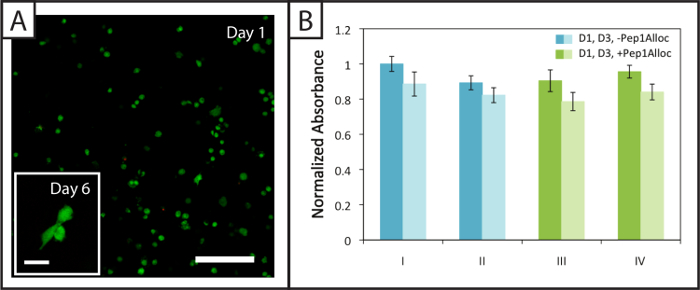
. Figura 4: Viabilidad y actividad metabólica de las células dentro de los hidrogeles photopatterned no degradables (A) Ejemplo confocal z-pila (z-proyección; 10X objetivo de inmersión en agua) de viables (verde; Ex / Em 488/525 nm) y muertos hMSCs (rojo; Ex / Em 543/580 nm) encapsulado dentro de los hidrogeles de 24 horas después de la encapsulación (barra de escala = 200 micras). Las células se extienden dentro de estos hidrogeles 6 días después (imagen de la inserción, Barra de escala = 50 micras) en la encapsulación. (B) Las células son metabólicamente activas 1 y 3 días (barras claras y oscuras, respectivamente) después de la encapsulación para los distintos polimerización (polimerización I = 1 min; II = 5 min de polimerización) y las condiciones de modelado (III = 30 min de incubación con Pep1Alloc ; IV = 90 min incubation con Pep1Alloc; tanto polimerizado y modelado con luz durante 1 min). Los datos mostrados ilustran la media (n = 3) con barras de error muestran el error estándar. Haga clic aquí para ver una versión más grande de esta figura.
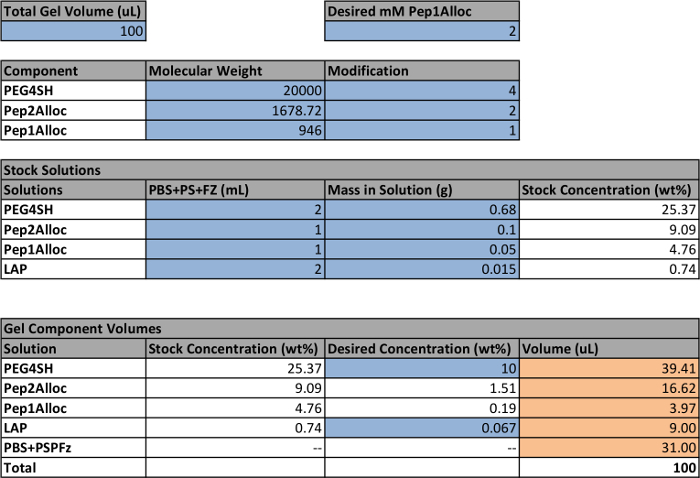
Tabla 1: Ejemplo de configuración para calcular los volúmenes de las soluciones madre para la fabricación de hidrogeles Las células dentro de la hoja de cálculo que se puso de relieve en azul indican los parámetros definidos por el usuario;. las otras cantidades se calculan sobre la base de estos valores. Los volúmenes finales de cada solución madre para hacer un gel de color naranja se resaltan. Los glóbulos blancos contienen las fórmulas utilizadas para calcular los volúmenes finales sobre la base de los parámetros definidos por el usuario. Tenga en cuenta que una concentración de 2,2 mM LAP es equivalente a 0,067% en peso.
Discusión
El procedimiento presentado aquí demuestra técnicas para photoencapsulate células dentro de los hidrogeles formados por tiol-eno química modular y posteriormente photopattern los geles con señales bioquímicas. El uso de la luz para formar inicialmente hidrogeles permite una mezcla homogénea y la suspensión de las células dentro de la solución de polímero antes de la polimerización. Los rápidos de polimerización '' cerraduras el gel en la forma del molde definida y encapsula las células dentro de la red de hidrogel suspendidos. Los geles también pueden moldearse en numerosas formas diferentes (por ejemplo, diapositivas de vidrio o puntas de jeringa) dependiendo de la aplicación final deseada. Por ejemplo, las células encapsuladas para la cultura en 3D hidrogeles fijados a portaobjetos de vidrio son particularmente útiles para aplicaciones de imagen como atenuación de la luz es limitada dentro de una muestra delgada. moldes de jeringa se pueden utilizar para una rápida encapsulación de células, lo que permite un mayor número de muestras para estar preparados en un tiempo corto (en comparación con portaobjetos de vidrios) que se puede utilizar para experimentos que requieren un gran número de células, tales como citometría de flujo o qPCR. Posteriormente, estos geles a continuación, pueden estar decoradas con señales bioquímicas para provocar respuestas celulares deseados tales como la diferenciación o la invasión 30,31.
Los ensayos para la viabilidad y la actividad metabólica indican la supervivencia de las células para el sistema de materiales y condiciones de modelado presentados. Tenga en cuenta que la actividad metabólica se monitorizó hasta el día 3 en geles no degradables (RGKGRK péptido de reticulación) para evaluar los efectos iniciales de condiciones de polimerización y los patrones sobre la función celular. Además, un ensayo de integridad de la membrana (viabilidad / citotoxicidad tinción vivo / muerto) de hMSC a 1 y 6 días después de la encapsulación en geles formulados con un péptido de reticulación degradable (GPQGIWGQ) admite que las células permanecen viables y se extendió a 1 semana de cultivo. La viabilidad de las líneas celulares adicionales se ha informado de las condiciones photopatterning 32 similares a lasutilizado aquí y puede ser evaluado por el sistema de hidrogel se describe mediante la tinción en vivo / muerto y ensayos de actividad metabólica. Si bien no hemos observado problemas con la viabilidad celular usando este sistema de materiales y procedimientos conexos (4.2 y 4.3), algunos tipos de células pueden ser sensibles a liberar radicales libres y / o exposición a la luz. En este caso, los usuarios pueden considerar el uso de sistemas de materiales no fotoiniciadas, como azida-alquino, 12 FXIII, 31 o químicas de la formación del hidrogel a base de Alder Diels. 33
técnicas fáciles de detectar y cuantificar los patrones de señales bioquímicas dentro de geles también son presentados (3.1 y 3.2). Ensayo de Ellman es de particular interés debido a que los reactivos están disponibles comercialmente y no se requieren etapas de procesamiento adicionales o sintético reactivos más costosos (por ejemplo, péptido marcado fluorescentemente). ensayo de Ellman se puede utilizar para determinar con precisión la modificación de tioles libres con señales bioquímicas bajo different photopatterning condiciones, así como para visualizar rápidamente patrones. Para la cuantificación de la incorporación de péptidos, la concentración de grupos funcionales tiol antes y después de patrón, como una medida cuantitativa de incorporación de péptido, se evalúa directamente con el ensayo de Ellman. Aunque este tipo de cuantificación se puede hacer con péptidos fluorescentemente marcada, 34 de cuantificación basado en formación de imágenes requiere más manejo y análisis de los pasos que consumen mucho tiempo (por ejemplo, la síntesis de un péptido marcado fluorescentemente y la generación de una curva de calibración para relacionar la fluorescencia a concentración de péptido usando análisis de imagen). Para imágenes péptido incorporación, reactivo de Ellman se puede aplicar directamente a las muestras inmediatamente y visualiza. Si bien limitado a los ejes x e y planos para la visualización de patrón, la técnica se puede utilizar como un método simple, rutina para determinar si se han modelado matrices que contienen grupos tiol libres. Es importante observar que el ensayo de Ellman no es ccitocompatible onsidered, lo que si bien se puede utilizar para observar y cuantificar photopatterns, no puede realizarse en la presencia de células. Para la formación de imágenes patrón en tres dimensiones y en la presencia de células, la conjugación de péptidos fluorescentes dentro de matrices de hidrogel sigue siendo un enfoque poderoso y ampliamente utilizado. Resolución de patrones puede ser evaluada en los ejes X, Y y Z. aviones usando microscopía confocal, y este método es citocompatible para que las células dentro de las regiones estampadas o regiones no se pueden identificar con dibujos. En su conjunto, las técnicas de ensayo y basados en la imagen de Ellman son herramientas complementarias para los investigadores evaluar cuantitativa y cualitativamente la photopatterning de señales bioquímicas dentro del sistema de materiales.
Photoclick, o más fotoiniciada ampliamente, químicas para la formación y modificación de hidrogeles en la presencia de células son numerosas. Las piezas de fundición, encapsulación, y el patrón técnicas presentadas aquí no son limITED al sistema material descrito y se puede aplicar a las químicas basadas en la luz alternativas, tales como tiol-alquino, 35 azida-alquino, 4 y otras químicas de tiol-eno (por ejemplo, tiol-norborneno), 10,13, así como con fotoiniciadores diferentes, tales como Irgacure 2959, eosina y, y camforquinona. Tenga en cuenta, los usuarios pueden necesitar para ajustar los parámetros de procedimiento (por ejemplo, tiempos de incubación, los tiempos de polimerización, la densidad celular) para garantizar que las condiciones siguen siendo citocompatible para estos otros sistemas. Dado que el proceso requiere patrones de difusión del péptido (s) alloc modificado en el hidrogel (2.2.3-2.2.5), este proceso puede resultar más útil para la adición de restos de unión a integrina (por ejemplo, péptidos o proteína de la matriz extracelular fragmentos) al hidrogel, donde se requiere la unión del ligando a la red para la generación de fuerzas de tracción por la activación celular y la integrina completo. 36 Nota, para biomoléculas que puedenser igualmente activo en solución o en el momento de inmovilización (por ejemplo, factores de crecimiento o citoquinas), la etapa de incubación para la difusión resto (~ 1 h) en el hidrogel podría conducir a eventos que convoluta resultados de modelado de señalización. Se han establecido otros métodos para la inmovilización del factor de crecimiento o el secuestro local para su patrón. 37-39 Además, la resolución de patrón viene dado por el control de la exposición a la luz. Aquí, máscaras permiten la creación de patrones a través de la profundidad de gel y en las direcciones x e y aviones; sin embargo, un mayor control espacial sobre los patrones de señales bioquímicas dentro de los geles se puede lograr con los métodos alternativos de irradiación, tales como el uso de un microscopio confocal de dos fotones para generar patrones en el x-, y-, y z- aviones. 34,40 finalmente, es importante señalar que mientras que el sistema de material utilizado en este procedimiento sólo se modifica inicialmente con las señales bioquímicas, químicas Photoclick ortogonales se podrían usar para permitir alteraciones en las propiedades de la matriz a través del tiempo. 12 El procedimiento y las técnicas que aquí se presentan se suman la diversidad de los enfoques actuales para la creación de matrices sintéticas con propiedades bien definidas y controladas por espaciotemporalmente. En particular, la disponibilidad comercial de los reactivos y materiales utilizados en este procedimiento será útil para una amplia gama de investigadores interesados en el uso de biomateriales a base de hidrogel para aplicaciones en cultivo celular controlada.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Este trabajo fue apoyado por los programas de Delaware Cobre, en el descubrimiento de medicamentos y en Biomateriales avanzados financiados por los Premios de Desarrollo Institucional del Instituto Nacional de Ciencias Médicas Generales de los Institutos Nacionales de Salud (P20GM104316 y GM110758-01 P30, respectivamente), el Pew Charitable Trusts (00026178), el Premio Nacional de Carrera Science Foundation (DMR-1253906), el Fondo Burroughs Wellcome (1.006.787), y el programa Nacional de Ciencia Fundación IGERT SBE2 en la Universidad de Delaware (beca para L. Sawicki). Los autores agradecen al Centro de Delaware BIOimagen Instituto de Biotecnología de la Universidad de Delaware para la formación y el acceso a la microscopía confocal, Sra. Katherine Wiley de asistencia durante la filmación del video, el Sr. Mateo Rehmann generosamente para proporcionar hMSCs aisladas de la médula ósea, el profesor Christopher J . Kloxin y el Sr. Stephen Ma generosamente para proporcionar máscaras, y el Prof. Wilfred Chen por el uso del lector de placas automático.
Materiales
| Name | Company | Catalog Number | Comments |
| Custom Peptides | Various Vendors | ---- | Peptides may also be synthesized via standard SPPS techniques with materials from vendors including ChemImpex and ChemPep. |
| 4-arm PEG Thiol, MW 20k | JenKem USA | 4ARM-SH | Listed under Multi-arm Homofunctional PEGs. PEG4SH may also be synthesized as previously referenced. |
| Lithium Phenyl-2,4,6-trimethylbenzoylphosphinate | Colorado Photopolymer Solutions | Li-TPO | |
| Dimethyl Phenylphosphonite | Sigma Aldrich | 149470 | Caution, causes severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| 2,4,6-Trimethylbenzoyl Chloride | Sigma Aldrich | 682519 | Caution, causes severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Lithium Bromide | Sigma Aldrich | 213225 | |
| 2-Butanone | Sigma Aldrich | 360473 | |
| Flask, Round Bottom, 100 ml | Chemglass | CG-1506-05 | |
| 80 ml Filter Funnel, Buchner, Medium Frit | Chemglass | CG-1402-L-02 | Filter paper inside a regular glass funnel may be used if desired. |
| Magnetic Stir Bars | Various Vendors | ---- | |
| Magnetic Stirring and Hot Plate | Various Vendors | ---- | |
| Vacuum Dessicator | Various Vendors | ---- | |
| Deuterium Oxide | Acros Organics | 16630 | |
| Dulbecco's Phosphate Buffered Saline | ThermoFisher Scientific | 14190-250 | |
| Penicillin Streptomycin | ThermoFisher Scientific | 15070-063 | |
| Fungizone | ThermoFisher Scientific | 15290-018 | |
| Trypsin-EDTA (0.5%), no phenol red | ThermoFisher Scientific | 15400-054 | Select appropriate medium and trypsin depending on cell type. |
| Microcentrifuge tubes | Various Vendors | ---- | 1.5 or 2 ml sizes, sterile. |
| BD Syringe with Slip (Luer) Tips (Without Needle) | Fisher Scientific | 14-823-16H | Product number listed here is for a 1 ml syringe (16H), Various sizes are available (14-823-XX). |
| Fisherfinest Premium Plain Glass Microscope Slides | Fisher Scientific | 12-544-1 | |
| High-Purity Silicone Rubber, 0.010" Thick, 6" x 8" Sheet, 55A Durometer (Gasket) | McMaster Carr | 87315K62 | |
| Ethanol, 200 Proof | Decon Labs | 2701 | |
| RainX, Original | Amazon | 800002243 | May be purchased from other vendors. |
| Sigmacote | Sigma Aldrich | SL2 | |
| Photomasks | Advance Reproductions Corporation | ---- | Photomask Division, different designs may be printed as desired. |
| DTNB; Ellman's Reagent; 5,5-dithio-bis(2-nitrobenzoic acid) | ThermoFisher Scientific | PI-22582 | |
| Sodium Phosphate Dibasic | Sigma Aldrich | S5136 | |
| Ethylenediaminetetraacetic acid | Sigma Aldrich | E6758 | |
| Sodium Hydroxide, Pellets/Certified ACS | Fisher Scientific | S318 | |
| Orthophosphoric Acid | Alfa Aesar | 33266 | |
| Cysteine Hydrochloride Monohydrate | Sigma Aldrich | C7880 | |
| LIVE/DEAD Viability/Cytotoxicity Kit, for mammalian cells | ThermoFisher Scientific | L-3224 | |
| CellTiter 96 Aqueous One Solution Assay | Promega | G3582 | |
| Centrifuge | Various Vendors | ---- | Capable of speeds at 90-110 x g. |
| Multiwell Plate Reader | Various Vendors | ---- | Capable of reading absorbance at 405 nm in a 96-well plate. |
| Epifluorescent or Confocal Microscope | Various Vendors | ---- | To visualize peptide patterns and cells within hydrogels. |
| Omnicure Exfo Series 2000 | Excelitas Technologies | ---- | Alternate light systems may be used to polymerize hydrogels. |
| Zeiss Zen Lite Software | Zeiss | ---- | Available at zeiss.com; compatible with images taken on Zeiss microscopes |
| ImageJ | NIH | ---- | Available at imagej.nih.gov; applicable for general image analysis |
Referencias
- Kolb, H. C., Finn, M. G., Sharpless, K. B. Click Chemistry: Diverse Chemical Function from a Few Good Reactions. Angew Chemie - Int Ed. 40 (11), 2004-2021 (2001).
- Xi, W., Scott, T. F., Kloxin, C. J., Bowman, C. N. Click Chemistry in Materials Science. Adv Funct Mater. 24 (18), 2572-2590 (2014).
- Azagarsamy, M. A., Anseth, K. S. Bioorthogonal click chemistry: An indispensable tool to create multifaceted cell culture scaffolds. ACS Macro Lett. 2 (1), 5-9 (2013).
- Adzima, B. J., Tao, Y., Kloxin, C. J., DeForest, C. A., Anseth, K. S., Bowman, C. N. Spatial and temporal control of the alkyne-azide cycloaddition by photoinitiated Cu(II) reduction. Nat Chem. 3 (3), 256-259 (2011).
- Hoyle, C. E., Bowman, C. N. Thiol-ene click chemistry. Angew Chemie - Int Ed. 49 (9), 1540-1573 (2010).
- Fan, Y., Deng, C., Cheng, R., Meng, F., Zhong, Z. In situ forming hydrogels via catalyst-free and bioorthogonal "tetrazole-Alkene" photo-click chemistry. Biomacromolecules. 14 (8), 2814-2821 (2013).
- Burdick, J. A., Murphy, W. L. Moving from static to dynamic complexity in hydrogel design. Nat Commun. 3, 1269 (2012).
- Rehmann, M. S., Kloxin, A. M. Tunable and dynamic soft materials for three-dimensional cell culture. Soft Matter. 9 (29), 6737-6746 (2013).
- Yang, T., Long, H., Malkoch, M., Gamstedt, K. E., Berglund, L., Hult, A. Characterization of well-defined poly(ethylene glycol) hydrogels prepared by thiol-ene chemistry. J Polym Sci Part A Polym Chem. 49 (18), 4044-4054 (2011).
- Fairbanks, B. D., et al. A versatile synthetic extracellular matrix mimic via thiol-norbornene photopolymerization. Adv Mater. 21 (48), 5005-5010 (2009).
- Roberts, J. J., Bryant, S. J. Comparison of photopolymerizable thiol-ene PEG and acrylate-based PEG hydrogels for cartilage development. Biomaterials. 34 (38), 9969-9979 (2013).
- DeForest, C. A., Polizzotti, B. D., Anseth, K. S. Sequential click reactions for synthesizing and patterning three-dimensional cell microenvironments. Nat Mater. 8 (8), 659-664 (2009).
- Lin, C. C., Ki, C. S., Shih, H. Thiol-norbornene photoclick hydrogels for tissue engineering applications. J Appl Polym Sci. 132 (8), (2015).
- Sawicki, L. A., Kloxin, A. M. Design of thiol-ene photoclick hydrogels using facile techniques for cell culture applications. Biomater Sci. 2 (11), 1612-1626 (2014).
- Patterson, J., Hubbell, J. A. Enhanced proteolytic degradation of molecularly engineered PEG hydrogels in response to MMP-1 and MMP-2. Biomaterials. 31 (30), 7836-7845 (2010).
- Fairbanks, B. D., Singh, S. P., Bowman, C. N., Anseth, K. S. Photodegradable, photoadaptable hydrogels via radical-mediated disulfide fragmentation reaction. Macromolecules. 44 (8), 2444-2450 (2011).
- Fairbanks, B. D., Schwartz, M. P., Bowman, C. N., Anseth, K. S. Photoinitiated polymerization of PEG-diacrylate with lithium phenyl-2,4,6-trimethylbenzoylphosphinate: polymerization rate and cytocompatibility. Biomaterials. 30 (35), 6702-6707 (2009).
- Yang, F., et al. The effect of incorporating RGD adhesive peptide in polyethylene glycol diacrylate hydrogel on osteogenesis of bone marrow stromal cells. Biomaterials. 26 (30), 5991-5998 (2005).
- Wacker, B. K., et al. Endothelial cell migration on RGD-peptide-containing PEG hydrogels in the presence of sphingosine 1-phosphate. Biophys J. 94 (1), 273-285 (2008).
- Wilson, M. J., Liliensiek, S. J., Murphy, C. J., Murphy, W. L., Nealey, P. F. Hydrogels with well-defined peptide-hydrogel spacing and concentration: impact on epithelial cell behavior. Soft Matter. 8 (2), 390-398 (2012).
- Qiong Liu, S., et al. Injectable biodegradable polyethylene glycol/ RGD peptide hybrid hydrogels for in vitro chondrogenesis of human mesenchymal stern cellsa. Macromol Rapid Commun. 31 (13), 1148-1154 (2010).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix Elasticity Directs Stem Cell Lineage Specification. Cell. 126 (4), 677-689 (2006).
- Levental, I., Georges, P. C., Janmey, P. A. Soft biological materials and their impact on cell function. Soft Matter. 3 (3), 299-306 (2007).
- Lin, C. C., Raza, A., Shih, H. PEG hydrogels formed by thiol-ene photo-click chemistry and their effect on the formation and recovery of insulin-secreting cell spheroids. Biomaterials. 32 (36), 9685-9695 (2011).
- Khetan, S., Burdick, J. Cellular encapsulation in 3D hydrogels for tissue engineering. J Vis Exp. (32), e1590 (2009).
- Bryant, S. J., Nuttelman, C. R., Anseth, K. S. Cytocompatibility of UV and visible light photoinitiating systems on cultured NIH/3T3 fibroblasts in vitro. J Biomater Sci Polym Ed. 11 (5), 439-457 (2000).
- Burdick, J. A., Chung, C., Jia, X., Randolph, M. A., Langer, R. Controlled Degradation and Mechanical Behavior of Photopolymerized Hyaluronic Acid Networks Controlled Degradation and Mechanical Behavior of Photopolymerized Hyaluronic Acid Networks. Society. 6 (1), 386-391 (2005).
- Marsano, E., Gagliardi, S., Ghioni, F., Bianchi, E. Behaviour of gels based on (hydroxypropyl) cellulose methacrylate. Polymer (Guildf). 41 (21), 7691-7698 (2000).
- Bryant, S. J., Anseth, K. S. Photopolymerization of Hydrogel Scaffolds. Scaffolding Tissue Eng. , 71-90 (2005).
- Khetan, S., Burdick, J. A. Patterning hydrogels in three dimensions towards controlling cellular interactions. Soft Matter. 7 (3), 830-838 (2011).
- Mosiewicz, K. A., et al. In situ cell manipulation through enzymatic hydrogel photopatterning. Nat Mater. 12 (11), 1072-1078 (2013).
- Williams, C. G., Malik, A. N., Kim, T. K., Manson, P. N., Elisseeff, J. H. Variable cytocompatibility of six cell lines with photoinitiators used for polymerizing hydrogels and cell encapsulation. Biomaterials. 26 (11), 1211-1218 (2005).
- Nimmo, C. M., Owen, S. C., Shoichet, M. S. . Diels - Alder Click Cross-Linked Hyaluronic Acid Hydrogels for Tissue Engineering. , 824-830 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Fairbanks, B. D., Sims, E. A., Anseth, K. S., Bowman, C. N. Reaction rates and mechanisms for radical, photoinitated addition of thiols to alkynes, and implications for thiol-yne photopolymerizations and click reactions. Macromolecules. 43 (9), 4113-4119 (2010).
- Tibbitt, M. W., Anseth, K. S. Hydrogels as extracellular matrix mimics for 3D cell culture. Biotechnol Bioeng. 103 (4), 655-663 (2009).
- Wylie, R. G., et al. Spatially controlled simultaneous patterning of multiple growth factors in three-dimensional hydrogels. Nat Mater. 10 (10), 799-806 (2011).
- Pompe, T., Salchert, K., Alberti, K., Zandstra, P., Werner, C. Immobilization of growth factors on solid supports for the modulation of stem cell fate. Nat Protoc. 5 (6), 1042-1050 (2010).
- Hudalla, G. A., Murphy, W. L. Biomaterials that regulate growth factor activity via bioinspired interactions. Adv Funct Mater. 21 (10), 1754-1768 (2011).
- Lee, S. H., Moon, J. J., West, J. L. Three-dimensional micropatterning of bioactive hydrogels via two-photon laser scanning photolithography for guided 3D cell migration. Biomaterials. 29 (20), 2962-2968 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados