Method Article
Evaluación de la contaminación del ADN en las muestras de RNA basadas en ADN Ribosomal
En este artículo
Resumen
Aquí, presentamos un protocolo de rastreo genómica contaminación del ADN (gDNA) en las muestras de RNA. El método presentado utiliza cebadores específicos para la región del espaciador transcrito interno (ITS) de genes ribosómicos (rDNA) del ADN. El método es adecuado para la detección fiable y sensible de la contaminación del ADN en la mayoría de eucariotas y procariotas.
Resumen
Un método ampliamente utilizado para la cuantificación de cambios de expresión génica y abundancia de la transcripción es la PCR en tiempo real cuantitativa de la reverso-transcripción (RT-qPCR). Proporciona resultados precisos, sensibles, fiables y reproducibles. Varios factores pueden afectar la sensibilidad y especificidad del RT-qPCR. Residual de ADN genómico (gDNA) contaminación de las muestras de RNA es uno de ellos. En el análisis de expresión génica, amplificación no específica debido a la contaminación del gDNA sobrestiman la abundancia de niveles de transcripción y puede afectar los resultados de la RT-qPCR. Por lo general, se detecta gDNA por qRT-PCR usando la cartilla pares hibridarlo con regiones intergénicas o un intrón del gen de interés. Por desgracia, las anotaciones del intrón/del exón aún no se conocen para todos los genes de vertebrados, bacterias, Protista, hongos, plantas y especies de metazoarios invertebrados.
Aquí presentamos un protocolo para la detección de contaminación gDNA en RNA de muestras mediante el uso de ADN ribosomal (ADNr)-imprimadores base. El método se basa en las características únicas del rDNA: su naturaleza multigénica, secuencias altamente conservadas y alta frecuencia en el genoma. También como caso de estudio, un conjunto único de cartillas fueron diseñados basándose en la región conservada de ADN ribosomal (ADNr) de la familia Poaceae. La universalidad de estos pares de cartilla fue probada por electroforesis en gel análisis y agarosa curva de fusión. Aunque nuestro método explica cómo primers basados en rADN pueden ser aplicados para el ensayo de contaminación gDNA en la familia Poaceae, podría utilizarse fácilmente para otras especies de célula procariota y eucariotas
Introducción
Explorando la regulación transcripcional de los interesantes conjuntos de genes o redes de señalización es fundamental para comprender los complejos mecanismos moleculares implicados en acontecimientos biológicos1. En la actualidad, análisis de qPCR es el más ampliamente utilizado enfoque para estudios de expresión génica que pueden dirigirse a ADN (el genoma) o RNA (transcriptoma) que permiten análisis metiloma y transcriptoma, respectivamente. Transcripción reversa (RT) seguida por qPCR es ampliamente utilizada para el análisis del transcriptoma que miden los niveles de expresión de genes en diversas áreas de investigación biológica2. En comparación con otros métodos como el hibridación norteño tradicional, detección específica de tejido mediante in situ hibridación, ensayos de protección de la ribonucleasa (RPA) y semi-RT-PCR, la exactitud, conveniencia, velocidad y alcance amplio y dinámico de ensayos de qPCR son altamente notable3,4. Hay varios factores importantes que tienen que ser considerados para una cuantificación fiable de ARN mensajero (ARNm), incluyendo la calidad y la cantidad de ARN a partir de material. Además, la amplificación no específica la eficacia del RT-qPCR y eficiencia PCR tienen que ser considerados5,6.
La presencia del gDNA es un problema inherente durante la extracción de RNA debido, en parte, las propiedades físicas y químicas similares de ADN y ARN7. Debido a la identidad de la secuencia del gDNA y ADN complementario (cDNA) derivado de las muestras del mRNA, la amplificación no específica puede ocurrir, que influirá en la precisión de los resultados de la RT-qPCR. GDNA restantes dará lugar a la sobrestimación de la abundancia del mRNA de destino en gene expresión análisis8.
Básicamente, los productos no específicos surge sobre todo de formación de dímeros de primer o amplificación inespecífica de fondo debido a gDNA, ambos de los cuales pueden evaluarse mediante el uso de muestras de control apropiado. Dichas muestras son ningún control de plantilla (NTC) y sin control de la transcriptasa reversa (NRT), respectivamente. Puesto que los niveles de contaminación del gDNA en las muestras estudiadas son diferentes y la sensibilidad hacia gDNA difiere mucho entre los genes analizados, los controles de la intervención NRT se requieren para cada par de muestra/ensayo. Aunque esto aumenta considerablemente el costo y el trabajo en estudios de generación de perfiles RT-qPCR, estos controles son necesarios7,9.
Métodos alternativos relacionados con la contaminación del gDNA incluyen el uso de pares de primer recocido a regiones intergénicas o un intrón del gene de interés10y el uso de cebadores que flanquean un gran intrón o abarcan a un empalme exón-exón, es decir los sitios de recocido son ausentes en el mRNA maduro secuencia1,4. Sin embargo, las anotaciones del intrón/del exón de los genes de muchos vertebrados, bacterias, Protista, hongos, planta y especies de metazoarios invertebrados se conocen todavía. Además, muchos organismos eucariotas con pseudogenes derivados de eventos de duplicación. Además, primer diseño en intrones no garantiza la no amplificación del gDNA. Como la cromatina accesibilidad de regiones genómicas a DNasa varía, se recomienda para el diseño de pares diferentes cartilla dirigida a diversos cromosomas10.
Los genomas de organismos eucariotas pueden abarcar hasta 1 mil copias de genes rDNA codifican subunidades de ribosomas necesarios para la formación de los ribosomas. Estos genes de ADNr se organizan a menudo en arreglos simples o tandem repetición11. RRNAs policistrónico (figura 1), incluyendo la subunidad grande (LSU) y la subunidad pequeña (SSU) son transcritos por la polimerasa de ARN I (pol RNA I). El pre-rRNAs resultantes se procesan aún más mediante la eliminación de las dos regiones del espaciador transcrito interno ITS1 e ITS2. Como productos finales, tres maduran rRNAs, 17-18S rRNA (SSU), 5.8S y 28S 25 rRNA (LSU) son generados12. genes del rDNA son representantes típicos de una familia multigénica con secuencias altamente conservadas. Ocurren con una alta frecuencia en el genoma y están potencialmente presentes en más de una localización cromosómica13. El procesamiento del rRNA y la degradación de los espaciadores transcritos son un proceso rápido en el nucleolo. Debido al alto grado de repetitividad, el cociente del número de copias genómicas y premolecules de RNA detectables es inferior en comparación con las secuencias del intrón de la bajo-copia y unspliced precursores. Estas características hacen que genes rDNA idóneo para la detección fiable y altamente sensible de contaminación gDNA en eucariotas y procariotas más3.
Aquí se describe un novedoso procedimiento para detectar contaminación gDNA en las muestras de RNA. Un conjunto de cebadores universales basados en la secuencia del rDNA conservado se presenta para los ensayos de gDNA en varias especies de Poaceae. La especificidad y la universalidad de los iniciadores de la propuesta se analizaron por análisis de la curva de fusión usando la DNA como plantilla. Nuestro protocolo no sólo es aplicable para la Poaceae, pero fácilmente podría adaptarse a otras especies de eucariotas y procariotas.
Protocolo
Nota: Puede utilizarse cualquier tejido.
1. extracción
- Poner 100 mg de muestras de tejido en un tubo de 2.0 mL, agregue dos bolas de acero inoxidable de 5 mm y homogeneizar el tejido a 25-30 Hz durante 30 s (duración de homogeneización y frecuencia dependiendo del tipo de tejido) para ARN y ADN.
- Aislar el ARN total según las instrucciones del fabricante.
- Aislar ADN total según las instrucciones del fabricante.
- Controlar la pureza y la cantidad de las muestras de RNA mediante la medición de absorbancia a 260 y 280 nm.
- Controlar la pureza y la cantidad de las muestras de ADN midiendo absorbancia a 260 y 280 nm.
Nota: Mientras que los ácidos nucleicos absorben la luz con una longitud de onda de 260 nm (A260), la absorbancia de la luz en la longitud de onda 280 (A280) puede utilizarse para cuantificar la cantidad de proteínas y fenoles presentan en la muestra. Por lo tanto, la relación A260/A280 nm puede utilizarse para evaluar la pureza del ADN y ARN extraído de una muestra. A260/280 valores en el rango de ≥1.8 y > 2.0 se considera generalmente para ser "puro" para ADN y ARN, respectivamente. Valores más bajos de A260/280 pueden indicar contaminación por proteínas o productos químicos orgánicos. - Verificar la calidad del ADN mediante la ejecución de una electroforesis en agarosa al 0,7%. Preparar el gel y en 1 x TRIS-bórico EDTA-buffer (TBE: 89 mM Tris, ácido bórico de 89 mM y EDTA 2 mM) a 100 V por 30 min alta calidad gDNA aparece como un agudo, alto peso molecular (HMW) banda con sin extensiones en el rango de moléculas de bajo peso molecular (LMW).
- Compruebe el RNA aislado para cantidad, pureza e integridad bajo desnaturalizar condiciones mediante una electroforesis en gel de agarosa guanidina tiocianato (GTC) o viruta de electroforesis capilar, según las instrucciones del fabricante.
- Preparar el gel GTC agregando 5 mM GTC a un gel de agarosa al 1% x TBE 1 estándar después de enfriar el agar hasta 60 ° C.
Nota: GTC es tóxico, así que dispensar en una campana de humos y usar equipo de protección personal. - Preparación de RNA desnaturalización buffer de carga: 95% formamida, 10 mM EDTA pH 8.0, azul de bromofenol 0.1% 0.1% xileno cyanole y bromuro de etidio 10 μl.
Nota: Formamida con bromuro de etidio son tóxico y deben ser dispensados en una campana de humos. - Carga 1-5 μg de ARN total en RNA desnaturalización buffer de carga, calor la mezcla durante 5 minutos a 70 ° C, coloque en hielo antes de cargar en un gel y luego separar RNA en gel de GTC a 100 V por 45 min carga de ADN o ARN de peso molecular marcador como estándar junto con la muestra de RNA.
- Teñir los geles con bromuro de etidio y visualizar las bandas utilizando sistemas de captura de imagen bajo luz ultravioleta. En eucariotas, ARN total intacto en desnaturalizar condiciones mostrará por lo menos dos nítida y clara rRNA bandas (28S y 18S) con una relación 2:1 intensidad.
- Preparar el gel GTC agregando 5 mM GTC a un gel de agarosa al 1% x TBE 1 estándar después de enfriar el agar hasta 60 ° C.
- Eliminar restos de gDNA por el tratamiento con DNasa (DNasa I libre de ARNasas). Añadir a un tubo libre de Rnasa en el volumen total de 10 μl: 0.1 - 1 μg de ARN total, una unidad de la DNasa I y 1 μl de reacción 10 x tampón con MgCl2. Incubar la mezcla durante 30 min a 37 ° C. Terminar la reacción agregando 1 μl de EDTA 50 mM y la incubación a 65 ° C durante 10 minutos.
- Eliminar los restos de RNA del gDNA extractos utilizando libre de DNasa Rnasa A, según protocolo del fabricante. Añadir 5 μl de Rnasa 10 mg/mL de ADN total y se incuba a 37 ° C para los extractos de tienda de ARN y ADN de 1 h. a-80 ° C.
2. el primer diseño de rDNA región para gDNA ensayo
Nota: La secuencia completa de rDNA contiene dos regiones (ITS1 e ITS2), que se eliminan en la molécula de ARNr maduros por una serie de segmentación endonucleolítica y luego degradación (figura 1).
- Recuperar la secuencia de nucleótidos de rDNA de NCBI (http://www.ncbi.nlm.nih.gov) para las especies de interés. La mejor palabra clave para buscar la base de datos es "espaciador transcrito interno."
- De entrada la secuencia de nucleótidos de destino en una búsqueda de BLASTn para encontrar regiones del espaciador transcrito interno (STI), SSU y LSU conserva regiones.
- Seleccione cartillas que cualquiera flanquean una secuencia de STI o que amplifican secuencias de STI que no están presentes en el ARNr maduros.
- Diseño de cebadores que flanquean las secuencias de ITS1, ITS2: alinear las regiones conservadas de diversas especies de ClustalW. Diseño de cebadores específicos para la región que flanqueaba después análisis específico/especie de taxa con AlleleID software. El primer dos pares amplificación SSU-5.8S y 5.8S-LSU amplicones pueden ser diseñado en base a las regiones que flanqueaban del ITS1 e ITS2, respectivamente. Porque estos amplicones se que abarca toda la región ITS, se incrementará la longitud del amplicón para por lo menos 300 bp en amplicones del gDNA. Este aumento reduce la sensibilidad.
- Flanqueando ITS1: Seleccione SSU y 5.8S secuencia rRNA. Las cartillas para Poaceae son: SSU, SF: CGTAACAAGGTTTCCGTAGGTG, GGTTCACGGGATTCTGCAAT R:. Este par de cartilla (SF: adelante y atrás R:) amplifica la región parcial de SSU, la larga duración de ITS1 y la región parcial de 5.8S rDNA.
- Flanqueando el ITS2: Seleccione 5.8S y secuencia LSU. Las cartillas para Poaceae son: secuencia consenso de F: ATTGCAGAATCCCGTGAACC LSU, LR: TGCTTAAAYTCAGCGGGTAGYC. Este par de primer amplifica la región parcial de 5.8S, la larga duración de ITS2 y la región parcial de LSU (figura 1).
Nota: En el caso del primer diseño basado en ITSs que flanquean la región, las áreas altamente conservadas de SSU, 5.8S y LSU se identificaron. Los iniciadores de avance y retroceso de 5.8S rRNA fueron diseñados en base a un motivo conservado en plantas con flores14. Los iniciadores de avance y retroceso fueron diseñados en base LSU y SSU conservaron regiones en Poaceae, respectivamente. La divergencia de SSU y LSU cartillas para cada especie se da en la tabla 1.
- Cebadores de amplificación de una secuencia de ITSs: en este protocolo, iniciadores ITS1 de diseño basaban en Aeluropus su secuencia (número NCBI taxid: 110873). Para imprimaciones, use: adelante: GGTATGGCGTCAAGGAACACT, revés: ATAGCATCGCTGCAAGAGGT. Según los amplicones generada por el primer pares en silico, el tamaño debe variar de 60 a 200 bp. Este es también el tamaño recomendado para el análisis de qPCR.
- Diseño de cebadores que flanquean las secuencias de ITS1, ITS2: alinear las regiones conservadas de diversas especies de ClustalW. Diseño de cebadores específicos para la región que flanqueaba después análisis específico/especie de taxa con AlleleID software. El primer dos pares amplificación SSU-5.8S y 5.8S-LSU amplicones pueden ser diseñado en base a las regiones que flanqueaban del ITS1 e ITS2, respectivamente. Porque estos amplicones se que abarca toda la región ITS, se incrementará la longitud del amplicón para por lo menos 300 bp en amplicones del gDNA. Este aumento reduce la sensibilidad.
- Recoger los iniciadores teniendo en cuenta estas recomendaciones: contenido de GC: 40-60%, primer longitud: longitud de producto PCR baja, 18-23: 60-160 bp (especialmente para su cartilla), temperatura (Tm) de fusión: 60 ° C, la final Tm para ambos primers no difieran más de 5 ° C y el primitivo ERS no son complementario a los mismos o socios iniciadores.
- Compruebe el primer especificidad y número de copia. Realizar análisis en silico de la secuencia de las cartilla programa de cartilla-blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
- Abra la página de presentación del Primer chorro. Entre dos secuencias de la cartilla en la sección de parámetros de la cartilla de la forma. En la especificidad de par primer sección parámetros, introduzca un organismo nombre (o nombre de grupo de organismo) y seleccione el base de datos del genoma. Estos ajustes dan la información de especificidad acerca de parámetros de secuencia y cartilla de destino, incluyendo producto longitud, posición en el cromosoma y copiar el número.
3. realizar paso de qPCR para la validación de los Primers basados en el rDNA con plantillas de la DNA
Nota: La funcionalidad de los cebadores diseñados debe validarse mediante la realización de qPCR utilizando gDNA como plantilla. Para realizar varias reacciones paralelas y reducir los errores de pipeteo, se recomienda la preparación de la mezcla principal. Para una mezcla principal, preparar un volumen equivalente a la cantidad total de mezcla de reacción más el ~ 10%.
- Preparar una mezcla maestra mezclando todos los componentes de reacción excepto la plantilla de la DNA en un tubo de reacción de PCR. Como falta de una reacción para preparar la mezcla maestra de lujo: 5 μl SYBR green (SYBR) master mix (x 2), primer μl 0,3 (0,3 μm cada del primer avance y retroceso) y ajustar el volumen final de 10 μl con agua libre de ARNasa. Utilice aproximadamente ≤ 200 ng en 1 μl del gDNA plantilla para el análisis.
Nota: Descongelar, montar y mantener todos los reactivos, los componentes y mezclas de reacción sobre hielo. - Parte alícuota de la mezcla de maestra en una placa de 96 pocillos óptica. Pipeta 1 μl del gDNA a cada pozo y luego cubrir con la placa óptica de película. Girar y colocar en cycler.
- Ejecutar el ensayo qPCR en un termociclador en tiempo real bajo las siguientes condiciones: 10 min a 95 ° C seguido de 40 ciclos de 95 ° C por 15 s y 60 ° C durante 1 minuto realizar adquisición de datos en el recocido/extensión de 60 ° C la paso.
- Después del procedimiento de amplificación, someter todas las reacciones de PCR para un análisis de la curva fusión con medición continua de la fluorescencia de 55 ° C a 95 ° C. Por lo general, recoge un punto de datos cada ciclo por un progresivo aumento de la temperatura de 0,5 ° C por ciclo.
Nota: Incluir al menos 2 controles no es de plantilla (NTC) para cada mezcla maestro primer par. Realizar todos los ensayos en al menos tres repeticiones. - Confirmar la especificidad de la cartilla mediante el análisis de la curva de fusión. Analizar las curvas con el ciclo umbral único y método de ajuste de curva resta.
Nota: La aparición de un fuerte pico individual indica un amplicón individual uniforme. Primer dímero productos pueden aparecen como picos individuales a temperaturas más bajas. - Validar el tamaño de cada amplicón por electroforesis en gel de agarosa.
- Preparar un gel de agarosa al 3% mediante la mezcla de agarosa al 3 g con 100 mL de tampón TBE (TBE: 89 mM Tris, ácido bórico de 89 mM y EDTA 2 mM).
- Mezclar 5-10 μl del producto de PCR con ADN y 1-2 μl de 6 x de buffer de carga. Producto de la polimerización en cadena junto a una escalera de DNA en gel de agarosa al 3% de la carga. Llevar a cabo una separación electroforética en 1 x de tampón EDTA TRIS-bórico a 100 V durante 45 minutos.
- Teñir los geles con bromuro de etidio o cualquier otro agente intercalante y visualizar las bandas utilizando sistemas de captura de imagen bajo luz ultravioleta.
Nota: Agentes intercalante de ADN (p. ej., bromuro de etidio) son cancerígenos y deben manejarse con cuidado y dispensados por separado. La aparición de una única banda aguda (con respecto a tamaño y sin primer-dimer o amplificación de fondo artificial) confirma la especificidad de los productos.
4. gDNA procedimiento de análisis de contaminación con ARN plantillas
Nota: Después del tratamiento con DNasa, el RNA purificado muestra es probada por cebadores específicos de ADNr. Debido a la transformación de la función como del intrón de la ITSs cuando estas regiones se utilizan para la amplificación, no hay señal de amplificación debe ser detectada en las muestras de RNA de DNA libre. Basados en esto, si se detecta una señal de amplificación en qPCR o una banda en el gel de agarosa observada con el tamaño esperado (estimado por análisis de silico ), que esto debe ser debido a la contaminación del gDNA. Los pasos realizados en esta sección, son similares a la sección 3, excepto que el cDNA de todas las muestras se utiliza como plantilla en vez de gDNA.
- Preparar una mezcla maestra mezclando todos los componentes de reacción excepto plantilla de RNA en un tubo de reacción de PCR. Amo la combinación de mezcla para una reacción: 5 μl SYBR master mix (x 2), primer μl 0,3 (0,3 μm cada mezcla primer avance y retroceso) y ajustar el volumen final de 10 μl con agua libre de ARNasa. Utilice aproximadamente 500 ng plantilla RNA 1 volumen μL para el análisis.
- Parte alícuota de la mezcla principal en una placa de 96 pocillos óptica. Pipetee 1 RNA μl a cada pozo y luego cubrir por la placa óptica de película. Centrífuga y el lugar en cycler.
Nota: Incluir al menos dos controles NTC y dos controles positivos gDNA para cada ensayo. Realizar todos los ensayos en tres repeticiones técnicas. - Ejecutar el ensayo qPCR en un termociclador en tiempo real bajo las siguientes condiciones: 10 min a 95 ° C seguido de 40 ciclos de 95 ° C por 15 s y 60 ° C durante 1 minuto realizar adquisición de datos en el recocido/extensión de 60 ° C la paso.
- Después del procedimiento de amplificación, someter todas las reacciones de PCR para un análisis de la curva fusión con medición continua de la fluorescencia de 55 ° C a 95 ° C. Generalmente, recoger un punto de datos cada ciclo por un progresivo aumento de la temperatura de 0.5 ° C por ciclo.
- Ver todos los productos PCR de ejecución en agarosa al 3% gel de electroforesis.
Nota: La aparición de cualquier banda o pico de la reacción de NTC se relaciona probablemente con formación de primer-dimer que generalmente se observa a temperaturas bajas en la curva de fusión, mientras que la presencia de alguna banda o pico en las muestras de RNA es el resultado de contaminación gDNA. Se recomienda primero probar todas las muestras de RNA cebadores basados en el rDNA, y entonces no DNA muestras contaminadas se utilizan para aplicaciones posteriores, tales como análisis de expresión génica, síntesis de cDNA, etcetera.
5. paso RT-PCR de cDNA síntesis y análisis de qPCR
- Descongele la DNasa-tratamiento RNA y los reactivos de síntesis de cDNA a temperatura ambiente. Después de descongelar, desactivación de los reactivos. Añadir 1 μg de ARN y 1 μl de Oligo (dT) 18 cartilla en un tubo libre de nucleasas. Ajustar el volumen total de 12 μl con agua libre de ARNasa, mezclar suavemente y luego almacenar en hielo.
- Estructuras secundarias de plantilla del RNA del derretimiento por incubando la reacción a 65 ° C por 5 min vuelta abajo y enfríe el frasco en el hielo.
- Preparar la mezcla principal de reacción (volumen final de 20 μl de cada reacción) como sigue: 1 μl de transcriptasa reversa (200 U/μL), 4 μL de tampón de reacción (x 5), 1 μl de inhibidor de Rnasa (20 U/μL) y 2 μl de dNTP Mix (10 mM). Mezclar suavemente y enfriar el frasco en el hielo. Añadir 19 μl al tubo preparado que contiene el ARN.
- Incubar la reacción durante 60 min a 42 ° C y luego incubar a 70 ° C por 5 min terminar la actividad de la transcriptasa reversa. Lugar las reacciones de RT en el hielo y proceder a análisis de expresión génica mediante procedimiento de rutina qPCR (como se explica en la sección 3 y 4).
Resultados
Proponemos el uso de cebadores basados en el rDNA para validar la ausencia de contaminación gDNA en las muestras de RNA de tejido foliar. El diagrama de flujo del análisis de qPCR análisis y gDNA contaminación se muestra en la figura 2. En el protocolo presentado, se utilizaron dos estrategias complementarias para diseño de base de rDNA: cartillas 1) específicos para cada especie se seleccionaron de ITSs secuencias y cartillas 2) universales fueron seleccionados de la ITSs que flanquean regiones. Para prueba de concepto, diseñamos cebadores específicos para Aeluropus littoralis y cebadores universales basados en especies de Poaceae, tal como se indica en el protocolo. El 5.8S cartillas de avance y retroceso fueron seleccionados en base a un motivo conservado de 14 par base (bp) que muestra la similitud entre las plantas con flores, briofitas y varias órdenes de algas y hongos14. Las características de los primers diseñados se dan en la tabla 2. La universalidad de SSU, 5.8S y LSU primer fueron comprobados por BLASTn y cartilla homología resultados se presentan en la figura 3 como una insignia de adorno. La lista de especies incluidas en el análisis de homología como los iniciadores divergentes para cada especie se dan en tabla 1. Primer especificidad fue del cheque por la explosión de la cartilla. Para las especies donde la secuencia del genoma entero está disponible, se estimó la localización cromosómica de genes de ADNr. Por ejemplo, en Oryza sativa y Arabidopsis thaliana, rDNA genes están localizados en dos cromosomas distintos y en Zea mays en tres cromosomas diferentes.
validación de qPCR de cartillas de rDNA se realizó con análisis de la curva de amplicones ITS1 e ITS2-flanco usando la DNA como plantilla de fusión. Tal como se presenta en figura 4 y figura 5, especificidad cartilla fue confirmada experimentalmente por la observación de un solo pico agudo con ninguna formación primer-dimer en diferentes especies de poáceas como Triticum aestivum, Hordeum vulgare, Oryza sativay en las dicotiledóneas Medicago sativa, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia fabay Arabidopsis thaliana. La prueba más de los productos de amplificación por separación electroforética tamaño mostraron una única banda. Como era de esperar, las bandas derivan de las muestras de las diferentes especies variadas en tamaño (figura 6A y 6B). Curiosamente, el uso de los iniciadores universales diseñados específicamente para las tres especies de Poaceae no sólo son útiles para otras especies de Poaceae, pero también para otras especies como a. thaliana y para un hongo endofítico viz. Piriformospora indica.
La validez de la cartilla específica diseñada (ITS1) también fue confirmada por qPCR en a. littoralis usando gDNA como plantilla. Se observó un solo pico con ninguna formación primer-dimer. Sorprendentemente, la cartilla de ITS1 a. littoralis (como el primer específico) genera una única banda sharp no sólo en a. littoralis, sino también para todas las demás especies de excepción de Nicotiana tabacum y Trifolium alexandrinum, que produjo dos bandas (figura 6). Realizó el ensayo de contaminación gDNA cebadores ITS o su acompañamiento en todas las muestras de RNA. Una representación esquemática de la placa de amplificación en el ensayo de contaminación gDNA y la interpretación de resultados se presentan en la figura 7.
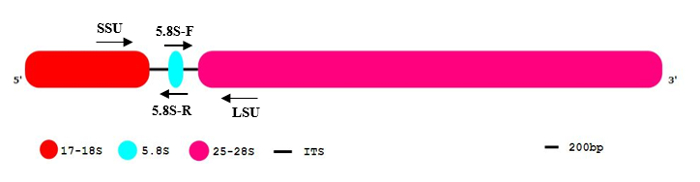
Figura 1: El patrón general de organización de la secuencia de ADNr eucariótico.
El segmento de ADNr eucariótico contiene 17-18 años (rojo), 5.8S (azul) y 25-28S rRNA (rosa). Los espaciadores transcritos internos (ITS) están indicados como líneas negras. 5´and 3´ indican la orientación de la molécula de ADN. Haga clic aquí para ver una versión más grande de esta figura.
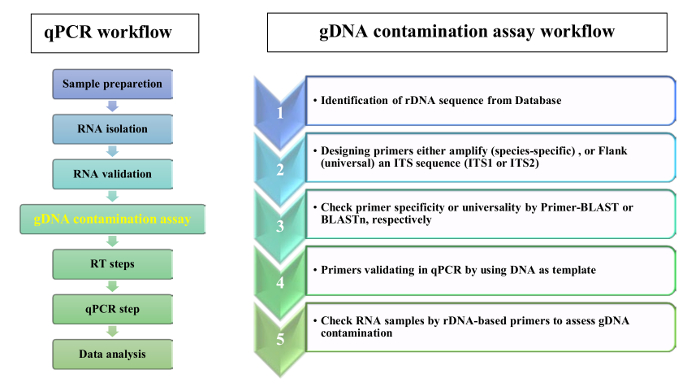
Figura 2: flujo de trabajo para un análisis de contaminación RT-qPCR y gDNA. Haga clic aquí para ver una versión más grande de esta figura.
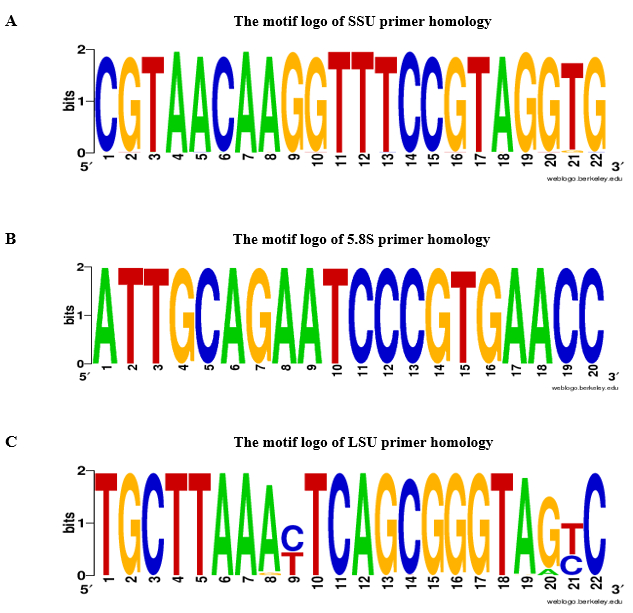
Figura 3: logo de adorno de A. SSU, B. 5.8S y homología de LSU C. cartilla. Para SSU, 5.8S, y cartillas LSU, el logo de adorno fue construido por BLASTn basado en 2.000 registros de planta verde (número NCBI taxid: 33090) con un corte e valor ≤10-10. A-adenina, T timina, G Guanina, C citosina. Haga clic aquí para ver una versión más grande de esta figura.
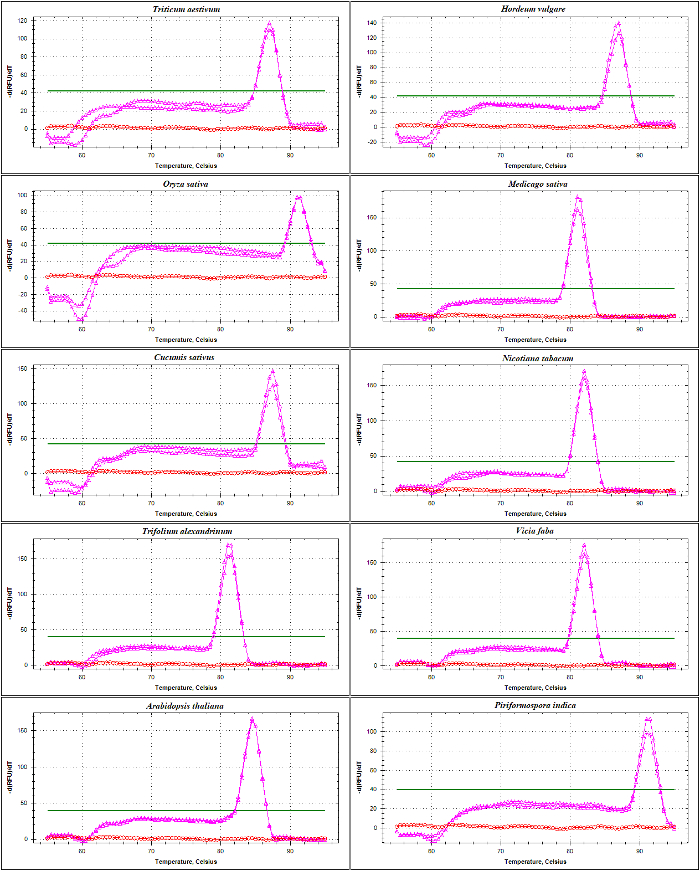
Figura 4: El derretimiento análisis de la curva de amplicones ITS1-acompañamiento en las diferentes especies.
Este amplicón, amplificado por SSU y 5.8S-imprimaciones R, contiene parte de la secuencia de la región de codificación de 17-18 años, toda la secuencia de ITS1 y secuencia parcial de 5.8S. Se muestran las curvas de fusión de amplicones generados (rosa) y NTC (rojo) de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, Arabidopsis thaliana y Piriformospora indica. La línea plana indica el umbral de la línea de base. Haga clic aquí para ver una versión más grande de esta figura.
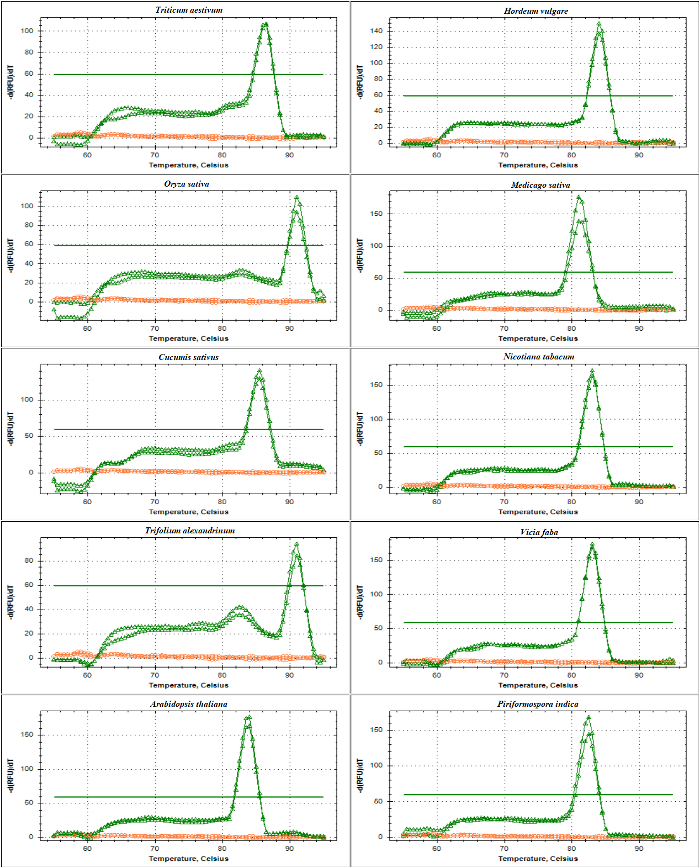
Figura 5: El análisis de curva derretimiento de amplicones que flanqueaban el ITS2 en diferentes especies.
Este amplicón generado por el uso de 5.8S-cartillas F y LSU. El amplicon descrito contiene secuencias de parte de 5.8 S, toda la secuencia de ITS2 y una secuencia parcial de 25-28S. Se muestran las curvas de fusión de los amplicones (verde) y NTC (rojo) generados a partir de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, Arabidopsis thaliana y Piriformospora indica. Haga clic aquí para ver una versión más grande de esta figura.
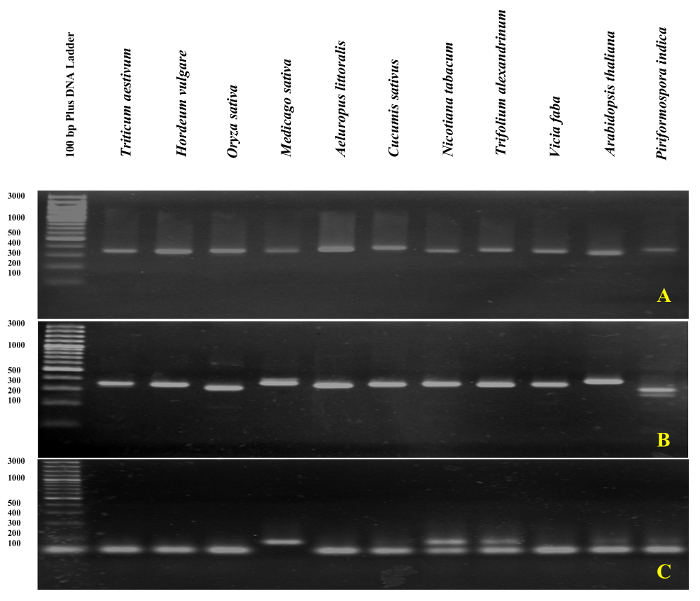
Figura 6: Análisis de gel agarosa rDNA-base del producto de PCR.
El amplicon de ITS1-flancos (A), flancos de ITS2 (B) y ITS1 (C) se ejecuta en gel de agarosa al 3%. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Intrón-como las características de la ITSs pueden considerarse para el diseño de primers que detectan contaminación gDNA.
Cualquier pico o banda con el tamaño esperado en análisis de qPCR indican contaminación gDNA en la muestra de RNA. UNK: muestra desconocida, pos: control positivo, NTC: control no es de plantilla. Haga clic aquí para ver una versión más grande de esta figura.
| Cartilla de | Género | ID de taxid | Especies | Primer divergente | ||
| SSU | Arabidopsis | 3701 | kamchatica, thaliana y lyrata | - | ||
| Vicia | 3904 | villosa, americana, unijuga, amoenane, amurensis, craccamal, pseudo-orobus, multicaulis, japonica, ramuliflora y faba | ||||
| Trifolium | 3898 | alexandrinum, montanum, resupinatum y repens | - | |||
| Nicociana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, moscada, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atenuar, acuminata, linearis, alata, sylvestris, rustica y suaveolens | - | |||
| Cucumis | 3655 | sandía, melo y sativus | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | No encuentra la cartilla | |||
| Medicago | 3877 | sativa, salto, pamphylica, lunata, rostrate, plicata y truncatula | - | |||
| Oryza | 4597 | sativa, glumipatula, rufipogon barthii glaberrima punteada, longistaminata, meridionalis, nivara, meridionalis y longistaminata | - | |||
| Triticum | 4564 | aestivum, urartu y monococcum | - | |||
| Hordeum | 4512 | bulbosum, marinum, vulgare, brevisubulatum y bogdanii | - | |||
| LSU | Arabidopsis | 3701 | petraea, thaliana y lyrata | TGCTTAAACTCAGCGGGTAATC | ||
| Vicia | 3904 | sylvatica, tetrasperma, sativa, sepium hirsutos, parviflora, cracca, lathyroides, orobus, orobus, bithynica y faba | TGCTTAAATTCAGCGGGTAGCC | |||
| Trifolium | 3898 | pretexto, nigrescens, resupinatum, occidentale, subterraneum, strictum, ochroleucon, glomeratum, Squamosum ornithopodioides y repens | TGCTTAAATTCAGCGGGTAGCC | |||
| Nicociana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, moscada, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atenuar, acuminata, linearis, alata, sylvestris y suaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| Cucumis | 3655 | Melo, ritchiei y javanica | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | lagopoide pungens y littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| Medicago | 3877 | ruthenica sativa, salto, arabica, polymorpha y mínimos | TGCTTAAATTCAGCGGGTAGCC | |||
| pamphylica, lunata, rostrate y plicata | TGCTTAAACTCAGCGGGTAGTC | |||||
| Oryza | 4597 | sativa, glumipatula, rufipogon, barthiial, glaberrima, australiensis, officinalis, australiensis, ridleyi, malampuzhaensis, alta, nivara, rufipogon, meridionalis y longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| Triticum | 4564 | aestivum, spelta, turgidum, dicoccoides, petropavlovskyi, urartu y monococcum | TGCTTAAACTCAGCGGGTAGTC | |||
| Hordeum | 4512 | vulgare, bulbosum, murinum, secalinum, brevisubulatum y bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| Una cartilla degenerada se define como sistema IUPAC para nomenclatura de nucleótidos | ||||||
Tabla 1: Lista de especies consideradas para recoger cartillas de ADNr.
Sitio de unión de SSU en comparación con el sitio de unión de LSU demostrada mayor homología de la secuencia sobre dado el género.
| Longitud del amplicón | Área de amplificación | Secuencia de | Nombre de la cartilla | Amplicones | |
| 332 - 405 bp | Secuencia parcial de SSU, toda la secuencia de ITS1 y secuencia parcial de 5.8S | CGTAACAAGGTTTCCGTAGGTG | SSU | ITS1-flancos | |
| GGTTCACGGGATTCTGCAAT | 5.8S-R | ||||
| 318 - 361 bp | Secuencia parcial de 5.8S, toda la secuencia de ITS2 y secuencia parcial de LSU | ATTGCAGAATCCCGTGAACC | 5.8S-F | ITS2-flancos | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100 - 200 bp | ITS1 | GGTATGGCGTCAAGGAACACT | ITS1-F | ITS1 | |
| ATAGCATCGCTGCAAGAGGT | ITS1-R | ||||
Tabla 2: Secuencias de la cartilla.
Discusión
Análisis de expresión génica mediante PCR cuantitativa se ha aplicado extensamente en los últimos años. La principal ventaja de este método automatizado, rápido y rentable es su resultado exacto y preciso. Sin embargo, obtener beneficios óptimos de estas ventajas requiere un claro entendimiento de la configuración de los parámetros utilizados para el experimento de qPCR. Para obtener un resultado confiable en análisis de expresión génica de qPCR, es necesario para evitar la amplificación no específica que surge del primer-dimer o gDNA contaminación en la muestra de RNA3,15. Se espera que los niveles de transcripción de RNA se debe subestimarse gDNA contaminación8. Aquí, las características únicas de un gen ADNr fue considerado para un ensayo de contaminación gDNA en las muestras de RNA.
Las propiedades básicas del rDNA en este protocolo: Genes ribosómicos consisten en el dos ITSs, es decir, ITS1 y ITS2 y los tres genes de la codificación del rRNA, 17-18 años, 5.8S y 28S 25 subunidad12. Las dos regiones ITS no son parte de la secuencia de codificación de las subunidades ribosomales. Se retiran por al menos tres actividades enzimáticas para procesar el precursor madurar rRNA: una actividad endonucleasa, helicasa y exonucleasa. Como el ARN ribosómico se transcribe como una transcripción policistrónico, un producto primario que contiene las ITSs está ciertamente presente. El proceso es muy rápido y tiene lugar en el nucleolo, y la cantidad de moléculas del precursor detectable con el su es por debajo del límite de detección del método de qPCR. Por lo tanto, cuando ITS1 o ITS2 se amplifican por su acompañamiento cartillas, amplificación no puede detectarse en las muestras de RNA a menos que se presente contaminación gDNA. Se estimó el número de genes de ADNr en el genoma de los organismos eucariotas para incluir hasta 1 mil copias, que están organizadas en matrices simples o tandem en los cromosomas11. En este protocolo, se propone una forma alternativa, en lugar de NRT, detectar gDNA contaminación, que se utiliza en cada análisis de reacción.
Beneficios y limitaciones con respecto a los métodos existentes: NRT se suele utilizar para probar si la muestra de RNA preparada es limpia o contaminada por gDNA. GDNA contaminación no se distribuye uniformemente entre diferentes muestras de RNA, y la sensibilidad de la reacción a gDNA es afectada significativamente por los genes analizados, NRT controles son necesarios para cada muestra/ensayo par7,15. Esto añadirá considerablemente costos y trabajo al manipular muchas muestras simultáneamente3,9. Otros métodos alternativos, documentados en la literatura incluyen el uso de primers específicos del intrón para la detección del gDNA o diseñar cebadores que flanquean un intrón o abarcan a un empalme exón-exón. Las limitaciones de estos métodos se derivan de la falta de información de secuencia del intrón, anotación incompleto de la estructura del intrón/del exón y la ausencia de intrones en los genes o pseudogenes de interés1,4,10 . Debido a la evolución, existen genes de ADNr como familias génicas multigénica y altamente conservado. Son muy abundantes en el genoma y presentes en diferentes cromosomas13. En comparación con otros genes de codificación o nonconding, los genes de ADNr muestran el mejor ajuste para la detección de contaminación gDNA. En análisis transcriptómico comparativa, la normalización de datos qPCR por calibrador de rRNA no se recomienda para algunas cuestiones, como las diferencias en cDNA preparación (priming de polyA vs random hexamer cebado), grandes diferencias en abundancia entre rRNA y mRNA , y la biogénesis diferentes que puede generar engaño10,16. Sin embargo, los problemas que acabamos de mencionar son una ventaja para el ensayo de contaminación gDNA. Por ejemplo, con respecto a la mayor abundancia de sitio targeting en el genoma y la localización en cromosomas diferentes, cebadores basados en el rDNA mejoran significativamente la sensibilidad de detección del gDNA en comparación con los métodos existentes.
Versatilidad de rDNA a otro organismo: genes de ADNr son una familia de genes estudiados identificada en la mayoría de los organismos. El método basado en el rDNA propuesto representa un sistema simple, altamente sensible y económico para los ensayos de contaminación gDNA que puede adaptarse fácilmente a otros organismos eucarióticos y procarióticos (protocolo 2 - 5). Como caso de estudio, hemos demostrado aquí la utilidad de este método en algunas especies Poceae (figura 4 y figura 5). Los cebadores utilizados muestran una alta tasa de transferencia a otras especies Poceae debido a la estructura altamente conservada de rDNA subunidades entre especies. Este problema se vuelve aún más importante cuando suficiente información de la secuencia genómica no está disponible para el primer diseño. Por lo tanto, pueden utilizarse cebadores que flanquean su diseñados para una especie en una especie relacionada. También, el 5.8S-cartillas F/R fueron seleccionados partiendo de un motivo conservado que muestra gran similitud en la mayoría de las plantas con flores14. Aunque técnicas de secuenciación de alto rendimiento aumentan permanentemente el número de genomas conocidos, no se realiza la anotación del exón-intrón de la mayoría de los organismos, y tan a menudo no es posible diseñar primers para atravesar una frontera exón-exón. Nuestro método explica cómo base de rDNA iniciadores pueden ser aplicados para el ensayo de contaminación gDNA en análisis de qPCR de procariotas y eucariotas, con el objetivo de eliminar los costosos controles NRT en cada combinación de ensayo/primer.
Divulgaciones
Los autores tienen intereses financieros que compiten.
Agradecimientos
Esta investigación fue apoyada por la genética y biotecnología agrícola Instituto de Tabarestan (GABIT), Sari ciencias agrícolas y recursos naturales Universidad (SANRU). El grupo junior de investigación genómica de estrés abiótico fue financiado por IZN (centro interdisciplinario de investigación de cultivos vegetales, Halle (Saale), Alemania. Agradecemos a Rhonda Meyer para la lectura crítica del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
Referencias
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18(2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51(2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51(2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7(2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880(2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados