Method Article
Valutazione della contaminazione da DNA a RNA campioni basati sul DNA Ribosomal
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per l'analisi genomica contaminazione da DNA (gDNA) in campioni di RNA. Il metodo proposto utilizza gli iniettori specifici per la regione di distanziatore interno trascritti (ITS) di geni ribosomali DNA (rDNA). Il metodo è adatto per il rilevamento affidabile e sensibile di contaminazione da DNA nella maggior parte dei eucarioti e procarioti.
Abstract
Un metodo ampiamente utilizzato per la quantificazione dei cambiamenti di espressione genica e abbondanze di trascrizione è d'inversione-trascrizione PCR quantitativa in tempo reale (RT-qPCR). Fornisce risultati accurati, sensibile, affidabile e riproducibile. Diversi fattori possono influenzare la sensibilità e la specificità del RT-qPCR. DNA genomico residuo (gDNA) contaminando campioni di RNA è uno di loro. Nell'analisi dell'espressione genica, amplificazione non specifico dovuto contaminazione gDNA sarà sopravvalutare l'abbondanza dei livelli della trascrizione e può influenzare i risultati di RT-qPCR. In genere, gDNA viene rilevato mediante qRT-PCR utilizzando primer coppie ricottura a regioni intergeniche o un introne del gene di interesse. Purtroppo, le annotazioni essone / dell'introne non sono ancora noti per tutti i geni da vertebrato, batteri, protisti, funghi, pianta e specie di Metazoi invertebrati.
Qui presentiamo un protocollo per rilevazione di contaminazione gDNA in RNA campioni usando il DNA ribosomiale (rDNA)-base di primer. Il metodo si basa su caratteristiche uniche di rDNA: loro natura multigenica, sequenze altamente conservate e ad alta frequenza nel genoma. Anche come caso di studio, un unico set di primers sono stati progettati in base alla regione conservata di DNA ribosomiale (rDNA) della famiglia delle graminacee. L'universalità di queste coppie di primer è stata testata da fondere curva analisi e agarosio elettroforesi del gel. Anche se il nostro metodo spiega come primer a base di rDNA possono essere applicate per il dosaggio di contaminazione gDNA della famiglia delle graminacee, potrebbe essere facilmente utilizzato per altre specie prokaryote e dell'eucariota
Introduzione
Esplorare la regolazione trascrizionale di interessante set di gene o reti di segnalazione è essenziale per comprendere i complessi meccanismi molecolari coinvolti in eventi biologici1. Attualmente, l'analisi qPCR è l'approccio più utilizzato per studi di espressione genica che possono essere destinati (genoma) di DNA o RNA (trascrittoma) che consentono analisi methylome e del trascrittoma, rispettivamente. Trascrizione inversa (RT) seguita da qPCR è ampiamente usata per l'analisi del trascrittoma che misurano i livelli di espressione genica in vari settori della ricerca biologica2. Rispetto ad altri metodi come l'ibridazione nordica tradizionale, il rilevamento specifico tessuto via in situ di ibridazione, nelle analisi di protezione della ribonucleasi (RPA) e semi-RT-PCR, la precisione, convenienza, velocità e ampia gamma dinamica del analisi qPCR-based sono altamente notevole3,4. Ci sono diversi fattori importanti che devono essere considerati per una quantificazione affidabile di RNA messaggero (mRNA), tra cui la qualità e la quantità di RNA materiale di partenza. Inoltre, amplificazione aspecifici, l'efficienza di RT-qPCR ed efficienza PCR devono essere considerati5,6.
La presenza di gDNA è un problema inerente durante l'estrazione di RNA, in parte, per le proprietà fisiche e chimiche simili di DNA e RNA7. A causa dell'identità di sequenza di DNA complementare (cDNA) derivate da campioni di mRNA e gDNA, amplificazione aspecifici può verificarsi, che influenzano l'accuratezza dei risultati di RT-qPCR. Il restante gDNA porterà alla sopravvalutazione dell'abbondanza del mRNA di destinazione nel gene espressione analisi8.
Fondamentalmente, l'amplicone non specifici si pone principalmente da formazione di primer-dimero o amplificazione di sfondo non specifico dovuto gDNA, entrambi i quali può essere valutati utilizzando appropriati campioni di controllo. Tali campioni sono senza controllo del modello (NTC) e nessun controllo della trascrittasi inversa (NRT), rispettivamente. Poiché i livelli di contaminazione gDNA nei campioni in fase di studio sono diversi e la sensibilità verso gDNA differisce notevolmente tra i geni analizzati, i controlli NRT sono necessari per ogni coppia di campione/dosaggio. Anche se questo aumenta notevolmente il costo e lavoro negli studi di analisi di RT-qPCR, questi controlli sono necessari7,9.
Metodi alternativi a che fare con gDNA contaminazione includono l'uso di coppie di primer annealing regioni intergeniche o un introne del gene di interesse10e l'uso degli iniettori che fiancheggiano un introne grande o estendersi su una giunzione esone-esone, cioè i siti di ricottura sono assenti nel mRNA maturo sequenza1,4. Tuttavia, le annotazioni di essone / dell'introne per tutti i geni da molti vertebrati, batteri, Protista, funghi, piante e specie di Metazoi invertebrati sono ancora noti. Inoltre, molti organismi eucarioti hanno pseudogeni dagli eventi di duplicazione. Ulteriormente, disegno di primer in introni non garantisce la non-amplificazione di gDNA. Come la cromatina accessibilità delle regioni genomiche di dnasi varia, si raccomanda di progettare coppie di primer diversi cromosomi differenti10di targeting.
I genomi di organismi eucariotici possono comprendere fino a un migliaio di copie di rDNA geni codificanti subunità ribosomali necessaria per formazione di ribosomi. Questi geni rDNA sono spesso organizzati in singole o ripetizione in tandem matrici11. I rRNAs Polycistronic (Figura 1) tra cui la grande unità secondaria (LSU) e la piccola unità secondaria (SSU) sono trascritti dalla RNA polimerasi I (RNA pol io). La risultante pre-rRNA è ulteriormente trattati eliminando le due regioni di distanziatore trascritto interno ITS1 e ITS2. Come prodotti finali, tre coppie i rRNAs, 17-18S rRNA (SSU), 5.8S e 25-28S rRNA (LSU) sono generati12. i geni rDNA sono tipici rappresentanti di una famiglia multigenica con sequenze altamente conservate. Si presentano con una frequenza elevata nel genoma e sono potenzialmente presenti in più di una posizione cromosomica13. L'elaborazione del rRNA e il degrado dei distanziali trascritti è un processo veloce nel nucleolo. A causa dell'elevato grado di ripetitività, il rapporto del numero di copie genomiche e premolecules di RNA non trasformati rilevabile è inferiore rispetto alle sequenze di basso-copia introne e precursori unspliced. Queste caratteristiche rendono geni rDNA adatti per rilevamento affidabile e altamente sensibile di gDNA contaminazione nella maggior parte dei eucarioti e procarioti3.
Qui è descritta una procedura innovativa per la rilevazione di contaminazione gDNA in campioni di RNA. Un set di primer universale basata sulla sequenza di rDNA conservato è presentato per gDNA saggi in diverse specie di Poaceae. La specificità e universalità dei primer proposti sono stati testati da analisi della curva di fusione utilizzando il DNA come un modello. Il nostro protocollo non è solo applicabile per Poaceae, ma potrebbe essere anche facilmente adattato ad altre specie eucariotici e procariotici.
Protocollo
Nota: Può essere utilizzato qualsiasi tessuto.
1. estrazione di acidi nucleici da
- Mettere 100 mg di campioni di tessuto in una provetta da 2,0 mL, aggiungere due perle di 5 mm in acciaio inox e omogeneizzare il tessuto a 25-30 Hz per 30 s (omogeneizzazione durata e frequenza a seconda del tipo di tessuto) per RNA e il DNA.
- Isolare il RNA totale secondo le istruzioni del produttore.
- Isolare il DNA totale secondo le istruzioni del produttore.
- Controllare la purezza e la quantità dei campioni di RNA misurando l'assorbanza a 260 e 280 nm.
- Controllare la purezza e la quantità dei campioni del DNA di misurare l'assorbanza a 260 e 280 nm.
Nota: Mentre gli acidi nucleici assorbono la luce con una lunghezza d'onda di 260 nm (A260), l'assorbanza della luce alla lunghezza d'onda 280 (A280) può essere utilizzato per quantificare la quantità di proteine e fenoli presentano nel campione. Di conseguenza, il rapporto A260/A280 nm può essere utilizzato per valutare la purezza di DNA e RNA Estratto da un campione. Valori di A260/280 nella gamma di ≥1.8 e > 2.0 sono generalmente considerati essere "puro" per DNA e RNA, rispettivamente. I valori più bassi di A260/280 possono indicare la contaminazione da proteine o di prodotti chimici organici. - Testare la qualità del DNA a elettroforesi del gel dell'agarosi 0,7%. Preparare il gel ed eseguire in 1x tampone EDTA TRIS-borico (TBE: 89 mM Tris, acido borico 89 mM e 2 mM EDTA) a 100 V per 30 min ad alta qualità gDNA appare come un acuto, ad alto peso molecolare (HMW) band con senza sbavature nella gamma di molecole a basso peso molecolare (LMW).
- Verifica RNA isolato per quantità, purezza e integrità sotto denaturare le condizioni di un'elettroforesi su gel di agarosio di guanidina tiocianato (GTC) o dal chip di elettroforesi capillare, secondo le istruzioni del produttore.
- Preparare il gel GTC aggiungendo 5 mM GTC ad un gel di agarosio all'1% x TBE 1 standard dopo il raffreddamento l'agar a 60 ° C.
Nota: GTC è tossico, quindi dispensarlo in una cappa aspirante e indossare adeguati dispositivi di protezione personale. - Preparare il RNA denaturare tampone di caricamento: 95% formammide, 10mm EDTA pH 8.0, blu di bromofenolo 0.1%, 0,1% xilene cyanole e bromuro di etidio 10 µ l.
Nota: Formammide ed etidio bromuro sono tossico e dovrebbe essere erogato in una cappa aspirante. - Carico 1-5 µ g di RNA totale nel RNA denaturare tampone di caricamento, calore la miscela per 5 min a 70 ° C, posizionarlo sul ghiaccio prima di caricarla su un gel e quindi separare RNA su gel di GTC a 100 V per 45 min. di carico del DNA o RNA di marker di peso molecolare come standard insieme a il campione di RNA.
- Macchiare i gel con bromuro di etidio e visualizzare le bande utilizzando sistemi di cattura di immagine sotto la luce ultravioletta. Negli eucarioti, RNA totale intatto eseguita alle condizioni di denaturazione mostrerà almeno due nitide e chiare rRNA bande (28S e 18S) con un rapporto di 2:1 intensità.
- Preparare il gel GTC aggiungendo 5 mM GTC ad un gel di agarosio all'1% x TBE 1 standard dopo il raffreddamento l'agar a 60 ° C.
- Rimuovere le tracce di gDNA dal trattamento con dnasi (dnasi ho RNAsi-libera). Aggiungere in una provetta di RNAsi-libera a 10 µ l di volume totale: 0,1 - 1 µ g di RNA totale, una unità della dnasi I e 1 µ l di reazione 10X tampone con MgCl2. Incubare la miscela per 30 min a 37 ° C. Terminare la reazione aggiungendo 1 µ l di 50 mM EDTA e incubando a 65 ° C per 10 min.
- Rimuovere tracce di RNA da gDNA estratti utilizzando gratis dnasi RNasi A, secondo il protocollo del produttore. Aggiungere 5 µ l di RNAsi 10 mg/mL di DNA totale e incubare a 37 ° C per gli estratti di DNA e RNA Store h. 1 a-80 ° C.
2. primer Design da rDNA regione per gDNA Assay
Nota: La sequenza di Full-Length di rDNA contiene due regioni (ITS1 e ITS2), che sono stati rimossi nella molecola del rRNA maturo da una serie di fenditure di sia e quindi degradato (Figura 1).
- Recuperare la sequenza nucleotidica di rDNA da NCBI (http://www.ncbi.nlm.nih.gov) per le specie di interesse. La migliore parola chiave per la ricerca del database è "trascritto distanziatore interno."
- Ingresso la sequenza nucleotidica di destinazione in una ricerca di BLASTn per trovare aree di distanziatore interno trascritto (STI), SSU e LSU conservato regioni.
- Selezionare gli iniettori che sia fiancheggiano una sequenza ITSs o che amplificano ITSs sequenze che non sono presenti nel rRNA maturo.
- Progettazione degli iniettori che fiancheggiano le sequenze ITS1 e ITS2: allineare le regioni conservate da varie specie di ClustalW. Progettare gli iniettori specifici per la sua regione fiancheggiante dopo analisi specifiche/cross-specie taxa con software AlleleID. Il primer due paia d'amplificazione SSU-5.8S e 5.8S-LSU ampliconi possono essere progettato basato su regioni fiancheggianti di ITS1 e ITS2, rispettivamente. Perché questi ampliconi sono che attraversa tutta la regione ITS, la lunghezza di amplicon sarà aumentata per almeno 300 bp in ampliconi di gDNA. Questo aumento riduce la sensibilità.
- Che fiancheggiano ITS1: Selezionare SSU e 5.8S sequenza del rRNA. Selezionati gli iniettori per Poaceae sono: SSU, SF: CGTAACAAGGTTTCCGTAGGTG, r: GGTTCACGGGATTCTGCAAT. Questa coppia di primer (SF: avanti e indietro r:) amplifica la regione parziale di SSU, il full-length di ITS1 e la regione parziale di 5.8S rDNA.
- Che fiancheggiano ITS2: Selezionare 5.8S e la sequenza LSU. Vengono selezionati gli iniettori per Poaceae: sequenza consenso di f: ATTGCAGAATCCCGTGAACC LSU, LR: TGCTTAAAYTCAGCGGGTAGYC. Questa coppia di primer amplifica la regione parziale di 5.8S, il full-length di ITS2 e la regione parziale di LSU (Figura 1).
Nota: Nel caso di disegno dell'iniettore sulla regione, le aree altamente conservate di SSU, fiancheggiante ITSs basata 5.8S e LSU sono stati identificati. I primer forward e reverse di 5.8S rRNA sono stati progettati in base a un motivo conservato in piante fiorite14. I primer forward e reverse sono stati progettati in base su SSU e LSU conservato regioni in Poaceae, rispettivamente. La divergenza degli iniettori SSU e LSU per ogni specie è indicata nella tabella 1.
- Primer di amplificazione di una sequenza di ITSs: In questo protocollo, progettazione ITS1 primer basato su Aeluropus sua sequenza (numero NCBI taxid: 110873). Per primer, utilizzare: avanti: GGTATGGCGTCAAGGAACACT, Reverse: ATAGCATCGCTGCAAGAGGT. Secondo gli ampliconi generati dal primer coppie in silico, le dimensioni dovrebbero variare da 60 a 200 bp. Questa è anche la dimensione consigliata per analisi qPCR.
- Progettazione degli iniettori che fiancheggiano le sequenze ITS1 e ITS2: allineare le regioni conservate da varie specie di ClustalW. Progettare gli iniettori specifici per la sua regione fiancheggiante dopo analisi specifiche/cross-specie taxa con software AlleleID. Il primer due paia d'amplificazione SSU-5.8S e 5.8S-LSU ampliconi possono essere progettato basato su regioni fiancheggianti di ITS1 e ITS2, rispettivamente. Perché questi ampliconi sono che attraversa tutta la regione ITS, la lunghezza di amplicon sarà aumentata per almeno 300 bp in ampliconi di gDNA. Questo aumento riduce la sensibilità.
- Scegliere i primers, tenendo conto di questi consigli: contenuto di GC: 40-60%, lunghezza dell'iniettore: 18-23 base, lunghezza del prodotto PCR: 60-160 bp (specialmente per la sua primer), temperatura (Tm) di fusione: 60 ° C, la Tm finale per entrambi gli iniettori non differiscono più di 5 ° C e il prim ERS non sono complementari a se stessi o collaborare primer.
- Verifica numero di specificità e copia di primer. Eseguire analisi in silico di sequenza del primer selezionato dal programma primer-blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
- Aprire la pagina di presentazione di Primer-BLAST. Inserire entrambe le sequenze primer nella sezione parametri di primer del modulo. Nella specificità della coppia di primer controllare la sezione parametri, immettere un nome di organismo (o nome del gruppo di organismo) e selezionare il Database del genoma. Queste impostazioni dare le informazioni di specificità su parametri di primer e sequenza di destinazione compreso la lunghezza del prodotto, posizione sul cromosoma e copiare il numero.
3. eseguire qPCR passo per la convalida dei primer a base di rDNA con modelli del DNA
Nota: La funzionalità degli iniettori progettati deve essere convalidata eseguendo qPCR utilizzando gDNA come modello. Per eseguire parecchie reazioni parallele e ridurre gli errori di pipettamento, la preparazione di un mix master è consigliata. Per un mix master, preparare un volume equivalente al numero totale di reazione mix plus ~ 10%.
- Preparare un mix master con tutti i componenti di reazione tranne il modello di DNA in una provetta di reazione di PCR. Secondo le necessità di una reazione per preparare il Master mix di alto livello: 5 µ l SYBR green (SYBR) master mix (2x), primer di 0,3 µ l (0,3 µM ciascuna di primer forward e reverse) e regolare il volume finale di 10 µ l con acqua RNAsi-libera. Utilizzare circa ≤ 200 ng in 1 µ l di gDNA modello per l'analisi.
Nota: Scongelare, assemblare e conservare tutti i reagenti, i componenti e le miscele di reazione sul ghiaccio. - Aliquotare il Master mix in un'ottica piastra a 96 pozzetti. Pipettare 1 µ l di gDNA in ciascun pozzetto e poi coprire con piastra ottico pellicola sigillante. Spin e posto in cycler.
- Eseguire il test di qPCR su un termociclatore in tempo reale le seguenti condizioni: 10 min a 95 ° C seguiti da 40 cicli di 95 ° C per 15 s e 60 ° C per 1 min. acquisizione dati Perform a 60 ° C ricottura/estensione passo.
- Dopo la procedura di amplificazione, sottoporre tutte le reazioni di PCR per un'analisi della curva di fusione con misurazione della fluorescenza continuo da 55 ° C a 95 ° C. In genere, raccogliere un punto dati ogni ciclo da un aumento graduale della temperatura di 0,5 ° C per ciclo.
Nota: Includere almeno 2 controlli non modello (NTC) per ogni mix master coppia di primer. Eseguire tutte le analisi in almeno tre repliche. - Confermare la specificità di primer tramite l'analisi della curva di fusione. Analizzare le curve con il ciclo soglia singola e metodo di adattamento curva sottratto.
Nota: L'aspetto di un singolo picco acuto indica un amplicone individuo uniforme. Prodotti di primer dimero possono apparire come singoli picchi a temperature più basse. - Convalidare la dimensione di ogni ampliconi mediante elettroforesi su gel di agarosio.
- Preparare un gel di agarosio al 3% di miscelazione dell'agarosi 3G con 100 mL di tampone TBE (TBE: 89 mM Tris, acido borico 89 mM e 2 mM EDTA).
- Mescolare 5-10 µ l di prodotto di PCR con il DNA e 1-2 µ l di tampone di caricamento: 6x. Caricare il prodotto di PCR a fianco di una scaletta di DNA su gel di agarosio al 3%. Eseguire una separazione elettroforetica in 1x tampone EDTA TRIS-borico a 100 V per 45 min.
- Macchiare i gel con bromuro di etidio o qualsiasi altro agente intercalante e visualizzare le bande utilizzando sistemi di cattura di immagine sotto la luce ultravioletta.
Nota: Gli agenti intercalante del DNA (per esempio, il bromuro di etidio) sono cancerogene e dovrebbero essere maneggiati con cura e dispensati separatamente. L'aspetto di un'unica banda tagliente (per quanto riguarda dimensioni e senza primer-dimero o amplificazione artificiale sfondo) conferma la specificità del amplicon.
4. gDNA procedura di dosaggio di contaminazione con modelli di RNA
Nota: Dopo il trattamento con DNasi, il RNA purificato campione viene testato da primer specifici rDNA. Grazie alla lavorazione dell'introne-come la caratteristica di ITSs quando queste regioni sono utilizzate per l'amplificazione, nessun segnale di amplificazione dovrebbe essere rilevato in campioni di RNA DNA-free. Sulla base di questo, se viene rilevato un segnale di amplificazione in qPCR o una fascia in gel di agarosio osservata con le dimensioni previste (stimate dall'analisi in silico ), che questo dovrebbe essere dovuto contaminazione gDNA. I passaggi eseguiti in questa sezione, sono simili alla sezione 3, salvo che il cDNA di tutti i campioni è utilizzato come modello anziché gDNA.
- Preparare un mix master con tutti i componenti di reazione tranne modello di RNA in un tubo di reazione di PCR. Master mix combinazione per una reazione: 5 µ l SYBR master mix (2x), primer di 0,3 µ l (0,3 µM ciascuna delle mix di primer forward e reverse) e regolare il volume finale di 10 µ l con acqua RNAsi-libera. Utilizzare circa 500 ng modello RNA in 1 µ l di volume per l'analisi.
- Aliquotare il mix master in un'ottica piastra a 96 pozzetti. Pipettare 1 µ l RNA in ciascun pozzetto e poi coprirlo dal piatto ottico pellicola sigillante. Centrifuga e posto in cycler.
Nota: Includere almeno due controlli NTC e due gDNA positivo per ogni dosaggio. Eseguire tutte le analisi in tre tecniche successive. - Eseguire il test di qPCR su un termociclatore in tempo reale le seguenti condizioni: 10 min a 95 ° C seguiti da 40 cicli di 95 ° C per 15 s e 60 ° C per 1 min. acquisizione dati Perform a 60 ° C ricottura/estensione passo.
- Dopo la procedura di amplificazione, sottoporre tutte le reazioni di PCR per un'analisi della curva di fusione con misurazione della fluorescenza continuo da 55 ° C a 95 ° C. Di solito, raccogliere un punto dati ogni ciclo da un aumento graduale della temperatura di 0,5 ° C per ciclo.
- Controllare tutti i prodotti di PCR eseguendo il 3% di agarosio elettroforesi del gel.
Nota: L'aspetto di qualsiasi band o picco nella reazione NTC è probabilmente correlato alla formazione di primer-dimero che è veduto solitamente a basse temperature in curva di fusione, mentre la presenza di qualsiasi band o picco nei campioni di RNA è il risultato di contaminazione gDNA. Si consiglia di testare prima tutti i campioni di RNA di primer a base di rDNA, e quindi non-DNA campioni contaminati sono utilizzati per applicazioni a valle come sintesi di cDNA, analisi dell'espressione genica, ecc.
5. fase RT-PCR per la sintesi del cDNA e qPCR analisi
- Scongelare la dnasi-trattati di RNA e i reagenti di sintesi di cDNA a temperatura ambiente. Dopo lo scongelamento, rallentare i reagenti. Aggiungere 1 µ g di RNA e 1 µ l di Oligo (dT) 18 primer in un tubo privo di nucleasi. Regolare il volume totale di 12 µ l con acqua RNAsi-libera, mescolare delicatamente e quindi memorizzare sul ghiaccio.
- Sciogliere strutture secondarie di modello di RNA incubando la reazione a 65 ° C per 5 min, Spin giù e raffreddare la fiala sul ghiaccio.
- Preparare il mix master di reazione (volume finale di 20 µ l per ogni reazione) come segue: 1 µ l della trascrittasi inversa (200 U / µ l), 4 µ l di tampone di reazione (5x), 1 µ l di RNasi inibitore (20 U / µ l) e 2 µ l di dNTP Mix (10 mM). Mescolare delicatamente e lasciare raffreddare la fiala sul ghiaccio. Aggiungere 19 µ l nella provetta preparata contenente il RNA.
- Incubare la reazione per 60 min a 42 ° C e poi incubare a 70 ° C per 5 min terminare l'attività della trascrittasi inversa. Mettere le reazioni di RT sul ghiaccio e procedere all'analisi dell'espressione genica mediante procedura ordinaria qPCR (come spiegato nella sezione 3 e 4).
Risultati
Proponiamo l'uso di primer a base di rDNA per convalidare l'assenza di contaminazione gDNA nei campioni di RNA di tessuto fogliare. Il diagramma di flusso di qPCR analisi e gDNA contaminazione test è illustrato nella Figura 2. Nel protocollo presentato, due strategie complementari sono state usate per il disegno di primer rDNA-base: 1) specie-specifici primer sono stati selezionati da ITSs sequenze e 2) universal primer sono stati selezionati da ITSs delle regioni fiancheggianti. Per proof-of-concept, abbiamo progettato gli iniettori specifici per Aeluropus littoralis e primer universale basato su specie di Poaceae, come specificato nel protocollo. Il 5.8S forward e reverse primer sono stati selezionati basato su un motivo conservato di 14 coppie di basi (bp) che mostra la somiglianza tra diversi ordini di alghe e funghi14, briofite e piante fiorite. Le caratteristiche degli iniettori progettati sono indicate nella tabella 2. L'universalità di SSU, 5.8S e LSU primer sono stati controllati da BLASTn e primer omologia risultati sono presentati in Figura 3 come un motivo logo. L'elenco delle specie incluse l'analisi di omologia, nonché i primers divergenti per ciascuna specie sono date tabella 1. Specificità di primer era check da Primer-BLAST. Per specie dove è disponibile la sequenza dell'intero genoma, è stata valutata la posizione cromosomica dei geni rDNA. Per esempio, in rDNA Oryza sativa e Arabidopsis thaliana, geni si trovano su due cromosomi diversi e in Zea mays su tre diversi cromosomi.
convalida di qPCR di primer a base di rDNA è stata eseguita con analisi della curva degli ampliconi ITS1 e ITS2-fianco utilizzando il DNA come un modello di fusione. Come presentato Figura 4 e Figura 5, specificità di primer è stata confermata sperimentalmente tramite l'osservazione di un singolo picco acuto senza formazione di primer-dimero in diverse specie di Poaceae compreso Triticum aestivum, Hordeum vulgare, Oryza sativae in dicotiledoni Medicago sativa, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia fabae Arabidopsis thaliana. L'ulteriore prova dei prodotti di amplificazione di separazione elettroforetica dimensioni ha mostrato una fascia unica. Come previsto, le bande derivano dai campioni di diverse specie variata in dimensione (Figura 6A e 6B). È interessante notare che, l'uso dei primer universale progettato specificamente per le tre specie di Poaceae non sono solo utili per altre specie di Poaceae, ma anche per altre specie di piante quali a. thaliana e un fungo endofito viz. Agaricomycetes indica.
La validità del primer specifici progettati (ITS1) è stata confermata anche da qPCR in a. littoralis utilizzando gDNA come modello. Un singolo picco senza formazione di primer-dimero è stato osservato. Sorprendentemente, il primer a. littoralis ITS1 (come il primer specifico) generato una singola banda forte non solo in a. littoralis, ma anche per tutte le altre specie testate ad eccezione di n. tabacum e Trifolium alexandrinum, che ha prodotto due bande (Figura 6). L'analisi di contaminazione gDNA è stata eseguita dai primer ITS o ITS-accompagnamento in tutti i campioni di RNA. Una rappresentazione schematica della piastra amplificazione nell'analisi della contaminazione gDNA e l'interpretazione dei risultati è presentato nella Figura 7.
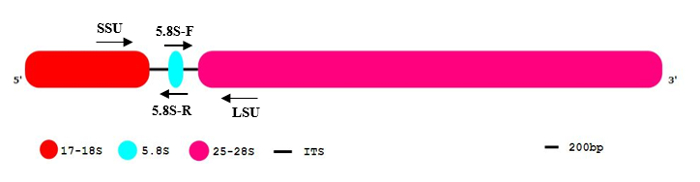
Figura 1: Il modello generale di organizzazione di sequenza eucariotiche rDNA.
Il segmento di rDNA eucariotiche contiene 17-18S (rosso), 5.8S (blu) e 25-28S rRNA (rosa). I distanziali interni trascritti (ITS) sono indicati come linee nere. 3 ´ 5´and indicare l'orientamento della molecola del DNA. Clicca qui per visualizzare una versione più grande di questa figura.
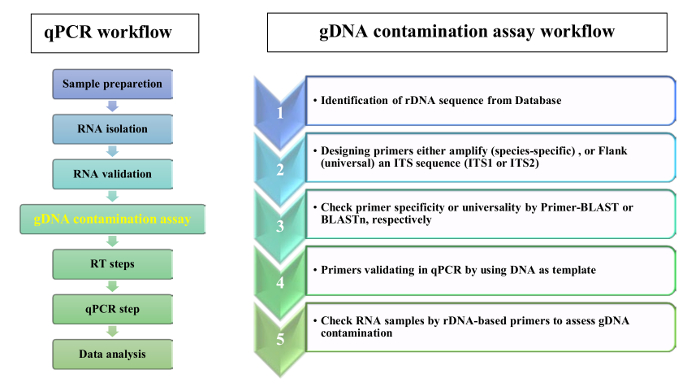
Figura 2: flusso di lavoro per un test di contaminazione RT-qPCR e gDNA. Clicca qui per visualizzare una versione più grande di questa figura.
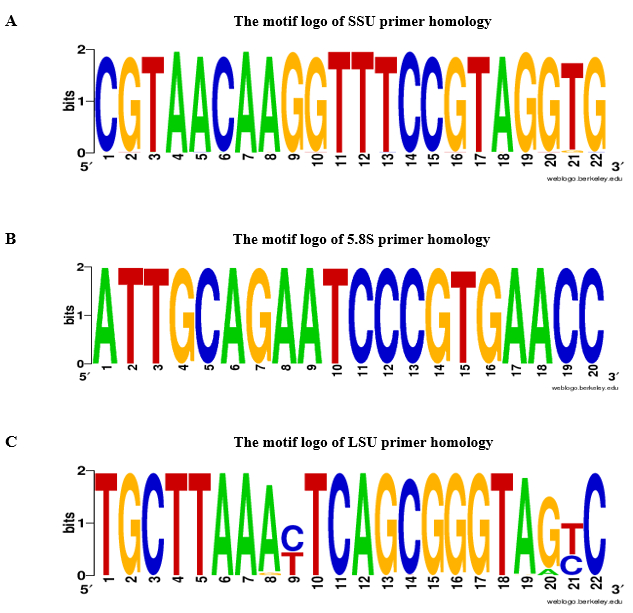
Figura 3: motivo logo di r. SSU, B. 5.8S e C. LSU primer omologia. Per SSU, 5.8S, e primer per LSU, il logo di motivo è stato costruito da BLASTn basata sui 2.000 record di pianta verde (numero taxid NCBI: 33090) con un cut-off e-valore ≤ 10-10. A-adenina, timina-T, G-guanina, citosina-C. Clicca qui per visualizzare una versione più grande di questa figura.
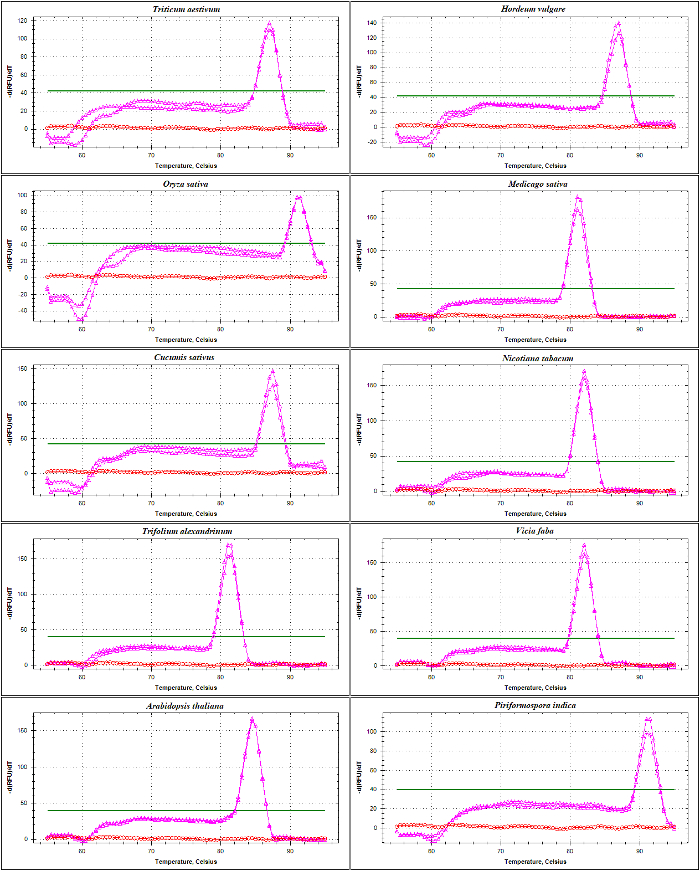
Figura 4: L'analisi della curva di fusione degli ampliconi ITS1-accompagnamento in specie diverse.
Questo amplicon, amplificato dalla SSU e 5.8S-primer R, contiene parte della sequenza della regione di codifica di 17-18 anni, l'intera sequenza di ITS1 e sequenza parziale di 5.8S. Vengono riportate le curve di fusione di ampliconi generati (rosa) e NTC (rosso) da Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, thaliana di Arabidopsis e Agaricomycetes indica. La linea piatta grassetto indica la soglia della linea di base. Clicca qui per visualizzare una versione più grande di questa figura.
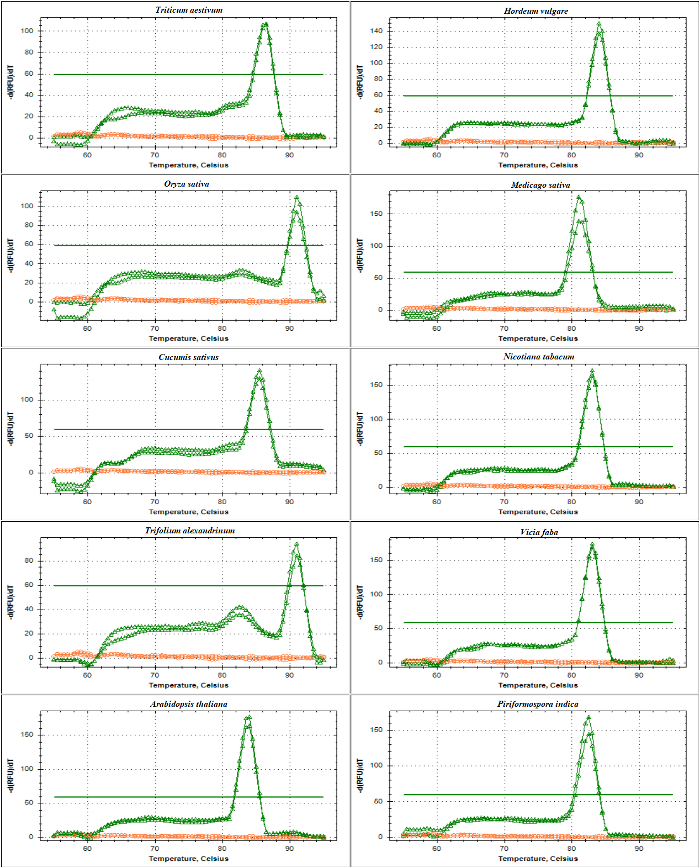
Figura 5: L'analisi della curva di fusione degli ampliconi ITS2-accompagnamento in specie diverse.
Questo amplicon viene generato mediante l'uso di 5.8S-primer F e LSU. L'amplicone descritto contiene sequenze di parte del 5.8 S, l'intera sequenza di ITS2 e una sequenza parziale di 25-28S. Vengono riportate le curve di fusione di ampliconi (verde) e NTC (rosso) generato da Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium alexandrinum, Vicia faba, Arabidopsis thaliana e Agaricomycetes indica. Clicca qui per visualizzare una versione più grande di questa figura.
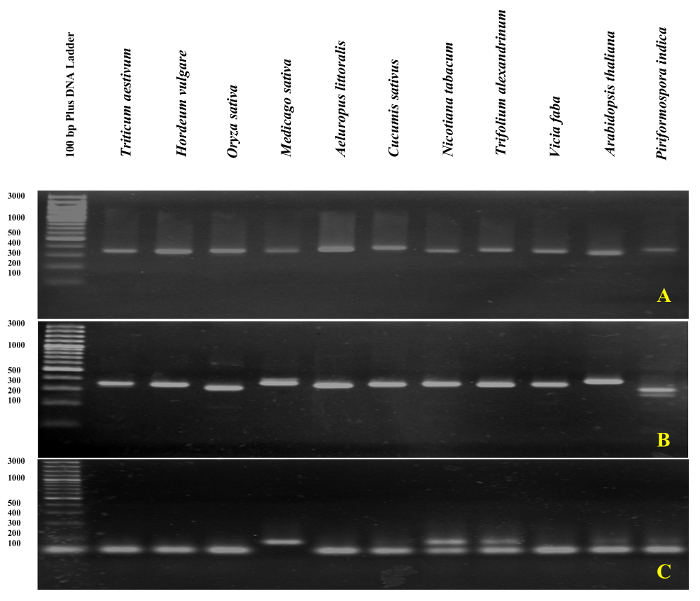
Figura 6: Analisi del gel dell'agarosi del prodotto PCR basati su rDNA.
L'amplicone di ITS1-fianchi (A), ITS2-fianchi (B) e ITS1 (C) sono stati analizzati su gel di agarosio al 3%. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7: Introne-come le caratteristiche di ITSs possono essere considerate per disegnare primers in grado di rilevare gDNA contaminazione.
Qualsiasi picco o la banda con le dimensioni previste nell'analisi qPCR indicare gDNA contaminazione del campione di RNA. UNK: campione sconosciuto, pos: controllo positivo, NTC: controllo non template. Clicca qui per visualizzare una versione più grande di questa figura.
| Primer | Genere | Taxid ID | Specie | Primer divergenti | ||
| SSU | Arabidopsis | 3701 | kamchatica, thaliana e lyrata | - | ||
| Vicia | 3904 | villosa, americana, unijuga, amoenane, amurensis, craccamal, pseudo-orobus, multicaulis, japonica, ramuliflora e faba | ||||
| Trifolium | 3898 | alexandrinum, montanum, resupinatum e repens | - | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, communis, nesophila, solanifolia, Tinospora, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, ondulata, obtusifolia, miersii, pauciflora, attenuare, acuminata, linearis, alata, sylvestris, rustica e suaveolens | - | |||
| Cucumis | 3655 | anguria, melo e sativus | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | Nessun primer trovato | |||
| Medicago | 3877 | Sativa, lupulina, pamphylica, lunata, rostrate, plicata e truncatula | - | |||
| Oryza | 4597 | Sativa, glumipatula, rufipogon barthii glaberrima punctata, longistaminata, meridionalis, nivara, meridionalis e longistaminata | - | |||
| Triticum | 4564 | aestivum, urartu e monococcum | - | |||
| Hordeum | 4512 | vulgare, bulbosum, marinum, brevisubulatum e bogdanii | - | |||
| LSU | Arabidopsis | 3701 | petraea, thaliana e lyrata | TGCTTAAACTCAGCGGGTAATC | ||
| Vicia | 3904 | sylvatica, tetrasperma, sativa, irsuto, sepium, parviflora, cracca, lathyroides, orobus, orobus, bithynica e faba | TGCTTAAATTCAGCGGGTAGCC | |||
| Trifolium | 3898 | finzioni, nigrescens, resupinatum, occidentale, subterraneum, strictum, ochroleucon, glomeratum, squamosum, ornithopodioides e repens | TGCTTAAATTCAGCGGGTAGCC | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, communis, nesophila, solanifolia, Tinospora, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, ondulata, obtusifolia, miersii, pauciflora, attenuare, acuminata, linearis, alata, sylvestris e suaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| Cucumis | 3655 | Melo, ritchiei e javanica | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | lagopoide, pungens e littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| Medicago | 3877 | Ruthenica, sativa, lupulina, arabica, polymorpha e minimi | TGCTTAAATTCAGCGGGTAGCC | |||
| pamphylica, lunata, rostrate e plicata | TGCTTAAACTCAGCGGGTAGTC | |||||
| Oryza | 4597 | Sativa, glumipatula, rufipogon, barthiial, glaberrima, australiensis, officinalis, australiensis, ridleyi, malampuzhaensis, alta, nivara, rufipogon, meridionalis e longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| Triticum | 4564 | aestivum, spelta, turgidum, dicoccoides, petropavlovskyi, urartu e monococcum | TGCTTAAACTCAGCGGGTAGTC | |||
| Hordeum | 4512 | vulgare, bulbosum, murinum, Secalinum, brevisubulatum e bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| Un primer degenerato è definito come sistema IUPAC per la nomenclatura del nucleotide | ||||||
Tabella 1: Elenco delle specie considerate per la raccolta di primer a base di rDNA.
Sito di legame di SSU rispetto al sito di legame di LSU ha mostrato maggiore omologia di sequenza sopra dato genere.
| Lunghezza di amplicon | Area di amplificazione | Sequenza | Nome di primer | Amplicon | |
| 332 - 405 bp | Sequenza parziale di SSU, tutta la sequenza di ITS1 e sequenza parziale di 5.8S | CGTAACAAGGTTTCCGTAGGTG | SSU | ITS1-fianchi | |
| GGTTCACGGGATTCTGCAAT | 5.8S-R | ||||
| 318 - 361 bp | Sequenza parziale di 5.8S, tutta la sequenza di ITS2 e sequenza parziale di LSU | ATTGCAGAATCCCGTGAACC | 5.8S-F | ITS2-fianchi | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100 - 200 bp | ITS1 | GGTATGGCGTCAAGGAACACT | ITS1-F | ITS1 | |
| ATAGCATCGCTGCAAGAGGT | ITS1-R | ||||
Tabella 2: Sequenze Primer.
Discussione
Analisi di espressione genica mediante PCR quantitativa sono stato ampiamente applicato negli ultimi anni. Il principale vantaggio di questo metodo rapido, conveniente ed automatizzato è il risultato di preciso e accurato. Tuttavia, guadagnando i benefici ottimali da questi vantaggi richiede una chiara comprensione del setup dei parametri utilizzati per l'esperimento di qPCR. Per ricevere un risultato affidabile in qPCR analisi dell'espressione genica, è necessario evitare l'amplificazione aspecifico che nasce dalla contaminazione primer-dimero o gDNA nel RNA campione3,15. Si prevede che i livelli di trascrizione di RNA saranno essere sopravvalutati sotto gDNA contaminazione8. Qui, le caratteristiche uniche di un gene di rDNA è stato considerato per un dosaggio di contaminazione gDNA nei campioni di RNA.
Proprietà di base della rDNA utilizzata nel presente protocollo: Geni ribosomali sono costituiti i due ITSs, vale a dire ITS1 e ITS2 e i tre geni codificanti di rRNA, 17-18 anni, 5.8S e 25-28S subunità12. Le due regioni ITS non sono parte della sequenza di codificazione delle subunità ribosomali. Che sono stati rimossi da almeno tre attività enzimatiche per elaborare il precursore maturare rRNA: un'attività endonucleasi, elicasi ed esonucleasi. Come il RNA ribosomiale (rRNA) è trascritto come una trascrizione di polycistronic, un prodotto primario contenente il ITSs è sicuramente presente. Il trattamento è molto veloce e si svolge nel nucleolo, e la quantità di molecole precursori rilevabile contenente l'ITS è inferiore al limite di rilevazione del metodo qPCR. Pertanto, quando ITS1 o ITS2 sono amplificati da ITS che fiancheggiano gli iniettori, nessuna amplificazione può essere rilevata in campioni di RNA se non gDNA contaminazione è presente. Il numero di rDNA geni nel genoma di organismi eucariotici è stato stimato per includere fino a un migliaio di copie, che è disposti in singole o in tandem matrici sui cromosomi11. In questo protocollo, vi proponiamo un modo alternativo, invece di NRT, per rilevare gDNA contaminazione, che è usato per ogni reazione/test.
Vantaggi e le limitazioni per quanto riguarda i metodi esistenti: NRT è in genere utilizzato per verificare se il campione di RNA preparato è pulita o contaminati da gDNA. Poiché gDNA contaminazione non è distribuita uniformemente tra i diversi campioni di RNA e la sensibilità di reazione ai gDNA è significativamente influenzata dai geni analizzati, NRT controlli sono necessari per ogni campione/dosaggio coppia7,15. Questo aggiungerà sostanzialmente costi e manodopera durante la manipolazione di molti campioni contemporaneamente3,9. Altri metodi alternativi documentati nella letteratura includono l'uso di introne primer specifici per il rilevamento di gDNA, o primer di progettazione che fiancheggiano un introne o estendersi su una giunzione esone-esone. Le limitazioni di questi metodi derivano dall'indisponibilità di informazioni di sequenza dell'introne, incompleta annotazione della struttura essone / dell'introne e l'assenza di introni nei geni o pseudogeni di interesse1,4,10 . A causa di evoluzione, geni rDNA esistano come famiglie geniche multigenica e altamente conservata. Sono molto abbondanti nel genoma e presenti su cromosomi diversi13. Rispetto ad altri geni di codificazione o nonconding, i geni rDNA Visualizza la misura migliore per la rilevazione della contaminazione gDNA. Nelle analisi trascrittomica comparativa, la normalizzazione dei dati di qPCR di calibratore rRNA non è raccomandata per alcuni problemi, ad esempio differenze nel cDNA preparazione (adescamento di polyA vs casuale hexamer priming), grandi differenze in abbondanza tra mRNA e rRNA , e biogenesi diversa che possono generare fuorvianti risultati10,16. Tuttavia, i problemi che abbiamo appena detto sono un vantaggio per il dosaggio di contaminazione gDNA. Ad esempio, per quanto riguarda la maggiore abbondanza di sito targeting nel genoma e localizzazione su cromosomi diversi, primer a base di rDNA migliorare significativamente la sensibilità di rilevamento di gDNA rispetto a metodi esistenti.
Versatilità di rDNA-based di altro organismo: geni rDNA sono una famiglia di ben studiati geni identificata nella maggior parte degli organismi. Il metodo di base di rDNA proposto rappresenta un sistema semplice, altamente sensibile ed economico per le analisi di contaminazione gDNA che può essere facilmente adattata ad altri organismi eucariotici e procariotici (protocollo n. 2 - 5). Come caso di studio, abbiamo dimostrato qui l'utilità di questo metodo in alcune specie di Poceae (Figura 4 e Figura 5). I primers utilizzati mostrano un alto tasso di trasferibilità ad altre specie di Poceae a causa della struttura altamente conservata di rDNA subunità tra specie. Questo problema diventa ancora più importante, quando non sono disponibile per il disegno dell'iniettore di informazioni di sequenza genomic sufficienti. Così, che fiancheggiano ITS primer disegnati per una specie può essere utilizzato in una specie affini. Inoltre, la 5.8S-primer F/R sono state raccolte basato su un motivo conservato che mostra l'alta somiglianza nella maggior parte delle piante da fiore14. Anche se tecniche di sequenziamento di throughput elevato permanentemente aumentano il numero di genomi noti, l'annotazione di esone-introne della maggior parte degli organismi non è stata completata, e così spesso non è possibile disegnare primers possono estendersi su un bordo di esone-esone. Il nostro metodo spiega come base di rDNA primer può essere applicato per il dosaggio di contaminazione gDNA nell'analisi qPCR di procarioti e negli eucarioti con l'obiettivo di eliminare costosi controlli NRT in ogni combinazione di dosaggio/primer.
Divulgazioni
Gli autori non hanno nessun concorrenti interessi finanziari.
Riconoscimenti
Questa ricerca è stata sostenuta dalla genetica e Agricultural Biotechnology Institute di Tabarestan (GAM), Sari Agricultural Sciences e Università di risorse naturali (SANRU). Il gruppo di ricerca junior Abiotic Stress genomica è stato finanziato da IZN (centro interdisciplinare per la ricerca delle piante delle colture, Halle (Saale), Germania. Ringraziamo Rhonda Meyer per la lettura critica del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
Riferimenti
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18(2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51(2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51(2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7(2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880(2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon