Method Article
Análisis Electrofisiológico de Cardiomiocitos Pluripotentes Humanos Pluripotentes (hPSC-CMs) Usando Matrices Multi-Electrodo (MEAs)
En este artículo
Resumen
La caracterización electrofisiológica de los cardiomiocitos derivados de las células madre pluripotentes humanas (hPSC-CMs) es crucial para el modelado de la enfermedad cardiaca y para la determinación de las respuestas de los fármacos. Este protocolo proporciona la información necesaria para disociar y planificar hPSC-CM en arrays de múltiples electrodos, medir su potencial de campo, y un método para analizar intervalos QT y RR.
Resumen
Los cardiomiocitos pueden derivarse ahora con alta eficiencia tanto de células madre pluripotentes humanas embrionarias como humanas (hPSC). Los cardiomiocitos derivados de hPSC (hPSC-CMs) son cada vez más reconocidos como de gran valor para el modelado de enfermedades cardiovasculares en seres humanos, especialmente síndromes de arritmia. También han demostrado su pertinencia como sistemas in vitro para predecir las respuestas a los fármacos, lo que los hace potencialmente útiles para la detección y el descubrimiento de fármacos, la farmacología de seguridad y tal vez, eventualmente, para la medicina personalizada. Esto se facilitaría derivando hPSC-CMs de pacientes o individuos susceptibles como hiPSCs. Para todas las aplicaciones, sin embargo, la medición precisa y el análisis de las propiedades eléctricas del hPSC-CM son esenciales para identificar los cambios debidos a las mutaciones del canal de iones cardíacos y / o fármacos que apuntan a los canales iónicos y pueden causar muerte súbita cardiaca. En comparación con la abrazadera de parche manual, los dispositivos con múltiples electrodos (MEA) ofrecen la ventaja dePermitiendo grabaciones de medio a alto rendimiento. Este protocolo describe cómo disociar cultivos de células 2D de hPSC-CM a pequeños agregados y células individuales y placas en MEAs para registrar su actividad eléctrica espontánea como potencial de campo. También se describen aquí métodos para analizar los datos registrados para extraer parámetros específicos, tales como los intervalos QT y RR. Se esperan cambios en estos parámetros en hPSC-CMs portadores de mutaciones responsables de arritmias cardíacas y después de la adición de fármacos específicos, permitiendo la detección de aquellos que portan un riesgo cardiotóxico.
Introducción
Las células madre pluripotentes humanas (hPSCs) tienen la capacidad de auto-renovarse y generar prácticamente cualquier tipo de célula del cuerpo humano a través de la diferenciación 1 , 2 . Se han descrito protocolos detallados sobre cómo diferenciar directamente los hPSCs en varios linajes cardíacos (ventricular, auricular, cardiomiocitos tipo marcapasos) 3 , 4 , 5 , 6 , 7 . Los cardiomiocitos son células eléctricamente activas y un conocimiento detallado de su actividad electrofisiológica puede ser extremadamente informativo para comprender el desarrollo del corazón y la enfermedad 8 . Los cardiomiocitos derivados del hiPSC específicos para pacientes (hiPSC-CMs) se han utilizado con éxito para modelar y estudiar las características celulares, moleculares y eléctricas de varias arritmias cardíacas, incluyendo el síndrome de QT largo(LQTS) 9 , 10 , 11 , 12 , 13 , síndrome de Brugada 14 , y taquicardia ventricular polimórfica catecolaminérgica 15 , 16 . Además, se han añadido múltiples fármacos a los hiPSC-CM enfermos para recapitular la intervención terapéutica y rescatar los fenotipos patológicos celulares 10 , 15 , 20 , 21 , 22 . Más recientemente, se han desarrollado plataformas de cribado basadas en WPSH-CMs, en respuesta a la necesidad de sistemas humanos para las primeras fases del descubrimiento de fármacos 23 , 24 , 25 , ya que los cardiomiocitos de roedores difieren profundamente de huEn la expresión del canal iónico y la biofísica 26 .
Para ello, se están desarrollando e implementando tecnologías adecuadas para aplicaciones de medio a alto rendimiento. Estos incluyen grabaciones ópticas de potencial de membrana, transitorios de Ca2 + y medidas de deformación, impedancia (como medida indirecta de la contractilidad celular) y mediciones de potencial de campo extracelular (FP) (para revisión véase la referencia 24 ). Los dispositivos de multielectrodos (MEA) permiten registrar las señales de forma de onda eléctrica (o FPs) generadas y formadas por monocapas o pequeños grupos de cardiomiocitos. El contorno FP se correlaciona con el potencial de acción cardiaco y, hasta cierto punto, con las grabaciones electrocardiográficas (ECG) 27 ; Típicamente muestran una subida rápida inicial correspondiente a la afluencia de Na + y la despolarización de la membrana (pico R / Q), una fase de onda lenta / meseta probablemente correspondiente a la Ca2+, y una fase de repolarización correspondiente a un flujo de K + predominante (pico T). Perturbación de la forma de onda FP se puede correlacionar con los cambios en las fases de potencial de acción específicos [ 28] .
Aunque las grabaciones de pinzas de los potenciales de acción podrían ser más informativas, especialmente para parámetros como la velocidad ascendente y el potencial de membrana en reposo, las mediciones manuales no son factibles para experimentos a escala de medio y alto rendimiento, mientras que la abrazadera automática ha sido recientemente aplicada a hPSC -CMs 29 . Sin embargo, dado que las grabaciones prolongadas sobre los AMA permiten la exposición tanto aguda como crónica a los compuestos, ahora es posible utilizar las plataformas hPSC-CM para el cribado de fármacos, el descubrimiento 24 , 30 y para la farmacología de seguridad 31 , 32 . Esto tiene la promesa de precisión futura o persoNalizada 33 .
El propósito de este protocolo es proporcionar la información necesaria para disociar y depositar hPSC-CM en chips MEA y medir su FP. En este procedimiento, se ha optimizado cada paso, asegurando una supervivencia y recuperación óptimas de las células después de la disociación, uniones óptimas a la placa MEA y un análisis estandarizado y cuantificación de los parámetros. En particular, se explican y ejemplifican el procedimiento para el registro extracelular de FP, el análisis de los intervalos QT y RR y la evaluación de los efectos de los fármacos.
Protocolo
1. Preparación de soluciones y reactivos
- Preparar el medio de cultivo hPSC-CM usando medio de insulina baja, albúmina de suero bovino, poli (alcohol vinílico), lípidos esenciales (LI-BPEL) 34 , 35 , 36 combinando los reactivos descritos en la Tabla 1 . Filtrar el medio a través de un filtro de poro de 0,22 μm y almacenar a 4 ° C durante un máximo de 2 semanas.
- Preparar la solución madre de fibronectina recombinante humana reconstituyendo 1 mg de fibronectina recombinante humana en 5 ml de agua destilada estéril para alcanzar una concentración de 200 μg / ml, alícuota y almacenar a -80 ° C.
- Preparar solución de detergente enzimático al 1% (p / v), para ayudar a eliminar las células residuales del chip de microarrays, disolviendo 1 g de detergente enzimático en polvo en 100 ml de solución caliente (~ 40-45 ° C) agua desionizada. Dejar que la solución se enfríe y almacenar a 4 ° C fO hasta un año.
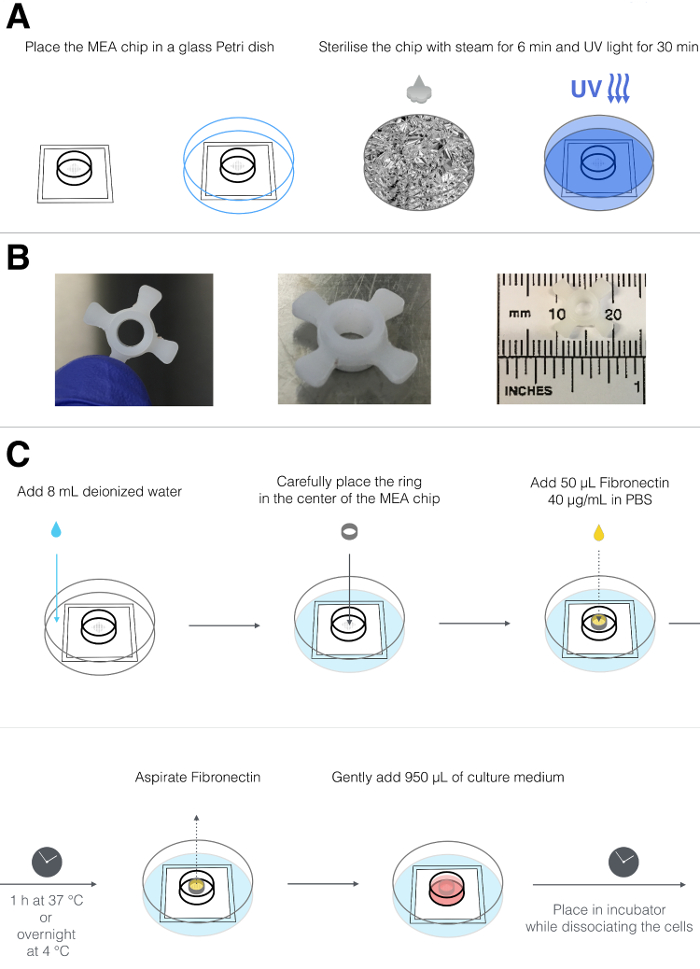
2. Esterilización de chips MEA (Figura 1A)
NOTA: Hay varias configuraciones diferentes de los MEA disponibles, con formatos de uno o varios pozos. El protocolo descrito aquí utiliza el MEA de cámara única que contiene 60 electrodos de registro en una disposición de rejilla de 8 x 8 (véase la Tabla de Materiales ). El diámetro del electrodo es de 30 μm y la distancia entre electrodos es de 200 μm. También está presente un electrodo de referencia.
- Enjuague bien el chip con agua desionizada.
- Poner los chips MEA dentro de una placa de Petri de vidrio que se puede autoclavar. Envuelva el plato en papel de aluminio.
- Esterilizar los chips en una olla a presión de laboratorio durante 6 min. Deje que los platos se enfríen antes de abrirlos.
NOTA: Alternativamente, los chips pueden esterilizarse sumergiéndolos en 1 mL de etanol al 80% (v / v) a RT durante 15-30 min. - Coloque las virutas en una campana de cultivo celular ySuperficie a luz UV durante aproximadamente 30 min.
3. Recubrimiento de chips MEA (Figura 1B y 1C)
- Coloque las virutas limpias dentro de una placa de Petri estéril estándar de 10 cm de diámetro.
- Añadir 8 ml de agua destilada estéril a la placa de Petri para formar una cámara humidificada, lo que evitará que el pequeño volumen de medio de cultivo en el chip se seque cuando se coloca en la incubadora.
- Utilice anillos de politetrafluoroetileno (PTFE) hechos a medida ( Figura 1B ) para asegurar el chapado de los cardiomiocitos en el centro del chip, donde se encuentra la matriz de electrodos. (Los anillos de PTFE se almacenan previamente en etanol al 80%). En la campana de cultivo, retire los anillos del etanol, colóquelos en una placa de Petri estéril sin tapa y deje que los anillos se sequen en la campana.
- Coloque un anillo seco dentro de un chip MEA usando pinzas esterilizadas con llama ( Figura 1C ).
NOTA: Alternativamente utilice etanol al 80% para lavar las pinzas y dejarN la campana de cultivo de tejidos antes de usarlos. - Descongelar una alícuota de stock de fibronectina recombinante humana (200 μg / mL en agua destilada) y diluir en PBS con Ca2 + y Mg2 + para obtener una solución de trabajo de 40 μg / mL. Cubra los electrodos añadiendo 50 μl de fibronectina 40 μg / mL dentro del anillo.
NOTA: El recubrimiento con fibronectina recombinante humana asegura la unión óptima de hPSC-CM. Sin embargo, pueden usarse otros tipos de proteínas de recubrimiento tales como fibronectina bovina o mezclas de proteínas de matriz extracelular. - Cierre la tapa de la placa de Petri de plástico de 10 cm de diámetro y transfiera cuidadosamente el plato que contiene el chip MEA en la incubadora. Incubar a 37 ° C durante al menos 1 h, o a 4 ° CO / N.
- Transferir el plato que contiene el chip MEA a la campana de cultivo celular. Antes de utilizar el chip MEA, aspirar los 50 μl de fibronectina con una pipeta P200 o un sistema de vacío en la campana, sin desplazar el anillo de PTFE. Use plaPara este paso. Asegúrese de que ningún objeto sólido ( por ejemplo, puntas de pipeta) toque el interior del plato ya que esto puede dañar los electrodos.
NOTA: Esto prolongará la vida de los chips MEA. - Agregue suavemente 950 μl de medio LI-BPEL (ver Tabla 1 y Tabla de Materiales ), asegurándose de que está uniformemente distribuido y que el anillo no flote. Devuelva el plato a la incubadora.
4. Disociación y placas de hPSC-CM (Figura 2)
NOTA: El protocolo aquí descrito hace uso de hPSC-CMs que se diferenciaron en un cultivo monocapa utilizando citoquinas 34 a ~ 18 días después de iniciar la diferenciación. Sin embargo, se ha demostrado que es adecuado para cualquier cultivo 2D y 3D hPSC-CM. Cuando se usan cultivos diferenciados en puntos de tiempo anteriores o posteriores, el ajuste del tiempo de incubación de la enzima de disociación (véase Tabla de Materiales ) puede ser neNecesario Los siguientes volúmenes están destinados a un solo pocillo de un formato de placa de 12 pocillos (3,8 cm $ ² $ ).
- Aspirar el medio del cultivo de hPSC-CMs.
- Mientras se trabaja bajo una campana de cultivo de tejidos, se añade 1-2 mL / pocillo de PBS sin Ca2 + / Mg2 + para lavar el cultivo. Aspirar el PBS.
- Añadir 500 μl / pocillo de la enzima de disociación. Incubar durante 5 min a 37 ° C.
- Añadir 1 ml de LI-BPEL / pocillo para diluir la enzima. Suavemente separe la monocapa de hPSC-CM rascándola suavemente usando una pipeta P1000. Recoger la suspensión celular en un tubo de 15 ml.
- Enjuague el pocillo con 1 mL de LI-BPEL para recoger todas las células y células restantes.
- Añadir otros 2-3 ml de LI-BPEL para alcanzar un volumen final de 5-6 ml y suavemente pipetear hacia arriba y hacia abajo de 3-5x con una pipeta de 5 ml para disociar grupos de células.
NOTA: La disociación en células individuales en este punto no es necesaria, ya que afectará a la supervivencia celular. La presencia de pequeños gruposGarantizar una mayor viabilidad celular. - Centrifugar las células a RT durante 3 min a 300 x g.
- Retire el sobrenadante, tratando de eliminar la mayor parte del exceso de líquido, pero sin desalojar el sedimento celular.
- Resuspender el sedimento de células en 250 μL de LI-BPEL (utilizando un P1000 y pipetear extremadamente suavemente).
- Distribuir ~ 50 μL de suspensión celular por MEA (hasta 5 MEAs pueden prepararse a partir de un pocillo de una placa de 12 pocillos) pipeteando la suspensión de células directamente en el centro del anillo de PTFE, encima de la matriz de electrodos.
NOTA: En esta fase las células son difíciles de contar debido a la presencia de grupos de células y el número total de células por MEA puede variar sustancialmente, dependiendo de la fuente de hPSC-CM utilizada. Durante el chapado, asegúrese de que la nube de células disociadas cubre el área de los electrodos. - Transfiera cuidadosamente los MEAs a la incubadora a 37 ° C y permita que las células conecten O / N
5. Eliminación del anillo y re medianoFresado (Figura 3)
- Al 1 día después del chapado, retire cuidadosamente el anillo en un ambiente estéril usando pinzas estériles ( Figura 3A-3B ).
- Enjuague el anillo en etanol al 80% (v / v) y guárdelo en un tubo de 50 ml que contiene 80% (v / v) de etanol fresco.
- Retire suavemente 500 μl de medio del chip MEA y añada 500 μl de LI-BPEL fresco.
- Transferir los MEAs a la incubadora a 37 ° C.
NOTA: Las células deben empezar a batir 1-7 días después de la eliminación del anillo y el cambio del medio ( Figura 3C ). - Medir la actividad eléctrica de los hPSC-CM en los MEA 1-7 días después de la eliminación del anillo.
6. Compruebe la calidad de la señal (Figura 4)
- Encienda el ordenador e inicie la suite de software vinculada a la configuración MEA: TCX-Control, MC_MEA Select y MC_Rack. Ajuste la temperatura a 37 ° C en TCX-Control para registrar mediciones a temperatura fisiológica.
- Retire el plato que contiene el chip MEA fDe la incubadora. Abra la tapa, saque el chip MEA y colóquelo en un pañuelo de papel para absorber el agua residual.
- Limpie con cuidado los contactos externos de la placa con un pañuelo de papel y límpielos con un hisopo de algodón humedecido con etanol al 100% (v / v) para eliminar cualquier resto de agua o desechos, lo que podría causar ruido de la señal.
- Transfiera la placa MEA a la etapa de la cabeza de grabación calentada (37 ° C) para detectar la actividad espontánea ( por ejemplo, hardware: consulte la Tabla de materiales , software: MC_Rack).
- Abrir MC_Rack: Haga clic en 'Editar' → 'Añadir MC_Card' para crear un nuevo protocolo. Utilice el menú desplegable 'Editar' para agregar diferentes ventanas de Grabador y Pantalla al protocolo.
NOTA: Se recomienda una frecuencia de muestreo de al menos 10 kHz. El protocolo que utilizamos contiene una herramienta de visualización de largo plazo, con un diseño completo de la totalidad del chip MEA, y un clasificador de Spike, esencial para capturar el efecto del fármaco iN tiempo real. El Spike Cutout se sintoniza con un 'Pre Trigger' de 20 ms, un 'Post Trigger' de 800 ms y un 'Dead Time' de 2 ms. El protocolo se puede guardar como archivo '.rck' y volver a cargarse antes de iniciar los experimentos.
- Abrir MC_Rack: Haga clic en 'Editar' → 'Añadir MC_Card' para crear un nuevo protocolo. Utilice el menú desplegable 'Editar' para agregar diferentes ventanas de Grabador y Pantalla al protocolo.
- Inicie el protocolo en modo de reproducción haciendo clic en el botón 'reproducir'.
NOTA: En este punto es posible volver a cargar el protocolo guardado en el paso 6.5. En MC_Rack, cargue el protocolo (extensión .rck) haciendo clic en 'Archivo' → 'Abrir'. - Si las señales muestran picos R claramente visibles y picos T, espere 10-15 minutos para concluir la fase de adaptación. En la Figura 4 se muestran ejemplos de trazas de calidad buena y mala.
NOTA: Si no se puede detectar ningún pico T en ninguno de los electrodos, no proceda con el experimento. Normalmente esto es el resultado de una actividad eléctrica deficiente de los hPSC-CM o de una mala fijación de las células a los electrodos.
7. Iniciar ExY grabación
- Haga clic en "registro" y luego "reproducir", y adquirir los datos durante 10 min en condiciones de línea de base para determinar el estado estacionario. Anote los electrodos que tienen la mejor señal para que puedan ser fácilmente identificados y exportados posteriormente para el análisis.
- Para la evaluación de la respuesta a fármacos, añada concentraciones crecientes de fármaco cada 10 min. Como ejemplo, agregue el bloqueador hERG E4031 a una concentración final de 1 μM. Para ello, eliminar 100 μL de medio y añadir el mismo volumen de 10 μ M E4031 disuelto en el medio.
NOTA: Como demostrado previamente por Cavero y colaboradores [ 31] , es importante una sabia elección del volumen en el que se disuelven los fármacos, ya que puede alterar profundamente la curva de respuesta al fármaco. - Repita el paso 7.2 para todas las otras concentraciones de droga de interés.
- Haga clic en 'detener' para concluir las grabaciones al final del protocolo.
8.MEA Limpieza para Reutilización
- Una vez finalizada la grabación del experimento, retire suavemente el medio con una pipeta P1000. No toque el interior del plato ya que esto puede dañar los electrodos. Deseche de acuerdo con las normas locales de seguridad.
- Enjuague los chips de MEA con agua desionizada usando una botella de lavado, y repita el paso de lavado 3-4x.
NOTA: En este punto no es necesario que las células estén completamente separadas del chip. - Añadir 1 mL de solución de detergente enzimático al 1% (v / v) en cada pocillo e incubar O / N a 4 ° C para permitir el desprendimiento celular y la eliminación celular.
- Un día más tarde, enjuague los chips MEA a fondo con agua desionizada para eliminar la solución de detergente enzimático y las células residuales y agregue 1 mL de agua desionizada. Los chips MEA limpios se pueden almacenar sumergidos en agua desionizada a 4 ° C.
9. Exportación de datos
- Abra el software MC_Data Tool vinculado a la configuración MEA.
- Haga clic en 'Archivo' → 'Abrir MCRE'.
- Haga clic en 'Herramientas' → 'Convertir MCD a ABF' .
- Seleccione los electrodos con las mejores señales de grabación que se van a exportar para el análisis. Seleccione el directorio en el que desea guardar los archivos exportados y haga clic en "Guardar". El procedimiento de exportación puede tardar varios minutos dependiendo del número de electrodos exportados y del tamaño total de los archivos de grabación.
10. Análisis de datos
- Descargue e instale un programa de adquisición y análisis de datos de electrofisiología ( por ejemplo, pClamp). Una vez completado, inicie el software de análisis ( por ejemplo, Clampfit).
- RR Intervalo de cálculo (Figura 5).
- Coloque dos cursores verticales para definir la región de interés en la traza. Seleccione 'Detección de eventos' → 'Búsqueda de umbral'. El cursor horizontal debe cruzar todos los eventos que deben ser cuantificados, como se muestra en la figura 5A .
- Haga clic en &# 39; Aceptar 'y luego' Aceptar toda la categoría '. A continuación, el software buscará en el rastreo todos los eventos adecuados. Las marcas azules se colocarán por encima de los eventos.
- Ajuste la sensibilidad de la selección automática ajustando los parámetros en la ventana de detección de eventos principales.
- Una vez que todos los eventos han sido identificados automáticamente, vaya a la ventana de resultados (Ventana → Resultados) y copie la columna titulada "Intervalo de Intervención", que contiene los datos de frecuencia ( Figura 5B ).
- Cálculo del intervalo QT (Figuras 6-9):
- Coloque un cursor justo antes y uno después de un único FP en la condición de estado estacionario. Seleccione 'Detección de eventos' → 'Crear plantilla' ( Figura 6 ) .
- Compruebe que el FP está correctamente identificado y haga clic en 'Añadir'. La plantilla se moverá al panel inferior.
- Guarde la plantilla como un ".atf"archivo. De esta forma, se ha creado un rastreo de plantilla que será buscado por el software a lo largo de toda la grabación ( Figura 7 ). Crear una plantilla para cada condición, ya que un efecto de drogas podría alterar la forma de la FP. Si es necesario, filtre ligeramente la traza para incluir un máximo de 10.000 puntos dentro de los dos cursores.
- Una vez que la plantilla se ha guardado con la extensión '.atf', seleccione 'Detección de eventos' → 'Búsqueda de plantillas' y cargue la plantilla.
- Ajuste el "Umbral de coincidencia de plantilla" para identificar correctamente todos los FP en el intervalo seleccionado.
- Una vez que todos los eventos se han identificado correctamente, guárdelos en un nuevo archivo .abf.
- Abra el archivo con el software de análisis y calcule automáticamente el 'Time of Peak' para los picos Q / R y T ( Figuras 8, 9 ). Si las trazas son muy ruidosas, aplique un filtro. Calcular el intervalo QT restando elValor Q / R del valor T en un editor de hojas de cálculo de elección.
Resultados
Un dıa después de la disociación y plaqueado, la capa de hPSC-CMs será visible como una pelıcula densa y blanca que cubre el centro de la cámara MEA ( Figura 3A ). Después de retirar el anillo ( Figura 3B ), La capa debe permanecer en su lugar y la inspección en un microscopio óptico mostrará los electrodos MEA cubiertos por la capa hPSC-CM (contracción) ( Figura 3C ). Debido al acoplamiento físico y eléctrico de las células, sólo se utilizará un electrodo (electrodo dorado) para el análisis.
Alternativamente, cuando se trabaja con estructuras tridimensionales, tales como cuerpos embrioides o microtiches, éstos pueden ser revestidos de manera que estén desacoplados físicamente y eléctricamente. La inspección visual en el microscopio podría confirmar que ninguna conexión física entre los grupos y las ondas R no sincronizadas en los MEAs no confirman ningún acoplamiento eléctrico. En este sentidoSe pueden analizar múltiples electrodos independientes.
Las grabaciones típicas de trazas FP se muestran en la Figura 4 . En particular, un trazo de buena calidad puede definirse por la presencia de un pico claro correspondiente a la afluencia de Na + y la despolarización de la membrana (pico R / Q), una fase clara de repolarización correspondiente al flujo de K + (pico T) Señal a ruido ( Figura 4A , a la izquierda: nota escala del eje y y Figura 4B ). Las trazas de mala calidad ( Figura 4A , centro) pueden ser el resultado del fallo de hPSC-CMs para unirse a la placa MEA o de actividad eléctrica débil del hPSC-CM. Esperar 1-3 días para un mejor acoplamiento puede mejorar la señal; Sin embargo, si no se observa mejoría de la señal, se recomienda excluir este MEA de los experimentos. Las trazas ruidosas ( Figura 4A , derecha) pueden analizarse después del filtrado.
El análisis exitoso del intervalo RR puede identificarse mediante inspección visual de la pantalla que muestra la detección de picos ( Figura 5A, 5B ). Dentro del intervalo de tiempo definido por los cursores verticales, deben estar presentes marcas azules correspondientes a cada pico. En caso de que el programa no identifique uno o más picos, intente mover el cursor horizontal y ejecutar el análisis de nuevo o ajustar los ajustes de detección. Análogamente, el análisis exitoso del intervalo QT puede ser identificado mediante inspección visual de la pantalla que muestra la detección de FP ( Figura 7 ). Dentro del intervalo de tiempo definido por los cursores verticales, deben estar presentes marcas azules correspondientes a cada detección de FP. En caso de que el programa no identifique uno o más FPs, intente redefinir la plantilla FP ( Figura 6 ) o ajuste la configuración de detección y vuelva a ejecutar el análisis.
Trazas de PF individuales extraídas o su promedio ( FigurE 8) se puede utilizar para obtener valores de intervalos QT con ajustes específicos como en la Figura 9 . El análisis de la relación QT-RR es aconsejable y es significativo en las líneas de hPSC enfermas y WT ( Figura 10A ) y para evaluar la necesidad y / o el efecto de las correcciones del intervalo QT ( Figura 10B-10D ). HPSC-CMs que llevan LQTS que causan mutaciones tienen intervalos QT prolongados en comparación con los controles de WT ( Figura 11A ]. El tratamiento de hPSC-CMs con un bloqueador de hERG resulta en la prolongación del intervalo QT ( Figura 11B ); Por el contrario, el tratamiento con un activador hERG da como resultado un acortamiento del intervalo QT ( Figura 11C ). Por último, el tratamiento con fármacos que afectan a la frecuencia de picado de hPSC-CM debe ser visible como un cambio en el intervalo RR ( Figura 11D , acortamiento de intervalo RR).
 Unesdoc.unesco.org unesdoc.unesco.org
Unesdoc.unesco.org unesdoc.unesco.org
Figura 1: Esterilización y recubrimiento de chips MEA. ( A ) Esquema que representa el proceso de esterilización incluyendo la colocación del chip MEA en una placa de Petri de vidrio autoclavable, envoltura en papel de aluminio, vaporización durante 6 min y exposición a UV durante 30 min. ( B ) Vista superior (panel izquierdo) y lateral (panel central) del anillo de PTFE personalizado; El anillo tiene un diámetro exterior de 1,2 cm, incluyendo 4 solapas que permiten su colocación en el centro del chip MEA y el diámetro interior es de 0,4 cm (panel derecho). ( C ) Esquema representativo de la preparación del chip MEA que incluye colocar el chip en una placa de Petri plástica estándar, añadir 8 ml de agua desionizada fuera de la cámara MEA, colocar el anillo de PTFE en el centro de la cámara y revestir el conjunto de electrodos con fibronectina . Después del tiempo de incubación, la fibronectina se elimina y se reemplaza con medio de cultivo.T = "_ blank"> Haga clic aquí para ver una versión más grande de esta figura.
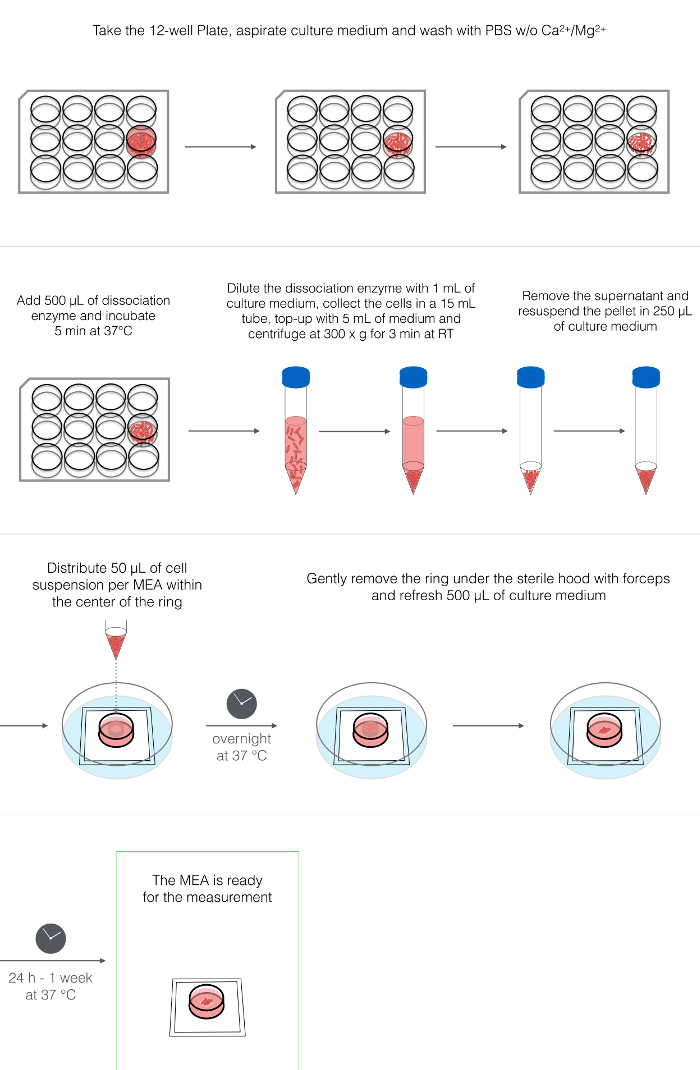
Figura 2: Disociación y Enchapado de hPSC-CMs. Esquema representativo de los procesos de disociación enzimática de hPSC-CM, centrifugación, resuspensión y plaqueado en el centro de la cámara MEA. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: MEA Chips con hPSC-CM Capa. ( A ) Vistas desde arriba del chip MEA que contiene el anillo y la capa de hPSC-CM. ( B ) vista lateral del chip MEA después de la eliminación del anillo. ( C ) Imagen de campo brillante de hPSC-CMs capa chapada en el micro-Matriz de electrodos; Aumento de 4X. Haga clic aquí para ver una versión más grande de esta figura.
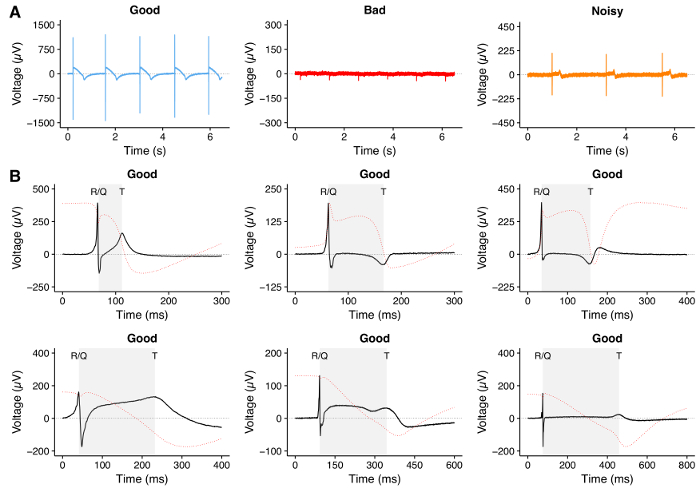
Figura 4: Registro MEA de hPSC-CM. ( A ) Trazas representativas registradas con el MEA que muestran una traza de buena calidad con picos R / Q y T claramente visibles con alta relación señal / ruido (izquierda), un trazo de mala calidad sin picos claramente visibles R / Q y T (medio) Y un rastro ruidoso con puntos R / Q y T claramente visibles, pero con baja relación señal / ruido (derecha). ( B ) Ejemplos representativos de trazas de FP de buena calidad con diferentes morfologías que pueden ser registradas durante experimentos MEA utilizando hPSC-CM. El área sombreada representa el intervalo QT medido durante el análisis. Dado que el FP en MEA se parece al primer derivativoE del potencial de acción 28 , hemos calculado la integral de la traza FP, mostrada como línea roja punteada, como demostración teórica de una elección de onda T próxima a una repolarización de potencial de acción completa. Haga clic aquí para ver una versión más grande de esta figura.
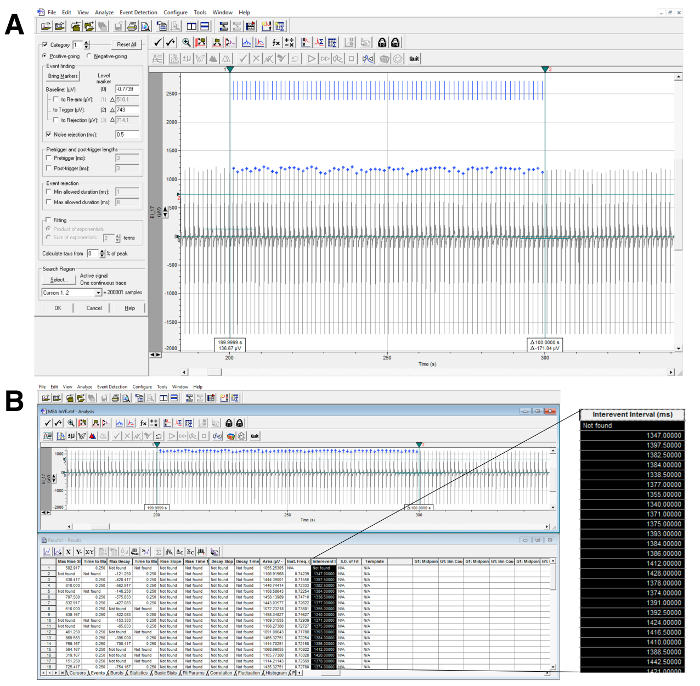
Figura 5: Cálculo del intervalo RR. ( A ) Ejemplo de análisis de intervalo RR con detección automática de picos (arriba) y extracción de datos (parte inferior) usando el software de análisis (ver Tabla de Materiales). Los cursores verticales identifican el intervalo de tiempo de interés y el cursor horizontal está cruzando todos los eventos que son detectados e identificados por marcas azules. ( B ) La columna ampliada muestra los datos extraídos utilizados para calcular el intervalo RR. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Creación de la plantilla FP. Ejemplo de selección de plantillas utilizando cursores verticales colocados antes y después de un único FP. Esta plantilla se utiliza para la identificación automática FP. Haga clic aquí para ver una versión más grande de esta figura.
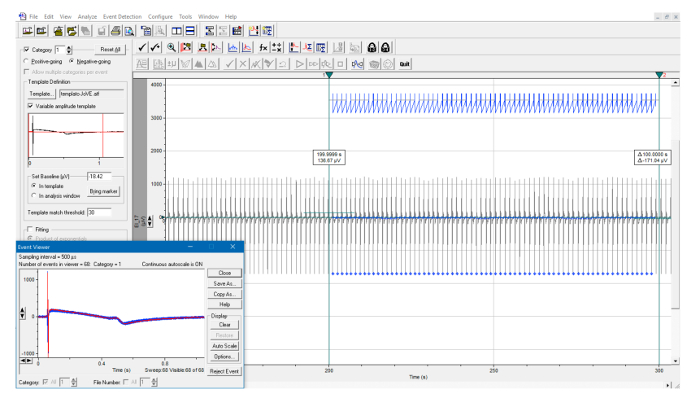
Figura 7: Identificación automática de la plantilla FP. Ejemplo de búsqueda de plantillas a lo largo de un intervalo definido por dos cursores verticales. Todos los eventos detectados se identifican con marcas azules y son automáticosY superpuesta en la inserción. Haga clic aquí para ver una versión más grande de esta figura.
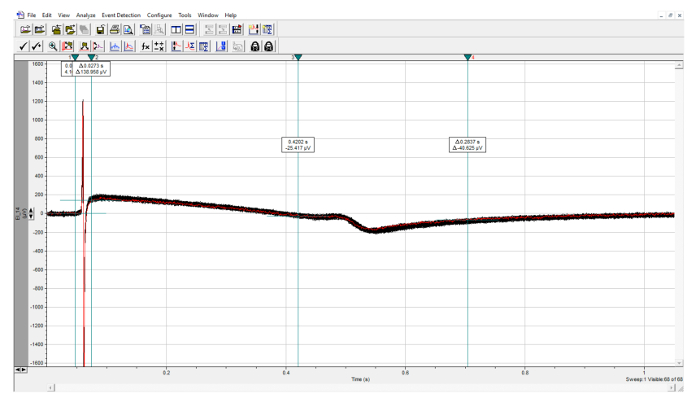
Figura 8: Cuantificación de los parámetros FP. Análisis de los eventos guardados, con el pico R identificado manualmente dentro de los dos primeros cursores y el pico T identificado manualmente dentro de los dos últimos cursores. Haga clic aquí para ver una versión más grande de esta figura.
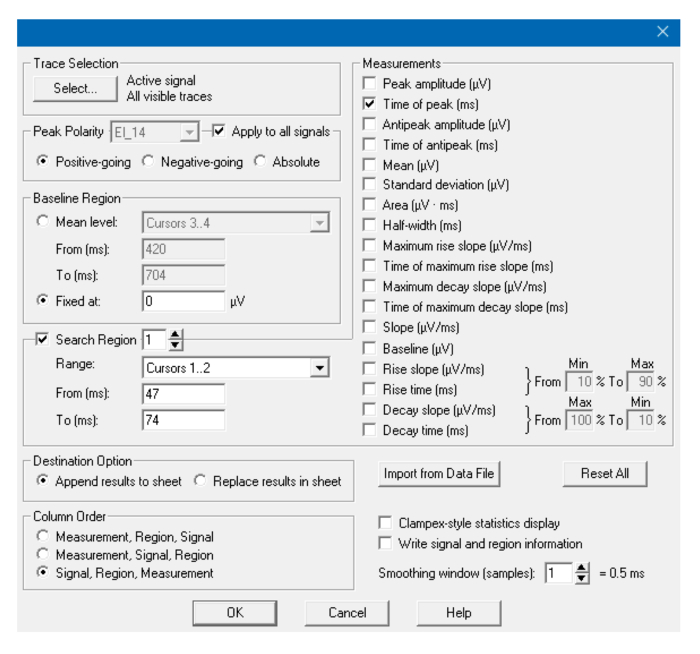
Figura 9: Ventana de análisis. Parámetros utilizados en la ventana Estadísticas para detectar el pico R. Para la detección del pico T, cambie los cursores y (si es necesario) poLaridad. Haga clic aquí para ver una versión más grande de esta figura.
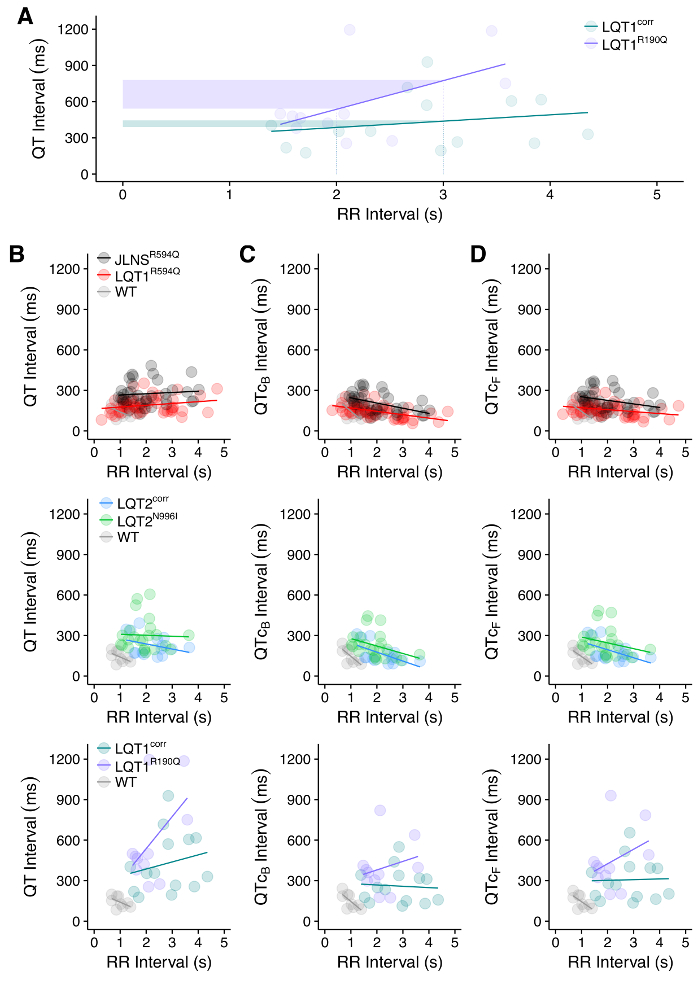
Figura 10: Relación QT-RR. ( A ) Ejemplo de relación diferente entre los intervalos QT y RR entre WT (LQT1 corr ) y QT de tipo QT 1 (LQT1 R190Q ) hPSC-CM. Las áreas sombreadas muestran que el mismo cambio en el intervalo RR genera un mayor cambio en el intervalo QT de la LQT1 línea enferma, lo que probablemente aumenta la susceptibilidad a la arritmia. ( B ) Relación entre los intervalos QT y RR no corregidos medidos en MEA en CMs de 7 hPSC líneas diferentes. El efecto de la corrección de QT para las fórmulas de Bazett ( C ) o de Fridericia ( D ) se muestra y es visible como cambio en la pendiente del QT-RR en Relación terval. Figuras adaptadas de la referencia 30 . Haga clic aquí para ver una versión más grande de esta figura.
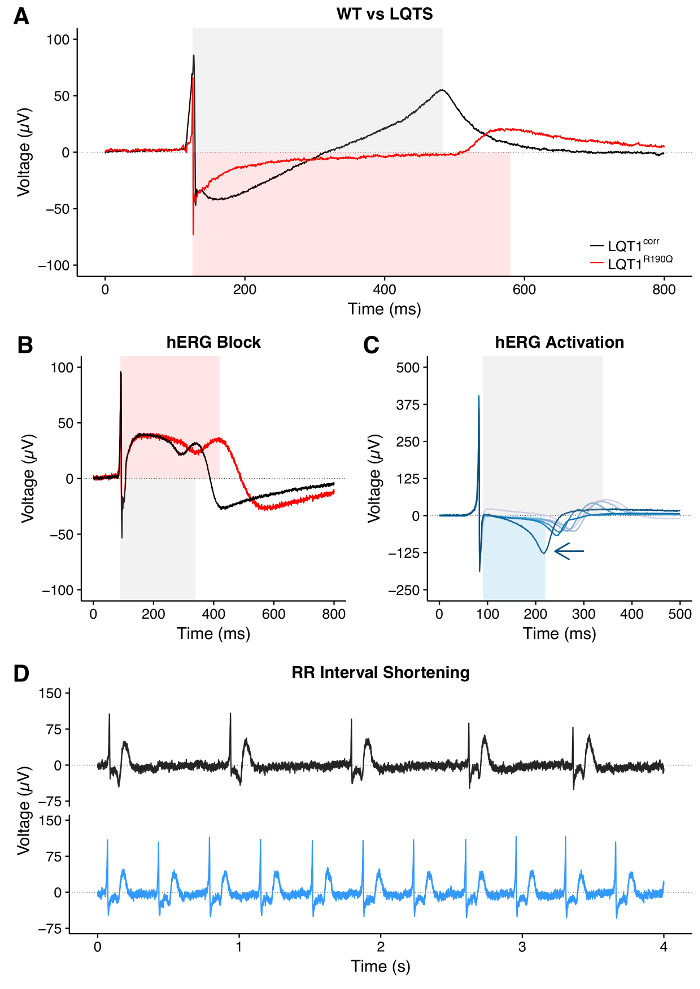
Figura 11: Variaciones del intervalo QT y RR de la enfermedad o la droga. ( A ) Ejemplo de prolongación del intervalo QT en hPSC-CM derivado de un paciente portador de una mutación del Síndrome de QT Longo (LQTS) en comparación con su control isotópico de WT. ( B ) Ejemplo de prolongación del intervalo QT en hPSC-CM tras bloqueo farmacológico de hERG. ( C ) Acortamiento del intervalo QT tras el tratamiento con dosis crecientes de activador de hERG. La flecha indica la dirección del acortamiento. ( D ) Ejemplo de acortamiento del intervalo RR inducido por fármacos. El Panel (C) fue adaptado de referencia> 30. Haga clic aquí para ver una versión más grande de esta figura.
| Baja insulina, BSA, poli (alcohol vinílico), lípidos esenciales (LI-BPEL) Medio | |||
| Componente | Cantidad para 100 mL | ||
| IMDM | 43 ml | ||
| F12 | 43 ml | ||
| Ácido Ascórbico 2-fosfato (5 mg / mL en agua destilada) | 1 ml | ||
| Suplemento de cultivo celular (sustituto directo de la L-glutamina) | 1 ml | ||
| Penicilina / Estreptomicina | 0,5Ml | ||
| Phenol Red | 1 mg | ||
| Hibridoma de Proteína Libre de Medio II (PFHMII) | 5 ml | ||
| BSA (10% en peso / vol en IMDM) | 2,5 ml | ||
| PVA (5% en peso / volumen en agua destilada) | 2,5 ml | ||
| Concentrado de lípidos químicamente definido (CDLC) | 1 ml | ||
| Insulina-Transferrina-Selenio-Etanolamina (ITS-X) 100X | 0,1 ml | ||
| Α-Monotioglicerol (13 μL en 1 mL de IMDM) | 0,3 ml | ||
| Combinar los reactivos, filtrar con un filtro de poro de 0,22 μm y almacenar el medio a 4 ° C durante un máximo de 2 semanas. | |||
Tabla 1: Composición media de Li-BPEL.
Discusión
Este protocolo muestra cómo disociar y preparar hPSC-CMs para medir su FP utilizando MEAs. Los hPSC-CM suelen mostrar actividad eléctrica espontánea, que puede medirse como FP y puede proporcionar datos significativos con respecto a la frecuencia de batir, la duración del intervalo QT y los eventos arrítmicos.
La disociación de los cultivos cardíacos diferenciados 2D es necesaria para recrear una capa de batido en el MEA y representa un paso crítico. El estrés mecánico mediante pipeteo repetido y / o tratamientos enzimáticos de disociación agresivos puede resultar en una alta mortalidad celular, falla en la fijación de la placa MEA y falta de actividad eléctrica espontánea. Este protocolo ha sido optimizado para cultivos monocapa. Sin embargo, se puede utilizar un enfoque similar para los cultivos tridimensionales ( por ejemplo, los cuerpos embrioides o EB) con modificaciones menores, tales como la recolección de EBs seguido de lavado con PBS y un tiempo de incubación más prolongado con la enzima disociadora. ImportarTanto en los cultivos diferenciados 2D como en los 3D, cuanto más antiguas son las células diferenciadas, mayor será el tiempo de incubación necesario para separar las células debido al aumento de la deposición de la matriz extracelular.
El protocolo descrito aquí para la cuantificación de parámetros de PF puede usarse para generar curvas dosis-respuesta para fármacos cardioactivos. Tal como ha sido descrito recientemente por Cavero et al. 31 , la concentración inicial de un fármaco podría afectar profundamente el resultado de una medición de MEA. Por lo tanto, para mejorar la precisión y la fiabilidad de los resultados, sugerimos lo siguiente: 1) en el caso de activadores / bloqueadores irreversibles, utilizar volúmenes relativamente grandes de medio que contiene el fármaco a ser probado. Más en detalle, eliminar 10-50% del volumen medio del chip MEA y añadir un volumen igual de medio en el que el fármaco se disolvió previamente a la concentración apropiada. En este caso, para calcular la concentración final de fármaco, esEl cambio en la concentración después de la remoción del medio. 2) En el caso de activadores / bloqueadores reversibles, añada 10 μl de cada dosis de fármaco a partir de una solución madre 100X.
La mayoría de los protocolos de diferenciación cardiaca tiene como resultado una población mixta variable de cardiomiocitos de tipo nodal, tipo auricular y ventricular, siendo el tipo ventricular el más representado. Esto podría constituir una limitación al modelar las enfermedades cardíacas que afectan a un subtipo específico de cardiomiocitos oa fármacos que actúan sobre canales de iones específicos de subtipos cardíacos. Aunque varios estudios han optimizado las condiciones para dirigir la especificación más controlada durante la diferenciación cardiaca [ 3 , 5 , 37 , 38] , su aplicación más amplia todavía está bajo investigación.
Además, la eficiencia variable de la diferenciación (en diferentes experimentos y en diFferent hPSC líneas) 39 , 40 , 41 , 42 , 43 , 44 . Las estrategias de enriquecimiento de cardiomiocitos basadas en la expresión de proteínas superficiales 35 , 45 (por selección de células asistida por florescence o por selección de gránulos magnéticos 46 , 47 ) y selección metabólica 44 , 48 pueden representar estrategias válidas que se pueden aplicar a cualquier Sin modificar) hPSC-line previa chapado de la hPSC-CM, para mejorar la señal eléctrica.
Aunque los hPSC-CM son notoriamente inmaduros en comparación con los cardiomiocitos adultos humanos 4 , 49 , han demostrado ser valiosos en rEcapitulando e identificando cambios específicos relacionados con la enfermedad ( p . Ej ., En las canalopatías) 19 , 20 , 50 y respuestas inducidas por fármacos ( por ejemplo, bloqueadores del canal iónico cardiaco) 4 , 51 . Además, las células inmaduras son más fáciles de disociar y recuperarse mejor que los cardiomiocitos adultos después de la disociación y el chapado 44 , por lo tanto, la inmadurez de hPSC-CM puede ser recompensada como una ventaja a este respecto. Sin embargo, para poder recapitular, por ejemplo . Las enfermedades cardíacas de inicio tardío y reproducir fielmente las respuestas de fármacos de los cardiomiocitos adultos, se debe obtener un estado hPSC-CM mecánico, metabólico y eléctrico más maduro. Los métodos para madurar estas células incluyen tiempo prolongado en el cultivo 52 , cepa mecánica 53 , estimulación eléctrica 54 , adición de pequeñasMoléculas 55 , cultivo 3D 56 , co-cultivo con otros tipos de células 57 , e incluso una combinación de estos enfoques 58 ; Hasta la fecha, ninguno de estos enfoques ha dado lugar a un fenotipo de tipo adulto.
Como parte de las características de inmadurez, los hPSC-CM muestran automaticidad eléctrica. Aquí se proporcionan detalles sobre cómo cuantificar con exactitud los intervalos QT y RR. Una limitación de la medición de la actividad eléctrica espontánea es que la comparación de intervalos QT puede ser difícil cuando los hPSC-CM muestran frecuencias de batido diferentes. En este caso, las fórmulas de Bazett o Fridericia pueden usarse para corregir el intervalo QT de la frecuencia. Sin embargo, como se informó previamente 30 , se recomienda realizar un análisis de regresión del eje mayor trazando el intervalo QT frente al intervalo RR para los datos crudos y corregidos, para excluir cualquier sesgo posible debido al método de corrección propiamente dicho.
El protocolo presentado aquí, junto con los métodos descritos anteriormente 59 , 60 , ayuda a la estandarización de los procedimientos y el análisis de los FP hPSC-CM, mejorando la reproducibilidad de los datos y permitiendo una mejor comparación de los resultados entre laboratorios.
Divulgaciones
CLM es el co-fundador y asesor de Pluriomics bv. Parte de los costos de publicación fueron cubiertos por Multi Channel Systems.
Agradecimientos
Este trabajo fue apoyado por las siguientes subvenciones: CVON (HUSTCARE): la Iniciativa Holandesa de Investigación Cardiovascular (la Fundación Holandesa del Corazón, la Federación Holandesa de Centros Médicos Universitarios, la Organización Holandesa de Investigación y Desarrollo en Salud y la Real Academia Holandesa de Ciencias); El Consejo Europeo de Investigación (ERCAdG 323182 STEMCARDIOVASC). Damos las gracias a E. Giacomelli (LUMC) por su ayuda con la diferenciación cardíaca hPSC.
Materiales
| Name | Company | Catalog Number | Comments |
| Biological safety cabinet/laminar flow-hood | Cleanair | ||
| MEA2100 in vitro recording system | Multi Channel Systems | ||
| CO2 cell-culture incubator | Sanyo | MCO-15A | |
| Centrifuge | Hitachi | himac-CT6EL | |
| Leica stereomicroscope | Leica Microsystems | MS5 | |
| Handheld pipetman (P-10 (10 μL), P-200 (200 μL), P-1000 (1,000 μL)) | Gilson International | ||
| Filter tips (10 μL, 200μL, 1,000μL) | Corning | 4807 (10 μL), 4810 (200μL), 4809 (1000μL) | |
| Disposable bottle top filter (0.22 μm pore size) | Millipore | SCGVU02RE | |
| Sterile plastic pipette | Greiner Bio-One | 606180 (5 mL), 607180 (10 mL), 760180 (25 mL) | |
| Tweezers | Dumont | ||
| Autoclavable Petri dishes | VWR/ Duran Group | 391-0860 | |
| MEA chip | Multi Channel Systems | MEA200/30iR-Ti-gr | |
| Phosphate-buffered Saline (PBS) calcium, magnesium | Thermo Fisher Scientific | 14040-091 | |
| Phosphate-Buffered Saline (PBS) no calcium, no magnesium | Thermo Fisher Scientific | 14190-169 | |
| Human recombinant fibronectin | Tebu-Bio | J64560 | |
| Custom-made polytetrafluoroethylene (PTFE) MEA rings | LUMC: department of Instrument Development | ||
| Dissociation enzyme - TrypLE Select 1x | Thermo Fisher Scientific | 12563-029 | |
| Tergazyme enzyme detergent | Sigma-Aldrich | Z273287-1EA | |
| Distilled Water | Thermo Fisher Scientific | 15230-089 | |
| Name | Company | Catalog Number | Comments |
| Reagents for LI-BPEL medium | |||
| Iscove's Modified Dulbecco's Medium (IMDM) | Thermo Fisher Scientific | 21056-023 | |
| F12 nutrient mixture (Ham) | Thermo Fisher Scientific | 31765-027 | |
| Ascorbic Acid 2-phosphate | Sigma-Aldrich | A8960 | |
| Glutamax | Thermo Fisher Scientific | 35050-038 | |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15070-063 | |
| Phenol Red | Sigma-Aldrich | P3532 | |
| Protein-free Hybridoma Medium-II (PFHMII) | Thermo Fisher Scientific | 12040-077 | |
| Bovine Serum Albumin (BSA) | Bovogen Biologicals Australia | BSAS05 | |
| Poly(Vinyl Alcohol) (PVA) | Sigma-Aldrich | P8136 | |
| Chemically-defined Lipid Concentrate (CDLC) | Thermo Fisher Scientific | 11905-031 | |
| Insulin-Transferrin-Selenium-Ethanolamine (ITS-X) 100x | Thermo Fisher Scientific | 51599-056 | |
| α-Monothioglycerol | Sigma-Aldrich | M6145 | |
| Name | Company | Software version | Comments |
| MC_Rack | Multi Channel Systems | 4.6.2 | Alternatively, data can be recorded using Cardio2D or MC_Experimenter (MultiChannel Systems) |
| TCX Control | Multi Channel Systems | 1.3.4 | |
| MEA Select | Multi Channel Systems | 1.3.0 | |
| MC_Data Tool | Multi Channel Systems | 2.6.15 | Alternatively, Multi Channel Data Manager (MultiChannel Systems) can be used when custom data export is required (HDF5, EDF, etc.) |
| Clampfit | Molecular Devices | 7.0.0 | Used in step 10.1 for analyzing, graphing, and formatting of all of data. To use Clampfit, download and install the electrophysiology data acquisition and pClam (latest version, 10.7.0), available on the Molecular Devices Website. Once complete, launch the software Clampfit. Alternatively, data can be analysed using Cardio2D (MultiChannel Systems) or MatLab custom code. |
Referencias
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Thomson, J. A., et al. Embryonic stem cell lines derived from human blastocysts. Science. 282 (5391), 1145-1147 (1998).
- Birket, M. J., et al. Expansion and patterning of cardiovascular progenitors derived from human pluripotent stem cells. Nat Biotechnol. 33 (9), 970-979 (2015).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochim Biophys Acta. 1863 (7 Pt B), 1728-1748 (2016).
- Devalla, H. D., et al. Atrial-like cardiomyocytes from human pluripotent stem cells are a robust preclinical model for assessing atrial-selective pharmacology. EMBO Mol Med. 7 (4), 394-410 (2015).
- Lewandowski, J., Kolanowski, T. J., Kurpisz, M. Techniques for the induction of human pluripotent stem cell differentiation towards cardiomyocytes. J Tissue Eng Regen Med. , (2016).
- Mummery, C. L., Zhang, J., Ng, E. S., Elliott, D. A., Elefanty, A. G., Kamp, T. J. Differentiation of human embryonic stem cells and induced pluripotent stem cells to cardiomyocytes: a methods overview. Circ Res. 111 (3), 344-358 (2012).
- Davies, M. P., An, R. H., Doevendans, P., Kubalak, S., Chien, K. R., Kass, R. S. Developmental changes in ionic channel activity in the embryonic murine heart. Circ Res. 78 (1), 15-25 (1996).
- Bellin, M., et al. Isogenic human pluripotent stem cell pairs reveal the role of a KCNH2 mutation in long-QT syndrome. EMBO J. 32 (24), 3161-3175 (2013).
- Moretti, A., et al. Patient-specific induced pluripotent stem-cell models for long-QT syndrome. N Engl J Med. 363 (15), 1397-1409 (2010).
- Ma, D., et al. Modeling type 3 long QT syndrome with cardiomyocytes derived from patient-specific induced pluripotent stem cells. Int J Cardiol. 168 (6), 5277-5286 (2013).
- Yazawa, M., et al. Using induced pluripotent stem cells to investigate cardiac phenotypes in Timothy syndrome. Nature. 471 (7337), 230-U120 (2011).
- Limpitikul, W. B., et al. A Precision Medicine Approach to the Rescue of Function on Malignant Calmodulinopathic Long QT Syndrome. Circ Res. , (2016).
- Liang, P., et al. Patient-Specific and Genome-Edited Induced Pluripotent Stem Cell-Derived Cardiomyocytes Elucidate Single-Cell Phenotype of Brugada Syndrome. J Am Coll Cardiol. 68 (19), 2086-2096 (2016).
- Jung, C. B., et al. Dantrolene rescues arrhythmogenic RYR2 defect in a patient-specific stem cell model of catecholaminergic polymorphic ventricular tachycardia. EMBO Mol Med. 4 (3), 180-191 (2012).
- Novak, A., et al. Cardiomyocytes generated from CPVTD307H patients are arrhythmogenic in response to beta-adrenergic stimulation. J Cell Mol Med. 16 (3), 468-482 (2012).
- Bellin, M., Mummery, C. L. Inherited heart disease - what can we expect from the second decade of human iPS cell research. FEBS Lett. 590 (15), 2482-2493 (2016).
- Sallam, K., Li, Y., Sager, P. T., Houser, S. R., Wu, J. C. Finding the rhythm of sudden cardiac death: new opportunities using induced pluripotent stem cell-derived cardiomyocytes. Circ Res. 116 (12), 1989-2004 (2015).
- Sinnecker, D., Goedel, A., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes: a versatile tool for arrhythmia research. Circ Res. 112 (6), 961-968 (2013).
- Bellin, M., Marchetto, M. C., Gage, F. H., Mummery, C. L. Induced pluripotent stem cells: the new patient?. Nat Rev Mol Cell Biol. 13 (11), 713-726 (2012).
- Sinnecker, D., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes for drug development and toxicity testing. Pharmacol Ther. 143 (2), 246-252 (2014).
- Terrenoire, C., et al. Induced pluripotent stem cells used to reveal drug actions in a long QT syndrome family with complex genetics. J Gen Physiol. 141 (1), 61-72 (2013).
- Abi-Gerges, N., et al. Assessment of extracellular field potential and Ca2+ transient signals for early QT/pro-arrhythmia detection using human induced pluripotent stem cell-derived cardiomyocytes. J Pharmacol Toxicol Methods. 83, 1-15 (2016).
- Del Alamo, J. C., et al. High throughput physiological screening of iPSC-derived cardiomyocytes for drug development. Biochim Biophys Acta. 1863 (7 Pt B), 1717-1727 (2016).
- Blinova, K., et al. Comprehensive Translational Assessment of Human Induced Pluripotent Stem Cell Derived Cardiomyocytes for Evaluating Drug-Induced Arrhythmias. Toxicol Sci. , (2016).
- Nerbonne, J. M. Studying cardiac arrhythmias in the mouse--a reasonable model for probing mechanisms?. Trends Cardiovasc Med. 14 (3), 83-93 (2004).
- Halbach, M., Egert, U., Hescheler, J., Banach, K. Estimation of action potential changes from field potential recordings in multicellular mouse cardiac myocyte cultures. Cell Physiol Biochem. 13 (5), 271-284 (2003).
- Tertoolen, L. G., Braam, S. R., van Meer, B. J., Passier, R., Mummery, C. L. Interpretation of field potentials measured on a multi electrode array in pharmacological toxicity screening on primary and human pluripotent stem cell-derived cardiomyocytes. Biochem Biophys Res Commun. , (2017).
- Rajamohan, D., et al. Automated Electrophysiological and Pharmacological Evaluation of Human Pluripotent Stem Cell-Derived Cardiomyocytes. Stem Cells Dev. 25 (6), 439-452 (2016).
- Sala, L., et al. A new hERG allosteric modulator rescues genetic and drug-induced long-QT syndrome phenotypes in cardiomyocytes from isogenic pairs of patient induced pluripotent stem cells. EMBO Mol Med. 8 (9), 1065-1081 (2016).
- Cavero, I., Guillon, J. M., Ballet, V., Clements, M., Gerbeau, J. F., Holzgrefe, H. Comprehensive in vitro Proarrhythmia Assay (CiPA): Pending issues for successful validation and implementation. J Pharmacol Toxicol Methods. , (2016).
- Sala, L., Bellin, M., Mummery, C. L. Integrating cardiomyocytes from human pluripotent stem cells in safety pharmacology: has the time come?. Br J Pharmacol. , (2016).
- Chen, I. Y., Matsa, E., Wu, J. C. Induced pluripotent stem cells: at the heart of cardiovascular precision medicine. Nat Rev Cardiol. 13 (6), 333-349 (2016).
- Dambrot, C., et al. Strategies for rapidly mapping proviral integration sites and assessing cardiogenic potential of nascent human induced pluripotent stem cell clones. Exp Cell Res. 327 (2), 297-306 (2014).
- Elliott, D. A., et al. NKX2-5(eGFP/w) hESCs for isolation of human cardiac progenitors and cardiomyocytes. Nat Methods. 8 (12), 1037-1040 (2011).
- Ng, E. S., Davis, R., Stanley, E. G., Elefanty, A. G. A protocol describing the use of a recombinant protein-based, animal product-free medium (APEL) for human embryonic stem cell differentiation as spin embryoid bodies. Nat Protoc. 3 (5), 768-776 (2008).
- Karakikes, I., et al. Small molecule-mediated directed differentiation of human embryonic stem cells toward ventricular cardiomyocytes. Stem Cells Transl Med. 3 (1), 18-31 (2014).
- Zhang, Q., et al. Direct differentiation of atrial and ventricular myocytes from human embryonic stem cells by alternating retinoid signals. Cell Res. 21 (4), 579-587 (2011).
- Mummery, C., et al. Differentiation of human embryonic stem cells to cardiomyocytes: role of coculture with visceral endoderm-like cells. Circulation. 107 (21), 2733-2740 (2003).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Zhang, J., et al. Extracellular matrix promotes highly efficient cardiac differentiation of human pluripotent stem cells: the matrix sandwich method. Circ Res. 111 (9), 1125-1136 (2012).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/beta-catenin signaling under fully defined conditions. Nat Protoc. 8 (1), 162-175 (2013).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Dubois, N. C., et al. SIRPA is a specific cell-surface marker for isolating cardiomyocytes derived from human pluripotent stem cells. Nat Biotechnol. 29 (11), 1011-1018 (2011).
- Fuerstenau-Sharp, M., et al. Generation of highly purified human cardiomyocytes from peripheral blood mononuclear cell-derived induced pluripotent stem cells. PLoS One. 10 (5), e0126596 (2015).
- Schwach, V., Passier, R. Generation and purification of human stem cell-derived cardiomyocytes. Differentiation. 91 (4-5), 126-138 (2016).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
- Veerman, C. C., Kosmidis, G., Mummery, C. L., Casini, S., Verkerk, A. O., Bellin, M. Immaturity of human stem-cell-derived cardiomyocytes in culture: fatal flaw or soluble problem?. Stem Cells Dev. 24 (9), 1035-1052 (2015).
- Karakikes, I., Ameen, M., Termglinchan, V., Wu, J. C. Human induced pluripotent stem cell-derived cardiomyocytes: insights into molecular, cellular, and functional phenotypes. Circ Res. 117 (1), 80-88 (2015).
- Avior, Y., Sagi, I., Benvenisty, N. Pluripotent stem cells in disease modelling and drug discovery. Nat Rev Mol Cell Biol. 17 (3), 170-182 (2016).
- Otsuji, T. G., Minami, I., Kurose, Y., Yamauchi, K., Tada, M., Nakatsuji, N. Progressive maturation in contracting cardiomyocytes derived from human embryonic stem cells: Qualitative effects on electrophysiological responses to drugs. Stem Cell Res. 4 (3), 201-213 (2010).
- Mihic, A., et al. The effect of cyclic stretch on maturation and 3D tissue formation of human embryonic stem cell-derived cardiomyocytes. Biomaterials. 35 (9), 2798-2808 (2014).
- Lieu, D. K., et al. Mechanism-based facilitated maturation of human pluripotent stem cell-derived cardiomyocytes. Circ Arrhythm Electrophysiol. 6 (1), 191-201 (2013).
- Yang, X., et al. Tri-iodo-l-thyronine promotes the maturation of human cardiomyocytes-derived from induced pluripotent stem cells. J Mol Cell Cardiol. 72, 296-304 (2014).
- Mannhardt, I., et al. Human Engineered Heart Tissue: Analysis of Contractile Force. Stem Cell Reports. , (2016).
- Kim, C., et al. Non-cardiomyocytes influence the electrophysiological maturation of human embryonic stem cell-derived cardiomyocytes during differentiation. Stem Cells Dev. 19 (6), 783-795 (2010).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat Methods. 10 (8), 781-787 (2013).
- Clements, M. Multielectrode Array (MEA) Assay for Profiling Electrophysiological Drug Effects in Human Stem Cell-Derived Cardiomyocytes. Curr Protoc Toxicol. 68 (22), 1-22 (2016).
- Harris, K. A Human Induced Pluripotent Stem Cell-Derived Cardiomyocyte (hiPSC-CM) Multielectrode Array Assay for Preclinical Cardiac Electrophysiology Safety Screening. Curr Protoc Pharmacol. 71, 1-15 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados