Method Article
Analisi elettrofisiologica dei cardiomiociti derivanti da cellule staminali pluripotenti umane (hPSC-CMs) Utilizzando matrici multi-elettrodo (MEAs)
In questo articolo
Riepilogo
La caratterizzazione elettrofisiologica dei cardiomiociti derivata da cellule staminali pluripotenti umane (hPSC-CM) è fondamentale per la modellizzazione delle malattie cardiache e per la determinazione delle risposte di farmaco. Questo protocollo fornisce le informazioni necessarie per dissociare e piastrare hPSC-CM su matrici multi-elettrodi, misurare il loro potenziale di campo e un metodo per analizzare gli intervalli QT e RR.
Abstract
I cardiomiociti possono ora essere derivati ad alta efficienza sia dalle cellule staminali pluripotenziali embrionali umane che da quelle umane (hPSC). I cardiomiociti derivanti da hPSC (hPSC-CM) sono sempre più riconosciuti come aventi un grande valore per modellare le malattie cardiovascolari negli esseri umani, in particolare le sindromi di aritmia. Hanno anche dimostrato la loro rilevanza come sistemi in vitro per la previsione delle risposte alle droghe, che li rende potenzialmente utili per lo screening e la scoperta di farmaci, la farmacologia di sicurezza e forse anche per la medicina personalizzata. Questo sarebbe facilitato derivando hPSC-CM da pazienti o individui sensibili come hiPSCs. Per tutte le applicazioni, tuttavia, la misura precisa e l'analisi delle proprietà elettriche di hPSC-CM sono essenziali per identificare le modifiche dovute a mutazioni del canale cardiaco e / o farmaci che mirano a canali ionici e possono causare morte cardiaca improvvisa. Rispetto alla morsettiera manuale, i dispositivi multi-elettrodi (MEA) offrono il vantaggioConsentendo registrazioni a media e alta velocità. Questo protocollo descrive come dissociare le colture di cellule 2D di hPSC-CM a piccoli aggregati e singole cellule e piegarle su MEA per registrare la loro attività elettrica spontanea come potenziale di campo. Sono descritti anche metodi per l'analisi dei dati registrati per estrarre parametri specifici, come i QT e gli intervalli RR. Variazioni in questi parametri sarebbero previsti in hPSC-CM che trasportano mutazioni responsabili di aritmie cardiache e dopo l'aggiunta di farmaci specifici, permettendo di individuare quelli che portano un rischio cardiotossico.
Introduzione
Le cellule staminali pluripotenti umane (hPSCs) hanno la capacità di auto-rinnovare e generare praticamente qualsiasi tipo di cellule del corpo umano attraverso la differenziazione 1 , 2 . Sono stati descritti protocolli dettagliati su come dirigere la differenziazione degli hPSC in più linee cardiache (cardiomiociti ventricolari, atriali, pacemaker simili) , 3 , 4 , 5 , 6 , 7 . I cardiomiociti sono cellule elettricamente attive e la conoscenza dettagliata della loro attività elettrofisiologica può essere estremamente informativa per comprendere lo sviluppo e la malattia del cuore 8 . I cardiomiociti derivanti da HiPSC-specifici pazienti (hiPSC-CMs) sono stati utilizzati con successo per modellare e studiare le caratteristiche cellulari, molecolari e elettriche di numerose aritmie cardiache, tra cui la sindrome di Long QT(LQTS) 9 , 10 , 11 , 12 , 13 , sindrome di Brugada 14 e tachicardia ventricolare polimorfica cathecolaminergica 15 , 16 . Inoltre, sono stati aggiunti farmaci multipli a cibi hiPSC-CM per recapitolare l'intervento terapeutico e per salvare i fenotipi patologici cellulari 10 , 15 , 20 , 21 , 22 . Più di recente, sono state sviluppate piattaforme di screening basate su WT hiPSC-CM, in risposta alla necessità di sistemi umani nelle fasi iniziali della scoperta di farmaci 23 , 24 , 25 come i cardiomiociti roditori differiscono profondamente da huMans nell'espressione del canale ionico e della biofisica 26 .
A tal fine sono in fase di sviluppo e implementazione tecnologie adatte per applicazioni a media e alta velocità. Queste includono le registrazioni ottiche del potenziale di membrana, i transitori Ca 2+ e il ceppo, le misure di impedenza (come misura indiretta di contrattilità delle cellule) e le misure di potenziale extracellulare del campo (FP) (per la revisione vedere il riferimento 24 ). I dispositivi Multi-Electro Array (MEA) consentono la registrazione dei segnali elettrici (o FP) generati e modellati da monostrati o piccoli cluster di cardiomiociti. Il contorno FP è correlato con il potenziale cardiaco dell'azione e, in qualche misura, con le registrazioni dell'elettrocardiogramma (ECG) 27 ; Tipicamente mostrano una rapida accelerazione iniziale corrispondente all'influenza di Na + e alla depolarizzazione della membrana (picco di R / Q), una fase di slow wave / plateau probabilmente corrispondente alla Ca2+ e una fase di repolarizzazione corrispondente ad un efflusso predominante di K + (picco di T). La perturbazione della forma d'onda FP può essere correlata con i cambiamenti nelle fasi potenziali di azione specifiche 28 .
Sebbene le registrazioni di potenziali di azione delle pinze di patch potrebbero essere più informative, specialmente per i parametri come velocità di salto e potenziale di riposo, le misurazioni manuali non sono fattibili per esperimenti a scala media e ad alta velocità, mentre il morsetto di patch automatico è stato recentemente applicato a hPSC -CM 29 . Tuttavia, poiché le registrazioni prolungate su MEA permettono di esaminare sia l'esposizione acuta che cronica ai composti, è ora possibile utilizzare piattaforme hPSC-CM per screening dei farmaci, rilevazione 24 , 30 e per farmacologia di sicurezza 31 , 32 . Ciò mantiene la promessa di precisione futura o persoMedicina interna 34 .
Lo scopo di questo protocollo è fornire le informazioni necessarie per dissociare e placcare hPSC-CM sui chip MEA e misurare il loro FP. In questa procedura, ogni fase è stata ottimizzata, assicurando la sopravvivenza e il recupero delle cellule ottimali dopo la dissociazione, l'attaccamento ottimale delle cellule alla piastra MEA e l'analisi e la quantificazione standard dei parametri. In particolare vengono spiegati ed esemplificati la procedura per la registrazione FP extracellulare, l'analisi degli intervalli QT e RR e la valutazione degli effetti farmacologici.
Protocollo
1. Preparazione di soluzioni e reagenti
- Preparare il mezzo di coltura hPSC-CM utilizzando l'insulina, l'albumina bovina del siero, il polivinilalcool, i lipidi essenziali (LI-BPEL), media 34 , 35 , 36 combinando i reagenti descritti nella Tabella 1 . Filtrare il mezzo attraverso un filtro puro da 0,22 μm e conservare a 4 ° C per un massimo di 2 settimane.
- Preparare la soluzione umana ricombinante della fibronectina ricostituendo 1 mg di fibronectina ricombinante umana in 5 ml di acqua distillata sterile per raggiungere una concentrazione di 200 μg / ml, aliquota e conservare a -80 ° C.
- Preparare una soluzione detergente enzimatica (vedi tabella dei materiali ) per aiutare a rimuovere le cellule residui del microarray, sciogliendo 1 g di polvere detergente enzimatica in 100 ml di caldo (~ 40-45 ° C) Acqua deionizzata. Lasciare raffreddare la soluzione e conservare a 4 ° CO fino a un anno.
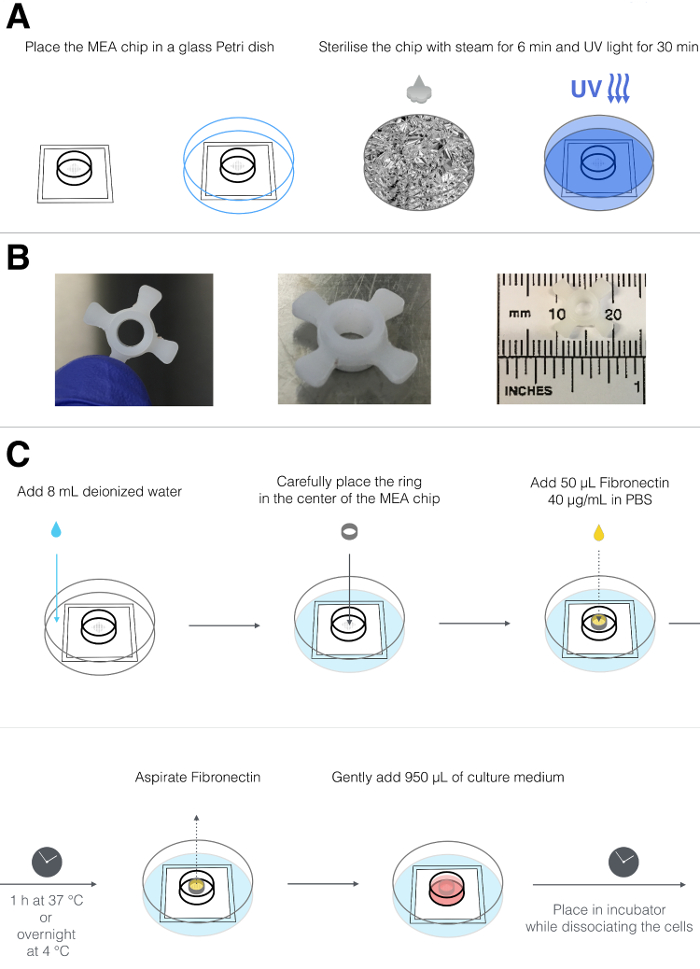
2. Sterilizzazione dei chip MEA (Figura 1A)
NOTA: Sono disponibili diverse configurazioni di MEA, con formati singolo o multi-well. Il protocollo qui descritto utilizza la camera singola MEA contenente 60 elettrodi di registrazione in una disposizione di griglia 8 x 8 (vedi tabella dei materiali ). Il diametro dell'elettrodo è di 30 μm e la distanza tra gli elettrodi è di 200 μm. È presente anche un elettrodo di riferimento.
- Sciacquare accuratamente il chip con acqua deionizzata.
- Metti i chip MEA all'interno di un piatto di vetro Petri che può essere autoclavato. Avvolgere il piatto in fogli di alluminio.
- Sterilizzare le patatine in una pentola a pressione di laboratorio per 6 min. Lasciare raffreddare i piatti prima di aprire.
NOTA: In alternativa, le patatine possono essere sterilizzate immersandole in 1 ml di etanolo al 80% in volume (v / v) a RT per 15-30 minuti. - Posizionare i chip in un cofano di cultura cellulare e esporre ilSuperficie per luce UV per circa 30 min.
3. Rivestimento di chip MEA (Figura 1B e 1C)
- Posizionare le patatine pulite all'interno di un piatto standard Petri da 10 cm Ø.
- Aggiungere 8 ml di acqua distillata sterile al piatto di Petri per formare una camera umidificata, che impedirà il piccolo volume di terreno di coltura sul chip da asciugare quando posto nell'incubatrice.
- Utilizzare anelli in politetrafluoroetilene su misura (PTFE) ( Figura 1B ) per assicurare la placcatura dei cardiomiociti al centro del chip, dove si trova l'array elettrodo. (Gli anelli in PTFE sono precedentemente conservati in etanolo al 80%.) Nella cappa di coltura, rimuovere gli anelli dall'etanolo, inserirli in un piatto di Petri sterile senza coperchio e lasciare asciugare gli anelli nel cofano.
- Posizionare un anello asciutto all'interno di un chip MEA utilizzando pinzette a fiamma sterilizzate ( Figura 1C ).
NOTA: In alternativa utilizzare l'etanolo al 80% per lavare le pinzette e lasciare asciugare iN il cofano della tessitura prima di utilizzarli. - Sciogliere una aliquota di materiale umano ricombinante di fibronectina (200 μg / mL in acqua distillata) e diluire in PBS con Ca 2+ e Mg 2+ per ottenere una soluzione di lavoro di 40 μg / mL. Coprire gli elettrodi aggiungendo 50 μL di fibronectina da 40 μg / mL all'interno dell'anello.
NOTA: Il rivestimento con fibrometina ricombinante umana assicura l'ottimale attacco hPSC-CM. Tuttavia, possono essere utilizzati altri tipi di proteine di rivestimento come la fibronectina bovina o le miscele di proteine della matrice extracellulare. - Chiudere il coperchio del piatto di Petri di plastica Ø 10 cm e trasferire con cura il piatto contenente il chip MEA nell'incubatrice. Incubare a 37 ° C per almeno 1 h, oa 4 ° CO / N.
- Trasferire il piatto contenente il chip MEA al cofano della cultura cellulare. Prima di usare il chip MEA, aspirare 50 μl di fibronectina usando una pipetta P200 o un sistema di vuoto nel cofano senza spostare l'anello PTFE. Utilizza plaSuggerimenti stic per questo passo. Assicurarsi che nessun oggetto solido ( ad esempio, punte pipette) tocchi l'interno del piatto in quanto ciò può danneggiare gli elettrodi.
NOTA: si estenderà la durata dei chip MEA. - Aggiungere delicatamente 950 μL di materiale LI-BPEL (vedere tabella 1 e tabella dei materiali ), assicurandosi che sia distribuita uniformemente e che l'anello non galleggi. Ritorna il piatto all'incubatore.
4. Dissociazione e placcatura di hPSC-CMs (Figura 2)
NOTA: Il protocollo qui descritto fa uso di hPSC-CMs che sono stati differenziati in una coltura monostrato utilizzando citochine 34 a ~ 18 giorni dopo l'inizio della differenziazione. Tuttavia, è risultato essere adatto a qualsiasi cultura 2D e 3D hPSC-CM. Quando si utilizzano culture differenziate a punti precedenti o successivi, la regolazione del tempo di incubazione dell'enzima di dissociazione (vedere tabella dei materiali ) potrebbe esseresario. I volumi seguenti sono intesi per un singolo pozzetto di un formato a piatto da 12 pozzetti (3,8 cm 2 ).
- Aspirare il mezzo dalla cultura hPSC-CMs.
- Mentre lavora sotto una cappa di coltura tissutale, aggiungere 1-2 mL / pozzetto di PBS senza Ca 2+ / Mg 2+ per lavare la coltura. Aspirare il PBS.
- Aggiungere 500 μl / pozzetto dell'enzima di dissociazione. Incubare per 5 minuti a 37 ° C.
- Aggiungere 1 mL LI-BPEL / pozzetto per diluire l'enzima. Staccare delicatamente il monostrato di hPSC-CM sciacquando delicatamente utilizzando una pipetta P1000. Raccogliere la sospensione cellulare in un tubo da 15 ml.
- Sciacquare il pozzetto con 1 ml di LI-BPEL per raccogliere tutte le cellule e le cellule rimanenti.
- Aggiungere un altro 2-3 mL di LI-BPEL per raggiungere un volume finale di 5-6 mL e pipettare leggermente 3-5 volte con una pipetta da 5 mL per dissociare i grumi di cellule.
NOTA: la dissociazione in singole cellule a questo punto non è necessaria, in quanto influisce sulla sopravvivenza cellulare. Presenza di piccoli cluster volontàGarantire una maggiore vitalità cellulare. - Centrifugare le cellule a RT per 3 min a 300 x g.
- Rimuovere il surnatante, cercando di rimuovere la maggior parte del liquido in eccesso ma senza dislocare il pellet cellulare.
- Resuspendere il pellet di cellule in 250 μl di LI-BPEL (utilizzando un P1000 e pipettare estremamente delicatamente).
- Distribuire ~ 50 μL di sospensione cellulare per MEA (fino a 5 MEAs possono essere preparate da un pozzetto di una piastra a 12 pozzetti) pipettando la sospensione cellulare direttamente al centro dell'anello PTFE, in cima all'arco di elettrodi.
NOTA: in questa fase le cellule sono difficili da contare a causa della presenza di cluster di cellule e il numero totale di cellule per MEA può variare sostanzialmente, a seconda della sorgente hPSC-CM utilizzata. Durante la placcatura, assicurarsi che la nube delle cellule dissociate copre l'area degli elettrodi. - Trasferire con cautela i MEA all'incubatore a 37 ° C e lasciare che le cellule fissino O / N
5. Rimozione anello e Re medioFreschezza (Figura 3)
- A 1 giorno dopo la placcatura, rimuovere attentamente l'anello in un ambiente sterile utilizzando pinze sterili ( Figura 3A-3B ).
- Sciacquare l'anello in etanolo al 80% (v / v) e conservarlo in un tubo da 50 ml contenente fresco etanolo al 80% (v / v).
- Rimuovere delicatamente 500 μL di mezzo dal chip MEA e aggiungere 500 μl di fresco LI-BPEL.
- Trasferire i MEA all'incubatore a 37 ° C.
NOTA: Le celle dovrebbero iniziare a battere 1-7 giorni dopo la rimozione degli anelli e il cambiamento medio ( Figura 3C ). - Misurare l'attività elettrica di hPSC-CM su MEA 1-7 giorni dopo la rimozione dell'anello.
6. Controllare la qualità del segnale (Figura 4)
- Accendere il computer e avviare la suite software collegata all'Area MEA: TCX-Control, MC_MEA Select e MC_Rack. Impostare la temperatura a 37 ° C in TCX-Control per registrare le misurazioni a temperatura fisiologica.
- Rimuovere il piatto contenente il chip MEA fL'incubatore. Aprire il coperchio, estrarre il chip MEA e collocarlo su un tessuto per assorbire l'acqua residua.
- Pulire con cautela i contatti esterni della piastra con un tessuto e pulirli utilizzando un tampone di cotone inumidito con etanolo al 100% (v / v) per rimuovere eventuali residui d'acqua o detriti, che potrebbero causare rumore del segnale.
- Trasferire la piastra MEA verso la fase di registrazione riscaldata (37 ° C) per rilevare l'attività spontanea ( es. Hardware: vedere tabella dei materiali software: MC_Rack).
- Apri MC_Rack: fai clic su 'Modifica' → 'Aggiungi MC_Card' per creare un nuovo protocollo. Utilizza il menu a discesa 'Modifica' per aggiungere diverse finestre di registrazione e di visualizzazione al protocollo.
NOTA: Si raccomanda una frequenza di campionamento di almeno 10 kHz. Il protocollo che utilizziamo contiene uno strumento di visualizzazione a lunga durata, con un layout completo di tutto il chip MEA e uno spike sorter, essenziale per catturare l'effetto farmaco iN in tempo reale. Il ritaglio di spike è sintonizzato con un 'pre-trigger' di 20 ms, un 'trigger post' di 800 ms e un 'dead time' di 2 ms. Il protocollo può essere salvato come file '.rck' e ricaricato prima di avviare gli esperimenti.
- Apri MC_Rack: fai clic su 'Modifica' → 'Aggiungi MC_Card' per creare un nuovo protocollo. Utilizza il menu a discesa 'Modifica' per aggiungere diverse finestre di registrazione e di visualizzazione al protocollo.
- Avviare il protocollo in modalità play cliccando sul pulsante 'play'.
NOTA: A questo punto è possibile ricaricare il protocollo salvato al punto 6.5. In MC_Rack, caricare il protocollo (estensione file .rck) facendo clic su 'File' → 'Apri'. - Se i segnali presentano picchi e picchi T chiaramente visibili, attendere 10-15 minuti per concludere la fase di adattamento. Esempi di tracce di qualità buone e cattive sono mostrate in Figura 4 .
NOTA: se non è possibile rilevare alcun picco di T in uno degli elettrodi, non procedere con l'esperimento. Di solito questo è il risultato di una scarsa attività elettrica degli hPSC-CM o un cattivo attaccamento delle celle agli elettrodi.
7. Avviare ExPeriment e registrazione
- Fare clic su "record" e quindi su "play" e acquisire i dati per 10 min sotto le condizioni di base per determinare lo stato stazionario. Annota gli elettrodi che hanno il segnale migliore in modo che possano essere facilmente identificati ed esportati in seguito per l'analisi.
- Per la valutazione del farmaco-risposta, aggiungere concentrazioni crescenti di farmaci ogni 10 minuti. Ad esempio, aggiungere il bloccatore hERG E4031 ad una concentrazione finale di 1 μM. A questo scopo, rimuovere 100 μl di mezzo e aggiungere lo stesso volume di 10 μM di E4031 sciolto nel mezzo.
NOTA: Come dimostrato in precedenza da Cavero e colleghi 31 , una scelta saggia del volume in cui i farmaci sono sciolti è importante, in quanto può alterare profondamente la curva di risposta ai farmaci. - Ripetere il passaggio 7.2 per tutte le altre concentrazioni di interesse.
- Fai clic su "stop" per concludere le registrazioni alla fine del protocollo.
8.MEA Pulizia per il riutilizzo
- Una volta terminata la registrazione dell'esperimento, rimuovere delicatamente il mezzo con una pipetta P1000. Non toccare l'interno del piatto in quanto ciò può danneggiare gli elettrodi. Scartare secondo le norme locali di sicurezza.
- Sciacquare le patatine MEA con acqua deionizzata usando una bottiglia di lavaggio e ripetere la fase di lavaggio 3-4x.
NOTA: a questo punto non è necessario che le celle siano completamente staccate dal chip. - Aggiungere 1 ml di soluzione detergente enzimatica 1% (v / v) in ogni pozzetto e incubare O / N a 4 ° C per consentire il distacco cellulare e la rimozione cellulare.
- Un giorno dopo, sciacquare accuratamente le patatine MEA con acqua deionizzata per rimuovere la soluzione detergente enzimatica e le cellule residue e aggiungere 1 mL di acqua deionizzata. I circuiti Clean MEA possono essere immagazzinati immersi in acqua deionizzata a 4 ° C.
9. Esportazione dei dati
- Aprire il software Strumento MC_Data collegato all'installazione MEA.
- Fai clic su 'File' → 'Apri MCD'.
- Fare clic su 'Strumenti' → 'Convertire MCD in ABF' .
- Selezionare gli elettrodi con i migliori segnali di registrazione da esportare per l'analisi. Scegli la directory in cui salvare i file esportati e fai clic su "Salva". La procedura di esportazione può richiedere diversi minuti a seconda del numero degli elettrodi esportati e della dimensione totale dei file di registrazione.
10. Analisi dei dati
- Scaricare e installare un programma di acquisizione e analisi dati di elettrofisiologia ( ad esempio, pClamp). Una volta completata, avviare il software di analisi ( ad esempio, Clampfit).
- Calcolo dell'intervallo RR (Figura 5).
- Posizionare due cursori verticali per definire la regione di interesse sulla traccia. Selezionare 'Rilevamento eventi' → 'Ricerca soglia'. Il cursore orizzontale deve attraversare tutti gli eventi che devono essere quantificati, come mostrato nella Figura 5A .
- Fare clic su &# 39; OK 'e quindi' Accetta la categoria intero '. Il software cercherà quindi la traccia per tutti gli eventi adatti. I puntini blu saranno posti sopra gli eventi.
- Regolare la sensibilità della selezione automatica impostando i parametri nella finestra di rilevazione degli eventi principali.
- Una volta che tutti gli eventi sono stati identificati automaticamente, passare alla finestra dei risultati (Finestra → Risultati) e copiare la colonna intitolata "Intervent Interval", che contiene i dati di frequenza ( Figura 5B ).
- Calcolo dell'intervallo QT (Figura 6-9):
- Posizionare un cursore prima e uno dopo un unico FP in condizione di stato stazionario. Selezionare 'Rilevazione eventi' → 'Crea modello' ( Figura 6 ) .
- Verifica che il FP sia identificato correttamente e fai clic su "Aggiungi". Il modello verrà spostato nel pannello inferiore.
- Salvare il modello come '.atf'file. In questo modo è stata creata una traccia di un modello che verrà ricercata dal software durante tutta la registrazione ( Figura 7 ). Crea un modello per ogni condizione, in quanto un effetto farmaco potrebbe alterare la forma del FP. Se necessario, filtrare leggermente la traccia per includere un massimo di 10.000 punti nei due cursori.
- Una volta che il modello è stato salvato con estensione ".atf", seleziona 'Rilevazione eventi' → 'Ricerca modello' e carica il modello.
- Regola la soglia "corrispondenza modello" per identificare correttamente tutti i FP nell'intervallo selezionato.
- Una volta che tutti gli eventi sono stati identificati correttamente, salvarli in un nuovo file '.abf'.
- Aprire il file con il software di analisi e calcolare automaticamente il tempo di picco per i Q / R ei picchi T ( figure 8, 9 ). Se le tracce sono molto rumorose, applicare un filtro. Calcola l'intervallo QT sottraendo il valoreValore Q / R dal valore T in un editor di fogli di calcolo di scelta.
Risultati
Un giorno dopo la dissociazione e la placcatura, lo strato di hPSC-CM sarà visibile come una pellicola densa e bianca che copre il centro della camera MEA ( Figura 3A ). Dopo la rimozione dell'anello ( figura 3B ), Lo strato dovrebbe rimanere in posizione e l'ispezione su un microscopio leggero mostrerà gli elettrodi MEA coperti dal layer (contrattuale) hPSC-CM ( figura 3C ). A causa dell'accoppiamento fisico ed elettrico delle celle, verrà utilizzato solo un elettrodo (elettrodo dorato) per l'analisi.
In alternativa, quando si lavora con strutture 3D, come corpi embrionali o microtissues, questi possono essere placcati in modo che siano fisicamente e elettricamente disaccoppiati. L'ispezione visiva al microscopio potrebbe confermare che nessuna connessione fisica tra i cluster e le onde R non sincronizzate a MEA non confermi alcun accoppiamento elettrico. In questo cPossono essere analizzati diversi elettrodi indipendenti.
Le registrazioni tipiche delle tracce FP sono mostrate in Figura 4 . In particolare, una traccia di buona qualità può essere definita dalla presenza di un picco chiaro corrispondente all'influenza di Na + e dalla depolarizzazione della membrana (picco di R / Q), una chiara fase di repolarizzazione corrispondente all'efflusso K + (picco T) Rapporto di segnale / rumore ( figura 4A , sinistra: scala della nota dell'asse y e figura 4B ). Le tracce di cattiva qualità ( Figura 4A , mezzo) possono essere il risultato di un guasto di hPSC-CM da attaccare alla piastra MEA o di una debole attività elettrica hPSC-CM. L'attesa di 1-3 giorni per un migliore attaccamento può migliorare il segnale; Tuttavia, se non è visibile alcun miglioramento del segnale, si consiglia di escludere questa MEA dagli esperimenti. Le tracce rumorose ( Figura 4A , destra) possono essere analizzate dopo il filtraggio.
L'analisi di successo dell'intervallo RR può essere identificata mediante ispezione visiva dello schermo che mostra la rilevazione di picco ( Figura 5A, 5B ). All'interno dell'intervallo di tempo definito dai cursori verticali, devono essere presenti i segni blu corrispondenti ad ogni picco. Nel caso in cui il programma non identifica uno o più picchi, provare a spostare il cursore orizzontale e eseguire nuovamente l'analisi o regolare le impostazioni di rilevamento. Allo stesso modo, l'analisi di successo dell'intervallo QT può essere identificata mediante ispezione visiva dello schermo che mostra la rilevazione FP ( Figura 7 ). All'interno dell'intervallo di tempo definito dai cursori verticali, devono essere presenti segni blu corrispondenti a ciascun rilevamento FP. Nel caso in cui il programma non identifica uno o più FP, provare a ridefinire il modello FP ( Figura 6 ) o regolare le impostazioni di rilevamento e rieseguire l'analisi.
Le tracce individuali FP estratte o la loro media ( FigE 8) può essere utilizzato per ottenere valori di intervallo QT con impostazioni specifiche come in Figura 9 . L'analisi della relazione QT-RR è consigliabile ed è significativa nelle linee hPSC malate e WT ( Figura 10A ) e per valutare la necessità e / o l'effetto delle correzioni dell'intervallo QT ( Figura 10B-10D ). Le hPSC-CM che trasportano le mutazioni che causano LQTS hanno intervalli QT prolungati rispetto ai controlli WT ( Figura 11A ). Il trattamento di hPSC-CM con un bloccante hERG produce un prolungamento dell'intervallo QT ( Figura 11B ); Al contrario, il trattamento con un attivatore hERG determina l'abbreviamento dell'intervallo QT ( Figura 11C ). Infine, il trattamento con farmaci che influenzano la frequenza di battitura di hPSC-CM dovrebbe essere visibile come una variazione dell'intervallo RR ( Figura 11D , abbreviazione dell'intervallo RR).
 />
/>
Figura 1: Sterilizzazione e rivestimento di chip MEA. ( A ) Schema che rappresenta il processo di sterilizzazione, incluso il posizionamento del chip MEA in un piatto di vetro Petri autoclavabile, avvolgente in foglio di alluminio, vaporante per 6 minuti e esposizione a UV per 30 minuti. ( B ) Viste in alto (pannello sinistro) e laterali (pannello centrale) di un anello PTFE personalizzato; L'anello ha un diametro esterno di 1,2 cm, di cui 4 flap che permettono il posizionamento al centro del chip MEA e il diametro interno è di 0,4 cm (pannello destro). ( C ) Schema che rappresenta la preparazione del chip MEA, inclusa la collocazione del chip in un piatto di plastica standard di Petri, aggiungendo 8 ml di acqua deionizzata all'esterno della camera MEA, posizionando l'anello di PTFE al centro della camera e rivestendo l'array elettrodo con fibronectina . Dopo il tempo di incubazione, la fibronectina viene rimossa e sostituita con mezzo di coltura.T = "_ blank"> Fare clic qui per visualizzare una versione più grande di questa figura.
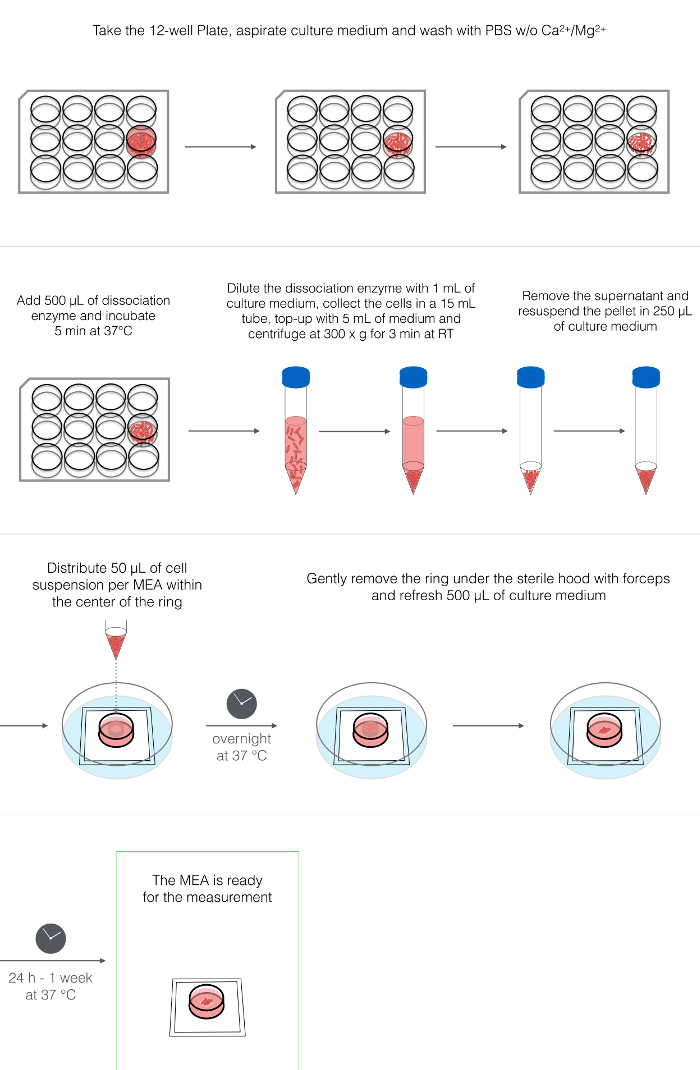
Figura 2: Dissociazione e placcatura di hPSC-CM. Schema rappresentativo dei processi di dissociazione enzimatica, centrifugazione, resuspensione e placcatura di hPSC-CMs al centro della camera MEA. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Chips MEA con layer hPSC-CM. ( A ) Vista superiore del chip MEA contenente l'anello e lo strato di hPSC-CM. ( B ) vista laterale del chip MEA dopo rimozione dell'anello. ( C ) Immagine del campo luminoso dello strato hPSC-CMs placcato sul micro-Elettrodo array; Ingrandimento 4X. Clicca qui per visualizzare una versione più grande di questa figura.
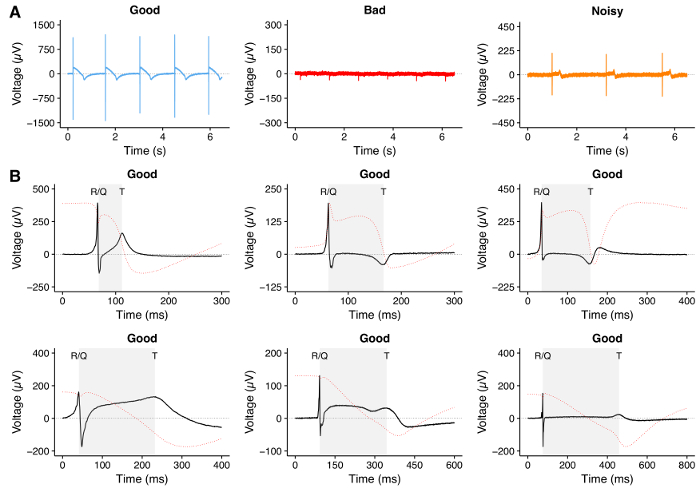
Figura 4: Registrazione MEA di hPSC-CM. ( A ) Le tracce rappresentative registrate con il MEA mostrano una traccia di buona qualità con picchi di R / Q e T chiaramente visibili con un elevato rapporto segnale / rumore (a sinistra), una traccia di cattiva qualità senza picchi di R / Q e T chiaramente visibili, E una traccia rumorosa con picchi R / Q e T chiaramente visibili ma con basso rapporto segnale / rumore (a destra). ( B ) Esempi rappresentativi di tracce FP di buona qualità con diverse morfologie che possono essere registrate durante gli esperimenti MEA usando hPSC-CM. L'area ombreggiata rappresenta l'intervallo QT misurato durante l'analisi. Poiché il FP a MEA assomiglia al primo derivatoE del potenziale d'azione 28 , abbiamo calcolato l'integrale della traccia FP, indicata come linea tratteggiata rossa, come dimostrazione teorica di una scelta dell'onda T vicina a una completa repolarizzazione potenziale d'azione. Clicca qui per visualizzare una versione più grande di questa figura.
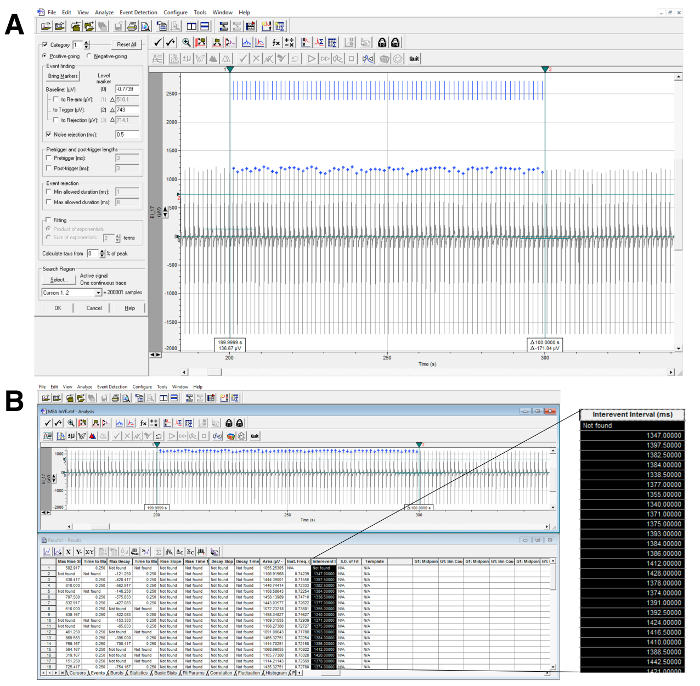
Figura 5: Calcolo dell'intervallo RR. ( A ) Esempio di analisi di intervallo RR con rilevazione automatica del picco (superiore) e estrazione dei dati (in basso) utilizzando il software di analisi (vedere tabella dei materiali). I cursori verticali identificano l'intervallo di tempo di interesse e il cursore orizzontale attraversa tutti gli eventi rilevati e identificati da segni blu. ( B ) La colonna ingrandita mostra i dati estratti utilizzati per calcolare l'intervallo RR. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Creazione di un modello FP. Esempio di selezione dei modelli mediante cursori verticali posizionati prima e dopo un unico FP. Questo modello viene utilizzato per l'identificazione automatica FP. Clicca qui per visualizzare una versione più grande di questa figura.
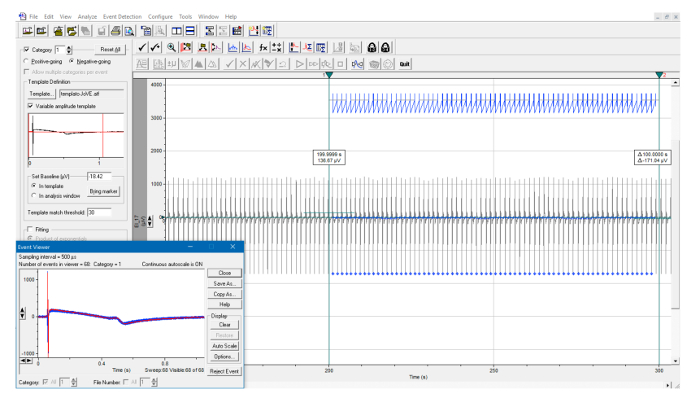
Figura 7: Identificazione automatica del modello FP. Esempio di ricerca di modelli durante un intervallo definito da due cursori verticali. Tutti gli eventi rilevati sono identificati da segni blu e sono automaticallY sovrapposti nell'inserto. Clicca qui per visualizzare una versione più grande di questa figura.
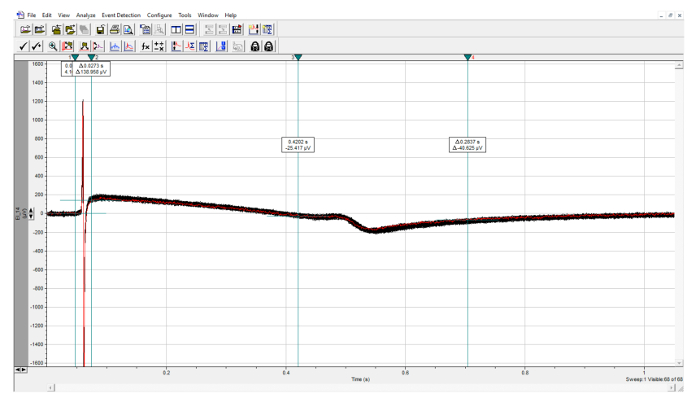
Figura 8: Quantificazione dei parametri FP. Analisi degli eventi salvati, con il picco R individuato manualmente nei primi due cursori e il picco T individuato manualmente negli ultimi due cursori. Clicca qui per visualizzare una versione più grande di questa figura.
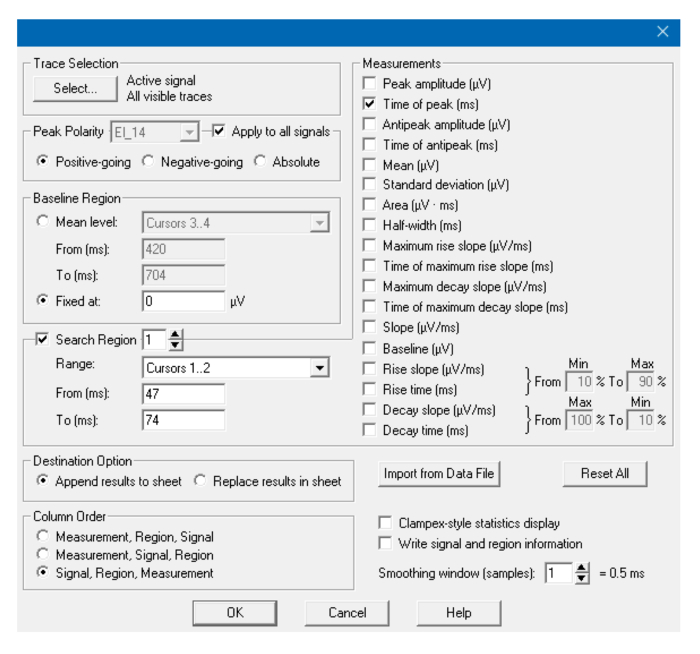
Figura 9: finestra di analisi. Parametri utilizzati nella finestra Statistica per rilevare il picco di R. Per rilevare il picco di T, cambiare i cursori e (se necessario) porità. Clicca qui per visualizzare una versione più grande di questa figura.
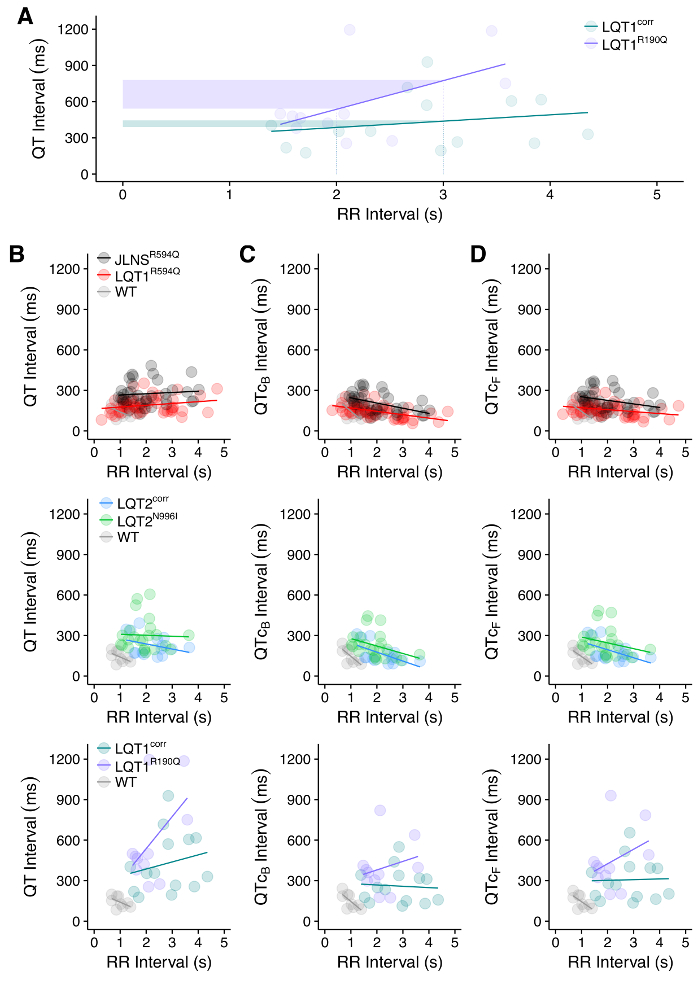
Figura 10: Relazione QT-RR. ( A ) Esempio di relazione differente tra intervalli QT e RR tra hTc-CM e WT (LQT1 corr ) e Long QT sindrome tipo 1 (LQT1 R190Q ). Le aree ombreggiate mostrano che lo stesso spostamento nell'intervallo RR genera un cambiamento maggiore nell'intervallo QT della linea malata LQT1, probabilmente aumentando la suscettibilità delle aritmie. ( B ) Rapporto tra gli intervalli QT e RR non corretti misurati a MEA in CM da 7 diverse linee hPSC. L'effetto della correzione QT per le formula di Bazett ( C ) o Fridericia ( D ) è mostrato e visibile come variazione del pendio del QT-RR in Rapporto taral. Figure adattate dal riferimento 30 . Clicca qui per visualizzare una versione più grande di questa figura.
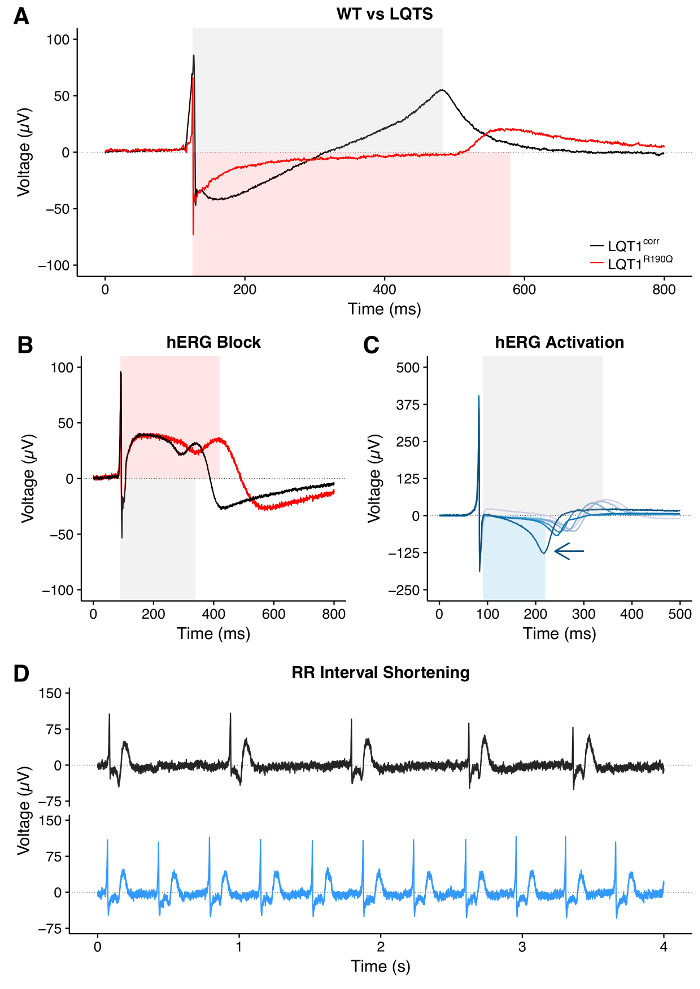
Figura 11: Variazioni intervallate QT e RR dell'intervallo delle malattie o dei farmaci. ( A ) Esempio di prolungamento dell'intervallo QT in hPSC-CM derivato da un paziente portante una mutazione Long-QT (LQTS) rispetto al suo controllo isogeno WT. ( B ) Esempio di prolungamento dell'intervallo QT in hPSC-CM sul blocco hERG farmacologico. ( C ) l'intervallo QT si riduce al momento del trattamento con dosi crescenti di attivatore hERG. La freccia indica la direzione dell'accelerazione. ( D ) Esempio di riduzione dell'intervallo RR indotto dalla droga. Il pannello (C) è stato adattato dal riferimento> 30. Clicca qui per visualizzare una versione più grande di questa figura.
| Bassa insulina, BSA, polivinilalcool, lipidi essenziali (LI-BPEL) Medio | |||
| Componente | Quantità per 100 ml | ||
| IMDM | 43 mL | ||
| F12 | 43 mL | ||
| Acido Ascorbico 2-fosfato (5 mg / mL in acqua distillata) | 1 ml | ||
| Supplemento alla coltura cellulare (sostituto diretto per L-glutammina) | 1 ml | ||
| Penicillina / streptomicina | 0.5mL | ||
| Fenolo rosso | 1 mg | ||
| Protein Free Ibridoma Medio-II (PFHMII) | 5 mL | ||
| BSA (10% wt / vol in IMDM) | 2,5 mL | ||
| PVA (5% in peso / vol in acqua distillata) | 2,5 mL | ||
| Concentrato lipidico definito chimicamente (CDLC) | 1 ml | ||
| Insulina-transferrina-selenio-etanolammina (ITS-X) 100X | 0,1 mL | ||
| Α-Monothioglycerol (13 μL in 1 mL di IMDM) | 0,3 mL | ||
| Combinare i reagenti, filtrare con un filtro puro da 0,22 μm e memorizzare il supporto a 4 ° C per un massimo di 2 settimane. | |||
Tabella 1: Composizione media di Li-BPEL.
Discussione
Questo protocollo mostra come dissociare e preparare hPSC-CM per misurare il loro FP utilizzando MEAs. HPSC-CM mostrano solitamente attività elettrica spontanea, che può essere misurata come FP e può fornire dati significativi rispetto alla frequenza battente, alla durata dell'intervallo QT e agli eventi aritmici.
La dissociazione di culture differenziate cardiache 2D è necessaria per ricreare un livello di battimento sulla MEA e rappresenta un passo critico. La tensione meccanica mediante pipettature ripetute e / o trattamenti enzimatici di dissociazione aggressiva può portare ad una elevata mortalità cellulare, a non attaccarsi alla piastra MEA e alla mancanza di attività elettrica spontanea. Questo protocollo è stato ottimizzato per le culture monolayer. Tuttavia, un approccio simile può essere utilizzato per colture tridimensionali (3D) ( ad esempio, corpi embriodali o EB) con modifiche minori, come la raccolta delle EBs seguita da lavaggio PBS e un tempo di incubazione più lungo con l'enzima dissociante. ImportareIn entrambe le colture differenziate 2D e 3D, le vecchie cellule differenziate, il tempo di incubazione più lungo richiesto potrebbe essere quello di staccare le cellule a causa della maggiore deposizione di matrici extracellulari.
Il protocollo qui descritto per la quantificazione dei parametri FP può essere utilizzato per generare curve di risposta dose per i farmaci cardioattivi. Come recentemente descritto da Cavero et al. 31 , la concentrazione iniziale di un farmaco potrebbe influire profondamente sull'esito di una misurazione MEA. Pertanto, per migliorare l'accuratezza e l'affidabilità dei risultati, suggeriamo quanto segue: 1) in caso di attivatori / bloccanti irreversibili, utilizzare volumi relativamente grandi di terreno contenente il farmaco da testare. Più in dettaglio, rimuovere il 10-50% del volume medio dal chip MEA e aggiungere un uguale volume di mezzo in cui il farmaco è stato precedentemente disciolto alla concentrazione appropriata. In questo caso, per calcolare la concentrazione finale del farmaco, è fondamentale considerareLa variazione della concentrazione dopo la rimozione media. 2) In caso di attivatori / bloccanti reversibili, aggiungere 10 μL di ciascuna dose di farmaco da una soluzione di riserva 100X.
La maggior parte dei protocolli di differenziazione cardiaca produce una popolazione misti variabile di cardiomiociti simil-nodali, atriali e ventricolari, con il tipo ventricolare più rappresentato. Ciò potrebbe costituire una limitazione quando modella le malattie cardiache che interessano un sottotipo specifico di cardiomiociti o farmaci che agiscono su canali ionici specifici del sottotipo cardiaco. Anche se diversi studi hanno ottimizzato le condizioni per dirigere una specifica più controllata durante la differenziazione cardiaca 3 , 5 , 37 , 38 , la loro più ampia applicabilità è ancora in fase di indagine.
Inoltre, l'efficienza variabile della differenziazione (in diversi esperimenti e in diLinee hPSC fferenti) potrebbero essere osservate 39 , 40 , 41 , 42 , 43 , 44 . Le strategie di arricchimento dei cardiomiociti basate sull'espressione di proteine superficiali 35 , 45 (mediante selezione delle cellule assistite da florescenza o mediante selezione del tallone magnetico 46 , 47 ) e la selezione metabolica 44 , 48 possono rappresentare strategie valide che possono essere applicate a qualsiasi (geneticamente modificato o Non modificata) hPSC-line prima del plating degli hPSC-CM, per migliorare il segnale elettrico.
Sebbene gli hPSC-CM siano notoriamente immaturi rispetto ai cardiomiociti adulti umani 4 , 49 , essi hanno dimostrato di essere preziosi in rEcapitolando e identificando cambiamenti specifici correlati alle malattie ( es . Nelle cancopatie) 19 , 20 , 50 e risposte indotte da farmaci ( ad esempio bloccanti del canale cardiaco) 4 , 51 . Inoltre, le cellule immature sono più facili da dissociare e recuperano meglio dei cardiomiociti adulti dopo dissociazione e placcatura 44, quindi, l'immaturietà hPSC-CM può essere ricompensata come un vantaggio a questo proposito. Tuttavia, per poter ricapitolare ad es . Le malattie cardiache in ritardo di insorgenza e riprodurre fedelmente le risposte di farmaci dei cardiomiociti adulti dovrebbero essere ottenuti un stato hPSC-CM più meccanico, metabolico e elettrico più maturo. I metodi per maturare queste cellule includono tempo prolungato nella coltura 52 , ceppo meccanico 53 , stimolazione elettrica 54 , aggiunta di piccoleMolecole 55 , coltura 3D 56 , co-coltura con altri tipi di cellule 57 e anche una combinazione di questi approcci 58 ; Finora, nessuno di questi approcci ha portato ad un fenotipo adulto.
Come parte delle caratteristiche di immaturity, hPSC-CMs mostrano automaticità elettrica. Qui vengono forniti i dettagli su come quantificare con precisione gli intervalli QT e RR. Una limitazione della misurazione dell'attività elettrica spontanea è che il confronto tra gli intervalli QT può essere difficile quando le hPSC-CM mostrano diverse frequenze di battitura. In questo caso, le formule di Bazett o Fridericia possono essere utilizzate per correggere l'intervallo QT per la frequenza. Tuttavia, come indicato in precedenza 30 , consigliamo di eseguire un'analisi di regressione di Axis Major plutando intervallo QT rispetto all'intervallo RR per i dati grezzi e corretti, per escludere eventuali pregiudizi dovuti al metodo di correzione stesso.
Il protocollo qui presentato, unitamente ai metodi descritti in precedenza 59 , 60, aiuta la standardizzazione delle procedure e l'analisi dei processori FP hPSC-CM, migliorando la riproducibilità dei dati e consentendo un migliore confronto dei risultati interlaboratorio.
Divulgazioni
CLM è co-fondatore e consulente di Pluriomics bv. Una parte dei costi di pubblicazione è stata coperta da Multi Channel Systems.
Riconoscimenti
Questo lavoro è stato sostenuto dalle seguenti sovvenzioni: CVON (HUSTCARE): l'Olanda Iniziativa di Ricerca CardioVascolare (la Fondazione olandese di cuore, la Federazione olandese dei centri medici universitari, l'Organizzazione olandese per la ricerca e lo sviluppo sanitario e la Royal Netherlands Academy of Sciences); Il Consiglio europeo della ricerca (ERCAdG 323182 STEMCARDIOVASC). Ringraziamo E. Giacomelli (LUMC) per aiutare con la differenziazione cardiaca hPSC.
Materiali
| Name | Company | Catalog Number | Comments |
| Biological safety cabinet/laminar flow-hood | Cleanair | ||
| MEA2100 in vitro recording system | Multi Channel Systems | ||
| CO2 cell-culture incubator | Sanyo | MCO-15A | |
| Centrifuge | Hitachi | himac-CT6EL | |
| Leica stereomicroscope | Leica Microsystems | MS5 | |
| Handheld pipetman (P-10 (10 μL), P-200 (200 μL), P-1000 (1,000 μL)) | Gilson International | ||
| Filter tips (10 μL, 200μL, 1,000μL) | Corning | 4807 (10 μL), 4810 (200μL), 4809 (1000μL) | |
| Disposable bottle top filter (0.22 μm pore size) | Millipore | SCGVU02RE | |
| Sterile plastic pipette | Greiner Bio-One | 606180 (5 mL), 607180 (10 mL), 760180 (25 mL) | |
| Tweezers | Dumont | ||
| Autoclavable Petri dishes | VWR/ Duran Group | 391-0860 | |
| MEA chip | Multi Channel Systems | MEA200/30iR-Ti-gr | |
| Phosphate-buffered Saline (PBS) calcium, magnesium | Thermo Fisher Scientific | 14040-091 | |
| Phosphate-Buffered Saline (PBS) no calcium, no magnesium | Thermo Fisher Scientific | 14190-169 | |
| Human recombinant fibronectin | Tebu-Bio | J64560 | |
| Custom-made polytetrafluoroethylene (PTFE) MEA rings | LUMC: department of Instrument Development | ||
| Dissociation enzyme - TrypLE Select 1x | Thermo Fisher Scientific | 12563-029 | |
| Tergazyme enzyme detergent | Sigma-Aldrich | Z273287-1EA | |
| Distilled Water | Thermo Fisher Scientific | 15230-089 | |
| Name | Company | Catalog Number | Comments |
| Reagents for LI-BPEL medium | |||
| Iscove's Modified Dulbecco's Medium (IMDM) | Thermo Fisher Scientific | 21056-023 | |
| F12 nutrient mixture (Ham) | Thermo Fisher Scientific | 31765-027 | |
| Ascorbic Acid 2-phosphate | Sigma-Aldrich | A8960 | |
| Glutamax | Thermo Fisher Scientific | 35050-038 | |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15070-063 | |
| Phenol Red | Sigma-Aldrich | P3532 | |
| Protein-free Hybridoma Medium-II (PFHMII) | Thermo Fisher Scientific | 12040-077 | |
| Bovine Serum Albumin (BSA) | Bovogen Biologicals Australia | BSAS05 | |
| Poly(Vinyl Alcohol) (PVA) | Sigma-Aldrich | P8136 | |
| Chemically-defined Lipid Concentrate (CDLC) | Thermo Fisher Scientific | 11905-031 | |
| Insulin-Transferrin-Selenium-Ethanolamine (ITS-X) 100x | Thermo Fisher Scientific | 51599-056 | |
| α-Monothioglycerol | Sigma-Aldrich | M6145 | |
| Name | Company | Software version | Comments |
| MC_Rack | Multi Channel Systems | 4.6.2 | Alternatively, data can be recorded using Cardio2D or MC_Experimenter (MultiChannel Systems) |
| TCX Control | Multi Channel Systems | 1.3.4 | |
| MEA Select | Multi Channel Systems | 1.3.0 | |
| MC_Data Tool | Multi Channel Systems | 2.6.15 | Alternatively, Multi Channel Data Manager (MultiChannel Systems) can be used when custom data export is required (HDF5, EDF, etc.) |
| Clampfit | Molecular Devices | 7.0.0 | Used in step 10.1 for analyzing, graphing, and formatting of all of data. To use Clampfit, download and install the electrophysiology data acquisition and pClam (latest version, 10.7.0), available on the Molecular Devices Website. Once complete, launch the software Clampfit. Alternatively, data can be analysed using Cardio2D (MultiChannel Systems) or MatLab custom code. |
Riferimenti
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Thomson, J. A., et al. Embryonic stem cell lines derived from human blastocysts. Science. 282 (5391), 1145-1147 (1998).
- Birket, M. J., et al. Expansion and patterning of cardiovascular progenitors derived from human pluripotent stem cells. Nat Biotechnol. 33 (9), 970-979 (2015).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochim Biophys Acta. 1863 (7 Pt B), 1728-1748 (2016).
- Devalla, H. D., et al. Atrial-like cardiomyocytes from human pluripotent stem cells are a robust preclinical model for assessing atrial-selective pharmacology. EMBO Mol Med. 7 (4), 394-410 (2015).
- Lewandowski, J., Kolanowski, T. J., Kurpisz, M. Techniques for the induction of human pluripotent stem cell differentiation towards cardiomyocytes. J Tissue Eng Regen Med. , (2016).
- Mummery, C. L., Zhang, J., Ng, E. S., Elliott, D. A., Elefanty, A. G., Kamp, T. J. Differentiation of human embryonic stem cells and induced pluripotent stem cells to cardiomyocytes: a methods overview. Circ Res. 111 (3), 344-358 (2012).
- Davies, M. P., An, R. H., Doevendans, P., Kubalak, S., Chien, K. R., Kass, R. S. Developmental changes in ionic channel activity in the embryonic murine heart. Circ Res. 78 (1), 15-25 (1996).
- Bellin, M., et al. Isogenic human pluripotent stem cell pairs reveal the role of a KCNH2 mutation in long-QT syndrome. EMBO J. 32 (24), 3161-3175 (2013).
- Moretti, A., et al. Patient-specific induced pluripotent stem-cell models for long-QT syndrome. N Engl J Med. 363 (15), 1397-1409 (2010).
- Ma, D., et al. Modeling type 3 long QT syndrome with cardiomyocytes derived from patient-specific induced pluripotent stem cells. Int J Cardiol. 168 (6), 5277-5286 (2013).
- Yazawa, M., et al. Using induced pluripotent stem cells to investigate cardiac phenotypes in Timothy syndrome. Nature. 471 (7337), 230-U120 (2011).
- Limpitikul, W. B., et al. A Precision Medicine Approach to the Rescue of Function on Malignant Calmodulinopathic Long QT Syndrome. Circ Res. , (2016).
- Liang, P., et al. Patient-Specific and Genome-Edited Induced Pluripotent Stem Cell-Derived Cardiomyocytes Elucidate Single-Cell Phenotype of Brugada Syndrome. J Am Coll Cardiol. 68 (19), 2086-2096 (2016).
- Jung, C. B., et al. Dantrolene rescues arrhythmogenic RYR2 defect in a patient-specific stem cell model of catecholaminergic polymorphic ventricular tachycardia. EMBO Mol Med. 4 (3), 180-191 (2012).
- Novak, A., et al. Cardiomyocytes generated from CPVTD307H patients are arrhythmogenic in response to beta-adrenergic stimulation. J Cell Mol Med. 16 (3), 468-482 (2012).
- Bellin, M., Mummery, C. L. Inherited heart disease - what can we expect from the second decade of human iPS cell research. FEBS Lett. 590 (15), 2482-2493 (2016).
- Sallam, K., Li, Y., Sager, P. T., Houser, S. R., Wu, J. C. Finding the rhythm of sudden cardiac death: new opportunities using induced pluripotent stem cell-derived cardiomyocytes. Circ Res. 116 (12), 1989-2004 (2015).
- Sinnecker, D., Goedel, A., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes: a versatile tool for arrhythmia research. Circ Res. 112 (6), 961-968 (2013).
- Bellin, M., Marchetto, M. C., Gage, F. H., Mummery, C. L. Induced pluripotent stem cells: the new patient?. Nat Rev Mol Cell Biol. 13 (11), 713-726 (2012).
- Sinnecker, D., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes for drug development and toxicity testing. Pharmacol Ther. 143 (2), 246-252 (2014).
- Terrenoire, C., et al. Induced pluripotent stem cells used to reveal drug actions in a long QT syndrome family with complex genetics. J Gen Physiol. 141 (1), 61-72 (2013).
- Abi-Gerges, N., et al. Assessment of extracellular field potential and Ca2+ transient signals for early QT/pro-arrhythmia detection using human induced pluripotent stem cell-derived cardiomyocytes. J Pharmacol Toxicol Methods. 83, 1-15 (2016).
- Del Alamo, J. C., et al. High throughput physiological screening of iPSC-derived cardiomyocytes for drug development. Biochim Biophys Acta. 1863 (7 Pt B), 1717-1727 (2016).
- Blinova, K., et al. Comprehensive Translational Assessment of Human Induced Pluripotent Stem Cell Derived Cardiomyocytes for Evaluating Drug-Induced Arrhythmias. Toxicol Sci. , (2016).
- Nerbonne, J. M. Studying cardiac arrhythmias in the mouse--a reasonable model for probing mechanisms?. Trends Cardiovasc Med. 14 (3), 83-93 (2004).
- Halbach, M., Egert, U., Hescheler, J., Banach, K. Estimation of action potential changes from field potential recordings in multicellular mouse cardiac myocyte cultures. Cell Physiol Biochem. 13 (5), 271-284 (2003).
- Tertoolen, L. G., Braam, S. R., van Meer, B. J., Passier, R., Mummery, C. L. Interpretation of field potentials measured on a multi electrode array in pharmacological toxicity screening on primary and human pluripotent stem cell-derived cardiomyocytes. Biochem Biophys Res Commun. , (2017).
- Rajamohan, D., et al. Automated Electrophysiological and Pharmacological Evaluation of Human Pluripotent Stem Cell-Derived Cardiomyocytes. Stem Cells Dev. 25 (6), 439-452 (2016).
- Sala, L., et al. A new hERG allosteric modulator rescues genetic and drug-induced long-QT syndrome phenotypes in cardiomyocytes from isogenic pairs of patient induced pluripotent stem cells. EMBO Mol Med. 8 (9), 1065-1081 (2016).
- Cavero, I., Guillon, J. M., Ballet, V., Clements, M., Gerbeau, J. F., Holzgrefe, H. Comprehensive in vitro Proarrhythmia Assay (CiPA): Pending issues for successful validation and implementation. J Pharmacol Toxicol Methods. , (2016).
- Sala, L., Bellin, M., Mummery, C. L. Integrating cardiomyocytes from human pluripotent stem cells in safety pharmacology: has the time come?. Br J Pharmacol. , (2016).
- Chen, I. Y., Matsa, E., Wu, J. C. Induced pluripotent stem cells: at the heart of cardiovascular precision medicine. Nat Rev Cardiol. 13 (6), 333-349 (2016).
- Dambrot, C., et al. Strategies for rapidly mapping proviral integration sites and assessing cardiogenic potential of nascent human induced pluripotent stem cell clones. Exp Cell Res. 327 (2), 297-306 (2014).
- Elliott, D. A., et al. NKX2-5(eGFP/w) hESCs for isolation of human cardiac progenitors and cardiomyocytes. Nat Methods. 8 (12), 1037-1040 (2011).
- Ng, E. S., Davis, R., Stanley, E. G., Elefanty, A. G. A protocol describing the use of a recombinant protein-based, animal product-free medium (APEL) for human embryonic stem cell differentiation as spin embryoid bodies. Nat Protoc. 3 (5), 768-776 (2008).
- Karakikes, I., et al. Small molecule-mediated directed differentiation of human embryonic stem cells toward ventricular cardiomyocytes. Stem Cells Transl Med. 3 (1), 18-31 (2014).
- Zhang, Q., et al. Direct differentiation of atrial and ventricular myocytes from human embryonic stem cells by alternating retinoid signals. Cell Res. 21 (4), 579-587 (2011).
- Mummery, C., et al. Differentiation of human embryonic stem cells to cardiomyocytes: role of coculture with visceral endoderm-like cells. Circulation. 107 (21), 2733-2740 (2003).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Zhang, J., et al. Extracellular matrix promotes highly efficient cardiac differentiation of human pluripotent stem cells: the matrix sandwich method. Circ Res. 111 (9), 1125-1136 (2012).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/beta-catenin signaling under fully defined conditions. Nat Protoc. 8 (1), 162-175 (2013).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Dubois, N. C., et al. SIRPA is a specific cell-surface marker for isolating cardiomyocytes derived from human pluripotent stem cells. Nat Biotechnol. 29 (11), 1011-1018 (2011).
- Fuerstenau-Sharp, M., et al. Generation of highly purified human cardiomyocytes from peripheral blood mononuclear cell-derived induced pluripotent stem cells. PLoS One. 10 (5), e0126596 (2015).
- Schwach, V., Passier, R. Generation and purification of human stem cell-derived cardiomyocytes. Differentiation. 91 (4-5), 126-138 (2016).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
- Veerman, C. C., Kosmidis, G., Mummery, C. L., Casini, S., Verkerk, A. O., Bellin, M. Immaturity of human stem-cell-derived cardiomyocytes in culture: fatal flaw or soluble problem?. Stem Cells Dev. 24 (9), 1035-1052 (2015).
- Karakikes, I., Ameen, M., Termglinchan, V., Wu, J. C. Human induced pluripotent stem cell-derived cardiomyocytes: insights into molecular, cellular, and functional phenotypes. Circ Res. 117 (1), 80-88 (2015).
- Avior, Y., Sagi, I., Benvenisty, N. Pluripotent stem cells in disease modelling and drug discovery. Nat Rev Mol Cell Biol. 17 (3), 170-182 (2016).
- Otsuji, T. G., Minami, I., Kurose, Y., Yamauchi, K., Tada, M., Nakatsuji, N. Progressive maturation in contracting cardiomyocytes derived from human embryonic stem cells: Qualitative effects on electrophysiological responses to drugs. Stem Cell Res. 4 (3), 201-213 (2010).
- Mihic, A., et al. The effect of cyclic stretch on maturation and 3D tissue formation of human embryonic stem cell-derived cardiomyocytes. Biomaterials. 35 (9), 2798-2808 (2014).
- Lieu, D. K., et al. Mechanism-based facilitated maturation of human pluripotent stem cell-derived cardiomyocytes. Circ Arrhythm Electrophysiol. 6 (1), 191-201 (2013).
- Yang, X., et al. Tri-iodo-l-thyronine promotes the maturation of human cardiomyocytes-derived from induced pluripotent stem cells. J Mol Cell Cardiol. 72, 296-304 (2014).
- Mannhardt, I., et al. Human Engineered Heart Tissue: Analysis of Contractile Force. Stem Cell Reports. , (2016).
- Kim, C., et al. Non-cardiomyocytes influence the electrophysiological maturation of human embryonic stem cell-derived cardiomyocytes during differentiation. Stem Cells Dev. 19 (6), 783-795 (2010).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat Methods. 10 (8), 781-787 (2013).
- Clements, M. Multielectrode Array (MEA) Assay for Profiling Electrophysiological Drug Effects in Human Stem Cell-Derived Cardiomyocytes. Curr Protoc Toxicol. 68 (22), 1-22 (2016).
- Harris, K. A Human Induced Pluripotent Stem Cell-Derived Cardiomyocyte (hiPSC-CM) Multielectrode Array Assay for Preclinical Cardiac Electrophysiology Safety Screening. Curr Protoc Pharmacol. 71, 1-15 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon