Method Article
Analyse électrophysiologique des cardiomyocytes à base de cellules souches pluripotentes humaines (HPSC-CM) utilisant des matrices multi-électrodes (MEA)
Dans cet article
Résumé
La caractérisation électrophysiologique des cardiomyocytes dérivés de cellules souches pluripotentes humaines (CMH-CM) est cruciale pour la modélisation de la maladie cardiaque et pour la détermination des réponses aux médicaments. Ce protocole fournit les informations nécessaires pour dissocier et plaquer les CMH-CM sur les réseaux multi-électrodes, mesurer leur potentiel de terrain et une méthode pour analyser les intervalles QT et RR.
Résumé
Les cardiomyocytes peuvent maintenant être dérivés avec une efficacité élevée à la fois des cellules souches pluripotentes induites par l'embryon humain et humain (HPSC). Les cardiomyocytes dérivés du HPSC (CMH-CM) sont de plus en plus reconnus comme ayant une grande valeur pour la modélisation des maladies cardiovasculaires chez les humains, en particulier les syndromes d'arythmie. Ils ont également démontré leur pertinence en tant que systèmes in vitro pour prédire les réponses aux médicaments, ce qui les rend potentiellement utiles pour le dépistage et la découverte de médicaments, la pharmacologie de sécurité et éventuellement pour la médecine personnalisée. Cela serait facilité en dérivant les CMH-CM de patients ou d'individus sensibles en tant que hiPSC. Cependant, pour toutes les applications, une mesure et une analyse précises des propriétés électriques du système hPSC-CM sont essentielles pour identifier les changements dus aux mutations des canaux ioniques cardiaques et / ou des médicaments qui ciblent les canaux ioniques et peuvent provoquer une mort cardiaque soudaine. Par rapport aux branchements manuels, les appareils multi-électrodes (MEA) offrent l'avantage dePermettant des enregistrements de débit moyen à élevé. Ce protocole décrit comment dissocier les cultures de cellules 2D de CMH-CM à de petits agrégats et des cellules individuelles et les plaquer sur des AME pour enregistrer leur activité électrique spontanée en tant que potentiel de terrain. Les méthodes d'analyse des données enregistrées pour extraire des paramètres spécifiques, comme les intervalles QT et RR, sont également décrites ici. Des changements dans ces paramètres seraient attendus dans les CMH-CM portant des mutations responsables des arythmies cardiaques et après l'ajout de médicaments spécifiques, permettant de détecter ceux qui présentent un risque cardiotoxique.
Introduction
Les cellules souches pluripotentes humaines (HPSC) ont la capacité de se renouveler automatiquement et de générer pratiquement n'importe quel type de cellule du corps humain par la différenciation 1 , 2 . Des protocoles détaillés sur la façon de diriger la différenciation des HPSC en plusieurs lignées cardiaques (cardiomyocytes ventriculaires, auriculaires, stimulateurs cardiaques) ont été décrits 3 , 4 , 5 , 6 , 7 . Les cardiomyocytes sont des cellules électriquement actives et une connaissance approfondie de leur activité électrophysiologique peut être extrêmement instructive pour comprendre le développement du cœur et la maladie 8 . Les cardiomyocytes dérivés du hiPSC spécifiques au patient (hiPSC-CM) ont été utilisés avec succès pour modéliser et étudier les caractéristiques cellulaires, moléculaires et électriques de plusieurs arythmies cardiaques, y compris le syndrome de QT long(LQTS) 9 , 10 , 11 , 12 , 13 , syndrome de Brugada 14 et tachycardie ventriculaire polymorphe cathecolaminergique 15 , 16 . En outre, de multiples médicaments ont été ajoutés aux hiPSC-CM malades pour récapituler l'intervention thérapeutique et pour sauver les phénotypes pathologiques cellulaires 10 , 15 , 20 , 21 , 22 . Plus récemment, des plates-formes de dépistage basées sur WT hiPSC-CM ont été développées, en réponse à la nécessité de systèmes humains aux premières phases de la découverte de médicaments 23 , 24 , 25 car les cardiomyocytes de rongeurs diffèrent profondément de huHomme dans l'expression des canaux ioniques et la biophysique 26 .
À cette fin, des technologies adaptées à une application à moyen et à haut débit sont en cours de développement et de mise en œuvre. Il s'agit notamment des enregistrements optiques du potentiel de la membrane, des transitoires de Ca 2+ et de la contrainte, des mesures d'impédance (comme mesure indirecte de la contractilité cellulaire) et des mesures du potentiel du champ extracellulaire (FP) (pour examen, voir référence 24 ). Les appareils multi-électrodes (MEA) permettent d'enregistrer les signaux de forme d'onde (ou FP) générés et façonnés par des monocouches ou de petits groupes de cardiomyocytes. Le contour FP est en corrélation avec le potentiel d'action cardiaque et, dans une certaine mesure, avec les enregistrements d'électrocardiogramme (ECG) 27 ; Ils montrent généralement une augmentation rapide rapide correspondant à l'afflux de Na + et à la dépolarisation de la membrane (pic R / Q), une phase d'onde lente / plateau probablement correspondant au Ca2+ afflux et une phase de repolarisation correspondant à un effluent K + prédominant (pic T). La perturbation de la forme d'onde FP peut être corrélée avec les changements dans les phases potentielles d'action 28 .
Bien que les enregistrements de pinces de patch des potentiels d'action puissent être plus informatifs, en particulier pour les paramètres comme vitesse de descente et potentiel de membrane de repos, les mesures manuelles ne sont pas réalisables pour des expériences à échelle moyenne et élevée, alors que la pince à patch automatisée n'a été appliquée que récemment à hPSC -CM 29 . Cependant, étant donné que les enregistrements prolongés sur les AME permettent une exposition aiguë et chronique aux composés à étudier, il est maintenant possible d'utiliser les plates-formes HPSC-CM pour le dépistage des médicaments, la découverte 24 , 30 et la pharmacologie de sécurité 31 , 32 . Cela garantit la future précision ou la persoMédecine nalentielle 33 .
Le but de ce protocole est de fournir les informations nécessaires pour dissocier et déposer les hPSC-CM sur les puces MEA et mesurer leur FP. Dans cette procédure, chaque étape a été optimisée, assurant une survie et une récupération optimales des cellules après la dissociation, une fixation optimale des cellules à la plaque MEA et une analyse et une quantification normalisées des paramètres. En particulier, la procédure d'enregistrement FP extracellulaire, l'analyse des intervalles QT et RR et l'évaluation des effets des médicaments sont expliquées et illustrées.
Protocole
1. Préparation des solutions et des réactifs
- Préparer un milieu de culture HPSC-CM en utilisant de l'insuline faible, de l'albumine de sérum bovin, de l'alcool polyvinylique, des lipides essentiels (LI-BPEL) 34 , 35 , 36 en combinant les réactifs décrits dans le tableau 1 . Filtrer le milieu à travers un filtre à pores de 0,22 μm et conserver à 4 ° C pendant 2 semaines.
- Préparer une solution mère de fibronectine recombinante humaine en reconstituant 1 mg de fibronectine recombinante humaine dans 5 ml d'eau distillée stérile pour atteindre une concentration de 200 μg / mL, une aliquote et conserver à -80 ° C.
- Préparez une solution de détergent enzymatique à 1% (p / v) (voir le tableau des matériaux ), pour aider à éliminer les cellules résiduelles de la puce de microarray en dissolvant 1 g de poudre détergente enzymatique dans 100 ml de chaleur (~ 40-45 ° C) eau déminéralisée. Laisser refroidir la solution et la conserver à 4 ° C fOu jusqu'à un an.
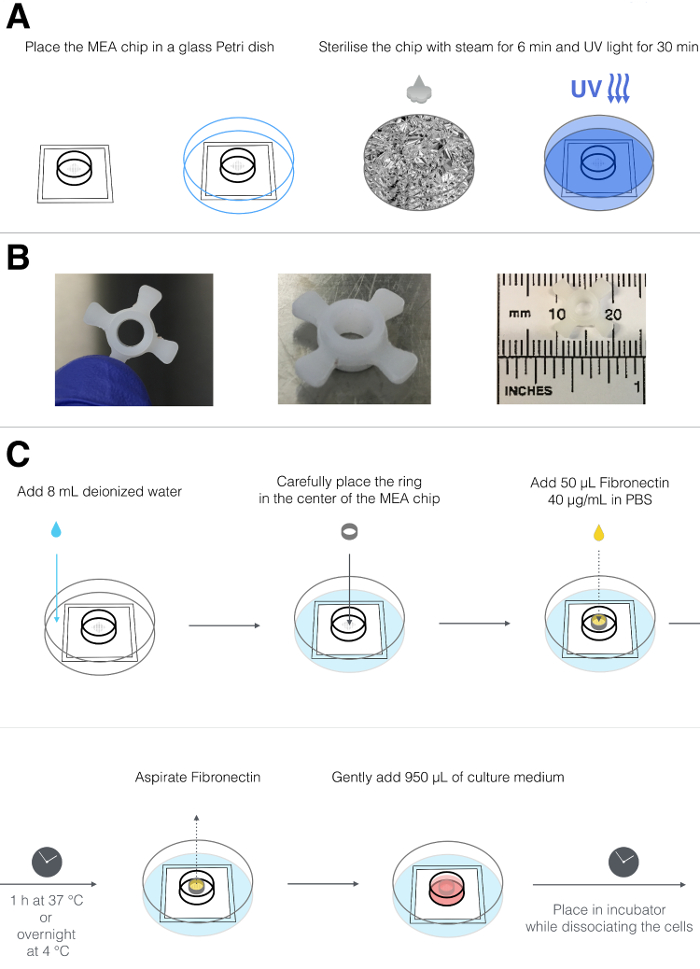
2. Stérilisation des chips MEA (figure 1A)
REMARQUE: plusieurs configurations différentes des MEA sont disponibles, avec des formats à un ou plusieurs puits. Le protocole décrit ici utilise le MEA à une seule chambre contenant 60 électrodes d'enregistrement dans un agencement de grille 8 x 8 (voir tableau de matériaux ). Le diamètre de l'électrode est de 30 μm et la distance entre les électrodes est de 200 μm. Une électrode de référence est également présente.
- Rincer soigneusement la puce avec de l'eau désionisée.
- Mettez les chips MEA dans une boîte en Pétri en verre qui peut être autoclavée. Enrouler le plat dans du papier d'aluminium.
- Stériliser les copeaux dans une autocuiseuse de laboratoire pendant 6 min. Laisser refroidir le plats avant de l'ouvrir.
NOTE: En variante, les puces peuvent être stérilisées en les immergeant dans 1 ml d'éthanol à 80% (v / v) à la température ambiante pendant 15 à 30 minutes. - Placez les copeaux dans un capot de culture cellulaire et exposez leSurface à la lumière UV pendant environ 30 min.
3. Revêtement des chips MEA (Figure 1B et 1C)
- Placez les puces propres dans une boîte de Petri stérile stéréo standard de 10 cm Ø.
- Ajouter 8 ml d'eau distillée stérile dans la boîte de Petri pour former une chambre humidifiée, ce qui empêchera le petit volume de milieu de culture sur la puce de sécher lorsqu'il est placé dans l'incubateur.
- Utilisez des anneaux en polytétrafluoroéthylène (PTFE) sur mesure ( Figure 1B ) pour assurer le placage des cardiomyocytes au centre de la puce, où se trouve le réseau d'électrodes. (Les anneaux de PTFE sont précédemment stockés dans 80% d'éthanol). Dans le capot de culture, retirer les anneaux de l'éthanol, les placer dans une boîte de Petri stérile sans couvercle et laisser sécher les anneaux dans le capot.
- Placez une bague sèche à l'intérieur d'une puce MEA à l'aide de pinces stérilisées à la flamme ( figure 1C ).
REMARQUE: Utilisez également 80% d'éthanol pour laver la pince et laisser sécher iDans le capot de culture des tissus avant de les utiliser. - Décongeler une aliquote de stock de fibronectine recombinante humaine (200 μg / mL dans de l'eau distillée) et diluer dans PBS avec Ca 2+ et Mg 2+ pour obtenir une solution de travail de 40 μg / mL. Enduire les électrodes en ajoutant 50 μL de 40 μg / mL de fibronectine à l'intérieur du cycle.
REMARQUE: Le revêtement utilisant une fibronectine recombinante humaine garantit une fixation optimale de l'hPSC-CM. Cependant, d'autres types de protéines de revêtement telles que la fibronectine bovine ou les mélanges de protéines de matrice extracellulaire peuvent être utilisés. - Fermez le couvercle de la boîte en Pétri plastique de 10 cm de diamètre et transférez soigneusement le plat contenant la puce MEA dans l'incubateur. Incuber à 37 ° C pendant au moins 1 h, ou à 4 ° CO / N.
- Transférer le plat contenant la puce MEA au capot de culture cellulaire. Avant d'utiliser la puce MEA, aspirer les 50 μL de fibronectine à l'aide d'une pipette P200 ou d'un système à vide dans le capot, sans déplacer l'anneau en PTFE. Use plaPour cette étape. Assurez-vous qu'aucun objet solide ( p. Ex., Pointes de pipette) ne touche l'intérieur du plat car cela peut endommager les électrodes.
REMARQUE: Cela prolongera la durée de vie des chips MEA. - Ajouter doucement 950 μL de milieu LI-BPEL (voir Tableau 1 et Tableau des matériaux ), en veillant à ce qu'il soit uniformément réparti et que l'anneau ne flotte pas. Retournez le plat à l'incubateur.
4. HPSC-CMs Dissociation and Plating (Figure 2)
REMARQUE: Le protocole décrit ici utilise des CMH-CM qui ont été différenciés dans une culture monocouche à l'aide de cytokines 34 à ~ 18 jours après le début de la différenciation. Cependant, il a été prouvé qu'il convient à toute culture HDPS et CM 2D et 3D. Lorsque l'on utilise des cultures différenciées à des moments antérieurs ou postérieurs, l'ajustement du temps d'incubation de l'enzyme de dissociation (voir le tableau des matériaux ) peut être néCessionnaire. Les volumes suivants sont destinés à un puit unique d'un format de plaque à 12 puits (3,8 cm 2 ).
- Aspirez bien le milieu de la culture de HPSC-CM.
- En travaillant sous un capot de culture tissulaire, ajouter 1-2 ml / puits de PBS sans Ca 2+ / Mg 2+ pour laver la culture. Aspirez le PBS.
- Ajouter 500 μl / puits de l'enzyme de dissociation. Incuber pendant 5 min à 37 ° C.
- Ajouter 1 mL de LI-BPEL / puits pour diluer l'enzyme. Détachez doucement la monocouche de hPSC-CM en grattant doucement à l'aide d'une pipette P1000. Recueillir la suspension cellulaire dans un tube de 15 ml.
- Rincer le puits avec 1 mL de LI-BPEL pour collecter toutes les cellules et les cellules restantes.
- Ajouter un autre 2-3 mL de LI-BPEL pour atteindre un volume final de 5-6 mL et faire pipeter doucement vers le haut et vers le bas 3-5x avec une pipette de 5 mL pour dissocier les touffes cellulaires.
NOTE: La dissociation en cellules simples à ce stade n'est pas nécessaire car elle affectera la survie cellulaire. La présence de petits grappesAssurer une plus grande viabilité cellulaire. - Centrifuger les cellules à la RT pendant 3 min à 300 x g.
- Retirer le surnageant, en essayant d'enlever la majeure partie du surplus de liquides, mais sans enlever la pastille cellulaire.
- Remettre en suspension la pastille cellulaire dans 250 μL de LI-BPEL (en utilisant un P1000 et pipeter extrêmement doucement).
- Distribuer ~ 50 μL de suspension cellulaire par MEA (jusqu'à 5 AME peuvent être préparés à partir d'un puits d'une plaque à 12 puits) en pipettant la suspension de cellules directement dans le centre de l'anneau en PTFE, sur le dessus de l'électrode.
REMARQUE: à ce stade, les cellules sont difficiles à compter en raison de la présence de grappes cellulaires et le nombre total de cellules par MEA peut sensiblement varier, selon la source de HPSC-CM utilisée. Pendant le placage, assurez-vous que le nuage de cellules dissociées recouvre la surface des électrodes. - Transférer soigneusement les MEA à l'incubateur à 37 ° C et permettre aux cellules de fixer O / N
5. Suppression de l'anneau et Medium ReFractionnement (Figure 3)
- Au 1 jour après le placage, retirer soigneusement l'anneau dans un environnement stérile en utilisant des pinces stériles ( Figure 3A-3B ).
- Rincer l'anneau dans de l'éthanol à 80% (v / v) et le ranger dans un tube de 50 ml contenant de l'éthanol à 80% (v / v).
- Retirez doucement 500 μL de milieu de la puce MEA et ajoutez 500 μL de LI-BPEL frais.
- Transférer les MEA à l'incubateur à 37 ° C.
REMARQUE: les cellules devraient commencer à battre de 1 à 7 jours après l'élimination de l'anneau et le changement moyen ( figure 3C ). - Mesurez l'activité électrique des CMH-CM sur les AME 1 à 7 jours après l'élimination du cycle.
6. Vérifier la qualité du signal (Figure 4)
- Allumez l'ordinateur et lancez la suite logicielle liée à la configuration MEA: TCX-Control, MC_MEA Select et MC_Rack. Réglez la température à 37 ° C dans TCX-Control pour enregistrer les mesures à la température physiologique.
- Retirer le plat contenant la puce MEA fRom de l'incubateur. Ouvrez le couvercle, sortez la puce MEA et placez-la sur un tissu pour absorber l'eau résiduelle.
- Essuyez soigneusement les contacts externes de la plaque avec un tissu et nettoyez-les à l'aide d'un coton-tige humidifié avec de l'éthanol à 100% (v / v) pour éliminer toute eau résiduelle ou débris, ce qui pourrait provoquer un bruit de signal.
- Transférez la plaque MEA à la tête d'enregistrement chauffée (37 ° C) pour détecter l'activité spontanée ( p. Ex., Matériel: voir la table des matières , logiciel: MC_Rack).
- Ouvrez MC_Rack: Cliquez sur 'Modifier' → 'Ajouter MC_Card' pour créer un nouveau protocole. Utilisez le menu déroulant 'Modifier' pour ajouter différents enregistreurs et fenêtres d'affichage au protocole.
REMARQUE: une fréquence d'échantillonnage d'au moins 10 kHz est recommandée. Le protocole que nous utilisons contient un outil d'affichage à long terme, avec une disposition complète de la puce MEA complète, et un classificateur Spike, essentiel pour capturer l'effet de drogue iEn temps réel. The Spike Cutout est syntonisé avec un 'Pre Trigger' de 20 ms, un 'Post Trigger' de 800 ms et un 'Dead Time' de 2 ms. Le protocole peut être enregistré sous le fichier '.rck' et rechargé avant de commencer les expériences.
- Ouvrez MC_Rack: Cliquez sur 'Modifier' → 'Ajouter MC_Card' pour créer un nouveau protocole. Utilisez le menu déroulant 'Modifier' pour ajouter différents enregistreurs et fenêtres d'affichage au protocole.
- Démarrez le protocole en mode lecture en cliquant sur le bouton 'play'.
REMARQUE: à ce stade, il est possible de recharger le protocole enregistré dans l'étape 6.5. Dans MC_Rack, chargez le protocole (extension du fichier .rck) en cliquant sur 'Fichier' → 'Ouvrir'. - Si les signaux affichent des pics R et des pics T clairement visibles, attendez 10 à 15 minutes pour conclure la phase d'adaptation. Des exemples de traces de bonne et de mauvaise qualité sont présentés à la figure 4 .
REMARQUE: Si aucun pic T ne peut être détecté dans aucune des électrodes, ne procédez pas à l'expérience. Habituellement, cela résulte d'une mauvaise activité électrique des CMH-CM ou d'une faible fixation des cellules aux électrodes.
7. Démarrez ExPeriment et enregistrement
- Cliquez sur "enregistrer", puis sur "jouer", et acquerrez des données pendant 10 min dans les conditions de base pour déterminer l'état stationnaire. Annotez les électrodes qui ont le meilleur signal afin qu'elles puissent être facilement identifiées et exportées plus tard pour l'analyse.
- Pour l'évaluation de la réponse au médicament, ajouter des concentrations croissantes de médicament à toutes les 10 minutes. Par exemple, ajoutez le bloqueur HERG E4031 à une concentration finale de 1 μM. Pour cela, retirer 100 μL de milieu et ajouter le même volume de 10 μM E4031 dissous dans le milieu.
NOTE: Comme le montrent déjà Cavero et ses collègues 31 , un choix judicieux du volume dans lequel les médicaments sont dissous est important, car cela peut modifier profondément la courbe de réponse au médicament. - Répétez l'étape 7.2 pour toutes les autres concentrations de médicament intéressantes.
- Cliquez sur 'arrêter' pour conclure les enregistrements à la fin du protocole.
8.MEA Cleaning for Reuse
- Une fois l'enregistrement de l'expérience terminé, retirez doucement le support avec une pipette P1000. Ne pas toucher l'intérieur du plat car cela peut endommager les électrodes. Jeter selon les règles locales de sécurité.
- Rincez les chips MEA avec de l'eau désionisée à l'aide d'un flacon de lavage et répétez l'étape de lavage 3-4x.
REMARQUE: à ce stade, il n'est pas nécessaire que les cellules soient totalement détachées de la puce. - Ajouter 1 ml de solution détergente enzymatique 1% (v / v) dans chaque puits et incuber O / N à 4 ° C pour permettre le détachement cellulaire et l'élimination des cellules.
- Un jour plus tard, rincer soigneusement les pains MEA avec de l'eau désionisée pour éliminer la solution détergente enzymatique et les cellules résiduelles et ajouter 1 ml d'eau désionisée. Les puces MEA propres peuvent être stockées immergées dans de l'eau désionisée à 4 ° C.
9. Exportation de données
- Ouvrez le logiciel MC_Data Tool lié à la configuration MEA.
- Cliquez sur 'Fichier' → 'Ouvrir MCRÉ'.
- Cliquez sur 'Outils' → 'Convertir MCD en ABF' .
- Sélectionnez les électrodes avec les meilleurs signaux d'enregistrement à exporter pour l'analyse. Choisissez le répertoire dans lequel enregistrer les fichiers exportés et cliquez sur 'Enregistrer'. La procédure d'exportation peut prendre plusieurs minutes selon le nombre d'électrodes exportées et la taille totale des fichiers d'enregistrement.
10. Analyse des données
- Téléchargez et installez un programme d'acquisition et d'analyse de données en électrophysiologie ( par exemple, pClamp). Une fois terminé, lancez le logiciel d'analyse ( p. Ex., Clampfit).
- Calcul de l'intervalle RR (Figure 5).
- Placez deux curseurs verticaux pour définir la région d'intérêt sur la trace. Sélectionnez 'Détection d'événement' → 'Recherche par seuil'. Le curseur horizontal doit traverser tous les événements qui doivent être quantifiés, comme le montre la figure 5A .
- Cliquez &# 39; OK ', puis "Accepter la catégorie entière". Le logiciel recherchera ensuite la trace pour tous les événements appropriés. Des marques bleues seront placées au-dessus des événements.
- Réglez la sensibilité de la sélection automatique en réglant les paramètres dans la fenêtre de détection d'événement principal.
- Une fois que tous les événements ont été identifiés automatiquement, passez à la fenêtre de résultat (Fenêtre → Résultats) et copiez la colonne intitulée «Intervalle Intervalle», qui contient les données de fréquence ( Figure 5B ).
- Calcul de l'intervalle QT (figures 6-9):
- Placez un curseur juste avant et un après un seul FP à l'état d'équilibre. Sélectionnez 'Détection d'événement' → 'Créer modèle' ( Figure 6 ) .
- Vérifiez que le FP est correctement identifié et cliquez sur 'Ajouter'. Le modèle sera déplacé vers le bas du panneau.
- Enregistrez le modèle en tant que '.atf'fichier. De cette façon, une trace de modèle a été créée qui sera recherchée par le logiciel tout au long de l'enregistrement complet ( Figure 7 ). Créez un modèle pour chaque condition, car un effet de médicament pourrait modifier la forme du FP. Si nécessaire, filtrez légèrement la trace pour inclure un maximum de 10 000 points dans les deux curseurs.
- Une fois que le modèle a été enregistré avec l'extension '.atf', sélectionnez 'Détection d'événement' → 'Recherche de modèle' et chargez le modèle.
- Réglez le "seuil de concordat du modèle" pour identifier correctement tous les FP dans l'intervalle sélectionné.
- Une fois que tous les événements ont été correctement identifiés, enregistrez-les dans un nouveau fichier '.abf'.
- Ouvrez le fichier avec un logiciel d'analyse et calculez automatiquement le 'Time of Peak' pour les pics Q / R et T ( Figures 8, 9 ). Si les traces sont très bruyantes, appliquez un filtre. Calculez l'intervalle QT en soustrayant leValeur Q / R de la valeur T dans un éditeur de tableur de choix.
Résultats
Un jour après la dissociation et le placage, la couche de hPSC-CM sera visible sous la forme d'un film blanc et dense couvrant le centre de la chambre MEA ( Figure 3A ). Après le retrait de l'anneau ( Figure 3B ), La couche doit rester en place et l'inspection sur un microscope optique montrera les électrodes MEA couvertes par la couche (contractuelle) hPSC-CM ( Figure 3C ). En raison du couplage physique et électrique des cellules, une seule électrode (électrode d'or) sera utilisée pour l'analyse.
Alternativement, lorsque vous travaillez avec des structures 3D, telles que des corps embryoïdes ou des micro-tissus, ceux-ci peuvent être plaqués de manière à ce qu'ils soient physiquement et électriquement désaccouplés. L'inspection visuelle au microscope pourrait confirmer qu'aucune connexion physique entre les grappes et les ondes R non synchronisées aux MEA ne confirme aucun couplage électrique. Dans ce cLes électrodes indépendantes multiples peuvent être analysées.
Les enregistrements typiques des traces de FP sont présentés à la figure 4 . En particulier, une trace de bonne qualité peut être définie par la présence d'un pic clair correspondant à l'afflux de Na + et à la dépolarisation de la membrane (pic R / Q), une phase de repolarisation claire correspondant à l'efflux K + (T peak) et à un haut Rapport signal sur bruit ( Figure 4A , gauche: note l'échelle des y et Figure 4B ). Des traces de mauvaise qualité ( Figure 4A , milieu) peuvent résulter d'une défaillance des CMH-CM pour s'attacher à la plaque MEA ou à une faible activité électrique du système hPSC-CM. En attendant 1-3 jours pour une meilleure fixation peut améliorer le signal; Cependant, si aucune amélioration du signal n'est visible, il est recommandé d'exclure ce MEA des expériences. Les traces bruyantes ( Figure 4A , à droite) peuvent être analysées après le filtrage.
L'analyse réussie de l'intervalle RR peut être identifiée par l'inspection visuelle de l'écran montrant la détection maximale ( Figure 5A, 5B ). Dans l'intervalle de temps défini par les curseurs verticaux, des marques bleues correspondant à chaque pic devraient être présentes. Dans le cas où le programme n'identifie pas un ou plusieurs pics, essayez de déplacer le curseur horizontal et de lancer l'analyse à nouveau ou ajuster les paramètres de détection. De même, une analyse réussie de l'intervalle QT peut être identifiée par l'inspection visuelle de l'écran montrant la détection FP ( Figure 7 ). Dans l'intervalle de temps défini par les curseurs verticaux, des marques bleues correspondant à chaque détection FP doivent être présentes. Dans le cas où le programme n'identifie pas un ou plusieurs FP, essayez de redéfinir le modèle FP ( Figure 6 ) ou ajustez les paramètres de détection et exécutez l'analyse à nouveau.
Des traces individuelles Extra extra ou leur moyenne ( FigurE 8) peut être utilisé pour obtenir des valeurs d'intervalle QT avec des paramètres spécifiques comme dans la Figure 9 . L'analyse de la relation QT-RR est souhaitable et est significative dans les lignes HPSC malades et WT ( Figure 10A ) et pour évaluer le besoin et / ou l'effet des corrections d'intervalle QT ( Figure 10B-10D ). Les HPSC-CM portant des mutations LQTS ont des intervalles QT prolongés par rapport aux contrôles WT ( Figure 11A ). Le traitement des HPSC-CM avec un inhibiteur hERG entraîne une prolongation de l'intervalle QT ( Figure 11B ); Inversement, le traitement par un activateur hERG entraîne un raccourcissement de l'intervalle QT ( Figure 11C ). Enfin, le traitement avec des médicaments affectant la fréquence de battement des CMH-CM devrait être visible comme un changement dans l'intervalle RR ( Figure 11D , raccourcissement de l'intervalle RR).
 />
/>
Figure 1: Stérilisation et revêtement des chips MEA. ( A ) Un schéma représentant le processus de stérilisation, y compris le fait de placer la puce MEA dans une boîte en Pétri en verre autoclavable, en enveloppant dans du papier d'aluminium, en vaporisant pendant 6 min et en exposant aux UV pendant 30 minutes. ( B ) Vue en haut (panneau gauche) et côté (panneau central) de l'anneau PTFE personnalisé; L'anneau a un diamètre extérieur de 1,2 cm, dont 4 volets qui permettent son positionnement au centre de la puce MEA et le diamètre intérieur est de 0,4 cm (panneau droit). ( C ) Schéma représentant la préparation de la puce MEA, y compris le placement de la puce dans une boîte de Petri en plastique standard, en ajoutant 8 ml d'eau désionisée à l'extérieur de la chambre MEA, en plaçant l'anneau de PTFE au milieu de la chambre et en enduisant le réseau d'électrodes avec la fibronectine . Après le temps d'incubation, la fibronectine est éliminée et remplacée par un milieu de culture.T = "_ blank"> Cliquez ici pour voir une version plus grande de cette figure.
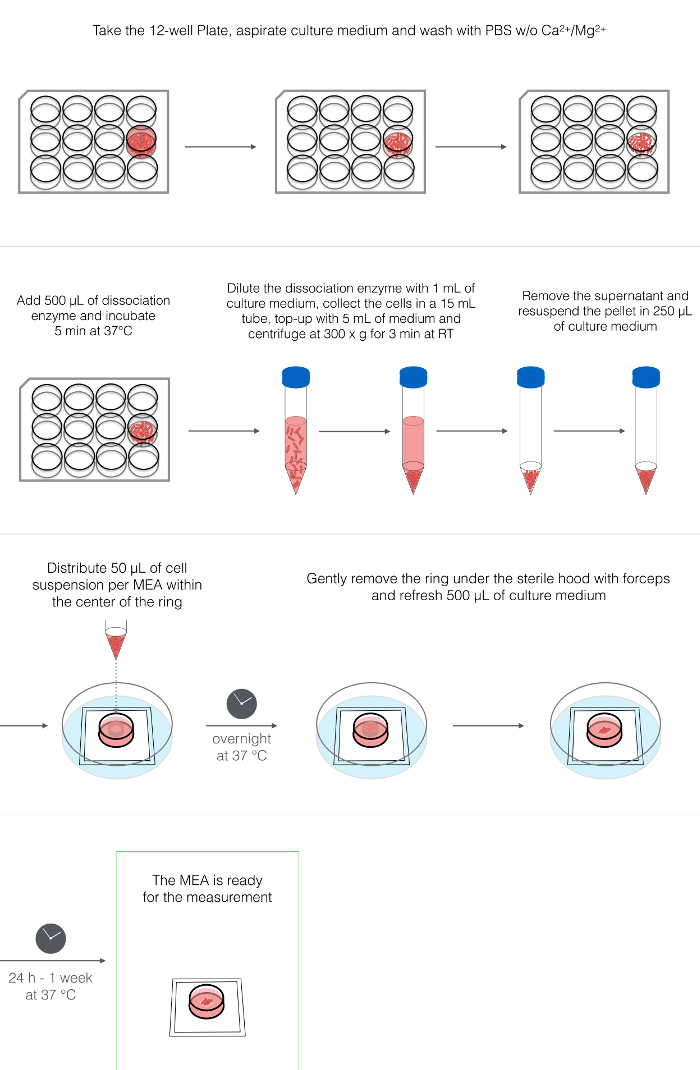
Figure 2: Dissociation et plaquage de hPSC-CM. Schéma représentant les procédés de dissociation enzymatique du HPSC-CM, centrifugation, remise en suspension et placage au centre de la chambre MEA. Cliquez ici pour voir une version plus grande de ce chiffre.

Figure 3: chips MEA avec couche HPSC-CM. ( A ) Vue de dessus de la puce MEA contenant l'anneau et la couche de hPSC-CM. ( B ) Vue latérale de la puce MEA après élimination de la bague. ( C ) Image de champ lumineux de la couche de CMH-CM appliquée sur le micro-Réseau d'électrodes; Augmentation de 4X. Cliquez ici pour voir une version plus grande de ce chiffre.
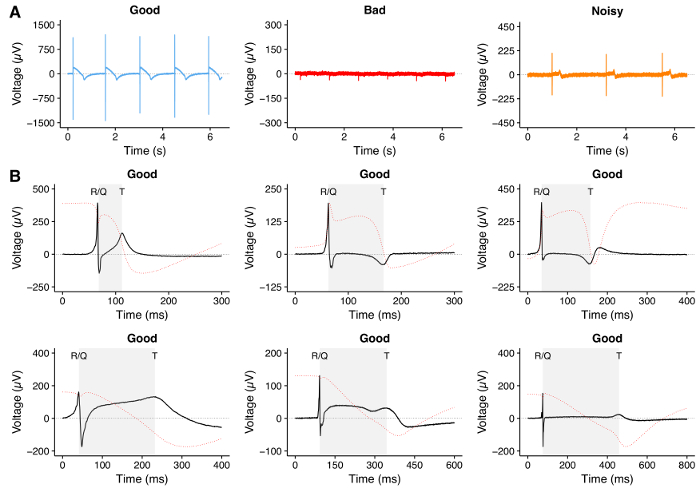
Figure 4: Enregistrement MEA de hPSC-CM. ( A ) Des traces représentatives enregistrées avec le MEA montrant une trace de bonne qualité avec des pics R / Q et T clairement visibles avec un rapport signal / bruit élevé (gauche), une trace de mauvaise qualité sans pics R / Q et T clairement visibles (milieu) Et une trace bruyante avec des pics R / Q et T clairement visibles mais avec un faible rapport signal / bruit (à droite). ( B ) Exemples représentatifs de traces de FP de bonne qualité avec différentes morphologies qui peuvent être enregistrées lors d'expériences de MEA utilisant des HPSC-CM. La zone ombrée représente l'intervalle QT mesuré lors de l'analyse. Comme le FP à MEA ressemble à la première dérivéeE du potentiel d'action 28 , nous avons calculé l'intégrale de la trace FP, représentée sous forme de ligne pointillée rouge, en tant que démonstration théorique d'un choix d'onde T près d'une repolarisation de potentiel d'action complète. Cliquez ici pour voir une version plus grande de ce chiffre.
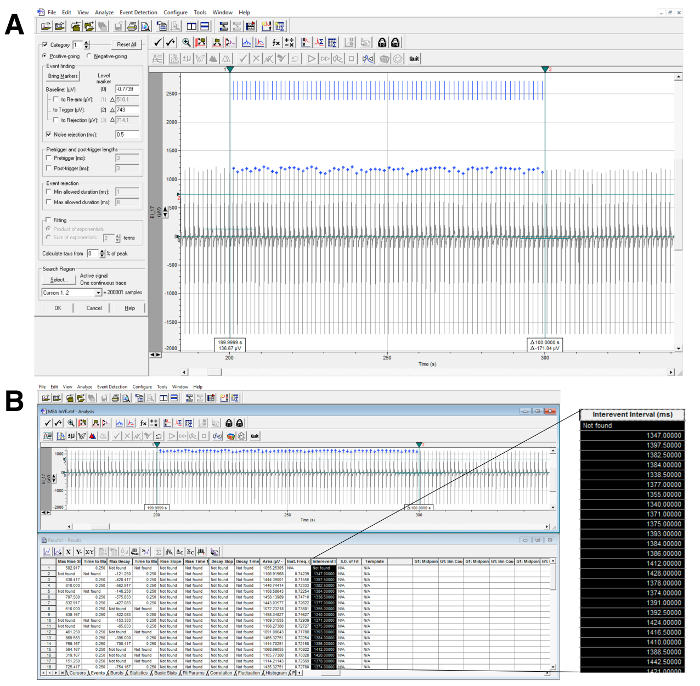
Figure 5: Calcul de l'intervalle RR. ( A ) Exemple d'analyse d'intervalle RR avec détection de crête automatique (en haut) et extraction de données (en bas) à l'aide du logiciel d'analyse (voir Tableau des matériaux). Les curseurs verticaux identifient l'intervalle de temps d'intérêt et le curseur horizontal traverse tous les événements détectés et identifiés par des marques bleues. ( B ) La colonne agrandie montre les données extraites utilisées pour calculer l'intervalle RR. Cliquez ici pour voir une version plus grande de ce chiffre.

Figure 6: Création du modèle FP. Exemple de sélection de modèle à l'aide de curseurs verticaux placés avant et après un FP unique. Ce modèle est utilisé pour l'identification automatique FP. Cliquez ici pour voir une version plus grande de ce chiffre.
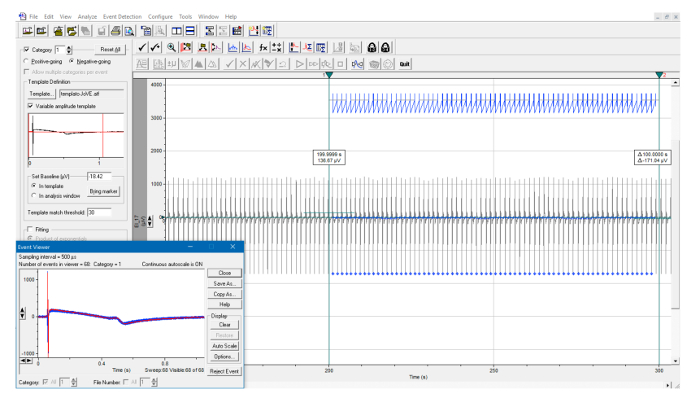
Figure 7: Identification automatique du modèle FP. Exemple de recherche de modèle dans un intervalle défini par deux curseurs verticaux. Tous les événements détectés sont identifiés par des marques bleues et sont automatiquementSuperposé à l'encart. Cliquez ici pour voir une version plus grande de ce chiffre.
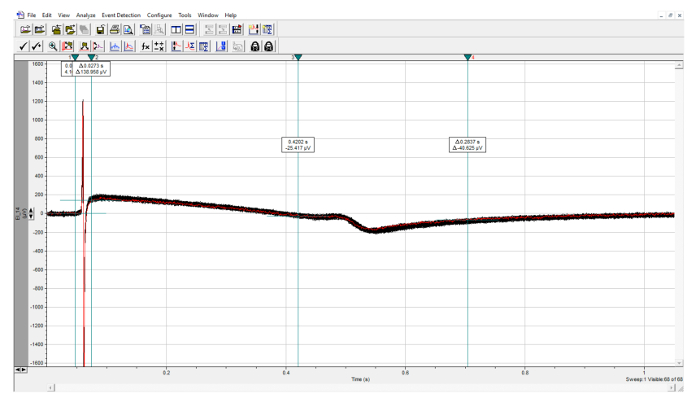
Figure 8: Quantification des paramètres FP. Analyse des événements enregistrés, avec le pic R défini manuellement dans les deux premiers curseurs et le pic T défini manuellement dans les deux derniers curseurs. Cliquez ici pour voir une version plus grande de ce chiffre.
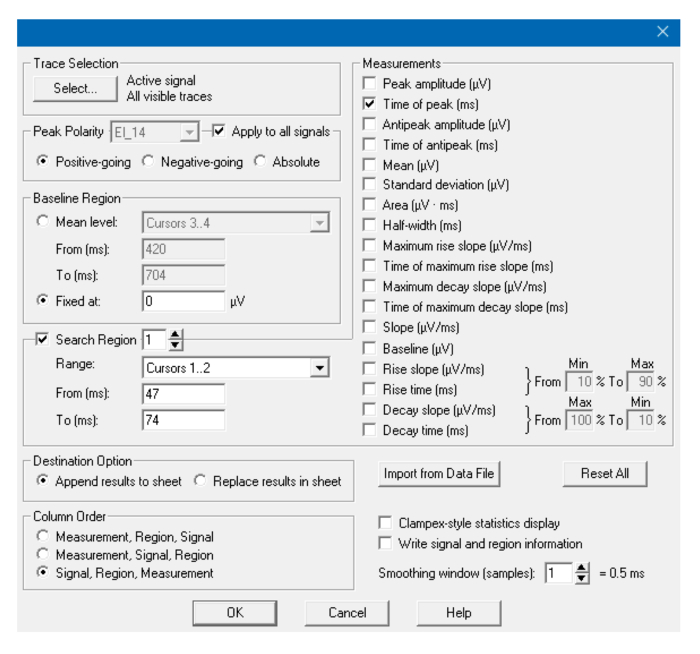
Figure 9: Fenêtre d'analyse. Paramètres utilisés dans la fenêtre Statistiques pour détecter le pic R. Pour la détection du pic T, changez les curseurs et (si nécessaire) poLarity. Cliquez ici pour voir une version plus grande de ce chiffre.
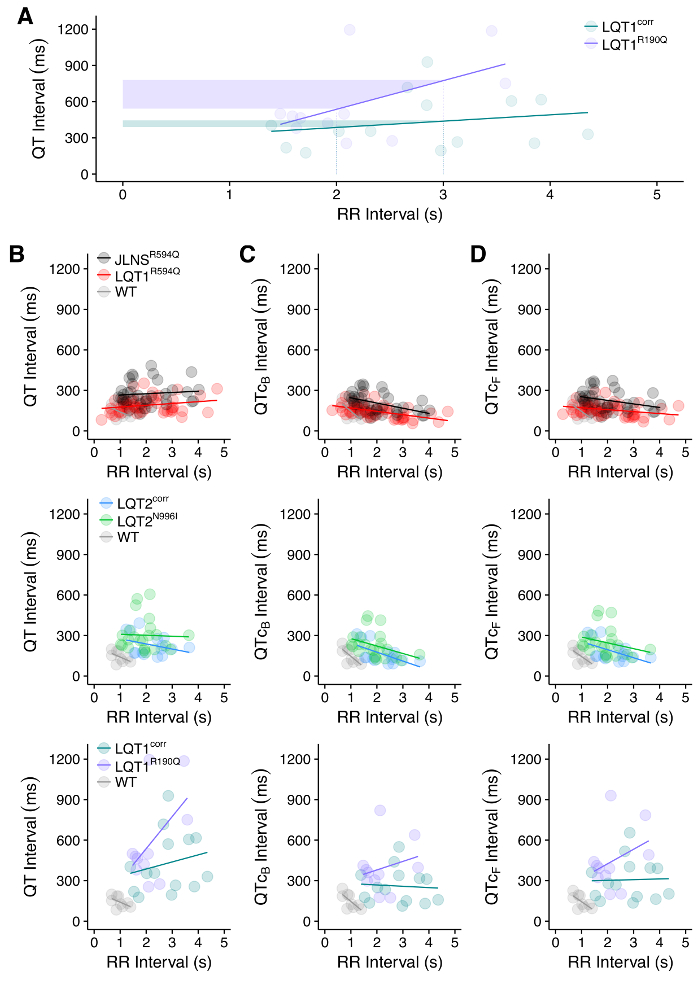
Figure 10: relation QT-RR. ( A ) Exemple de relation différente entre les intervalles QT et RR entre les HPSC-CM de WT (LQT1 corr ) et le syndrome QT long type 1 (LQT1 R190Q ). Les zones ombrées montrent que le même changement dans l'intervalle RR génère un changement plus important dans l'intervalle QT de la lignée malade LQT1, ce qui augmente probablement la susceptibilité à l'arythmie. ( B ) Relation entre les intervalles QT et RR non corrigés mesurés à MEA en CM à partir de 7 lignes HPSC différentes. L'effet de la correction QT pour les formules de Bazett ( C ) ou de Fridericia ( D ) est visible et visible comme changement de la pente du QT-RR dans Terval. Figures adaptées de la référence 30 . Cliquez ici pour voir une version plus grande de ce chiffre.
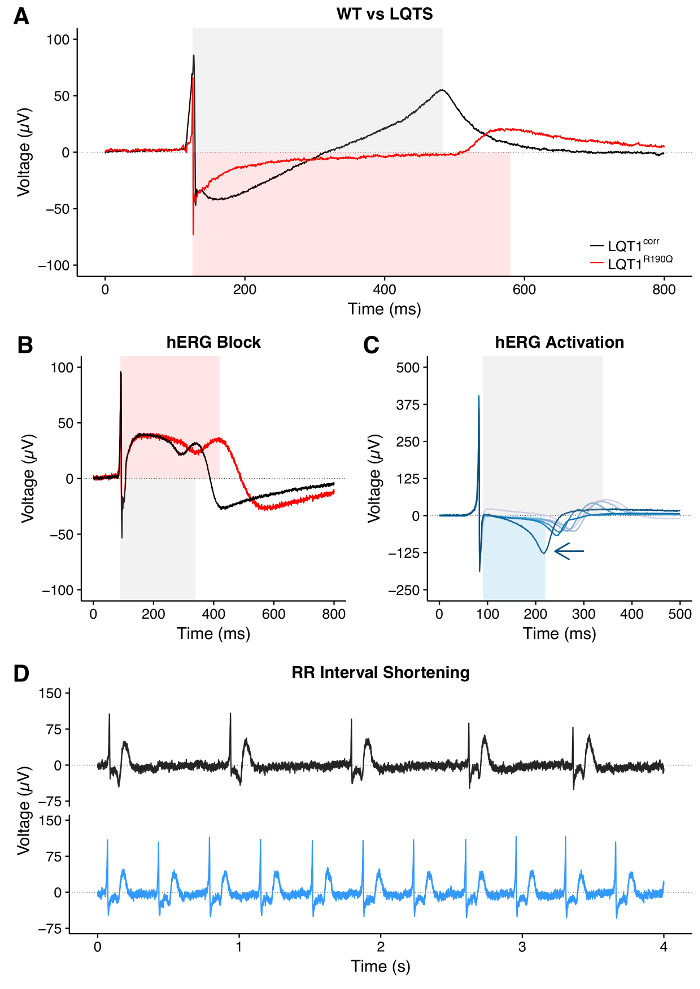
Figure 11: Variations d'intervalle QT et RR induites par la maladie ou le médicament. ( A ) Exemple de prolongement de l'intervalle QT dans hPSC-CM dérivé d'un patient porteur d'une mutation du syndrome à long-QT (LQTS) par rapport à son contrôle WT isogène. ( B ) Exemple de prolongement de l'intervalle QT dans hPSC-CM sur un bloc pharmacologique de hERG. ( C ) raccourcissement de l'intervalle QT lors du traitement avec des doses croissantes d'activateur hERG. La flèche indique la direction du shortening. ( D ) Exemple de raccourcissement de l'intervalle de RR induite par un médicament. Le panneau (C) a été adapté de la référence> 30. Cliquez ici pour voir une version plus grande de ce chiffre.
| Basses insulines, BSA, polyvinylalcool, lipides essentiels (LI-BPEL) Moyenne | |||
| Composant | Quantité pour 100 mL | ||
| IMDM | 43 mL | ||
| F12 | 43 mL | ||
| 2-phosphate d'acide ascorbique (5 mg / ml dans de l'eau distillée) | 1 mL | ||
| Supplément de culture cellulaire (substitut direct de la L-glutamine) | 1 mL | ||
| Pénicilline / Streptomycine | 0,5Ml | ||
| Phénol Rouge | 1 mg | ||
| Hybridome Medium-II sans protéines (PFHMII) | 5 ml | ||
| BSA (10% en poids / volume dans IMDM) | 2,5 mL | ||
| PVA (5% en poids / volume dans de l'eau distillée) | 2,5 mL | ||
| Concentré lipidique à définition chimique (CDLC) | 1 mL | ||
| Insuline-Transferrine-Sélénium-Ethanolamine (ITS-X) 100X | 0,1 mL | ||
| Α-monothioglycérol (13 μL dans 1 mL IMDM) | 0,3 mL | ||
| Mélanger les réactifs, filtrer avec un filtre à pores de 0,22 μm et conserver le milieu à 4 ° C pendant 2 semaines maximum. | |||
Tableau 1: Composition moyenne Li-BPEL.
Discussion
Ce protocole montre comment dissocier et préparer les CMH-CM pour mesurer leur FP à l'aide de MEA. Les CMH-CM affichent généralement une activité électrique spontanée, qui peut être mesurée comme FP et peut fournir des données significatives en ce qui concerne la fréquence de battement, la durée d'intervalle QT et les événements arythmiques.
La dissociation des cultures différenciées cardiaques 2D est nécessaire pour recréer une couche battante sur le MEA et représente une étape critique. Le stress mécanique par des pipettes répétées et / ou des traitements enzymatiques de dissociation agressive peut entraîner une mortalité cellulaire élevée, un échec à attacher à la plaque MEA et un manque d'activité électrique spontanée. Ce protocole a été optimisé pour les cultures monocouches. Cependant, une approche similaire peut être utilisée pour les cultures tridimensionnelles (3D) , par exemple, des corps embryoïdes ou EB) avec des modifications mineures, telles que la collecte des EBs suivie d'un lavage PBS et d'un temps d'incubation plus long avec l'enzyme dissociante. ImporterDe manière différente, dans les cultures différenciées en 2D et 3D, plus les cellules différenciées étaient anciennes, plus le temps d'incubation nécessaire serait de détacher les cellules en raison de l'augmentation du dépôt de matrice extracellulaire.
Le protocole décrit ici pour la quantification des paramètres FP peut être utilisé pour générer des courbes dose-réponse pour les médicaments cardio-actifs. Comme l'a décrit récemment Cavero et al. 31 , la concentration de départ d'un médicament pourrait affecter profondément le résultat d'une mesure MEA. Par conséquent, pour améliorer l'exactitude et la fiabilité des résultats, nous suggérons ce qui suit: 1) en cas d'activateurs / bloqueurs irréversibles, utiliser des volumes relativement importants de milieu contenant le médicament à tester. Plus en détail, retirez 10 à 50% du volume moyen de la puce MEA et ajoutez un volume égal de milieu dans lequel le médicament a été préalablement dissous à la concentration appropriée. Dans ce cas, pour calculer la concentration finale de médicament, il est essentiel de considérerLe changement de concentration après élimination du milieu. 2) En cas d'activateurs / bloqueurs réversibles, ajouter 10 μL de chaque dose de médicament à partir d'une solution stock 100X.
La majorité des protocoles de différenciation cardiaque aboutit à une population mixte variable de cardiomyocytes de type nodal, atrial et ventriculaire, le type ventriculaire étant le plus représenté. Cela pourrait constituer une limitation lors de la modélisation des maladies cardiaques affectant un sous-type de cardiomyocytes spécifique ou des médicaments agissant sur des canaux ioniques spécifiques aux sous-types cardiaques. Bien que plusieurs études aient des conditions optimisées pour diriger une spécification plus contrôlée pendant la différenciation cardiaque 3 , 5 , 37 , 38 , leur applicabilité plus large est encore à l'étude.
En outre, l'efficacité variable de la différenciation (dans différentes expériences et dansDes lignes HPSC compliquées) peuvent être observées 39 , 40 , 41 , 42 , 43 , 44 . Les stratégies d'enrichissement des cardiomyocytes basées sur l'expression des protéines de surface 35 , 45 (par le triage cellulaire assisté par fluorescence ou par la sélection des cordons magnétiques 46 , 47 ) et la sélection métabolique 44 , 48 peuvent représenter des stratégies valides qui peuvent être appliquées à Non modi fi é) le calage avant de la ligne HPSC des hPSC-CM, pour améliorer le signal électrique.
Bien que les CMH-CM soient notoirement immatures par rapport aux cardiomyocytes humains adultes 4 , 49 , ils se sont révélés utilesÉcapituler et identifier des changements spécifiques liés à la maladie ( p . Ex ., Dans les canalopathies) 19 , 20 , 50 et réponses induites par la drogue ( p . Ex ., Bloqueurs des canaux ioniques cardiaques) 4 , 51 . En outre, les cellules immatures sont plus faciles à dissocier et se rétablissent mieux que les cardiomyocytes adultes après la dissociation et le placage 44. Par conséquent, l'immaturité de l'HPSC-CM peut être récompensée en tant qu'avantage à cet égard. Cependant, pour pouvoir récapituler, par exemple . Les maladies cardiaques en retard tardif et reproduisent fidèlement les réponses aux médicaments des cardiomyocytes adultes, il faut obtenir un état mécanique, métabolique et électrique de l'état HPSC-CM. Les méthodes pour mûrir ces cellules comprennent un temps prolongé dans la culture 52 , la contrainte mécanique 53 , la stimulation électrique 54 , l'addition de petitesLes molécules 55 , la culture 3D 56 , la co-culture avec d'autres types de cellules 57 , et même une combinaison de ces approches 58 ; À ce jour, aucune de ces approches n'a conduit à un phénotype adulte.
Dans le cadre des caractéristiques d'immaturité, les CMH-CM montrent l'automatisme électrique. Ici, des détails sont fournis sur la façon de quantifier avec précision les intervalles QT et RR. Une limitation de la mesure de l'activité électrique spontanée est que la comparaison des intervalles QT peut être difficile lorsque les HPSC-CM affichent différentes fréquences de battement. Dans ce cas, les formules de Bazett ou Fridericia peuvent être utilisées pour corriger l'intervalle QT pour la fréquence. Cependant, comme indiqué précédemment 30 , nous recommandons vivement d'effectuer une analyse de régression des grands axes en traçant l'intervalle QT par rapport à l'intervalle RR pour les données brutes et corrigées, pour exclure tout biais possible en raison de la méthode de correction elle-même.
Le protocole présenté ici, ainsi que les méthodes décrites précédemment 59 , 60 , assurent la normalisation des procédures et l'analyse des FP de CMH-CM, améliorant la reproductibilité des données et permettant une meilleure comparaison des résultats inter-laboratoires.
Déclarations de divulgation
CLM est le cofondateur et conseiller de Pluriomics bv. Une partie des coûts de publication a été couverte par Multi Channel Systems.
Remerciements
Ce travail a été soutenu par les subventions suivantes: CVON (HUSTCARE): l'Initiative néerlandaise de recherche cardiovasculaire (la Fondation néerlandaise Heart, la Fédération néerlandaise des centres médicaux universitaires, l'Organisation néerlandaise pour la recherche et le développement en santé et l'Académie royale néerlandaise des sciences); Le Conseil européen de la recherche (ERCAdG 323182 STEMCARDIOVASC). Nous remercions E. Giacomelli (LUMC) d'avoir aidé à la différenciation cardiaque de l'HPSC.
matériels
| Name | Company | Catalog Number | Comments |
| Biological safety cabinet/laminar flow-hood | Cleanair | ||
| MEA2100 in vitro recording system | Multi Channel Systems | ||
| CO2 cell-culture incubator | Sanyo | MCO-15A | |
| Centrifuge | Hitachi | himac-CT6EL | |
| Leica stereomicroscope | Leica Microsystems | MS5 | |
| Handheld pipetman (P-10 (10 μL), P-200 (200 μL), P-1000 (1,000 μL)) | Gilson International | ||
| Filter tips (10 μL, 200μL, 1,000μL) | Corning | 4807 (10 μL), 4810 (200μL), 4809 (1000μL) | |
| Disposable bottle top filter (0.22 μm pore size) | Millipore | SCGVU02RE | |
| Sterile plastic pipette | Greiner Bio-One | 606180 (5 mL), 607180 (10 mL), 760180 (25 mL) | |
| Tweezers | Dumont | ||
| Autoclavable Petri dishes | VWR/ Duran Group | 391-0860 | |
| MEA chip | Multi Channel Systems | MEA200/30iR-Ti-gr | |
| Phosphate-buffered Saline (PBS) calcium, magnesium | Thermo Fisher Scientific | 14040-091 | |
| Phosphate-Buffered Saline (PBS) no calcium, no magnesium | Thermo Fisher Scientific | 14190-169 | |
| Human recombinant fibronectin | Tebu-Bio | J64560 | |
| Custom-made polytetrafluoroethylene (PTFE) MEA rings | LUMC: department of Instrument Development | ||
| Dissociation enzyme - TrypLE Select 1x | Thermo Fisher Scientific | 12563-029 | |
| Tergazyme enzyme detergent | Sigma-Aldrich | Z273287-1EA | |
| Distilled Water | Thermo Fisher Scientific | 15230-089 | |
| Name | Company | Catalog Number | Comments |
| Reagents for LI-BPEL medium | |||
| Iscove's Modified Dulbecco's Medium (IMDM) | Thermo Fisher Scientific | 21056-023 | |
| F12 nutrient mixture (Ham) | Thermo Fisher Scientific | 31765-027 | |
| Ascorbic Acid 2-phosphate | Sigma-Aldrich | A8960 | |
| Glutamax | Thermo Fisher Scientific | 35050-038 | |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15070-063 | |
| Phenol Red | Sigma-Aldrich | P3532 | |
| Protein-free Hybridoma Medium-II (PFHMII) | Thermo Fisher Scientific | 12040-077 | |
| Bovine Serum Albumin (BSA) | Bovogen Biologicals Australia | BSAS05 | |
| Poly(Vinyl Alcohol) (PVA) | Sigma-Aldrich | P8136 | |
| Chemically-defined Lipid Concentrate (CDLC) | Thermo Fisher Scientific | 11905-031 | |
| Insulin-Transferrin-Selenium-Ethanolamine (ITS-X) 100x | Thermo Fisher Scientific | 51599-056 | |
| α-Monothioglycerol | Sigma-Aldrich | M6145 | |
| Name | Company | Software version | Comments |
| MC_Rack | Multi Channel Systems | 4.6.2 | Alternatively, data can be recorded using Cardio2D or MC_Experimenter (MultiChannel Systems) |
| TCX Control | Multi Channel Systems | 1.3.4 | |
| MEA Select | Multi Channel Systems | 1.3.0 | |
| MC_Data Tool | Multi Channel Systems | 2.6.15 | Alternatively, Multi Channel Data Manager (MultiChannel Systems) can be used when custom data export is required (HDF5, EDF, etc.) |
| Clampfit | Molecular Devices | 7.0.0 | Used in step 10.1 for analyzing, graphing, and formatting of all of data. To use Clampfit, download and install the electrophysiology data acquisition and pClam (latest version, 10.7.0), available on the Molecular Devices Website. Once complete, launch the software Clampfit. Alternatively, data can be analysed using Cardio2D (MultiChannel Systems) or MatLab custom code. |
Références
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Thomson, J. A., et al. Embryonic stem cell lines derived from human blastocysts. Science. 282 (5391), 1145-1147 (1998).
- Birket, M. J., et al. Expansion and patterning of cardiovascular progenitors derived from human pluripotent stem cells. Nat Biotechnol. 33 (9), 970-979 (2015).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochim Biophys Acta. 1863 (7 Pt B), 1728-1748 (2016).
- Devalla, H. D., et al. Atrial-like cardiomyocytes from human pluripotent stem cells are a robust preclinical model for assessing atrial-selective pharmacology. EMBO Mol Med. 7 (4), 394-410 (2015).
- Lewandowski, J., Kolanowski, T. J., Kurpisz, M. Techniques for the induction of human pluripotent stem cell differentiation towards cardiomyocytes. J Tissue Eng Regen Med. , (2016).
- Mummery, C. L., Zhang, J., Ng, E. S., Elliott, D. A., Elefanty, A. G., Kamp, T. J. Differentiation of human embryonic stem cells and induced pluripotent stem cells to cardiomyocytes: a methods overview. Circ Res. 111 (3), 344-358 (2012).
- Davies, M. P., An, R. H., Doevendans, P., Kubalak, S., Chien, K. R., Kass, R. S. Developmental changes in ionic channel activity in the embryonic murine heart. Circ Res. 78 (1), 15-25 (1996).
- Bellin, M., et al. Isogenic human pluripotent stem cell pairs reveal the role of a KCNH2 mutation in long-QT syndrome. EMBO J. 32 (24), 3161-3175 (2013).
- Moretti, A., et al. Patient-specific induced pluripotent stem-cell models for long-QT syndrome. N Engl J Med. 363 (15), 1397-1409 (2010).
- Ma, D., et al. Modeling type 3 long QT syndrome with cardiomyocytes derived from patient-specific induced pluripotent stem cells. Int J Cardiol. 168 (6), 5277-5286 (2013).
- Yazawa, M., et al. Using induced pluripotent stem cells to investigate cardiac phenotypes in Timothy syndrome. Nature. 471 (7337), 230-U120 (2011).
- Limpitikul, W. B., et al. A Precision Medicine Approach to the Rescue of Function on Malignant Calmodulinopathic Long QT Syndrome. Circ Res. , (2016).
- Liang, P., et al. Patient-Specific and Genome-Edited Induced Pluripotent Stem Cell-Derived Cardiomyocytes Elucidate Single-Cell Phenotype of Brugada Syndrome. J Am Coll Cardiol. 68 (19), 2086-2096 (2016).
- Jung, C. B., et al. Dantrolene rescues arrhythmogenic RYR2 defect in a patient-specific stem cell model of catecholaminergic polymorphic ventricular tachycardia. EMBO Mol Med. 4 (3), 180-191 (2012).
- Novak, A., et al. Cardiomyocytes generated from CPVTD307H patients are arrhythmogenic in response to beta-adrenergic stimulation. J Cell Mol Med. 16 (3), 468-482 (2012).
- Bellin, M., Mummery, C. L. Inherited heart disease - what can we expect from the second decade of human iPS cell research. FEBS Lett. 590 (15), 2482-2493 (2016).
- Sallam, K., Li, Y., Sager, P. T., Houser, S. R., Wu, J. C. Finding the rhythm of sudden cardiac death: new opportunities using induced pluripotent stem cell-derived cardiomyocytes. Circ Res. 116 (12), 1989-2004 (2015).
- Sinnecker, D., Goedel, A., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes: a versatile tool for arrhythmia research. Circ Res. 112 (6), 961-968 (2013).
- Bellin, M., Marchetto, M. C., Gage, F. H., Mummery, C. L. Induced pluripotent stem cells: the new patient?. Nat Rev Mol Cell Biol. 13 (11), 713-726 (2012).
- Sinnecker, D., Laugwitz, K. L., Moretti, A. Induced pluripotent stem cell-derived cardiomyocytes for drug development and toxicity testing. Pharmacol Ther. 143 (2), 246-252 (2014).
- Terrenoire, C., et al. Induced pluripotent stem cells used to reveal drug actions in a long QT syndrome family with complex genetics. J Gen Physiol. 141 (1), 61-72 (2013).
- Abi-Gerges, N., et al. Assessment of extracellular field potential and Ca2+ transient signals for early QT/pro-arrhythmia detection using human induced pluripotent stem cell-derived cardiomyocytes. J Pharmacol Toxicol Methods. 83, 1-15 (2016).
- Del Alamo, J. C., et al. High throughput physiological screening of iPSC-derived cardiomyocytes for drug development. Biochim Biophys Acta. 1863 (7 Pt B), 1717-1727 (2016).
- Blinova, K., et al. Comprehensive Translational Assessment of Human Induced Pluripotent Stem Cell Derived Cardiomyocytes for Evaluating Drug-Induced Arrhythmias. Toxicol Sci. , (2016).
- Nerbonne, J. M. Studying cardiac arrhythmias in the mouse--a reasonable model for probing mechanisms?. Trends Cardiovasc Med. 14 (3), 83-93 (2004).
- Halbach, M., Egert, U., Hescheler, J., Banach, K. Estimation of action potential changes from field potential recordings in multicellular mouse cardiac myocyte cultures. Cell Physiol Biochem. 13 (5), 271-284 (2003).
- Tertoolen, L. G., Braam, S. R., van Meer, B. J., Passier, R., Mummery, C. L. Interpretation of field potentials measured on a multi electrode array in pharmacological toxicity screening on primary and human pluripotent stem cell-derived cardiomyocytes. Biochem Biophys Res Commun. , (2017).
- Rajamohan, D., et al. Automated Electrophysiological and Pharmacological Evaluation of Human Pluripotent Stem Cell-Derived Cardiomyocytes. Stem Cells Dev. 25 (6), 439-452 (2016).
- Sala, L., et al. A new hERG allosteric modulator rescues genetic and drug-induced long-QT syndrome phenotypes in cardiomyocytes from isogenic pairs of patient induced pluripotent stem cells. EMBO Mol Med. 8 (9), 1065-1081 (2016).
- Cavero, I., Guillon, J. M., Ballet, V., Clements, M., Gerbeau, J. F., Holzgrefe, H. Comprehensive in vitro Proarrhythmia Assay (CiPA): Pending issues for successful validation and implementation. J Pharmacol Toxicol Methods. , (2016).
- Sala, L., Bellin, M., Mummery, C. L. Integrating cardiomyocytes from human pluripotent stem cells in safety pharmacology: has the time come?. Br J Pharmacol. , (2016).
- Chen, I. Y., Matsa, E., Wu, J. C. Induced pluripotent stem cells: at the heart of cardiovascular precision medicine. Nat Rev Cardiol. 13 (6), 333-349 (2016).
- Dambrot, C., et al. Strategies for rapidly mapping proviral integration sites and assessing cardiogenic potential of nascent human induced pluripotent stem cell clones. Exp Cell Res. 327 (2), 297-306 (2014).
- Elliott, D. A., et al. NKX2-5(eGFP/w) hESCs for isolation of human cardiac progenitors and cardiomyocytes. Nat Methods. 8 (12), 1037-1040 (2011).
- Ng, E. S., Davis, R., Stanley, E. G., Elefanty, A. G. A protocol describing the use of a recombinant protein-based, animal product-free medium (APEL) for human embryonic stem cell differentiation as spin embryoid bodies. Nat Protoc. 3 (5), 768-776 (2008).
- Karakikes, I., et al. Small molecule-mediated directed differentiation of human embryonic stem cells toward ventricular cardiomyocytes. Stem Cells Transl Med. 3 (1), 18-31 (2014).
- Zhang, Q., et al. Direct differentiation of atrial and ventricular myocytes from human embryonic stem cells by alternating retinoid signals. Cell Res. 21 (4), 579-587 (2011).
- Mummery, C., et al. Differentiation of human embryonic stem cells to cardiomyocytes: role of coculture with visceral endoderm-like cells. Circulation. 107 (21), 2733-2740 (2003).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Zhang, J., et al. Extracellular matrix promotes highly efficient cardiac differentiation of human pluripotent stem cells: the matrix sandwich method. Circ Res. 111 (9), 1125-1136 (2012).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/beta-catenin signaling under fully defined conditions. Nat Protoc. 8 (1), 162-175 (2013).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Dubois, N. C., et al. SIRPA is a specific cell-surface marker for isolating cardiomyocytes derived from human pluripotent stem cells. Nat Biotechnol. 29 (11), 1011-1018 (2011).
- Fuerstenau-Sharp, M., et al. Generation of highly purified human cardiomyocytes from peripheral blood mononuclear cell-derived induced pluripotent stem cells. PLoS One. 10 (5), e0126596 (2015).
- Schwach, V., Passier, R. Generation and purification of human stem cell-derived cardiomyocytes. Differentiation. 91 (4-5), 126-138 (2016).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
- Veerman, C. C., Kosmidis, G., Mummery, C. L., Casini, S., Verkerk, A. O., Bellin, M. Immaturity of human stem-cell-derived cardiomyocytes in culture: fatal flaw or soluble problem?. Stem Cells Dev. 24 (9), 1035-1052 (2015).
- Karakikes, I., Ameen, M., Termglinchan, V., Wu, J. C. Human induced pluripotent stem cell-derived cardiomyocytes: insights into molecular, cellular, and functional phenotypes. Circ Res. 117 (1), 80-88 (2015).
- Avior, Y., Sagi, I., Benvenisty, N. Pluripotent stem cells in disease modelling and drug discovery. Nat Rev Mol Cell Biol. 17 (3), 170-182 (2016).
- Otsuji, T. G., Minami, I., Kurose, Y., Yamauchi, K., Tada, M., Nakatsuji, N. Progressive maturation in contracting cardiomyocytes derived from human embryonic stem cells: Qualitative effects on electrophysiological responses to drugs. Stem Cell Res. 4 (3), 201-213 (2010).
- Mihic, A., et al. The effect of cyclic stretch on maturation and 3D tissue formation of human embryonic stem cell-derived cardiomyocytes. Biomaterials. 35 (9), 2798-2808 (2014).
- Lieu, D. K., et al. Mechanism-based facilitated maturation of human pluripotent stem cell-derived cardiomyocytes. Circ Arrhythm Electrophysiol. 6 (1), 191-201 (2013).
- Yang, X., et al. Tri-iodo-l-thyronine promotes the maturation of human cardiomyocytes-derived from induced pluripotent stem cells. J Mol Cell Cardiol. 72, 296-304 (2014).
- Mannhardt, I., et al. Human Engineered Heart Tissue: Analysis of Contractile Force. Stem Cell Reports. , (2016).
- Kim, C., et al. Non-cardiomyocytes influence the electrophysiological maturation of human embryonic stem cell-derived cardiomyocytes during differentiation. Stem Cells Dev. 19 (6), 783-795 (2010).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat Methods. 10 (8), 781-787 (2013).
- Clements, M. Multielectrode Array (MEA) Assay for Profiling Electrophysiological Drug Effects in Human Stem Cell-Derived Cardiomyocytes. Curr Protoc Toxicol. 68 (22), 1-22 (2016).
- Harris, K. A Human Induced Pluripotent Stem Cell-Derived Cardiomyocyte (hiPSC-CM) Multielectrode Array Assay for Preclinical Cardiac Electrophysiology Safety Screening. Curr Protoc Pharmacol. 71, 1-15 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon