Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Actuador basado en las células musculares cardíacas y auto-estabilizante Biorobot - Parte 2
En este artículo
Resumen
En este estudio, un actuador biológico y un biorobot de natación autoestabilizante con brazos cantilever funcionalizados elastoméricos se siembran con cardiomiocitos, se cultivan y se caracterizan por sus propiedades bioquímicas y biomecánicas a lo largo del tiempo.
Resumen
En los últimos años se han desarrollado dispositivos híbridos que consisten en una célula viva o un componente de tejido integrado con una columna vertebral mecánica sintética. Estos dispositivos, llamados biorobots, son accionados únicamente por la fuerza generada por la actividad contráctil del componente vivo y, debido a sus muchas ventajas inherentes, podrían ser una alternativa a los robots convencionales totalmente artificiales. Aquí describimos los métodos para sembrar y caracterizar un actuador biológico y un biorobot que fue diseñado, fabricado y funcionalizado en la primera parte de este artículo en dos partes. El actuador biológico fabricado y los dispositivos biorobot compuestos de una base de polidimetilsiloxano (PDMS) y un cantilever de película delgada se funcionalizaron para la fijación celular con fibronectina. Después de la funcionalización, los cardiomiocitos neonatales de rata se sembraron en el brazo en voladizo PDMS a una alta densidad, dando como resultado una hoja de células confluentes. Los dispositivos se visualizaron todos los días y el movimiento del cantiSe analizaron los brazos de palanca. El segundo día después de la siembra observamos la flexión de los brazos en voladizo debido a las fuerzas ejercidas por las células durante las contracciones espontáneas. Tras el análisis cuantitativo de la flexión en voladizo, se observó un aumento gradual en el estrés superficial ejercido por las células a medida que maduraban con el tiempo. Del mismo modo, se observó el movimiento del biorobot debido a la actuación del brazo en voladizo PDMS, que actuó como una aleta. Tras la cuantificación de los perfiles de natación de los dispositivos, se observaron varios modos de propulsión, que fueron influenciados por el ángulo de reposo de la aleta. La dirección del movimiento y la frecuencia de golpeo también fueron determinadas por el ángulo de reposo de la aleta, y se observó una velocidad de nadada máxima de 142 μm / s. En este manuscrito, se describe el procedimiento para poblar los dispositivos fabricados con cardiomiocitos, así como para la evaluación del actuador biológico y la actividad biorobot.
Introducción
Los biorobots son dispositivos basados en células vivas que se incorporan dentro de una columna vertebral mecánica que normalmente está compuesta de materiales blandos y elásticos, tales como PDMS o hidrogeles 1 . Las células sufren contracciones rítmicas, espontáneamente o en respuesta a estímulos, y por lo tanto funcionan como un actuador. La energía generada por la contracción celular impulsa a varios biorobots. Las células del corazón de los mamíferos (cardiomiocitos) y las células del músculo esquelético se usan a menudo para el accionamiento del biorobot debido a sus propiedades contráctiles. Aparte de los cardiomiocitos y las células del músculo esquelético, se han utilizado otros tipos de células, tales como los tejidos musculares de insectos 2 y los tejidos musculares explantados 3 . Los tejidos musculares de los insectos permiten el funcionamiento de actuadores biológicos a temperatura ambiente.
La función y el rendimiento de un biorobot se determinan principalmente por la fuerza y consistencia del actuador biológico ( es decir,. Células musculares), mientras que la estructura de la columna vertebral mecánica determina principalmente los mecanismos de locomoción, estabilidad y potencia. Dado que estos dispositivos son impulsados exclusivamente por las fuerzas generadas por las células, no hay contaminantes químicos ni ruidos operativos. Por lo tanto, forman una alternativa energéticamente eficiente a otros robots convencionales. Varias fuentes de la literatura han discutido los diferentes métodos para integrar células vivas y tejidos en biorobots 1 , 4 , 5 . Los avances en microfabricación y técnicas de ingeniería de tejidos han permitido el desarrollo de biorobots que pueden caminar, agarrar, nadar o bombear 5 , 6 . En general, las células se cultivan directamente sobre el esqueleto mecánico (polimérico) como una lámina de células confluentes o se moldean en estructuras de actuación tridimensionales dentro de armazones tales como anillos y tiras. A menudo, los biorobots sonFabricadas con hojas de cardiomiocitos 6 , 7 , ya que estas células tienen una capacidad innata para exhibir una contracción espontánea sin estímulos externos. Por otro lado, los informes sobre las hojas de células del músculo esquelético son limitados debido a su necesidad de estímulos para iniciar las contracciones in vitro con el fin de iniciar la despolarización de la membrana [ 8] .
Este protocolo describe en primer lugar cómo sembrar cardiomiocitos en un actuador biológico funcionalizado hecho de un cantilever PDMS delgado. A continuación, se describe en detalle la siembra y el análisis de los perfiles de natación. El cantilever está funcionalizado con una proteína adhesiva celular tal como fibronectina y se siembra confluentemente con cardiomiocitos. A medida que las células sembradas en el dispositivo se contraen, hacen que el voladizo se doble y actúe así como un actuador. Con el tiempo, a medida que las células maduran, trazamos los cambios en el estrés superficial en el dispositivo analizando videos de laFlexión en voladizo. El actuador biológico desarrollado aquí puede usarse para determinar las propiedades contráctiles de cualquier tipo de célula, tales como los fibroblastos o las células de tallos pluripotentes inducidos, a medida que experimentan diferenciación.
Gran parte de la investigación anterior sobre biorobots se ha centrado en el desarrollo de actuadores biológicos, mientras que la optimización de la arquitectura biorobot y las capacidades funcionales se han descuidado en gran medida. Recientemente, algunos estudios han demostrado la implementación de modos de natación en biorobots que están inspirados en la naturaleza. Por ejemplo, los biorobots de natación con movimiento 6 basado en flagelos, propulsión 9 de medusas y rayos biohíbridos 4 han sido diseñados. A diferencia de otras obras en la literatura, aquí nos centramos en la variación de las propiedades de la columna vertebral mecánica para crear una estructura auto-estabilizadora. El biorobot desarrollado en este estudio es capaz de mantener un tono constante, roll, y imProfundidad de mersion como nada. Estos parámetros se pueden modificar variando el grosor de cada compuesto base. Los pasos de fabricación implicados en el desarrollo del actuador PDMS, el biorobot sumergible y la funcionalización del dispositivo se describen en detalle en la Parte 1 de este artículo en dos partes, así como en nuestro trabajo reciente 7 . Para el desarrollo de novelas, altamente eficientes biorobots para diversas aplicaciones, tales como entrega de carga.
El proceso de aislamiento seguido en este estudio es similar al proceso descrito en un trabajo anterior 10 , así como en trabajos publicados recientemente 7 . Los métodos de microfabricación utilizados para fabricar los actuadores PDMS y los dispositivos biorobot se describen en detalle en la Parte 1 de este manuscrito en dos partes. La sección de protocolo de este manuscrito describe los pasos implicados en la siembra de cardiomiocitos en el PDMS fabricado aCtuator y el biorobot siguiendo su funcionalización con proteínas adhesivas celulares.
Protocolo
Todos los procedimientos descritos aquí se han llevado a cabo usando un protocolo aprobado y de acuerdo con las regulaciones del Comité Institucional de Cuidado y Uso de Animales de la Universidad de Notre Dame.
1. Cultivo y siembra de células
- Antes de comenzar, preparar los artículos requeridos: un pequeño embudo, pipetas, y medio de Eagle modificado de Dulbecco caliente (DMEM) suplementado con 10% de suero bovino fetal (FBS) y 1% de antibiótico de penicilina (DMEM completo).
- Tome los frascos T-25 junto con el dispositivo funcionalizado (actuador biológico o el biorobot) dentro de él. Consulte la sección 4 de la Parte 1 de este manuscrito en dos partes para obtener detalles sobre la preparación, la funcionalización y el almacenamiento del dispositivo antes de la siembra de células.
- Preparar un embudo, que se puede hacer rodando una pequeña hoja de plástico cuadrado. Colóquelo sobre el actuador biológico o biorobot dentro del matraz T-25. Ajuste el diámetro del extremo más ancho para que se ajuste a todo el dispositivoY la altura para que encaje perfectamente cuando se aprieta la parte superior del matraz.
- Para los biorobots, utilice un imán para mantener el dispositivo en su lugar en la parte inferior del matraz durante todo el proceso de siembra.
Nota: En este caso, se utilizó un solo imán de disco de neodimio (1,26 "de diámetro), pero cualquier imán de tamaño y resistencia similar puede utilizarse también para mantener el biorobot con la base compuesta de níquel-PDMS magnético. - UV esterilizar la hoja de plástico durante al menos 30 minutos antes de su uso.
- Para los biorobots, utilice un imán para mantener el dispositivo en su lugar en la parte inferior del matraz durante todo el proceso de siembra.
- Asegúrese de que no haya un gran espacio entre la base del embudo y el matraz.
- Resuspender los cardiomiocitos en DMEM completo a una densidad de 1,6 x 10 7 células / ml y dejar caer lentamente 400 μl de suspensión sobre el dispositivo a través del embudo. Utilice un hemocitómetro o cualquier otro contador de células para determinar el número de células obtenidas.
- Lentamente mover el sistema de nuevo en la incubadora sin perturbar el dispositivo y el ingenio embudoHin Cultivo durante 24 ha 37 ° C.
- Después del período de incubación, retire lentamente el embudo, lave suavemente la muestra con PBS y rellene el matraz con 10 mL de DMEM nuevo completo.
Nota: Para los biorobots, retire el imán para que el dispositivo esté a flote.
2. Caracterización bioquímica
- Ensayo de flujo de calcio
Nota: El ensayo de flujo de calcio se lleva a cabo para evaluar la interconexión celular. El procedimiento para cargar las células con el colorante fluorescente, específico de ión calcio, sigue el proceso descrito en un protocolo previamente establecido 11 .- En primer lugar, preparar los materiales requeridos, éster de fluo-4-acetometil (AM) de calcio, poliol tensioactivo no iónico (véase la Tabla de Materiales ) y solución salina de Tyrode.
- Utilizando pinzas largas, transfiera suavemente el dispositivo del matraz de cultivo a una placa de Petri de 35 mm con 2 ml de solución salina de Tyrode Ción.
- En un tubo de centrífuga separado, tomar 1 ml de solución de Tyrode caliente (calentada a 37 ° C) y añadir 3-5 μL de colorante de calcio fluo-4 AM (concentración de trabajo: 3-5 μM) y una parte igual de tensioactivo no iónico Poliol (concentración de trabajo: 0,2%). Reemplazar la solución de muestra con una solución caliente de Tyrode suplementada con el colorante indicador de calcio, fluo-4 y 0,2% de tensioactivo no iónico. Incubar durante 25 - 30 min a 37 ° C.
- Retire la solución de colorante y lave suavemente la muestra con la solución fresca de Tyrode. Vuelva a incubar la muestra en 2 ml de DMEM fresco completo durante otros 30 min a 37 ° C antes de la obtención de imágenes.
Nota: Los resultados de este ensayo y el video relacionado se proporcionan en el trabajo publicado anteriormente 7 .
- Inmunofluorescencia
Nota: La inmunotinción doble de todas las muestras se realizó siguiendo el protocolo previamente establecido> 12.- En primer lugar, preparar un 10% de suero de cabra (GS) en solución salina tamponada con fosfato (PBS), paraformaldehído al 4% (PFA) en diH2O, 0,1% de detergente liso celular (ver Tabla de Materiales ) en PBS, anticuerpos primarios - anticuerpos monoclonales monoclonales anticuerpo monoclonal cardíaco troponina-I y anticuerpo monoclonal anti-conejo conexinas-43), anticuerpos secundarios (conjugado Alexa 594 y conjugado de cabra anti-IgG de ratón anti-IgG de ratón (Alexa 488) y DAPI.
Precaución : Paraformaldehído es cancerígeno. - Se retira la muestra de interés del matraz y se lava suavemente dos veces con PBS. Consulte la sección 4 de la Parte 1 de este manuscrito en dos partes para obtener detalles sobre la preparación de la muestra y su funcionalización.
- Añadir una gotita de PBS en una pequeña cubreobjetos (diámetro: 12 mm o 15 mm). Sujete suavemente la base del dispositivo con pinzas y corte los delgados brazos PDMS (cantilever, Figura 1 ) usando tijeras desde el extremo donde se conecta a la parte superior de la basemi. Coloque los brazos en voladizo sobre la gotita con el lado adherido a la celda hacia arriba. La gotita de PBS evitará que las células se sequen.
- Fijar las muestras con 4% PFA y realizar doble inmunoticción de las muestras, como se describe anteriormente [ 12] .
- Después de la inmunotinción, coloque las muestras sobre un portaobjetos de cristal limpio usando un reactivo de montaje antideslizante y deje a un lado, sin alteraciones, en la oscuridad durante 24 h.
- Repita el procedimiento para todas las muestras.
Nota: Los resultados de este ensayo y las imágenes relacionadas se discuten en profundidad en el trabajo previamente publicado 7 .
- En primer lugar, preparar un 10% de suero de cabra (GS) en solución salina tamponada con fosfato (PBS), paraformaldehído al 4% (PFA) en diH2O, 0,1% de detergente liso celular (ver Tabla de Materiales ) en PBS, anticuerpos primarios - anticuerpos monoclonales monoclonales anticuerpo monoclonal cardíaco troponina-I y anticuerpo monoclonal anti-conejo conexinas-43), anticuerpos secundarios (conjugado Alexa 594 y conjugado de cabra anti-IgG de ratón anti-IgG de ratón (Alexa 488) y DAPI.
3. Imágenes
- Coloque el frasco T-25 en posición vertical en una incubadora de CO 2 y prepare el sistema de imagen dentro de la incubadora. Grabe el dispositivo utilizando una cámara (consulte la Tabla de materiales ) con un objetivo zoom (consulte la tabla de materiales ). Para una fuente de luz,Utilice una tira de luces del LED.
Nota: Aquí se utilizó una tira de LEDs de luz blanca, pero también funcionarán los LED normales. - Conecte la cámara a un sistema operativo y abra el software específico de la cámara (consulte la Tabla de materiales ). Haga clic en la imagen de la cámara debajo de la pestaña "Archivo", en el panel superior, para abrir todas las opciones de la cámara, y elegir la cámara correcta.
- Elija "en vivo" en la lista de pestañas del panel superior dentro del software.
- Ponga la imagen en foco manualmente ajustando el dial de la lente. Seleccione "Recortar en la región de interés (ROI)" del panel superior. Luego, dibuje manualmente un rectángulo en el marco de video, encerrando el dispositivo de actuador biológico y los brazos en voladizo, para marcar el ROI.
Nota: La elección de un ROI adecuado minimiza el tamaño de los archivos de imagen.- En el caso de biorobots, capturar toda la pantalla con el fin de registrar el movimiento de natación del dispositivo.
NOTA: No es necesario hacer un ROI para los biorobots
- En el caso de biorobots, capturar toda la pantalla con el fin de registrar el movimiento de natación del dispositivo.
- Antes de iniciar la grabación, seleccione "Configuración de la cámara" en una de las pestañas del panel superior de la pantalla. Ajuste la velocidad de fotogramas ajustando la exposición y la relación de píxeles de la imagen en vivo deslizando la barra para cada uno o introduciendo manualmente los valores. Ajuste las velocidades de fotogramas a unos 30 ± 2 fps.
Nota: El cambio de la relación de exposición y de píxeles cambia el brillo y el contraste de la imagen en vivo. - Haga clic en el botón "Grabar" del panel superior del software para comenzar a grabar los vídeos de los actuadores con una resolución de 1.000 x 1.000 píxeles durante 30 s. Repita el proceso para todas las muestras.
4. Análisis de imagen de los actuadores biológicos en una base estacionaria
- Analizar las imágenes utilizando un software de programación ( por ejemplo, Matlab) ejecutando el script personalizado. Vea la Tabla de Materiales y el archivo suplementario para más detalles.
Nota: La secuencia de comandos muestra cada trama de los videos grabados, recibe la entrada del ratón del usuario para registrar la coordinación de los puntos del voladizo en las imágenes, calcula el diámetro y el centro del círculo que pasa los puntos introducidos a través del ajuste menos cuadrado y Exporta todos los datos introducidos y calculados para su uso posterior.- Abra el software de programación haciendo clic en el icono. Haga clic en "Archivo" -> "Abrir" en la barra de menú en la parte superior y seleccione el archivo de script. M para el análisis de imágenes. Asegúrese de que las imágenes TIFF grabadas están en la misma carpeta que el archivo .m. Haga clic en "Ejecutar" para ejecutar el script.
Nota: Aparecerá una pantalla interactiva para modificarla. - Pulse "play" para iniciar el programa actual. Haga clic en el botón "Abrir" y busque el archivo TIFF que va a ser analizado.
- Haga clic en "base & #34; Y luego haga clic en el punto donde el voladizo se adhiere a la base en la parte superior; Presione enter. Esto colocará un marcador cuadrado en la imagen para cada marco para indicar la ubicación de la base en voladizo.
- Haga clic en el botón "Escala" y luego haga clic manualmente en un borde de la cuenta de vidrio. Lleve el puntero del ratón al lado opuesto de la cuenta de vidrio y presione "Enter".
Nota: Deberá dibujar una línea que mida el diámetro del cordón de vidrio. Dado que el cordón de vidrio tiene 3 mm de diámetro, esto se relacionará con 3 mm con los píxeles mostrados. - Haga clic en el botón "Analizar". Haga clic a lo largo del voladizo a una corta distancia del primer marcador cuadrado que representa la base en voladizo.
- Haga clic en el botón "Analizar". Luego, haga clic a lo largo del voladizo a una corta distancia del primer marcador cuadrado que representa la base en voladizo. Continúe haciendo clic a lo largo del voladizo, incluyendo la punta, y presione Intro cuandohecho. Esto colocará una "x" en cada punto apretado en el cantilever.
Nota: Basándose en la coordinación del marcador cuadrado y en los marcadores x, el centro y el diámetro de un círculo se calcularán utilizando una función de ajuste de mínimos cuadrados (consulte el archivo suplementario adjunto para el guión utilizado). El círculo, que pasa por los marcadores x y el cuadrado, se superpondrá automáticamente en la imagen. - Compruebe si el círculo superpuesto traza correctamente el perfil en voladizo.
Nota: Cuando el voladizo es muy plano, es difícil juzgar si el perfil en voladizo es trazado correctamente. Consulte la Figura 3 . - Haga clic en el botón "siguiente marco". Esto cambiará a la siguiente trama en el archivo TIFF. La base y la escala ya se han establecido desde el paso anterior.
- Repita los pasos 4.1.5 a 4.1.7 hasta que se hayan completado todos los fotogramas del archivo TIFF. Una vez que todos los fotogramas han sido procesadosSsed, haga clic en el botón "Exportar".
Nota: Esto hará que un archivo de hoja de cálculo con el nombre de archivo TIFF para el voladizo que se acaba de analizar. Edite el nombre del archivo para incluir el lado (izquierdo o derecho) del voladizo que se analizó.
- Abra el software de programación haciendo clic en el icono. Haga clic en "Archivo" -> "Abrir" en la barra de menú en la parte superior y seleccione el archivo de script. M para el análisis de imágenes. Asegúrese de que las imágenes TIFF grabadas están en la misma carpeta que el archivo .m. Haga clic en "Ejecutar" para ejecutar el script.
- Cálculo del estrés en la hoja de cálculo.
- Calcule el esfuerzo superficial, "σ", en el voladizo usando la siguiente ecuación:
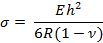
Donde E, R, v , yh son el módulo de Young, el radio de curvatura, la relación de Poisson y el espesor en voladizo, respectivamente.
Nota: El grosor del voladizo puede modificarse para cambiar la sensibilidad. En este estudio, los valores fueron los siguientes: E = 750 kPa, v = 0,49, yh = 25 μm 13 , 14 . - Calcular el esfuerzo superficial usando la ecuación en el paso 4.2.1. Abre el .Xls archivo de hoja de cálculo. Observe que la salida tiene varias columnas que muestran primero las coordenadas xey de base y círculo y luego el radio de curvatura. Calcular las coordenadas x e y de cada punto hecho clic en estas.
Nota: La representación gráfica de las tensiones sobre los fotogramas de imágenes de lapso de tiempo muestra los cambios en la fuerza ejercida sobre el voladizo a lo largo del tiempo. Las depresiones muestran la tensión sobre el voladizo durante la relajación de los cardiomiocitos o la tensión estática ejercida sobre el voladizo debido a las fuerzas de tracción de las células. Los picos muestran la tensión dinámica en el voladizo, que se ejerció por el golpe de los cardiomiocitos. Este valor corresponde a la cantidad máxima de fuerza generada por la contracción de los cardiomiocitos.
- Calcule el esfuerzo superficial, "σ", en el voladizo usando la siguiente ecuación:
5. Análisis de los biorobots de natación
- Registre la posición del biorobot usando el software de análisis de imagen.
Nota: Consulte la lista de Materiales para el softwareEs la sección.- Abra el software de análisis de imágenes ( por ejemplo, ImageJ). Pulse "Archivo" y "Abrir" y seleccione el archivo de vídeo biorobot natación. Haga clic en "Aceptar" y dejar que el programa cargue el archivo. Abra el software de hoja de cálculo.
- En el video biorobot cargado, encuentre una referencia de dimensiones conocidas ( por ejemplo, el cordón de vidrio de 3 mm de diámetro incrustado en el actuador biológico).
Nota: Cualquier objeto con dimensión conocida funcionará. Esto determinará las relaciones de píxeles a longitud en cada vídeo. - Utilice la herramienta "Recta" para dibujar una línea a través de la cuenta de vidrio. Haga clic en "Analizar" y seleccione "Establecer escala". Ajuste el campo "Distancia conocida" a "3000 μm" y haga clic en "Aceptar".
Nota: Esto establecerá las coordenadas x e y en micrómetros. - Elija un punto en el dispositivo que no oscile entre marcos para actuar como un marcador.
Nota:Se recomienda elegir una esquina de la base. - Apunte al punto seleccionado en 5.1.4 en el primer fotograma. Anote las coordenadas x e y en la hoja de cálculo.
- Vuelva a la ventana del software de análisis de imágenes y pulse la tecla de flecha derecha para cambiar al siguiente fotograma. Vuelva a apuntar al marcador (desde el paso 5.1.4) y anote las coordenadas x e y en la hoja de cálculo.
- Repita el paso 5.1.6 para todas las tramas.
- Calcular los parámetros de natación del biorobot utilizando la hoja de cálculo de coordenadas 7 .
- Calcular el período entre fotogramas de la velocidad de fotogramas conocida de cada vídeo.
- Calcule el cambio en las coordenadas x e y entre los fotogramas para encontrar la distancia recorrida, incluyendo la distancia total.
- Calcular la amplitud de la contracción desde el cambio máximo a lo largo del eje y. Determine la frecuencia de golpeo para cada biorobote de la inversa del periodo entre dos contracciones.
- doCalcular la velocidad de natación de cada dispositivo desde el tiempo total y la distancia movida en la dirección x.
- Repita el paso 5.2 para cada video biorobot que se analizó.
- Normalizar cada parámetro medido.
Nota: Normalizar todos los valores para visualizar mejor las diferencias. Este protocolo demuestra la normalización con respecto a un modo horizontal biorobot con baja frecuencia contracciones (LF horizontal) ( Figura 4 ] [ 7] .
6. Análisis de la expresión de proteínas
Nota: Las muestras montadas preparadas en los pasos 2.2.4 y 2.2.5 se visualizaron utilizando un microscopio confocal. Las imágenes se adquirieron a una amplificación de 20X, 40X y 60X secuencialmente en tres canales simultáneamente: 460 nm, 488 nm y 594 nm. Un conjunto de 5 imágenes fueron capturadas a una ampliación de 40X, de diferentes posiciones para cada muestra, y cada canal se guardó como un individuo.archivo. El ajuste de la exposición se determinó por la ampliación del objetivo utilizado y se estableció constante para todas las capturas en esa ampliación.
- Abra el software de análisis de imágenes y seleccione "Archivo" -> "Abrir" para cargar las imágenes.
- Dibuja un polígono rectangular en el marco de la imagen para marcar el ROI. Seleccione "Analizar" -> "Medir" para medir la intensidad fluorescente media.
- Repita el paso 6.2 para recoger mediciones de intensidad de todas las muestras y calcular la intensidad media respectiva para cada condición.
Nota: Aquí, una condición diferente se refiere a diferentes puntos temporales, como en, el día 1, el día 2 y hasta el día 6. - Exporte los resultados en una hoja de cálculo para un análisis estadístico adicional y para la producción de gráficos de datos.
Resultados
El actuador biológico constituido por un ligero voladizo PDMS (25 μm de espesor) y cardiomiocitos constituye el núcleo del biorobot de natación, como se muestra en el esquema y captura de pantalla de los dispositivos de la Figura 1 . Las células comienzan a mostrar contracciones después de 24 h en cultivo, y la flexión de los brazos en voladizo se observó el día 2. El perfil lateral del dispositivo se registró todos los días y el esfuerzo superficial se cuanti...
Discusión
El procedimiento descrito aquí describe un método de siembra exitoso para actuadores basados en PDMS y biorobots, lo que facilita la unión de cardiomiocitos. Además, se describió el proceso de adquisición de imágenes y el posterior análisis que caracteriza el comportamiento de las células y el rendimiento de los dispositivos.
Observamos la contracción espontánea de las células en los brazos en voladizo después de 24 h; La intensidad de las contracciones continuó aumentand...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
MT Holley es apoyado por el programa Graduate Fellows de la Junta de Regentes de Louisiana, y C. Danielson es apoyado por el Programa de Profesores del Instituto Médico Howard Hughes. Este estudio es apoyado por NSF Grant No: 1530884.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Cardiomyocytes (primary cardiac cells) | Charles River | NA | Isolated from 2-day old neonatal Sprague Dawley rats |
| Dulbecco’s modified eagle’s media (DMEM) | Hyclone Laboratories | 16750-074 | with 4500 mg/L glucose, 4.0 mM L-glutamine, and 110 mg/L sodium pyruvate |
| Fetalclone III serum | Hyclone industries, GE | 16777-240 | Fetal bovin serum (FBS) |
| Dulbecco’s phosphate buffer (PBS) | Sigma-Aldrich | D1408-100ML | |
| Penicillin-G sodium salt | Sigma-Aldrich | P3032 | |
| Goat serum | Sigma-Aldrich | G9023 | |
| 4,6-diamidino-2-phenylindole dihydrocholride powder (DAPI) | Sigma-Aldrich | D9542 | |
| Fibronectin from bovine plasma | Sigma-Aldrich | F1141 | Solution (1 mg/ml) |
| Calcein-AM and ethidium homodimer-1 kit (Live/Dead Assay) | Molecular Probes | L3224 | |
| Calcium Fluo-4, AM | Molecular Probes | F14217 | calcium indicator dye |
| Tyrodes salt solution | Sigma-Aldrich | T2397 | buffer solution |
| Pluronic F-127 | Molecular Probes | P3000MP | nonionic surfactant-20 % solution in Dimethylsiloxane (DMSO) |
| 16% Parafomaldehyde | Electron microscopy | 15710 | Caution: Irritant and combustible |
| Triton x-100 | Sigma-Aldrich | X-100 100 mL | cell lyses detergent, (4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether) |
| ProLong gold antifade reagent | Molecular Probes | P10144 | Mounting agent |
| Alexa Fluor 594 Phalloidin | Molecular Probes | A12381 | Actin filament marker |
| Goat anti-rabbit IgG (H+L) secondary antibody, Alexa Fluor 594 conjugate | Molecular Probes | A-11012 | |
| pha | Molecular Probes | A-11001 | |
| Anti-connexin 43 antibody | Abcam | ab11370 | Gap junction marker |
| Anti-cardiac troponin I antibody | Abcam | ab10231 | Contractile protein |
| 16% EM grade paraformaldehyde solution | Electron microscopy | 100503-916 | |
| Polydimethylsiloxane (PDMS) | Elsevier | Sylgard 184 | |
| Materials and Equipment | |||
| Camera | Thor Labs | DCC1545M | |
| LED light strip | NA | NA | Any white LED without spectrum emission |
| Confocal microscope | Nikkon C2 | NA | Confocal microscope with three filter set. |
| Zooming lens | Infinity | Model# 252120 | |
| Software | |||
| Matlab | Mathworks | NA | Used in Section 4) for biological actuator analysis. |
| Image J | National Institute of Health | NA | Java-based image processing software. Used in Section 5) for biorobot analysis. Free Image Processing and Analysis software in java. (https://imagej.nih.gov/ij/) |
| Thor Cam | Thor Labs | NA | Camera operating software |
Referencias
- Feinberg, A. W. Biological Soft Robotics. Annu. Rev. Biomed. Eng. 17, 243-265 (2015).
- Akiyama, Y., et al. Room Temperature Operable Autonomously Moving Bio-Microrobot Powered by Insect Dorsal Vessel Tissue. PLOS ONE. 7, 38274 (2012).
- Herr, H., Dennis, R. G. A swimming robot actuated by living muscle tissue. J. NeuroEng Rehabil. 1, 6 (2004).
- Park, S., et al. Phototactic guidance of a tissue-engineered soft-robotic ray. Science. 353 (6295), 158-162 (2016).
- Cvetkovic, C., et al. Three-dimensionally printed biological machines powered by skeletal muscle. Proc. Natl. Acad. Sci. 111, 10125-10130 (2014).
- Williams, B. J., Anand, S. V., Rajagopalan, J., Saif, M. T. A. A self-propelled biohybrid swimmer at low Reynolds number. Nat. Commun. 5, (2014).
- Holley, M. T., Nagarajan, N., Danielson, C., Zorlutuna, P., Park, K. Development and characterization of muscle-based actuators for self-stabilizing swimming biorobots. Lab. Chip. 16, 3473-3484 (2016).
- Hopkins, P. M. Skeletal muscle physiology. Contin Educ Anaesth Crit Care Pain. 6, 1-6 (2006).
- Nawroth, J., et al. A tissue-engineered jellyfish with biomimetic propulsion. Nat Biotechnol. 30 (8), 729-797 (2012).
- Ehler, E., Moore-Morris, T., Lange, S. Isolation and Culture of Neonatal Mouse Cardiomyocytes. J. Vis. Exp. JoVE. (79), e50154 (2013).
- Bers, D. M. Calcium Fluxes Involved in Control of Cardiac Myocyte Contraction. Circ. Res. 87, 275-281 (2000).
- Shin, S. R., et al. Carbon-Nanotube-Embedded Hydrogel Sheets for Engineering Cardiac Constructs and Biological actuators. ACS Nano. 7, 2369-2380 (2013).
- Park, J., et al. Real-Time Measurement of the Contractile Forces of Self-Organized Cardiomyocytes on Hybrid Biopolymer Microcantilevers. Anal. Chem. 77, 6571-6580 (2005).
- Tamayo, J., et al. Quantification of the surface stress in microcantilever biosensors: revisiting Stoney's equation. Nanotechnology. 23, 475702 (2012).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat. Methods. 10, 781-787 (2013).
- Louch, W. E., Sheehan, K. A., Wolska, B. M. Methods in Cardiomyocyte Isolation, Culture, and Gene Transfer. J. Mol. Cell. Cardiol. 51, 288-298 (2011).
- Alford, P. W., Feinberg, A. W., Sheehy, S. P., Parker, K. K. Biohybrid thin films for measuring contractility in engineered cardiovascular muscle. Biomaterials. 31, 3613-3621 (2010).
- Sfakiotakis, M., Lane, D. M., Davies, J. B. C. Review of fish swimming modes for aquatic locomotion. IEEE J. Ocean. Eng. 24, 237-252 (1999).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados