Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Herzmuskelzell-basierter Aktuator und selbststabilisierender Biorobot - Teil 2
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In dieser Studie werden ein biologischer Aktuator und ein selbststabilisierender, schwimmender Biorobot mit funktionalisierten elastomeren Cantileverarmen mit Kardiomyozyten ausgesät, kultiviert und charakterisiert für ihre biochemischen und biomechanischen Eigenschaften über die Zeit.
Zusammenfassung
In den letzten Jahren wurden Hybrid-Geräte entwickelt, die aus einer lebenden Zelle oder Gewebekomponente bestehen, die mit einem synthetischen mechanischen Rückgrat integriert ist. Diese Vorrichtungen, die sogenannten Biorobots, werden allein durch die Kraft, die aus der kontraktilen Aktivität der lebenden Komponente hervorgerufen wird, angetrieben und können aufgrund ihrer vielen inhärenten Vorteile eine Alternative zu herkömmlichen, vollständig künstlichen Robotern sein. Hier beschreiben wir die Methoden, um einen biologischen Aktuator und einen Biorobot zu seedieren und zu charakterisieren, der im ersten Teil dieses zweiteiligen Artikels entworfen, gefertigt und funktionalisiert wurde. Gefertigte biologische Aktuatoren und Biorobot-Vorrichtungen, die aus einer Polydimethylsiloxan-Basis (PDMS) und einem Dünnfilm-Cantilever zusammengesetzt waren, wurden für die Zelladhäsion mit Fibronektin funktionalisiert. Nach der Funktionalisierung wurden neonatale Ratten-Kardiomyozyten auf den PDMS-Cantilever-Arm mit hoher Dichte ausgesät, was zu einer konfluenten Zellfolie führte. Die Geräte wurden jeden Tag abgebildet und die Bewegung der CantiHebelarme wurde analysiert. Am zweiten Tag nach dem Seeding beobachteten wir die Biegung der Cantilever-Arme aufgrund der Kräfte, die von den Zellen während der spontanen Kontraktionen ausgeübt wurden. Bei der quantitativen Analyse der Cantilever-Biegung wurde eine allmähliche Zunahme der Oberflächenspannung, die von den Zellen ausgeübt wurde, während sie im Laufe der Zeit reiften, beobachtet. Ebenso beobachteten wir die Bewegung des Biorobots aufgrund der Betätigung des PDMS-Cantileverarms, der als Flosse fungierte. Bei der Quantifizierung der Schwimmprofile der Geräte wurden verschiedene Antriebsmodi beobachtet, die durch den Ruhewinkel der Rippe beeinflusst wurden. Die Bewegungsrichtung und die Schlagfrequenz wurden auch durch den Ruhewinkel der Rippe bestimmt und es wurde eine maximale Schwimmgeschwindigkeit von 142 μm / s beobachtet. In diesem Manuskript beschreiben wir die Vorgehensweise für die Bevölkeung der gefertigten Geräte mit Kardiomyozyten sowie für die Beurteilung der biologischen Aktor- und Biorobot-Aktivität.
Einleitung
Biorobots sind Geräte, die auf lebenden Zellen basieren, die in einem mechanischen Rückgrat eingebaut sind, das üblicherweise aus weichen, elastischen Materialien wie PDMS oder Hydrogelen besteht. Die Zellen unterliegen rhythmischen Kontraktionen, entweder spontan oder in Reaktion auf Reize und fungieren somit als Aktuator. Die aus der Zellkontraktion erzeugte Kraft treibt verschiedene Biorobots an. Säugetierherzzellen (Kardiomyozyten) und Skelettmuskelzellen werden häufig aufgrund ihrer kontraktilen Eigenschaften für die Biorobot-Betätigung verwendet. Abgesehen von Kardiomyozyten- und Skelettmuskelzellen wurden auch andere Zelltypen wie Insektenmuskelgewebe 2 und explantierte Muskelgewebe 3 verwendet. Insektenmuskelgewebe ermöglichen den Betrieb von biologischen Aktoren bei Raumtemperatur.
Die Funktion und die Leistungsfähigkeit eines Biorobots werden hauptsächlich durch die Festigkeit und Konsistenz des biologischen Aktors bestimmt ( dh. Muskelzellen), während die mechanische Grundgerüststruktur in erster Linie die Mechanismen der Fortbewegung, der Stabilität und der Kraft bestimmt. Da diese Geräte ausschließlich durch von Zellen erzeugte Kräfte angetrieben werden, gibt es keine chemischen Schadstoffe oder Betriebsgeräusche. Deshalb bilden sie eine energieeffiziente Alternative zu anderen konventionellen Robotern. Verschiedene Literaturquellen haben die verschiedenen Methoden zur Integration lebender Zellen und Gewebe in Bioroboter 1 , 4 , 5 diskutiert. Fortschritte in der Mikrofabrikation und Tissue Engineering Techniken haben die Entwicklung von Bioroboten, die zu Fuß, Griff, Schwimmen oder Pumpen 5 , 6 ermöglicht. Im Allgemeinen werden Zellen direkt auf das mechanische (polymere) Rückgrat als konfluierende Zellfolie kultiviert oder sie werden in dreidimensionale Betätigungsstrukturen in Gerüsten wie Ringen und Streifen geformt. Am häufigsten sind BiorobotsHergestellt unter Verwendung von Kardiomyozytenblättern 6 , 7 , da diese Zellen eine angeborene Fähigkeit haben, eine spontane Kontraktion ohne äußere Reize zu zeigen. Auf der anderen Seite sind Berichte über Skelettmuskelzellfolien aufgrund ihres Bedarfs an Stimuli begrenzt, um Kontraktionen in vitro einzuleiten , um die Membrandepolarisation zu initiieren 8 .
Dieses Protokoll beschreibt zunächst, wie man Kardiomyozyten auf einem funktionalisierten biologischen Aktuator aus einem dünnen PDMS-Cantilever aussieht. Es beschreibt dann im Detail die Aussaat und Analyse der Schwimmprofile. Der Cantilever ist mit einem Zellklebstoffprotein wie Fibronectin funktionalisiert und wird konsequent mit Kardiomyozyten ausgesät. Als die Zellen auf dem Gerät vertreten, veranlassen sie den Cantilever zu biegen und damit als Aktuator zu handeln. Im Laufe der Zeit, wie die Zellen reifen, verfolgen wir die Veränderungen der Oberflächenspannung auf dem Gerät durch die Analyse von Videos derFreitragende Biegung. Der hier entwickelte biologische Aktuator kann zur Bestimmung der kontraktilen Eigenschaften eines beliebigen Zelltyps, wie der Fibroblasten oder induzierten pluripotenten Stammzellen, verwendet werden, da sie einer Differenzierung unterliegen.
Ein Großteil der früheren Forschung auf Biorobots konzentrierte sich auf die Entwicklung biologischer Aktoren, während die Optimierung der Biorobot-Architektur und der funktionalen Fähigkeiten weitgehend vernachlässigt wurde. Vor kurzem haben einige Studien die Umsetzung von Schwimmmodi in Bioroboten gezeigt, die von der Natur inspiriert sind. Zum Beispiel wurden schwimmende Biorobots mit Flagella-basierter Bewegung 6 , Quallenantrieb 9 und Bio-Hybridstrahlen 4 entwickelt. Im Gegensatz zu anderen Werken in der Literatur konzentrieren wir uns hier auf die Veränderung der Eigenschaften des mechanischen Rückgrats, um eine selbststabilisierende Struktur zu schaffen. Der in dieser Studie entwickelte Biorobot ist in der Lage, eine konstante Tonhöhe, Rolle und Im aufrechtzuerhaltenMersionstiefe, wie sie schwimmt Diese Parameter können durch Variieren der Dicke jedes Basiskomposits modifiziert werden. Die Fertigungsschritte, die bei der Entwicklung des PDMS-Aktuators, des untertauchbaren Biorobots und der Funktionalisierung des Gerätes beteiligt sind, sind in Teil 1 dieses zweiteiligen Artikels sowie in unserer neueren Arbeit 7 ausführlich beschrieben. Die hier entwickelte Technik kann die Weg für die Entwicklung von neuartigen, hocheffizienten Bioroboten für verschiedene Anwendungen wie Frachtlieferung.
Der in dieser Studie verfolgte Isolierungsprozess ähnelt dem in einem früheren Werk 10 beschriebenen Verfahren sowie in der kürzlich veröffentlichten Arbeit 7 . Die für die Herstellung der PDMS-Aktoren und Biorobot-Geräte verwendeten Mikrofabrikationsverfahren sind in Teil 1 dieses zweiteiligen Manuskripts ausführlich beschrieben. Der Protokollabschnitt dieses Manuskripts beschreibt die Schritte, die bei der Aussaat von Kardiomyozyten auf das hergestellte PDMS a auftretenCtuator und der biorobot nach ihrer Funktionalisierung mit Zelladhäsivproteinen.
Protokoll
Alle hier beschriebenen Verfahren wurden mit einem genehmigten Protokoll und gemäß den Bestimmungen des Institutional Animal Care and Use Committee der Universität von Notre Dame durchgeführt.
1. Zellseeding und Kultur
- Bevor Sie beginnen, bereiten Sie die benötigten Gegenstände vor: einen kleinen Trichter, Pipetten und warmes Dulbecco's Modified Eagle Medium (DMEM), ergänzt mit 10% Fetal Bovine Serum (FBS) und 1% Penicillin Antibiotikum (DMEM komplett).
- Nehmen Sie T-25 Flaschen zusammen mit dem funktionalisierten Gerät (biologischer Aktor oder dem Biorobot) darin. Siehe Abschnitt 4 des Teils 1 dieses zweiteiligen Manuskripts für Einzelheiten über die Vorbereitung der Vorrichtung, die Funktionalisierung und die Lagerung vor dem Zelsen.
- Bereiten Sie einen Trichter vor, der durch Rollen einer kleinen, quadratischen Plastikfolie hergestellt werden kann. Legen Sie es über den biologischen Aktor oder Biorobot im T-25-Kolben. Stellen Sie den Durchmesser des breiteren Endes auf das gesamte Gerät einUnd die Höhe, so dass es eng anliegt, wenn die Oberseite des Kolbens angezogen ist.
- Für die Biorobots, verwenden Sie einen Magneten, um das Gerät an der Unterseite des Kolbens während des Saatprozesses zu halten.
Anmerkung : Hier wurde ein einzelner Neodym-Scheibenmagnet (1,26 "im Durchmesser) verwendet, aber jeder Magnet von ähnlicher Größe und Stärke kann auch verwendet werden, um den Biorobot mit der magnetischen Nickel-PDMS-Verbundbasis zu halten. - UV-Sterilisation der Plastikfolie für mindestens 30 Minuten vor Gebrauch.
- Für die Biorobots, verwenden Sie einen Magneten, um das Gerät an der Unterseite des Kolbens während des Saatprozesses zu halten.
- Stellen Sie sicher, dass es keine große Lücke zwischen der Basis des Trichters und der Flasche gibt.
- Die Kardiomyozyten in DMEM mit einer Dichte von 1,6 x 10 & sup7; Zellen / ml resuspendieren und langsam 400 & mgr; l Suspension auf dem Gerät durch den Trichter fallen lassen. Verwenden Sie ein Hämocytometer oder einen anderen Zellzähler, um die Anzahl der erhaltenen Zellen zu bestimmen.
- Ziehen Sie das System langsam in den Inkubator zurück, ohne das Gerät und den Trichter zu störenHin. Kultur für 24 h bei 37 ° C.
- Nach der Inkubationszeit langsam den Trichter entfernen, die Probe vorsichtig mit PBS waschen und den Kolben mit 10 ml frischem DMEM abfüllen.
Hinweis: Für die Biorobots, entfernen Sie den Magneten, so dass das Gerät flott ist.
2. Biochemische Charakterisierung
- Calciumfluss-Test
Hinweis: Der Calcium-Flux-Assay wird durchgeführt, um die Zell-Interkonnektivität zu beurteilen. Das Verfahren zum Beladen der Zellen mit dem fluoreszierenden, Calciumionen-spezifischen Farbstoff folgt dem in einem zuvor etablierten Protokoll 11 beschriebenen Verfahren.- Zuerst die erforderlichen Materialien, Calciumfluo-4-acetomethyl (AM) ester, nichtionisches Tensidpolyol (siehe Tabelle der Materialien ) und Tyrode- Salzlösung vorbereiten .
- Mit langen Pinzetten wird das Gerät vorsichtig aus dem Kulturkolben auf eine 35-mm-Petrischale mit 2 ml Tyrode-Salz-Solu übertragen .
- In einem separaten Zentrifugenröhrchen 1 ml Warm-Tyrode-Lösung (auf 37 ° C erwärmt) aufnehmen und 3-5 μl Lager-Calcium-Fluo-4-AM-Farbstoff (Arbeitskonzentration: 3-5 μM) und einen gleichen Teil des nichtionischen Tensids zugeben Polyol (Arbeitskonzentration: 0,2%). Ersetzen Sie die Probenlösung mit warmem Tyrode-Lösung, ergänzt mit dem Calcium-Indikator-Farbstoff, Fluo-4 und 0,2% nichtionischem Tensid. Inkubieren für 25 - 30 min bei 37 ° C.
- Entfernen Sie die Farbstofflösung und waschen Sie die Probe vorsichtig mit frischer Tyrode-Lösung. Re-inkubieren Sie die Probe in 2 ml frischem DMEM für weitere 30 min bei 37 ° C vor der Bildgebung.
Anmerkung: Die Ergebnisse dieses Assays und verwandten Videos werden in der zuvor veröffentlichten Arbeit zur Verfügung gestellt 7 .
- Immunfluoreszenz
Anmerkung : Die doppelte Immunfärbung aller Proben wurde nach vorher festgelegtem Protokoll durchgeführt> 12- Zuerst 10% Ziegenserum (GS) in phosphatgepufferter Kochsalzlösung (PBS), 4% Paraformaldehyd (PFA) in DiH 2 O, 0,1% Zelllyse-Detergens (siehe Tabelle der Materialien ) in PBS, primäre Antikörper (anti - monoklonale Antikörper-Herz-Troponin-I und anti-Kaninchen-monoklonale Antikörper-Connexine-43), sekundäre Antikörper (Alexa 594-Konjugat und Ziegen-anti-Maus-IgG (H + L) Alexa 488-Konjugat) und DAPI.
Achtung: Paraformaldehyd ist krebserregend. - Entfernen Sie die Probe von Interesse aus dem Kolben und waschen Sie es zweimal mit PBS. Siehe Abschnitt 4 in Teil 1 dieses zweiteiligen Manuskripts für Details zur Probenvorbereitung und Funktionalisierung.
- Füge ein Tröpfchen PBS auf ein kleines Deckglas (Durchmesser: 12 mm oder 15 mm). Halten Sie die Basis des Gerätes vorsichtig mit einer Pinzette und schneiden Sie die dünnen PDMS-Arme (Cantilever, Abbildung 1 ) mit einer Schere von dem Ende, an dem sie mit der Oberseite des Basels verbunden istE. Übertragen Sie die freitragenden Arme auf das Tröpfchen, wobei die zellseitige Seite nach oben zeigt. Das PBS-Tröpfchen verhindert, dass die Zellen trocknen.
- Fixieren Sie die Proben mit 4% PFA und führen Sie eine doppelte Immunfärbung der Proben durch, wie zuvor beschrieben 12 .
- Nach der Immunfärbung die Proben auf eine saubere Glasrutsche mit Anti-Fade-Montagereagenz aufsetzen und ungestört im Dunkeln für 24 h beiseite stellen.
- Wiederholen Sie den Vorgang für alle Proben.
Anmerkung : Die Ergebnisse dieses Assays und die zugehörigen Bilder werden in der zuvor veröffentlichten Arbeit ausführlich diskutiert 7 .
- Zuerst 10% Ziegenserum (GS) in phosphatgepufferter Kochsalzlösung (PBS), 4% Paraformaldehyd (PFA) in DiH 2 O, 0,1% Zelllyse-Detergens (siehe Tabelle der Materialien ) in PBS, primäre Antikörper (anti - monoklonale Antikörper-Herz-Troponin-I und anti-Kaninchen-monoklonale Antikörper-Connexine-43), sekundäre Antikörper (Alexa 594-Konjugat und Ziegen-anti-Maus-IgG (H + L) Alexa 488-Konjugat) und DAPI.
3. Bildgebung
- Den T-25-Kolben aufrecht in einen CO 2 -Inkubator stellen und das Imaging-System im Inkubator vorbereiten. Notieren Sie das Gerät mit einer Kamera (siehe Tabelle der Materialien ) mit einem Zoomobjektiv (siehe Tabelle der Materialien ). Für eine Lichtquelle,Verwenden Sie einen Streifen von LED-Leuchten.
Hinweis: Hier wurde ein Streifen von Weißlicht-LEDs verwendet, aber auch normale LEDs funktionieren. - Verbinden Sie die Kamera mit einem Betriebssystem und öffnen Sie die kameraspezifische Software (siehe Tabelle der Materialien ). Klicken Sie auf das Kamerabild unterhalb der Registerkarte "Datei", im oberen Bereich, um alle Kameraoptionen zu öffnen und die richtige Kamera auszuwählen.
- Wählen Sie "live" aus der Liste der Registerkarten auf der Oberseite innerhalb der Software.
- Manuell das Bild in den Fokus bringen, indem man das Objektiv wählt. Wählen Sie "Crop to region of interest (ROI)" aus dem oberen Panel. Dann manuell ein Rechteck in den Videorahmen ziehen, das die biologische Aktuatoreinrichtung und die freitragenden Arme umschließt, um den ROI zu markieren.
Hinweis: Die Auswahl eines geeigneten ROI minimiert die Größe der Bilddateien.- Im Falle von Bioroboten, erfassen Sie den ganzen Bildschirm, um die Schwimmbewegung des Gerätes aufzunehmen.
HINWEIS: Es besteht keine Notwendigkeit, einen ROI für die Biorobots zu zeichnen
- Im Falle von Bioroboten, erfassen Sie den ganzen Bildschirm, um die Schwimmbewegung des Gerätes aufzunehmen.
- Bevor Sie die Aufnahme starten, wählen Sie "Kameraeinstellungen" von einer der Registerkarten im oberen Bedienfeld auf dem Bildschirm. Legen Sie die Bildrate fest, indem Sie das Belichtungs- und Pixelverhältnis des Livebildes einstellen, indem Sie die Leiste für jedes oder durch manuelles Eingeben der Werte schieben. Stellen Sie die Bildraten auf ca. 30 ± 2 fps ein.
Hinweis: Das Ändern des Belichtungs- und Pixel-Verhältnisses verändert die Helligkeit und den Kontrast des Live-Bildes. - Klicken Sie auf die Schaltfläche "Record" von der Oberseite in der Software, um die Aufnahme der Videos der Aktoren mit 1.000 x 1.000 Pixel Auflösung für genau 30 s zu starten. Wiederholen Sie den Vorgang für alle Proben.
4. Bildanalyse der biologischen Aktoren auf einer stationären Basis
- Analysiere die Bilder mit einer Programmiersoftware ( zB Matlab) mit dem benutzerdefinierten Skript. Siehe die Tabelle der Materialien Rong > und die ergänzende Datei für weitere Details.
Anmerkung : Das Skript zeigt jeden Rahmen der aufgezeichneten Videos an, empfängt den Maus-Eingang des Benutzers, um die Koordination der Punkte des Cantilevers auf den Bildern aufzuzeichnen, berechnet den Durchmesser und die Mitte des Kreises, der die eingegebenen Punkte über die kleinste quadratische Anpassung passiert Exportiert alle eingegebenen und berechneten Daten zur weiteren Verwendung.- Öffnen Sie die Programmiersoftware, indem Sie auf das Symbol klicken. Klicken Sie auf "Datei" -> "Öffnen" aus der Menüleiste oben und wählen Sie die .m Skriptdatei für die Bildanalyse. Stellen Sie sicher, dass die aufgezeichneten TIFF-Bilder im selben Ordner wie die .m-Datei sind. Klicken Sie auf "Ausführen", um das Skript auszuführen.
Hinweis: Eine interaktive Anzeige erscheint zum Ändern. - Drücken Sie "play", um das aktuelle Programm zu starten. Klicken Sie auf die Schaltfläche "Öffnen" und suchen Sie die TIFF-Datei, die analysiert werden soll.
- Klicken Sie auf die "base & #34; Und klicken Sie dann auf den Punkt, an dem der Cantilever an der Basis an der Oberseite befestigt ist. Drücken Sie Enter. Dadurch wird eine quadratische Markierung auf das Bild für jeden Rahmen gesetzt, um die Lage der Cantilever-Basis zu bezeichnen.
- Klicken Sie auf die Schaltfläche "Scale" und klicken Sie dann manuell auf eine Kante der Glasperle. Bringen Sie den Mauszeiger auf die gegenüberliegende Seite der Glasperle und drücken Sie "Enter".
Hinweis: Dies sollte eine Linie zeichnen, die den Durchmesser der Glasperle misst. Da die Glasperle 3 mm im Durchmesser ist, bezieht sich dies auf 3 mm auf die angezeigten Pixel. - Klicken Sie auf die Schaltfläche "Analysieren". Klicken Sie auf den Cantilever in kurzer Entfernung von der ersten quadratischen Markierung, die die Cantilever-Basis darstellt.
- Klicken Sie auf die Schaltfläche "Analysieren". Dann klicken Sie auf den Cantilever in kurzer Entfernung von der ersten quadratischen Markierung, die die Cantilever-Basis darstellt. Fahren Sie weiter auf den Cantilever, einschließlich der Spitze, und drücken Sie Enter, wennerledigt. Hier wird ein "x" auf jeden Punkt gelegt, der auf den Cantilever geklickt wird.
Hinweis: Basierend auf der Koordination der quadratischen Markierung und der x-Marker werden die Mitte und der Durchmesser eines Kreises mit einer kleinsten quadratischen Anpassungsfunktion berechnet (siehe die beigefügte Zusatzdatei für das verwendete Skript). Der Kreis, der die x-Marker und den Quadratmacher übergibt, wird dem Bild automatisch überlagert. - Prüfen Sie, ob der überlagerte Kreis das freitragende Profil korrekt verfolgt.
Hinweis: Wenn der Cantilever sehr flach ist, ist es schwer zu beurteilen, ob das Cantilever-Profil korrekt verfolgt wird. Siehe Abbildung 3 . - Klicken Sie auf die Schaltfläche "Nächster Rahmen". Dies wechselt zum nächsten Frame in der TIFF-Datei. Die Basis und die Skala sind bereits aus dem vorherigen Schritt eingestellt.
- Wiederholen Sie die Schritte 4.1.5 bis 4.1.7, bis alle Frames in der TIFF-Datei abgeschlossen sind. Sobald alle Frames gewesen sindSsed, klicken Sie auf die Schaltfläche "Exportieren".
Hinweis: Damit wird eine Tabellenkalkulation mit dem TIFF-Dateinamen für den gerade analysierten Cantilever erstellt. Bearbeiten Sie den Dateinamen, um die Seite (links oder rechts) des Cantilevers zu analysieren.
- Öffnen Sie die Programmiersoftware, indem Sie auf das Symbol klicken. Klicken Sie auf "Datei" -> "Öffnen" aus der Menüleiste oben und wählen Sie die .m Skriptdatei für die Bildanalyse. Stellen Sie sicher, dass die aufgezeichneten TIFF-Bilder im selben Ordner wie die .m-Datei sind. Klicken Sie auf "Ausführen", um das Skript auszuführen.
- Berechnen von Stress in der Kalkulationstabelle
- Berechnen Sie die Oberflächenspannung, "σ", auf dem Cantilever mit der folgenden Gleichung:
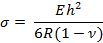
Wobei E, R, v und h der Elastizitätsmodul, der Krümmungsradius, das Poissonsche Verhältnis und die Auslegerdicke sind.
Hinweis: Die Dicke des Cantilevers kann geändert werden, um die Empfindlichkeit zu ändern. In dieser Studie waren die Werte wie folgt: E = 750 kPa, v = 0,49 und h = 25 μm 13 , 14 . - Berechnen Sie die Oberflächenspannung mit der Gleichung in Schritt 4.2.1. Öffne das .Xls Spreadsheet-Datei. Beachten Sie, dass die Ausgabe mehrere Spalten hat, die zuerst die x- und y-Koordinaten von Basis und Kreis und dann den Krümmungsradius anzeigen. Berechnen Sie die x- und y-Koordinaten jedes darauf angeklickten Punktes.
Anmerkung : Das Plotten der Spannungen über die Zeitraffer-Bildrahmen zeigt Veränderungen in der Kraft, die auf den Ausleger über die Zeit ausgeübt wird. Die Mulden zeigen die Belastung des Auslegers während der Entspannung der Kardiomyozyten oder die statische Belastung, die auf den Cantilever aufgrund von Zelle Zugkräften ausgeübt wird. Die Peaks zeigen den dynamischen Stress auf den Cantilever, der durch das Schlagen der Kardiomyozyten ausgeübt wurde. Dieser Wert entspricht der maximalen Kraftmenge, die durch die Kontraktion der Kardiomyozyten erzeugt wird.
- Berechnen Sie die Oberflächenspannung, "σ", auf dem Cantilever mit der folgenden Gleichung:
5. Analyse der Schwimmen Biorobots
- Notieren Sie die Position des Biorobots mit der Bildanalyse-Software.
Hinweis: Siehe Materialliste für die in thIst Abschnitt.- Öffnen Sie die Bildanalyse-Software ( zB ImageJ). Drücken Sie "Datei" und "Öffnen" und wählen Sie die schwimmende Biorobot-Videodatei. Klicken Sie auf "OK" und lassen Sie das Programm die Datei laden. Öffnen Sie die Tabellenkalkulationssoftware.
- Im beladenen Biorobot-Video finden Sie einen Hinweis auf bekannte Abmessungen ( z. B. die Glasperle mit 3 mm Durchmesser , die in den biologischen Aktuator eingebettet wurde).
Hinweis: Jedes Objekt mit bekannter Dimension wird funktionieren. Dies bestimmt die Pixel-zu-Länge-Verhältnisse in jedem Video. - Verwenden Sie das "Gerade" Werkzeug, um eine Linie über die Glasperle zu ziehen. Klicken Sie auf "Analysieren" und wählen Sie "Skala einstellen". Setzen Sie das Feld "Bekannte Distanz" auf "3.000 μm" und klicken Sie auf "Ok".
Hinweis: Damit werden die x- und y-Koordinaten auf Mikrometer eingestellt. - Wählen Sie einen Punkt auf dem Gerät, das nicht zwischen Frames wackelt, um als Marker zu fungieren.
Hinweis:Es empfiehlt sich, eine Ecke der Basis zu wählen. - Zeigen Sie auf den ausgewählten Punkt in 5.1.4 auf dem ersten Frame. Notieren Sie die x- und y-Koordinaten auf der Kalkulationstabelle.
- Wechseln Sie zurück zum Bildanalyse-Softwarefenster und drücken Sie die rechte Pfeiltaste, um zum nächsten Frame zu wechseln. Zeigen Sie auf den Marker erneut (ab Schritt 5.1.4) und notieren Sie die x- und y-Koordinaten auf der Kalkulationstabelle.
- Wiederholen Sie Schritt 5.1.6 für alle Frames.
- Berechnen Sie die Schwimmparameter des Biorobots mit der Kalkulationstabelle der Koordinaten 7 .
- Berechnen Sie die Periode zwischen den Frames aus der bekannten Bildrate jedes Videos.
- Berechnen Sie die Änderung der x- und y-Koordinaten zwischen den Frames, um die bewegte Distanz zu finden, einschließlich der Gesamtstrecke.
- Berechnen Sie die Amplitude der Kontraktion von der maximalen Veränderung entlang der y-Achse. Bestimmen Sie die Schlagfrequenz für jeden Biorobot aus der Umkehrung der Periode zwischen zwei Kontraktionen.
- CBerechnen Sie die Schwimmgeschwindigkeit jedes Gerätes von der Gesamtzeit und dem Abstand, der in der x-Richtung bewegt wird.
- Wiederholen Sie Schritt 5.2 für jedes Biorobot-Video, das analysiert wurde.
- Normalisieren Sie jeden gemessenen Parameter.
Hinweis: Normalisieren Sie alle Werte, um die Unterschiede besser zu visualisieren. Dieses Protokoll zeigt die Normalisierung in Bezug auf einen horizontalen Modus Biorobot mit niederfrequenten Kontraktionen (horizontale LF) (Abbildung 4 ) 7 .
6. Analyse der Protein-Expression
Hinweis: Die in den Schritten 2.2.4 und 2.2.5 hergestellten Messproben wurden mit einem konfokalen Mikroskop abgebildet. Bilder wurden bei 20X, 40X und 60X Vergrößerung sequentiell in drei Kanälen gleichzeitig aufgenommen: 460 nm, 488 nm und 594 nm. Ein Satz von 5 Bildern wurde bei 40facher Vergrößerung von verschiedenen Positionen für jede Probe erfasst und jeder Kanal wurde als individueller TIFF gespeichertDatei. Die Belichtungseinstellung wurde durch die Vergrößerung des verwendeten Objektivs bestimmt und wurde für alle Fänge bei dieser Vergrößerung konstant gesetzt.
- Öffnen Sie die Bildanalyse-Software und wählen Sie "Datei" -> "Öffnen", um die Bilder zu laden.
- Zeichnen Sie ein rechteckiges Polygon auf dem Bildrahmen, um den ROI zu markieren. Wählen Sie "Analysieren" -> "Messen", um die mittlere Fluoreszenzintensität zu messen.
- Wiederholen Sie Schritt 6.2, um Intensitätsmessungen von allen Proben zu sammeln und die jeweilige mittlere Intensität für jede Bedingung zu berechnen.
Anmerkung : Hier bezieht sich eine andere Bedingung auf verschiedene Zeitpunkte, wie in, Tag 1, Tag 2 und bis Tag 6. - Exportieren Sie die Ergebnisse in einer Tabelle für eine weitere statistische Analyse und für die Erstellung von Datenplots.
Ergebnisse
Der biologische Aktuator aus einem dünnen PDMS-Cantilever (25 μm Dicke) und Kardiomyozyten bildet den Kern des schwimmenden Biorobots, wie im schematischen und Screenshot der Geräte in Abbildung 1 gezeigt . Die Zellen beginnen nach 24 h in der Kultur Kontraktionen zu zeigen, und das Biegen der freitragenden Arme wurde am Tag 2 beobachtet. Das Seitenprofil der Vorrichtung wurde jeden Tag aufgezeichnet, und die Oberflächenspannung wurde aus der Biegung der Auslegerarme...
Diskussion
Das hier beschriebene Verfahren beschreibt eine erfolgreiche Seeding-Methode für PDMS-basierte Aktoren und Biorobots, die die Anbindung von Kardiomyozyten erleichtert. Weiterhin wurde der Prozess der Bilderfassung und die anschließende Analyse, die das Verhalten der Zellen charakterisiert, und die Leistungsfähigkeit der Geräte beschrieben.
Wir beobachteten spontane Kontraktion der Zellen an den freitragenden Armen nach 24 h; Die Intensität der Kontraktionen setzte sich im Laufe der Zeit...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
MT Holley wird unterstützt von der Graduate Fellows Programm der Louisiana Board of Regents, und C. Danielson wird von der Howard Hughes Medical Institute Professors Programm unterstützt. Diese Studie wird unterstützt von NSF Grant No: 1530884.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Cardiomyocytes (primary cardiac cells) | Charles River | NA | Isolated from 2-day old neonatal Sprague Dawley rats |
| Dulbecco’s modified eagle’s media (DMEM) | Hyclone Laboratories | 16750-074 | with 4500 mg/L glucose, 4.0 mM L-glutamine, and 110 mg/L sodium pyruvate |
| Fetalclone III serum | Hyclone industries, GE | 16777-240 | Fetal bovin serum (FBS) |
| Dulbecco’s phosphate buffer (PBS) | Sigma-Aldrich | D1408-100ML | |
| Penicillin-G sodium salt | Sigma-Aldrich | P3032 | |
| Goat serum | Sigma-Aldrich | G9023 | |
| 4,6-diamidino-2-phenylindole dihydrocholride powder (DAPI) | Sigma-Aldrich | D9542 | |
| Fibronectin from bovine plasma | Sigma-Aldrich | F1141 | Solution (1 mg/ml) |
| Calcein-AM and ethidium homodimer-1 kit (Live/Dead Assay) | Molecular Probes | L3224 | |
| Calcium Fluo-4, AM | Molecular Probes | F14217 | calcium indicator dye |
| Tyrodes salt solution | Sigma-Aldrich | T2397 | buffer solution |
| Pluronic F-127 | Molecular Probes | P3000MP | nonionic surfactant-20 % solution in Dimethylsiloxane (DMSO) |
| 16% Parafomaldehyde | Electron microscopy | 15710 | Caution: Irritant and combustible |
| Triton x-100 | Sigma-Aldrich | X-100 100 mL | cell lyses detergent, (4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether) |
| ProLong gold antifade reagent | Molecular Probes | P10144 | Mounting agent |
| Alexa Fluor 594 Phalloidin | Molecular Probes | A12381 | Actin filament marker |
| Goat anti-rabbit IgG (H+L) secondary antibody, Alexa Fluor 594 conjugate | Molecular Probes | A-11012 | |
| pha | Molecular Probes | A-11001 | |
| Anti-connexin 43 antibody | Abcam | ab11370 | Gap junction marker |
| Anti-cardiac troponin I antibody | Abcam | ab10231 | Contractile protein |
| 16% EM grade paraformaldehyde solution | Electron microscopy | 100503-916 | |
| Polydimethylsiloxane (PDMS) | Elsevier | Sylgard 184 | |
| Materials and Equipment | |||
| Camera | Thor Labs | DCC1545M | |
| LED light strip | NA | NA | Any white LED without spectrum emission |
| Confocal microscope | Nikkon C2 | NA | Confocal microscope with three filter set. |
| Zooming lens | Infinity | Model# 252120 | |
| Software | |||
| Matlab | Mathworks | NA | Used in Section 4) for biological actuator analysis. |
| Image J | National Institute of Health | NA | Java-based image processing software. Used in Section 5) for biorobot analysis. Free Image Processing and Analysis software in java. (https://imagej.nih.gov/ij/) |
| Thor Cam | Thor Labs | NA | Camera operating software |
Referenzen
- Feinberg, A. W. Biological Soft Robotics. Annu. Rev. Biomed. Eng. 17, 243-265 (2015).
- Akiyama, Y., et al. Room Temperature Operable Autonomously Moving Bio-Microrobot Powered by Insect Dorsal Vessel Tissue. PLOS ONE. 7, 38274 (2012).
- Herr, H., Dennis, R. G. A swimming robot actuated by living muscle tissue. J. NeuroEng Rehabil. 1, 6 (2004).
- Park, S., et al. Phototactic guidance of a tissue-engineered soft-robotic ray. Science. 353 (6295), 158-162 (2016).
- Cvetkovic, C., et al. Three-dimensionally printed biological machines powered by skeletal muscle. Proc. Natl. Acad. Sci. 111, 10125-10130 (2014).
- Williams, B. J., Anand, S. V., Rajagopalan, J., Saif, M. T. A. A self-propelled biohybrid swimmer at low Reynolds number. Nat. Commun. 5, (2014).
- Holley, M. T., Nagarajan, N., Danielson, C., Zorlutuna, P., Park, K. Development and characterization of muscle-based actuators for self-stabilizing swimming biorobots. Lab. Chip. 16, 3473-3484 (2016).
- Hopkins, P. M. Skeletal muscle physiology. Contin Educ Anaesth Crit Care Pain. 6, 1-6 (2006).
- Nawroth, J., et al. A tissue-engineered jellyfish with biomimetic propulsion. Nat Biotechnol. 30 (8), 729-797 (2012).
- Ehler, E., Moore-Morris, T., Lange, S. Isolation and Culture of Neonatal Mouse Cardiomyocytes. J. Vis. Exp. JoVE. (79), e50154 (2013).
- Bers, D. M. Calcium Fluxes Involved in Control of Cardiac Myocyte Contraction. Circ. Res. 87, 275-281 (2000).
- Shin, S. R., et al. Carbon-Nanotube-Embedded Hydrogel Sheets for Engineering Cardiac Constructs and Biological actuators. ACS Nano. 7, 2369-2380 (2013).
- Park, J., et al. Real-Time Measurement of the Contractile Forces of Self-Organized Cardiomyocytes on Hybrid Biopolymer Microcantilevers. Anal. Chem. 77, 6571-6580 (2005).
- Tamayo, J., et al. Quantification of the surface stress in microcantilever biosensors: revisiting Stoney's equation. Nanotechnology. 23, 475702 (2012).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat. Methods. 10, 781-787 (2013).
- Louch, W. E., Sheehan, K. A., Wolska, B. M. Methods in Cardiomyocyte Isolation, Culture, and Gene Transfer. J. Mol. Cell. Cardiol. 51, 288-298 (2011).
- Alford, P. W., Feinberg, A. W., Sheehy, S. P., Parker, K. K. Biohybrid thin films for measuring contractility in engineered cardiovascular muscle. Biomaterials. 31, 3613-3621 (2010).
- Sfakiotakis, M., Lane, D. M., Davies, J. B. C. Review of fish swimming modes for aquatic locomotion. IEEE J. Ocean. Eng. 24, 237-252 (1999).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten